基于转录组的赤斑门条天牛嗅觉相关基因分析
2024-01-01杨华芳张祖兵朱家颖柏天琦张宏瑞




摘要:【目的】鉴定赤斑白条天牛(Batocera rufomaculata)嗅觉相关基因,筛选其中的差异表达基因(DEGs),为探 明赤斑白条天牛嗅觉机制和基于嗅觉识别的天牛生物防治新途径研究提供理论支撑。【方法】采用Illumina NovaSeq 6000测序平台对赤斑白条天牛触角、跗节和残体进行转录组测序,利用测序数据组装、注释、数据库序列检索、序列比 对及差异基因分析等生物信息学分析方法挖掘嗅觉相关基因。【结果】转录组测序共获得137405条Unigenes,平均 长度774 bp,N50为1462bp,GC含量42.93%。通过同源比对共鉴定出289个嗅觉基因,包括45个OBPs、22个CSPs、50 个ORs、20个IRs、26个GRs、7个SNMPs、8个CXEs、82个CYPs、19个AOXs和10个GSTs基因。通过比较所有样本的转 录组,共筛选到8861个DEGs,其中2713个上调表达、6148个下调表达。基因功能注释和信号通路富集分析结果表明,有3613个DEGs注释到GO功能的生物学过程、细胞组分和分子功能,其中以生物学过程的占比最高,为34.87%;有4002个DEGs注释到KEGG数据库,参与96条通路。进一步对嗅觉基因进行分析发现,有114个嗅觉基因表 达差异显著,其中79个上调表达、35个下调表达;OBP22、OBP29、CSP10、CSP15、SNMP1、OR41、CXE2和CYP10基因 在触角中的表达量均高于其他组织,CSP10和CSP15基因在雌虫中偏好表达,CYP34基因在雄性中偏好表达,且在跗 节中高表达。【结论】筛选到赤斑白条天牛OBPs、CSPs、ORs、IRs、GRs、SNMPs、CXEs、CYPs、AOXs和GSTs等嗅觉相 关基因。推测触角中高表达的OBPs和CSPs基因对赤斑白条天牛雌雄虫识别同类异性释放的信息素或寄主植物释放的挥发物起关键作用,在雌虫中偏好表达的CSPs基因可能在雌虫寻找产卵场所过程中发挥重要作用。
关键词:赤斑白条天牛;转录组;高通量测序;嗅觉相关基因;差异基因
文章编号:2095-1191(2024)03-0699-11
中图分类号:S433.5
文献标志码:A
Analysis of olfactory related genes in Batocera rufomaculatabased on transcriptomeYANG Hua-fang1.2, ZHANG Zu-bing1*, ZHU Jia-ying3, BAI Tian-qi4, ZHANG Hong-rui2*
(1Yunnan Institute of Tropical Crops, Jinghong, Yunnan 666100, China; 2Plant Protection College, Yunnan Agricultural University/State Key Laboratory for Conservation and Utilization of Bio-resources in Yunnan, Kunming, Yunnan 650201, China; 3Southwest Forestry University/Key Laboratory of Forest Disaster Warning and Control of Yunnan Province, Kunming, Yunnan 650224, China; 4Tropical and Subtropical Cash Crop Research Institute, YunnanAcademy of Agricultural Sciences, Baoshan, Yunnan 678099, China)
Abstract:[Objective ] The olfactory related genes in Batocera rufomaculata were identified and the differentially expressed genes (DEGs) were screened to provide theoretical support for the exploration of olfactory mechanism and new approaches to biological control of B. rufomaculata based on olfactory recognition. [Method] The transcriptome sequences of antennae, tarsus and the body without antennae and tarsus of B. rufomaculata were sequenced using Illumina NovaSeq 6000 sequencing. Bioinformatics analysis methods such as sequencing data assembly, annotation, database sequence retrieval, sequence alignment and differential gene analysis were used to mine olfactory related genes. [Result】In total, 137405 unigenes were obtained by transcriptome sequencing with an average length of 774 bp, N50 of 1462 bp,and GC content of 42.93%. Through homologous alignment, a total of 289 olfactory related genes were identified, including 45 OBPs, 22 CSPs, 50 ORs, 20 IRs, 26 GRs, 7 SNMPs, 8 CXEs, 82 CYPs, 19 AOXs, 10 GSTs genes. By comparing the transcriptome of all samples, a total of 8861 DEGs were screened,of which 2713 were up-regulated and 6148 were down-regulated. The results of gene function annotation and pathway enrichment analysis showed that 3613 DEGs were annotated to the biological process, cellular component and molecular function of GO function, among which the biological process accounted for the highest proportion (34.87%). A total of 4002 DEGs were annotated to the KEGG database and involved in 96 pathways. Further analysis of olfactory related genes showed that 114 olfactory genes expression were significantly different, of which 79 were up-regulated and 35 were down-regulated. The expression levels of OBP22, OBP29, CSP10, CSP15, SNMP1, OR41, CXE2 and CYP10 genes in antennae were higher than those in other tissues. CSP10 and CSP15 genes were preferentially expressed in females. CYP34 gene was preferentially expressed in males and highly expressed in the tarsus. [Conclusion]Olfactory related genes in B. rufomaculata such as OBPs, CSPs, ORs, IRs, GRs, SNMPs, CXEs, CYPs, AOXs, and GSTs are screened. It is speculated that the highly expressed OBPs and CSPs genes in the antennae play a crucial role in the recognition of pheromones released by the opposite sex of the same species or volatiles released by host plants. The CSPs gene that are preferentially expressed in females may play an important role in the process of females looking for oviposition sites.
Key words: Batocera rufomaculata; transcriptome; high-throughput sequencing; olfactory related genes; differential genes
Foundation items: China Agriculture Research System(CARS-11-YNZZB)
0 引言
【研究意义】昆虫触角是接收外界化学信号的主 要器官,在长期进化过程中形成了高度灵敏和极其 复杂的嗅觉系统(Brito et al.,2016)。昆虫通过自身 灵敏的嗅觉系统感受寄主植物释放的气味物质和异 性释放的性信息物质,精准定位寄主和寻找配偶,最 终完成取食、交配和产卵活动(Kaupp,2010)。辣木 (Moringa oleifera)是辣木科辣木属多年生乔木,具有较高的食用和药用价值。赤斑白条天牛(Batocera rufomaculata)是辣木上的重要蛀干害虫之一,在西 双版纳地区发现为害已有10余年,严重威胁辣木产 业的发展,危害严重时造成重大经济损失。赤斑白 条天牛能准确识别辣木并进行取食及产卵危害,与 其灵敏的嗅觉系统息息相关。深入研究赤斑白条天 牛嗅觉系统,有助于分析其定位寄主和寻找配偶时 识别信息物质及其化学通讯与感受机制,而大量嗅 觉相关基因是进行分子机制研究的前提(巩雪芳等, 2020)。因此,鉴定获得大量的嗅觉基因,对研究基 因功能、揭示嗅觉识别机制以及筛选分子靶标等均 具有重要意义。【前人研究进展】完整的嗅觉感受过
程需要多种嗅觉基因相互协作,包括气味结合蛋白 (Odorant binding proteins,OBPs)、化学感受蛋白(Chemosensory proteins,CSPs)、气味受体(Olfactory receptors,ORs)、味觉受体(Gustatory receptors,GRs)、 感觉神经元膜蛋白(Sensory neuron membrane pro- teins,SNMPs)、离子型受体(Ionotropic receptors,IRs)和气味降解酶(Odorant-degrading enzymes,ODEs)(胡颖颖等,2013)。昆虫嗅觉识别气味分子的整个过程归纳为:气味分子经嗅觉感器微孔进入感器淋巴液,被OBP或CSP识别后结合并运载至嗅觉受体神经元(ORN)树突膜,激活膜上的OR、IR或GR,同 时将化学信号转化为电信号传递至触角叶,进而激 活脑部中枢神经系统,经大脑整合后发出指令,使昆 虫产生特定的行为反应,嗅觉信号转导后,在SNMP的作用下,气味分子脱离受体部位,迅速被ODEs降解失活,终止气味物质对ORs的刺激,保持受体活性 (陈聪,2021)。随着基因组学、转录组学、生物信息 学及基因测序技术的发展,大部分昆虫的嗅觉基因 陆续被鉴定报道。从亚洲小车蝗(Oedaleus asiaticus) 中鉴定到17个CSPs、60个ORs、6个IRs和3个SNMPs基因(周渊涛,2019);从绿眼赛茧蜂(Zele chloroph- thalmus)中鉴定到14个OBPs、6个CSPs、83个ORs、22个IRs、23个GRs和3个SNMPs基因(王予彤等,2021);从桃红颈天牛(Aromia bungii)中鉴定到52个 OBPs、17个CSPs、45个ORs、2个IRs、6个GRs、3个SNMPs和72个ODEs基因(吴振晨,2022);从叉角厉 蝽(Eocanthecona furcellata)中鉴定到10个OBPs、 32个CSPs、48个ORs、17个IRs、21个GRs和6个SNMPs基因(赵航等,2022);从眉斑并脊天牛(Glenea can-tor)中鉴定到29个OBPs、14个CSPs、13个ORs、18个IRs和2个SNMPs基因(Wu et al.,2022);从罗氏夜 蛾(Mythimna loreyi)中鉴定到33个OBPs、16个CSPs、 63个ORs、24个IRs和2个SNMPs基因(Zhang et al., 2022);从齿带卷叶象(Podabrus annulatus)中鉴定到 11个OBPs、6个CSPs、50个ORs、6个IRs、25个GRs和3个SNMPs基因(Wang et al.,2023)。此外,关于 嗅觉基因的组织表达特性也有大量研究,如咖啡脊 虎天牛(Anoplophora chinensis)CSP2基因在雌雄虫 触角、头和足中均高表达,CSP4、CSP5和CSP9基因在触角中高表达,在头的表达量次之,在腹部有少量 表达(詹文会,2018);花椒窄吉丁(Agrilus zanthoxy-lumi)OBP1和OBP2基因在雌成虫触角中不表达,OBP3基因在雄成虫触角中高丰度表达(巩雪芳等,2020);米蛾(Corcyra cephalonica)的PBPs和GOBPs 基因均在成虫触角上大量表达,并且GOBP2基因 仅在成虫触角中表达(李鹏燕等,2021);异色瓢虫(Harmonia axyridis)0BP2/3/5/12/15、CSP4/9/15、OR13 和OR14基因在雌雄成虫触角中的表达量显著高于 头、胸、腹、足和翅等其他组织(渠成,2021);小贯小 绿叶蝉(Empoasca onukii)的12个ORs和8个IRs基 因主要在雌雄成虫触角中显著表达,3个GRs基因 在各组织中表达较广泛(张信哲,2022)。可见,昆虫 嗅觉基因主要分布在触角、头和足等嗅觉组织中,而 在胸部和腹部等非嗅觉组织中分布较少。【本研究切 入点】昆虫嗅觉基因在身体的分布特征暗示着不同 的生物学功能,明确这些基因在组织上的分布特性 有助于探究昆虫的嗅觉分子机制。赤斑白条天牛凭 借高度专一灵敏的嗅觉系统长期在辣木上钻蛀危 害,幼虫期长且生活隐秘,成虫鞘翅坚硬,目前未能 探索出有效的防治方法。关于赤斑白条天牛的研究还停留在生物学方面,深入研究还处于空白,嗅觉机 制研究尚未见报道。【拟解决的关键问题】基于转录 组测序技术,鉴定赤斑白条天牛嗅觉相关基因,筛选 差异表达基因(DEGs)并进行功能注释和富集分析, 为探明赤斑白条天牛嗅觉机制和基于嗅觉识别的天 牛生物防治新途径研究提供理论支撑。
1材料与方法
1.1试验材料
赤斑白条天牛采自云南省景洪市热带作物科学 研究所辣木基地。采集受赤斑白条天牛危害的辣木 树干,置于田间养虫笼(150cm×150cm×100cm)中 饲养,定期检查并收集羽化的成虫,将成虫放入室 内养虫箱(60cm×30cm×30cm)中用辣木嫩枝喂食 备用。
1.2总RNA提取与质检
取羽化后未交配的赤斑白条天牛雌雄虫各 6头,雌雄分开,每2头1个处理;分别取触角、跗节 和残体(除触角和跗节外的其他身体组织),设 3个生物学重复,于液氮中充分研磨,用TRIzol法提取总RNA。用核酸蛋白仪[NanoDrop 2000,赛默飞世尔科技(中国)公司]检测RNA浓度和纯度、自动化电泳仪(Agilent 2100,上海艾研生物科技有限公司)检测RNA的完整性。
1.3cDNA文库构建与测序
RAN样品检测合格后,用带有多聚胸腺嘧啶的 磁珠富集真核生物mRNA;加入RNA裂解缓冲液 将mRNA打断成短片段,以mRNA为模板,用六碱基随机引物合成第一链cDNA;加入缓冲液、脱氧核糖核苷三磷酸、DNA聚合酶I和核糖核酸酶H合成第二链cDNA;用核酸纯化试剂纯化双链cDNA。纯化的双链cDNA先进行末端修复、加A尾并连接测 序接头;用核酸纯化试剂进行片段大小选择。进行 PCR扩增,并用核酸纯化试剂纯化PCR产物,得到 最终的文库。文库构建完成后,先使用核酸蛋白定 量仪(Qubit 2.0,上海艾研生物科技有限公司)进行初步定量,稀释文库至1.5ng/uL,随后使用自动化电泳仪(Agilent 2100,上海艾研生物科技有限公司) 对文库的插入片段进行检测,插入片段符合预期后, 使用实时荧光定量方法对文库的有效浓度进行准确 定量(文库有效浓度gt;2×10°mol/L),以保证文库质 量;文库检测合格后,使用Illumina NovaSeq 6000测序平台进行测序。cDNA文库构建与测序委托北京 诺禾致源生物信息科技有限公司完成。
1.4测序数据过滤与组装
对测序所得Raw reads进行过滤、去除带接头和 低质量的Reads后得到Clean reads,用Trinity软件对 Clean reads进行拼接得到转录本(Transcripts),利用 Corset进行聚类去冗余得到单基因(Unigenes)。
1.5嗅觉相关基因鉴定与序列分析
在NCBI数据库中分别以OBP(odorant-binding protein)、 CSP (chemosensory protein)、 OR (odorant receptor)、 GR (gustatory receptor)、 SNMP (sensory neu-ron membrane protein)、IR (ionotropic receptor)、 ODE(odorant-degrading enzymes)为关键词进行检 索,下载昆虫嗅觉相关基因的氨基酸序列。以所 下载的序列为种子序列,赤斑白条天牛Unigenes序列为目标序列,在TBtools中进行BLAST本地比对,获得高相似性的Unigenes,再运用在线网页展开BLASTx比对验证,最终确定是否为嗅觉基因。分 别使用在线软件ORFfinder(https://www.ncbi.nlm. nih.gov/orffinder/)寻找嗅觉相关基因的开放阅读框 (ORF),ExPASy(https://web.expasy.org/compute_pi/)计算蛋白质等电点和分子量,SignalP(https://services. healthtech.dtu.dk/services/SignalP-6.0/)预测嗅觉基 因的信号肽,TMHMM 2.0(https://services.health-tech.dtu.dk/services/TMHMM-2.0/)预测蛋白跨膜结构域。
1.6DEGs分析与功能富集
利用TBtools软件,以Unigenes的Count矩阵作 为输入文件,设置赤斑白条天牛样本归组信息和雌 雄虫组织样本差异比较组合,用DESeq2包进行 DEGs分析,Volcano Plot绘制DEGs火山图,Interac- tive Venn Graph绘制DEGs韦恩图;将1.5中鉴定到的嗅觉基因和所有DEGs求交集,筛选出差异表达显著的嗅觉相关基因,根据FPKM值绘制聚类热图;利用在线工具eggNOG-mapper(http://eggnog-mapper. embl.de/)对Unigenes进行基因功能注释,用eggNOG- mapper Helper整理基因功能注释结果,用GO enrichment进行GO功能注释分析,用KEGG enrich- ment进行KEGG信号通路富集分析,利用在线数据 分析可视化平台(https://www.bioinformatics.com.cn/) 对GO功能注释和KEGG信号通路富集结果进行可 视化。
2结果与分析
2.1赤斑白条天牛转录组测序质量统计结果
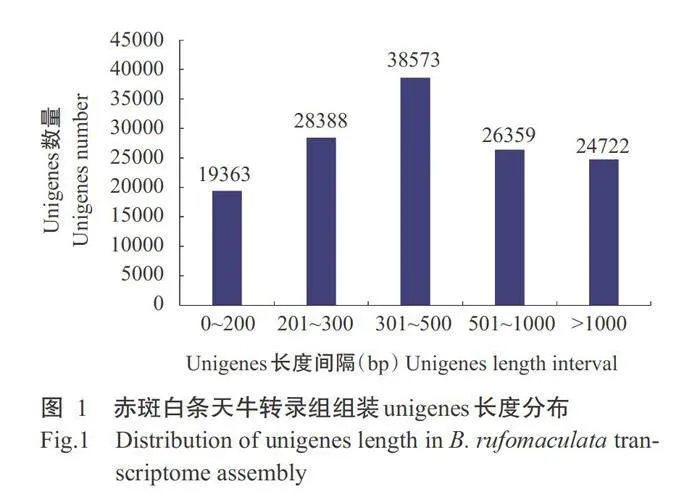
由表1可知,转录组测序碱基数达111.32Gb,共获得784288598条Raw reads,过滤掉带接头和低质量的Reads后,共得到742106736条高质量的Clean reads。运用Trinity对总Clean reads进行组装后得到 795957条Transcripts,总长度378815177 bp,平均长度476bp;以Corset聚类得到137405条Unigenes,总长度106341931bp,平均长度774bp,N50为1462bp,GC含量为42.93%,有89654条Unigenes长度大于 500bp,占总数的65.25%(表1和图1)。
2.2嗅觉相关基因鉴定及分析结果
通过同源比对共鉴定出289个嗅觉相关基因,包括45个OBPs、22个CSPs、50个ORs、20个IRs、26个 GRs、7个SNMPs、8个CXEs、82个CYPs、19个AOXs和10个GSTs基因。其中,42个OBPs、16个CSPs、 44个ORs、11个IRs、24个GRs、4个SNMPs、7个CXEs、49个CYPs、11个AOXs和8个GSTs基因具有完整ORF,ORF长度在90~2805bp,编码29~934个氨基酸,等电点4.05~11.71,分子量3.406~106.017kD;有26个OBPs、7个CSPs、2个IR、1个GR、1个SNMP和3个CXE基因能预测到N-端信号肽;有4个OBPs、 46个ORs、16个IRs、22个GRs、2个SNMPs、1个CXE和53个CYPs基因能预测跨膜结构域,数量为 1~9个。
2.3DEGs分析结果
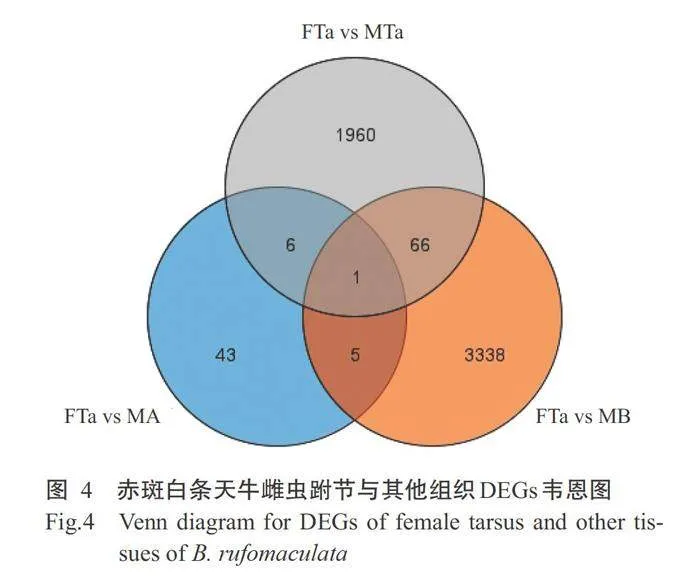
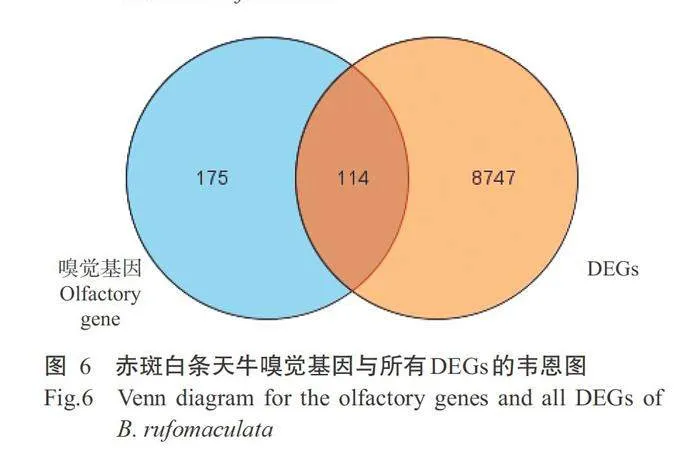
对赤斑白条天牛雌雄虫各组织进行DEGs分 析,共筛选到8861个DEGs,其中2713个上调表达、 6148个下调表达,上、下调表达基因数目差异较大。 雌虫触角与雌虫残体、雌虫跗节、雄虫触角、雄虫残 体和雄虫跗节5个比较组合特有DEGs分别为421、60、2、2744和2131个,共有DEGs 2个(图2);雌虫残体 与雌虫跗节、雄虫触角、雄虫残体和雄虫跗节4个比 较组合特有DEGs分别为136、150、1613和2902个,共有DEGs65个(图3);雌虫跗节与雄虫触角、雄虫 跗节和雄虫残体3个差异比较组合特有DEGs分别 为43、1960和3338个,共有DEGs1个(图4):雄虫触角与雄虫残体和雄虫跗节2个差异比较组合特 有DEGs分别为1621和295个,共有DEGs 154个 (图5);在289个嗅觉基因中有114个基因表达差 异显著,其中79个上调表达,35个下调表达(图6), 并且在雌雄虫各组织的表达表现为触角gt;跗节gt;残 体(图7)。
2.4114个差异表达嗅觉相关基因的表达模式分析
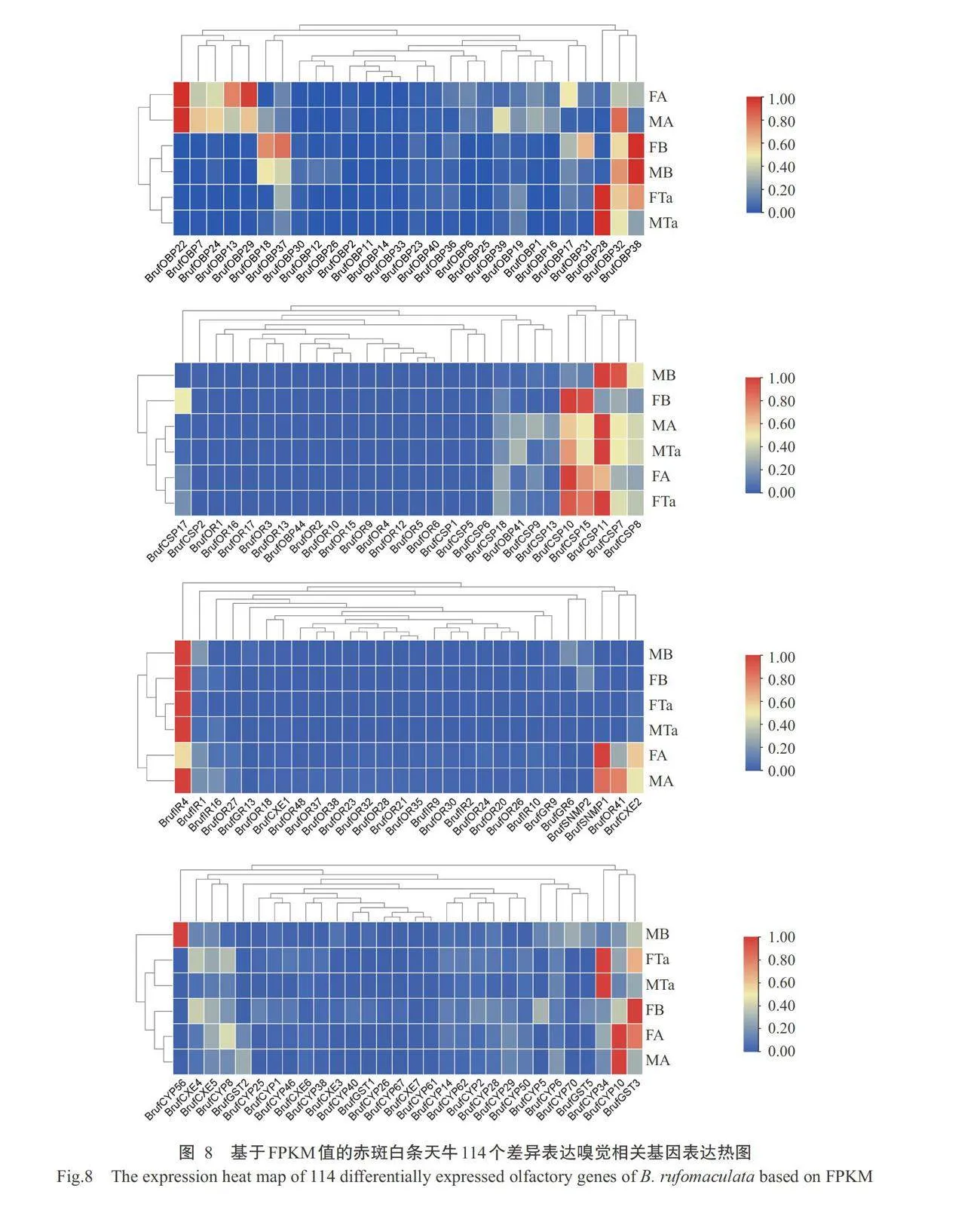
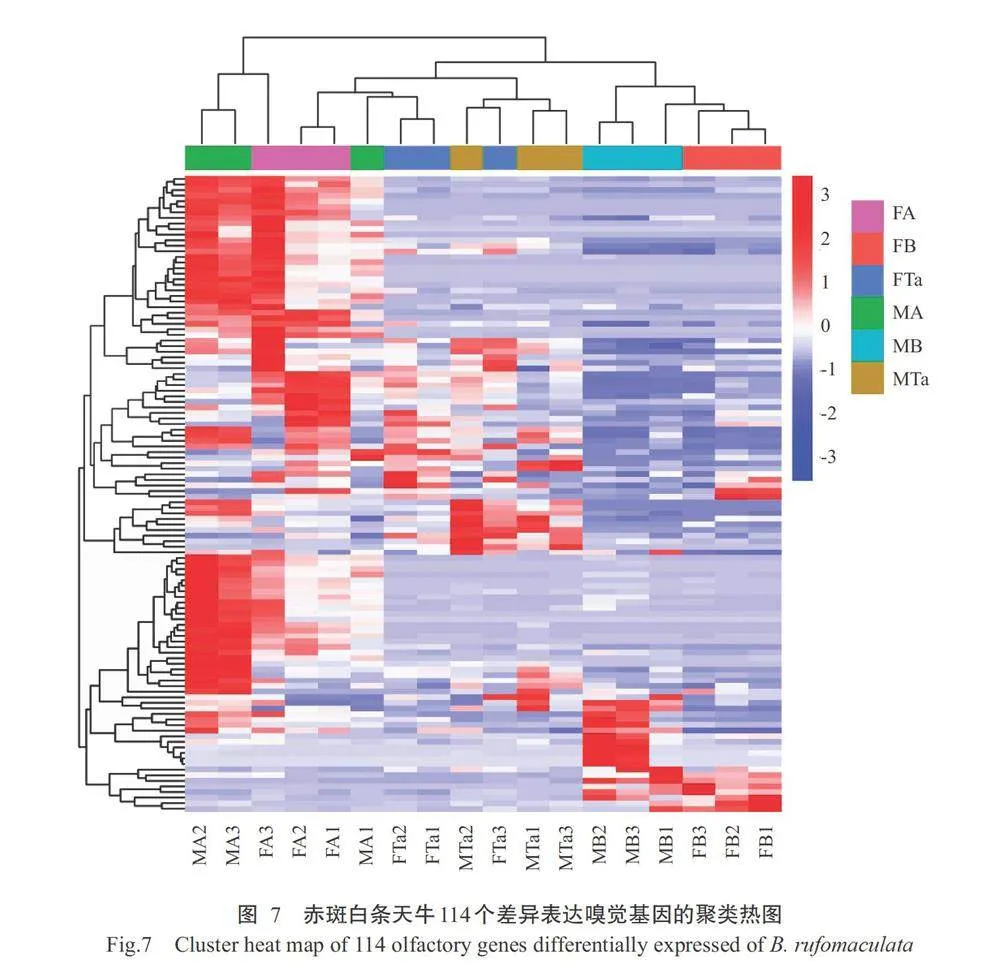
基于FPKM值,利用TBtools软件绘制114个差异表达嗅觉相关基因的表达模式热图,结果(图8)显示,OBP22基因在雌雄虫触角高表达,OBP28基因在雌雄虫跗节高表达,OBP38基因在雌雄虫残体高表达,OBP13和OBP29基因在雌虫触角高表达,OBP18/31/37和CSP17基因在雌虫残体高表达,OBP32基因在各个组织中均有表达,且在雄虫触角和雄虫残体表达量较高;CSP7/8/10/11/15基因呈现出广泛的表达谱,在各组织中均有表达,其中,CSP7基因在雄虫残体高表达,CSP8基因在雄虫残体表达量较其他组织高,CSP10和CSP15基因除在雄虫残 体低表达外,在其他组织中均高表达,且雌虫组织中 的表达量高于雄虫,CSP11基因在雄虫触角、跗节、 残体及雌虫跗节均高表达;IR4基因除在雌虫触角表 达较低外,在其余组织均高表达,SNMPI基因在雌雄虫触角高表达,OR41基因在雄虫触角高表达, CXE2基因在雌虫触角高表达;CYP10基因在雌雄虫 触角高表达,CYP34基因在雌雄虫跗节高表达,CYP56基因仅在雄虫残体高表达,GST3基因在各组 织中均有表达,其中在雌虫残体的表达量最高,雌虫 组织中的表达量高于雄虫。
2.5DEGs的GO功能注释和KEGG信号通路富集 分析结果
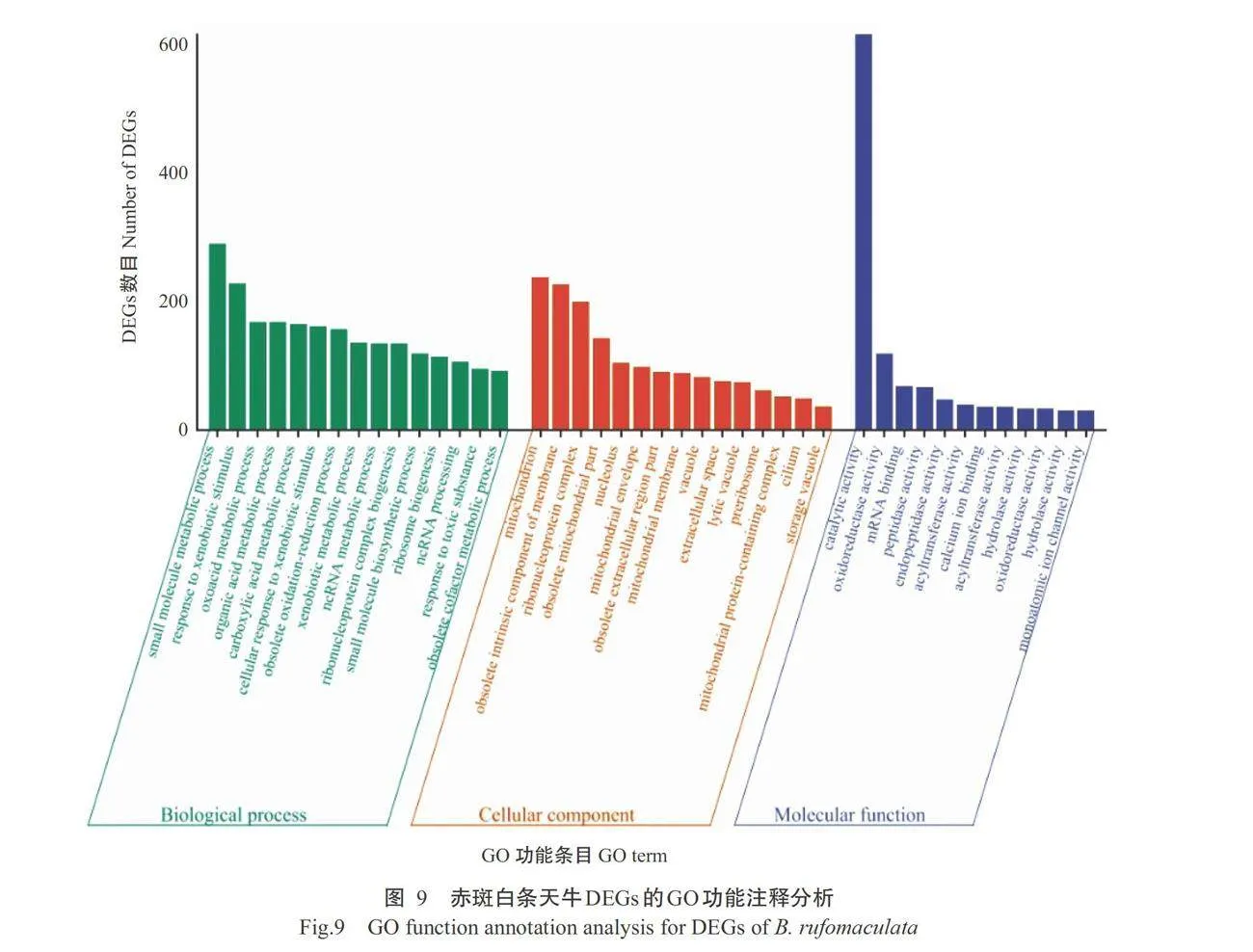
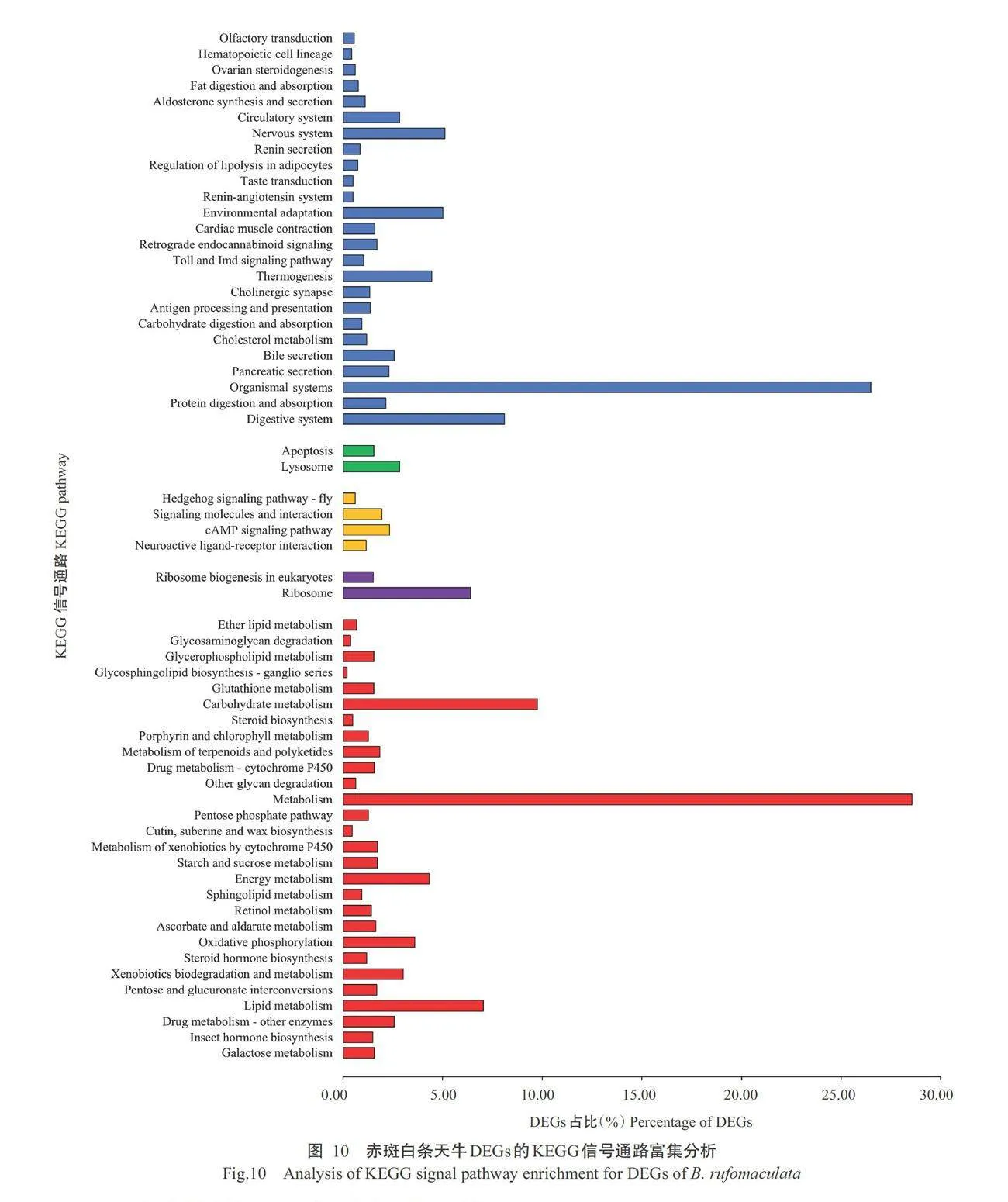
3613个DEGs注释到GO功能的生物学过程(Bio- logical process,BP)、细胞组分(Cellular component,CC)和分子功能(Molecular function,MF)3个分类 中,其中1260个注释到生物学过程(占比34.87%), 1214个注释到细胞组分(占比33.60%)、1139个注释 到分子功能(占比31.53%);在三大类的亚类中,所 注释到的基因数目最多的分别为小分子代谢过程(Small molecule metabolic process)、线粒体(Mito- chondrion)和催化活性(Catalytic activity),分别为 290、237和618个(图9)。4002个DEGs注释到KEGG的五大功能分类中,分别为新陈代谢(Metabolism)、 遗传信息处理(Genetic information processing)、环 境信息处理(Environmental information processing)、 细胞过程(Cellular process)和生物系统(Organismal system),注释到的DEGs分别为3343、760、241、174和2945个;DEGs共参与96条代谢通路,其中有1137个DEGs参与到新陈代谢通路中,占注释总数 的28.41%(图10)。
3讨论
嗅觉对昆虫的生存与繁殖具有非常重要的生物 学意义,嗅觉相关基因在昆虫嗅觉识别过程中发挥着重要作用,是昆虫与寄主植物互作过程中重要的 分子靶标(陈聪,2021)。赤斑白条天牛是危害辣木 的重要蛀干害虫,为获得其嗅觉相关基因,本研究利 用Illumina测序技术对赤斑白条天牛进行转录组测 序,筛选鉴定嗅觉基因并对差异表达显著的嗅觉相 关基因进行分析,揭示了嗅觉基因在触角、跗节、残 体以及雌雄虫之间的整体表达模式。通过序列的预 处理、Trinity拼接、GO功能注释和KEGG信号通路 富集分析等步骤,获得了转录组和嗅觉相关蛋白信 息。测序得到的赤斑白条天牛转录组数据以及基因 功能注释信息一方面为下一步的分子生物学研究打 下了基础,另一方面也为化学生态学相关研究提供 了先决条件(Zhu et al.,2017)。
本研究基于赤斑白条天牛转录组测序数据,利 用BLAST同源比对,共鉴定到289个嗅觉相关基因,包括45个OBPs、22个CSPs、50个ORs、20个IRs、26个GRs、7个SNMPs、8个CXEs、82个CYPs、19个AOXs和10个GSTs基因。学者们在赤斑白条 天牛近缘种中也鉴定到不同类型和数量的嗅觉相 关基因,如在星天牛(A.chinensis)中鉴定到52个OBPs、14个CSPs、53个ORs、17个GRs、4个IRs、3个SNMPs、25个CarEs和43个GSTs基因(孙龙, 2018);在咖啡脊虎天牛(Xylotrechus grayii)中鉴定到34个OBPs、15个CSPs、60个ORs和4个SNMPs基因(詹文会,2018);在桑天牛(Apriona germari)中鉴定到62个OBPs、25个CSPs、42个ORs、3个IRs、13个GRs和5个SNMPs基因(钱佳丽,2020);在桃红颈天 牛(A.bungii)中鉴定到52个OBPs、17个CSPs、45个ORs、2个IRs、6个GRs、3个 SNMPs、24个CarEs、32个CYPs、2个AOX和14个GSTs基因(吴振晨,2022)等。很显然,不同昆虫中鉴定到的嗅觉基因数目不 尽相同,但总体上看,获得的嗅觉基因中OBPs和ORs基因数目均较多,可能与OBPs及ORs的具体功能相关。对获得的Unigenes进行DEGs分析,共筛选到8861个DEGs,其中包含114个表达差异显著的嗅 觉基因,114个嗅觉基因中有79个呈上调表达,35个 呈下调表达,上、下调表达基因数目差异较大。将 DEGs进行基因功能注释和信号通路富集分析,分别 有3613和4002个DEGs注释到GO和KEGG数据库 中,共参与96条代谢通路,2大数据库的功能分类中 注释到基因数目最多的分别是催化活性功能和新陈 代谢通路,推测这些DEGs与天牛生长发育以及某 些重要物质的合成密切相关,与大多数研究结果吻 合(郑海霞等,2018;胡佳萌等,2019;李鹏燕等, 2021;张信哲,2022)。利用FPKM值分析嗅觉基因的表达模式并进行可视化,发现赤斑白条天牛OBP7/13/22/24/29、SNMP1、OR41、CXE2和CYP10基因只在雌雄虫触角表达,OBP28和CYP34基因只在雌雄虫跗节表达,OBP38基因在雌雄虫残体高表达,CSP17和GST3基因在雌虫残体高表达,CSP7和CYP56基因在雄虫残体高表达,而OBP18/37/32、CSP7/8/10/11/15、IR4和GST3基因具有广泛的表达谱。就表达量来看,OBP22、OBP29、CSP10、CSP15、SNMP1、OR41、 CXE2和CYP10基因在触角的表达量均高于其他组 织,且CSP10和CSPI5基因在雌虫的表达量高于雄虫,CYP34基因在雌虫的表达量低于雄虫,可见,嗅 觉基因的表达具有明显的性别差异和组织差异。其 他昆虫的部分嗅觉基因如腰带长体茧蜂(Macrocen- trus cingulum)OBPs基因(Ahmed,2014)、美国白蛾 (Hyphantria cunea)OBPs基因(康克,2016)、玉米象(Sitophilus zeamais)OBPs和CSPs基因(沈忱,2019) 及眉斑并脊天牛(Glenea cantor)OBPs和CSPs基因 (Wu et al.,2022)等也均表现为在触角中高表达,本 研究结果与前述研究相似,可能与触角作为主要的 感觉器官,触角上密布嗅觉感受器行使重要的嗅觉 感受功能相关;大蜡螟(Galleria mellonell)OBP基因 在雌成虫中的表达量显著高于雄成虫(蒋秀云,2021),罗氏夜蛾(Mythimna loreyi)部分OBPs、CSPs 和ORs基因的表达均存在明显的性别偏好(Zhanget al.,2022),表明嗅觉基因的表达存在组织差异性 和性别差异性,推测在赤斑白条天牛触角中高表达 的嗅觉基因可能在触角感受外界过程中发挥重要作 用,存在性别偏好表达的嗅觉基因可能在识别异性 和感受种内性信息素时发挥特殊作用。
结论
筛选到赤斑白条天牛OBPs、CSPs、ORs、IRs、GRs、 SNMPs、CXEs、CYPs、AOXs和GSTs等嗅觉相关基 因。推测触角中高表达的OBPs和CSPs基因对赤斑 白条天牛雌雄虫识别同类异性释放的信息素或寄主植物释放的挥发物起关键作用,在雌虫中偏好表 达的CSPs基因可能在雌虫寻找产卵场所过程中发 挥重要作用。
参考文献(References):
陈聪.2021.香樟齿喙象信息化学物质的活性组分鉴定及其 关键气味结合蛋白的功能分析[D].南京:南京林业大
学.[Chen C.2021.Identification of active components of semiochemicals and functional analysis of key odorant- binding proteins in Pagiophloeus tsushimanus[J]. Nanjing:
Nanjing Forestry University.] doi: 10.27242/d.cnki. gnjlu.2021.000005.
巩雪芳,谢寿安,杨平,郭莉,陈迪,车显荣,王延来,吕淑杰.
2020.花椒窄吉丁触角转录组及嗅觉相关基因分析[J].
昆虫学报,63(10):1159-1170.[Gong XF,Xie S A,Yang P, Guo L, Chen D, Chen X R, Wang Y L, Lü S J. 2020. Analysis of the antennal transcriptome and olfaction- related genes of Agrilus zanthoxylumi (Coleoptera: Bupres- tidae) [J]. Acta Entomologica Sinica, 63 (10) : 1159-1170.] doi:10.16380/j.kcxb.2020.10.001.
胡佳萌,徐丹萍,卓志航,杨伟,杨桦,郑奕然.2019.云斑天牛 成虫触角转录组及嗅觉相关基因分析[J].应用昆虫学
报,56(5):1037-1047.[HuJM,Xu D P,Zhuo ZH,Yang W, Yang Y, Zheng Y R. 2019. Analysis of antennal tran-
scriptome and olfaction-related genes of adult Batocera horsfieldi (Hope) [J]. Chinese Journal of Applied Entomology, 56 (5) : 1037-1047.] doi: 10.7679/j. issn. 2095-1353.2019.114.
胡颖颖,徐书法,AbebeJ.Wubie,李薇,国占宝,周婷.2013.昆虫嗅觉相关蛋白及嗅觉识别机理研究概述[J].基因组学
与应用生物学,32(5):667-676.[Hu Y Y,Xu S F,Wubie A J, Li W, Guo Z B, Zhou T. 2013. Research advance of olfactory proteins and olfactory mechanism in insects[J].
Genomics and Applied Biology, 32 (5) : 667-676.] doi:
10.3969/gab.032.000667.
蒋秀云.2021.大蜡螟嗅觉相关基因鉴定、序列特征与表达模 式分析[D].合肥:安徽农业大学.[Jiang XY.2021.Iden- tification, sequence characteristics, and expression pattern analysis of olfactory-related genes from Galleria mel- lonella [D]. Hefei: Anhui Agricultural University.] doi: 10.26919/d.cnki.gannu.2021.000059.
康克.2016.美国白蛾嗅觉基因的鉴定及表达研究[D].合肥:安徽农业大学.[Kang K.2016.Identification and expression of olfactory genes in Hyphantria cunea (Drury)
[D]. Hefei:Anhui Agricultural University.]
李鹏燕,张秋婷,黄铮昱,黄华,陈伟平,廖永林.2021.米蛾嗅
觉基因的转录组分析[J].应用昆虫学报,58(6):1347-
1355. [Li P Y,Zhang Q T,Huang Z Y,Huang H,Chen W P, Liao Y L. 2021. Analysis of the olfactory genes of Corcyra cephalonica (Lepidoptera: Pyralidae) [J]. Chinese Journal of Applied Entomology, 58 (6) : 1347-1355.] doi:
10.7679/j.issn.2095-1353.2021.135.
钱佳丽.2020.桑天牛嗅觉相关基因的鉴定及嗅觉受体的表 达分析[D].合肥:安徽农业大学.[QianJL.2020.Iden- tification of olfactory genes and expression profile of olfac- tory receptor genes in Apriona germari (Hope) [D]. Hefei:
Anhui Agricultural University.] doi: 10.26919/d.cnki.gannu.
2020.000242.
渠成.2021.异色瓢虫嗅觉相关蛋白基因的鉴定及功能研究
[D].泰安:山东农业大学.[Qu C.2021.Identification and functional characterization of olfactory relative protein genes in Harmonia axyridis (Coleoptera: Coccinellidae) ] [D]. Tai' an: Shandong Agricultural University.] doi: 10.
27277/d.cnki.gsdnu.2021.000042.
沈忱.2019.玉米象嗅觉相关基因的鉴定与表达模式分析
[D].合肥:安徽农业大学.[Shen C.2019.Identification and expression profiling of olfactory-related genes in Sitophilus zeamais (Coleoptera:Curculionidea) using qRT- PCR[D]. Hefei: Anhui Agricultural University.] doi: 10.
26919/d.cnki.gannu.2019.000080.
孙龙.2018.星天牛嗅觉相关基因的鉴定与表达分析[D].合
肥:安徽农业大学.[Sun L.2018.Identification and expres- sion profile analysis of olfactory genes in Anoplophora chi- nensis (Forster) [D]. Hefei:Anhui Agricultural University.]
Tofael Ahmed.2014.腰带长体茧蜂嗅觉相关蛋白基因的鉴定
和功能验证[D].北京:中国农业科学院.[Ahmed T.
2014. Identification and functional characterization of olfactory related genes in Macrocentrus cingulum[D]. Beijing: Chinese Academy of Agricultural Sciences Dissertation.]
王予彤,徐林波,段立清,于良斌,崔进,曹媛媛,赵一安.
2021.绿眼赛茧蜂转录组分析及嗅觉相关蛋白基因的鉴 定[J].应用昆虫学报,58(4):846-855.[Wang YT,Xu L B, Duan L Q, Yu L B, Cui J, Cao Y Y, Zhao Y A. 2021. Transcriptome sequencing and identification of the Zele chlorophthalmus olfactory related protein gene [J]. Chinese Journal of Applied Entomology, 58 (4) : 846-855.] doi: 10.
7679/j.issn.2095-1353.2021.082.
吴振晨.2022.桃红颈天牛触角嗅觉基因的鉴定与表达分析
[D].合肥:安徽农业大学.[Wu Z C.2022.Identification and expression profile of chemosensory genes in Aromia bungii (Faldermann) antennal transcriptome [D]. Hefei:
Anhui Agricultural University.] doi: 10.26919/d.cnki. gan-
nu.2022.000868.
詹文会.2018.金银花蛀干害虫咖啡脊虎天牛嗅觉相关基因 鉴定及表达谱分析[D].贵阳:贵州师范大学.[Zhan W H. 2018. Identification of olfactory related genes and expression profile analysis in Xylotrechus grayi of Honey- suckle stem pest [D]. Guiyang: Guizhou Normal Unive- rsity.]
张信哲.2022.小贯小绿叶蝉嗅觉受体基因的鉴定与表达分
析[D].泰安:山东农业大学.[Zhang XZ.2022.Identifi- cation and expression analysis of olfactory receptor genes
in Empoasca onuki Matsuda[D]. Tai'an: Shandong Agricultural University.] doi: 10.27277/d.cnki.gsdnu.2022.000950.
赵航,吴国星,汤永玉,张曼,梁晨,邢孔政,兰明先,高熹.
2022.叉角厉蝽触角转录组及嗅觉相关基因分析[J].
环境昆虫学报,44(5):1205-1217.[Zhao H,Wu GX, Tang Y Y,Zhang M,Liang C,Xing K Z,Lan M X,Gao X.
2022. Analysis of the antennal transcriptome and olfactionrelated genes of Eocanthecona furcellata [J]. Journal of Environmental Entomology, 44 (5) : 1205-1217.] doi: 10.3969/j.issn.1674-0858.2022.05.15.
郑海霞,张耀文,张仙红,杨美红.2018.绿豆象触角转录组及 嗅觉相关基因的分析[J].昆虫学报,61(2):168-177.
[Zheng H X, Zhang Y W,Zhang X H, Yang M H. 2018. Analysis of the antennal transcriptome and olfactionrelated genes of Callosobruchus chinensis (Coleoptera: Bruchuidae) [J]. Acta Entomologica Sinica, 61 (2) : 168177.] doi: 10.16380/j.kcxb.2018.02.003.
周渊涛.2019.亚洲小车蝗嗅觉相关蛋白的鉴定及功能研究 [D].呼和浩特:内蒙古农业大学.[Zhou Y T.2019.Iden- tification and function analysis of olfactory proteins in Oedaleus asiaticus (Orthopera: Acrididae) [D]. Hohhot: Inner Mongolia Agricultural University.]
Brito N F, Moreira M F, Melo A C. 2016. A look inside odorantbindingproteins in insect chemoreception [J]. Journal of Insect Physiology,95 (12) :51-65. doi: 10.1016/j.jinsphys.2016.09.008.
Kaupp U B. 2010. Olfactory signalling in vertebrates and
insects: Differences and commonalities[J]. Nature Reviews Neuroscience, 11: 188-200.
Wang X X, Liu H Y, Xie G L, Wang W K, Yang Y X. 2023. Identification and expression analyses of the olfactoryrelated genes in different tissues' transcriptome of a predacious soldier beetle,Podabrus annulatus (Coleoptera,Cantharidae)[J]. Archives of Insect Biochemistry and Physiology,112:e21997. doi: 10.1002/arch.21997.
Wu G X,Su R R,Ou Y H,Zheng X L,Lu W,Wang X Y. 2022. Antennal transcriptome analysis and identification of olfactory genes in Glenea cantor Fabricius (Cerambycidae: Lamiinae)[J]. Insects, 13(6): 553. doi: 10.3390/insects
13060553. Zhang Y Y, Guo J M,Wei Z Q,Zhang X T,Liu S R,Guo H F, Dong S L. 2022. Identification and sex expression profiles of olfactory-related genes in Mythimna loreyi based on antennal transcriptome analysis[J]. Journal of Asia-Pacific Entomology , 25 (3) : 101934. doi : 10.1016/j.aspen.2022.
101934. Zhu J, Arena S,Spinelli S, Liu D Z,Zhang G Q, Wei R P,Cambillau C, Scaloni A, Wang G R, Pelosi P. 2017. Reverse chemical ecology: Olfactory proteins from the giant panda and their interactions with putative pheromones and bamboo volatiles [J]. Proceedings of the National Academy of Sciences of the United States of America, 114(46): E9802-
E9810. doi:10.1073/pnas.1711437114.
(责任编辑麻小燕)
