巴氏新小绥螨转录组SSR位点分析及开发
2024-01-01刘辉李霞乌恩常静李志平霍志家李桂英武建华






摘要:【目的】开发巴氏新小绥螨(Neoseiulus barkeri)高效氯氟氰菊酯抗药和敏感品系的微卫星(SSR)分子标记, 为其抗药性基因遗传分析和抗药性品系的快速鉴定及筛选提供理论基础。【方法】应用MISA对巴氏新小绥螨转录组 已注释的Unigenes数据进行搜索,高通量挖掘该螨的SSR位点。使用Primer 5.0进行SSR引物设计,随机挑选其中 25对引物对巴氏新小绥螨高效氯氟氰菊酯抗性和敏感品系SSR位点基因进行PCR扩增,用重复性和稳定性高的12对引物进行实时荧光定量PCR分析基因表达。【结果】2个品系共获得52741条Unigenes,且SSR位点数量和分布密度一致。共筛选出5483个SSR位点,分布在4276条Unigenes中,发生频率为8.11%。主要重复类型为三核苷酸重复,占SSR总数的43.75%。共发现81种重复基元,其中单核苷酸重复基元A/T出现频率最高,占总量的19.65%。基于筛选出的SSR,共设计出4570对SSR引物,随机挑选的25对引物中有23对可扩增出巴氏新小绥螨高效氯氟氰菊酯抗性和敏感品系的目的基因片段。实时荧光定量PCR扩增结果显示,巴氏新小绥螨高效氯氟氰菊酯抗性品系有8个SSR基因位点的表达量高于敏感品系,其中TRINITY_DN24111_cO_g1基因的相对表达量为4.11,极显著高于敏感品系(1.06) (Plt;0.001,下同);有4个SSR位点基因的相对表达量低于敏感品系,其中TRINITY_DN20624_cO_g1基因的相对表达量为0.68,极显著低于敏感品系(1.01)。【结论】巴氏新小绥螨转录组SSR位点多态性丰富,已筛选开发出的12对在巴 氏新小绥螨抗性和敏感品系中表达量不同的SSR特异性引物可应用于对各地巴氏新小绥螨种群对高效氯氟氰菊酯 抗药性的快速检测。
关键词:巴氏新小绥螨;SSR;高通量测序;序列分析;表达量
中图分类号:S476.2
文章编号:2095-1191(2024)03-0689-10
文献标志码:A
Analysis and development of SSR loci based on transcriptome of Neoseiulus barkeriLIU Hui1, LI Xia1, WU En2, CHANG Jing1*, LI Zhi-ping2, HUO Zhi-jia1,LI Gui-ying3, WU Jian-hua4
(1College of Horticulture and Plant Protection, Inner Mongolia Agricultural University, Hohhot, Inner Mongolia 010019, China;2Agriculture and Animal Husbandry Technology Promotion Center of Inner Mongolia AutonomuousRegion, Hohhot, Inner Mongolia 010020, China;3Hohhot Agriculture and Animal Husbandry TechnologyPromotion Center, Hohhot, Inner Mongolia 010020, China;4Bayannnur Modern Agriculture and Animal Husbandry Development Center, Bayannnur, Inner Mongolia 015001, China)
Abstract: 【Objective】 The aim of this study was to develop microsatellite (SSR) molecular markers for Lambda- cyhalothrin resistant and susceptible strains of Neoseiulus barkeri, and to provide theoretical basis for genetic analysis of resistance genes and rapid identification and screening of resistant strains. 【Method】All unigenes from the transcriptome of N. barkeri were scanned using MISA software and its microsatellite (SSR) loci was screened by high-throughput. SSR primers were designed using Primer 5.0 software, 25 of these primer pairs were randomly selected for PCR amplification of the SSR loci genes for Lambda-cyhalothrin resistance and susceptible strains, and real-time fluorescence quantitative PCR gene expression analysis was conducted with 12 primer pairs with high reproducibility and stability. 【Result 】A totalof 52741 unigenes were obtained from the two strains, and the number and density of SSR loci were consistent. A total of 5483 SSR loci were detected from transcriptome, whose occurrence frequency was up to 8.11%, the analysis found that these loci were distributed in 4276 unigenes. SSR repeat types was mainly trinucleotide repeats, which accounted for 43.75% of the total number of SSR. A total of 81 kinds of repeat motifs were found, A/T was the most dominant motif in nucleotide repeats, accounting for 19.65% of the total, and 4570 SSR primers were designed, 23 pairs out of all 25 pairs randomly selected SSR primers could amplify the target gene fragments of both Lambda-cyhalothrin resistant and susceptible strains of N. barkeri. The results of real-time fluorescence quantitative PCR showed that the expression of 8 SSR loci genes of the Lambda-cyhalothrin resistant strains was higher than that of the susceptible strains , and the relative expression of TRINITY_DN24111_cO_gl gene was 4.11, extremely significantly higher than that of the susceptible strains(1.06) (Plt;0.001, the same below). There were 4 SSR loci genes with lower relative expression than the susceptible strains, the relative expression of TRINITY_DN20624_c0_gl gene was 0.68, extremely significant smaller than the susceptible strains (1.01). 【Conclusion】The SSR loci in the transcriptome of N. barkeri are rich in information.The screened and developed 12 SSR specific primer pairs that express at different levels in N. barkeri resistant and sensitive lines can be applied to the rapid detection of Lambda-cyhalothrin resistance of N. barkeri.
Key words: Neoseiulus barkeri;SSR;high-throughput sequencing;sequence analysis;expression level
Foundation items: Natural Science Foundation of Inner Mongolia Autonomuous Region (2023LHMS03016) ; Post-graduate Research Innovation Project of Inner Mongolia Autonomuous Region (S20231111Z)
0 引言
【研究意义】“以虫治虫”的生物防治方法是高效 且对环境友好的害虫综合防治方法之一。目前,全 球有230多种商品化天敌昆虫,其中蜱螨类天敌有 30余种(Chen et al.,2023),巴氏新小绥螨(Neosei- ulus barkeri)在许多农业生态系统中广泛应用于小 型害虫、螨及线虫的防治(王蔓等,2019;侯栋元等, 2020;王恩东等,2020;张丹等,2021;Azandeme-Hounmalon et al.,2022),被称为最有效的蜱螨类生物防治天敌之一(Tian et al.,2019)。但巴氏新小绥 螨对多种杀虫(螨)剂都非常敏感(张倩等,2022),经 常在化学药剂防治害虫(螨)的同时被杀伤,甚至死 亡。因此,室内筛选培育抗药性巴氏新小绥螨品系 并在田间释放,对螨虫防控具有极高的应用价值。 为实现这一目标,本研究在室内筛选巴氏新小绥 螨高效氯氟氰菊酯抗性品系的基础上,对巴氏新小 绥螨抗性和敏感品系进行转录组高通量测序,并对 其简单重复序列(Simple sequence repeats,SSR)特征 进行分析和基因验证,研究结果对巴氏新小绥螨 SSR分子标记开发、抗药性遗传功能分析和抗药性捕食螨快速鉴定及筛选等具有重要意义。【前人研究 进展】SSR又称微卫星(Microsatellite)DNA或简单 序列长度多态性(Simple sequence length polymor- phism),是由1~6个核苷酸基序多次串联重复而形 成的一段DNA序列(Kalia et al.,2011;刘昭阳等,2016),广泛存在于真核生物的核基因组中(Jarneand Lagoda,1996),该基因具有共显性遗传、高多态 性、变异性和跨物种转移能力的适宜性特征,可用于 许多生物类群的群体遗传学和生态学研究(柯富士,2013; Nigam et al., 2017; Simonato et al., 2019) 。Ouyang等(2021)对桔小实蝇进行转录组测序,并对SSR进行筛选和验证,结果显示,随机选择的80对SSR引物扩增出条带清晰、多态性良好的引物41对,为开发高多态性SSR分子标记研究桔小实蝇群体遗传结构和遗传多样性打下了基础。Yang等 (2021)根据已筛选获得的刺槐叶瘿蚊广腹细蜂(Platygaster robiniae)的SSR分子标记,选取采自丹 东和成都的2个地理分离群体对其遗传分化情况进 行研究,结果发现,所应用的SSR中有34个具有多 态性,平均存在4个基因型,且2个种群中的部分个 体单独聚类在1个分支上,表明2个群体间存在基因 流动和遗传分化。目前许多昆虫在转录组测序基础 上成功筛选到SSR位点,如桔小实蝇(Bactrocera dorsalis)(魏丹丹等,2014)、东方粘虫(Mythimna separata)(李微等,2017)、印度谷螟(Plodia interpunc- tella)(唐培安等,2017)、甜菜夜蛾(Spodoptera exigua)(王明明等,2021)、褐软蜡蚧(Coccus hesperi- dum)(李冬芝等,2022)、中华大仰蝽(Notonecta chi- nensis)(李敏等,2022)和李白盾蚧(Pseudaulacaspis prunicola)(杜炫星等,2023)等,这些研究结果将为各类昆虫种群遗传结构、昆虫特定基因定位及基因 功能分析等研究打下良好基础。【本研究切入点】本 研究团队前期筛选获得了巴氏新小绥螨高效氯氟氰 菊酯的抗药性品系,并应用杂交回交方式初步分析 了其遗传方式(常静等,2021),但该方法只能初步推 测害虫(螨)的抗药性是单基因控制还是多基因控 制,缺乏真正的分子水平验证,且用时长,数据处理 繁琐。【拟解决的关键问题】应用MISA对巴氏新小 绥螨高效氯氟氰菊酯抗性和敏感品系转录组数据中的SSR位点进行挖掘,筛选出与巴氏新小绥螨抗药性形成密切相关的SSR分子标记,为巴氏新小绥螨 抗药性基因遗传分析和抗药性品系的快速鉴定及筛 选提供理论基础。
1材料与方法
1.1试验材料
巴氏新小绥螨敏感品系(CS):于内蒙古农业大 学园艺与植物保护学院有害昆虫生物防治实验室, 在不接触任何杀虫(螨)剂的条件下,采用甜果螨(Carpoglyphus lactis)饲养繁殖120余代。巴氏新小绥螨高效氯氟氰菊酯抗性品系(CR):从亲代中挑选巴氏新小绥螨雌成螨,以亚致死浓度(LC2)的高效 氯氟氰菊酯原药处理雌成螨,经过21代筛选,获得 相对于敏感种群57.9倍抗性的抗药性品系。
1.2DNA提取
分别收集2个品系巴氏新小绥螨雌成螨,使用 天根DP304动物基因组DNA提取试剂盒提取巴氏新小绥螨基因组DNA,提取的DNA经NanoDropOne/OneC微量核酸蛋白浓度测定仪[赛默飞世尔科 技(中国)有限公司)]检测合格后,于-20°℃保存备用。
1.3转录组数据
巴氏新小绥螨转录组第2代高通量测序委托生 工生物工程(上海)股份有限公司完成。
1.4SSR筛选
应用MISA从巴氏新小绥螨高效氯氟氰菊酯敏 感品系的Unigenes中筛选SSR位点,对筛选的SSR 进行统计分析。筛选标准:单碱基重复次数≥10、 2碱基重复次数≥6、3~6碱基重复次数≥5,复合型 SSR间隔不超过100bp。
1.5SSR位点验证
1.5.1引物设计
基于筛选的SSR,使用Primer5.0进行SSR引物设计。引物设计标准:引物长度16~22bp;产物长度111~261bp;引物退火温度53~ 60℃。为验证所设计的引物能否稳定扩增,从设计 出的引物中随机挑选25对引物进行验证。引物由 北京擎科生物科技有限公司合成。
1.5.2PCR反应PCR反应
体系20.0μL:巴氏新 小绥螨DNA模板2.0 uL,2×Tag Master Mix 10.0 uL,上、下游引物(10umol/L)各0.8μL,ddH2O补足至20.0μL。扩增程序:95℃预变性5min;95℃30s,55℃45s,72℃1min,进行35个循环;72℃延伸 5min,4℃保存。PCR产物用1%琼脂糖凝胶电泳 检测。
1.5.3实时荧光定量PCR检测
使用MonAmpTM SYBR®Green qPCR Mix试剂盒(莫纳生物科技有限公司)进行实时荧光定量PCR检测。反应体系20.0 uL:MonAmpTM SYBR® Green qPCR Mix 10.0 uL,上、下游引物(10umol/L)各0.4μL、cDNA模板2.0μL,ddH2O7.2μL。扩增程序:95℃预变性30s;95℃10s,60°℃30s,进行40个循环。使用2AAa法计算 12个基因的相对表达量。
2结果与分析
2.1巴氏新小绥螨SSR基元数量及分布密度
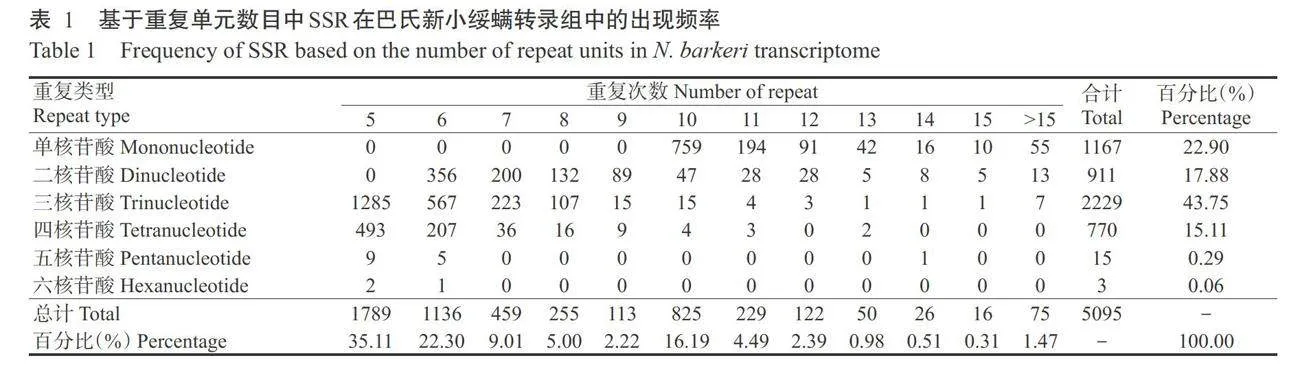
巴氏新小绥螨高效氯氟氰菊酯抗性和敏感品系 转录组测序组装结果显示,2个品系共获得52741条 Unigenes,且SSR位点数量和分布密度一致。应用MISA对其进行检索,共找到5483个SSR位点,分布 在4276条Unigenes中,发生频率(含有SSR的Unige-nes数量与总Unigenes数量之比)为8.11%。其中含1个SSR位点的Unigenes序列有3487条,含2个及以上SSR位点的Unigenes序列有789条,以复合型形式存在的SSR序列有388条。
由表1可看出,巴氏新小绥螨转录组SSR重复 类型丰富,共存在6种核苷酸重复,其中三核苷酸重 复在总SSR中出现频率最高,为43.75%;其次是单核苷酸,占SSR总数的22.90%;五核苷酸和六核苷 酸重复的含量少,仅为0.29%和0.06%。巴氏新小绥螨转录组SSR位点各重复类型的重复次数各不相 同,重复次数主要集中在5~15次,所占比例为98.51%; 大于15次的仅占总数的1.47%。其中各重复类型重 复次数为5次的SSR占比最大,为35.11%。
在巴氏新小绥螨转录组数据中,SSR基元长度 主要集中于10~20bp(图1)。其中,序列长度为13~ 20bp的SSR有2929个,所占比例最多,为53.42%;其次是长度为10~12bp的SSR,有1400个,占SSR总 数的25.53%;序列长度大于20bp的数量最少,仅有 1154个,占SSR总数的21.05%,且多数为复合型SSR。
2.2巴氏新小绥螨SSR分布特点
在巴氏新小绥螨转录组SSR中共获得81种重 复基元。单核苷酸重复中,有A/T、C/G2种基元,其中A/T基元在巴氏新小绥螨转录组SSR中的出现频率最高,占总数的19.65%;二核苷酸重复中,有AT/TA、AC/GT、AG/CT、TC/GA、TG/CA和CG/GC共6种基元,其中TC/GA和AG/CT基元占比较多,分 别占总数的5.36%和5.30%;三核苷酸重复中包含30种基元,其中CAG/CTG、TGC/GCA和TTG/CAA基元占比较多,分别占总数的4.04%、3.96%和3.10%; 四核苷酸重复中的基元类型最多,共有37种,但各类 基元均占比较小;五核苷酸重复有AAAAC/AACAA/ ATTTA、 AAGTG/TCGAC/TGGTA 和 TGATT/CGGGA/ CGCCG等5种基元类型,六核苷酸重复仅有ATGGTT/ATGGAG/GTAAAG1种基元类型,各类型五核 苷酸基元与六核苷酸基元占比相同,均为总量的 0.06%(表2)。
2.3巴氏新小绥螨SSR引物设计与PCR验证
根据已设置好的参数,共设计出4570对SSR引 物。随机挑选25对引物对巴氏新小绥螨高效氯氟 氰菊酯抗性和敏感品系DNA进行扩增,结果(表3 和图2)显示,23对引物在巴氏新小绥螨高效氯氟氰菊酯抗性和敏感品系中均可成功扩增出与预期片段 大小一致的特异性片段,TRINITY_DN25202_cO_g9(编号13)和TRINITY_DN22589_cO_g1(编号17)均 未扩增出特异性条带。
2.4巴氏新小绥螨高效氯氟氰菊酯抗性和敏感 品系SSR位点基因相对表达量分析结果
选取表3中前12对引物,应用实时荧光定量PCR 测定巴氏新小绥螨抗药和敏感品系位点SSR基因表达情况,结果(图3)显示,12对引物在巴氏新小绥螨 高效氯氟氰菊酯抗性和敏感品系中扩增出的基因相 对表达量各不相同。高效氯氟氰菊酯抗性品系的TRINITY_DN20904_cO_g1 TRINITY_DN24134_c1 g2、TRINITY_DN23943_c1_g1、TRINITY_DN24111c0_g1、TRINITY_DN26079_c0_g1、TRINITY_DN22 260_cO_gl、TRINITY_DN25142_cl_g3和TRINITY DN16203_c0_g1等8个基因的相对表达量高于敏感 品系,其中TRINITY_DN24111_cO_g1基因的相对表达量为4.11,极显著(Plt;0.001,下同)高于敏感品系(1.06)。抗性品系的TRINITY_DN20624_c0_g1、 TRINITY_DN20334_c0_g1、 TRINITY_DN14906_cO g1和TRINITY_DN3574_c0_g1等4个基因的相对表达 量低于敏感品系,其中TRINITY_DN20624_c0_g1基因 的相对表达量为0.68,极显著低于敏感品系(1.01)。
3讨论
SSR分子标记挖掘及其在昆虫进化过程和多样 性研究以及控制害虫保护益虫等方面具有重要的应 用价值。本研究通过高通量测序技术获得巴氏新小 绥螨转录组52741条Unigenes,在其中的4276条Unigenes中找到5483个SSR位点,SSR发生频率为8.11%。通常不同物种的SSR发生频率存在较大差 异,这与物种本身的特异性密切相关,同时也因高通 量测序标准不同、SSR筛选时参数设置不同或数据库挖掘工具不同而不同(Deng et al.,2018)。如扶桑绵粉蚧(Phenacoccus solenopsis Tinsley)为5.79% (罗梅等,2014)、桔小实蝇(Bactrocera dorsalis)为4.00%(魏丹丹等,2014)、沙葱萤叶甲(Galeruca dau-rica)为4.53%(张鹏飞等,2016)、齿缘刺猎蝽(Sclo- mina erinacea)为4.99%(黎东海和赵萍,2019),这些 昆虫的SSR发生频率均较低;而窄足真蚋[Simulium (Eusimulium)angustipes](郭欢等,2018)、意大利蝗(Calliptamus italicus)(桑迪等,2020)和灰茶尺蠖(Ectropis grisescens)(王定锋等,2021)的SSR发生 频率均较高,分别为36.05%、17.58%和27.59%。
前人研究发现,具有大量短重复基元的物种具 有相对较高的进化水平(Tóth et al.,2000)。本研究结果显示,巴氏新小绥螨转录组SSR重复类型丰富,单核苷酸重复、二核苷酸重复、三核苷酸重复和四核 苷酸重复占所有重复类型的99.64%,具有绝对优 势,可见巴氏新小绥螨处于较高的生物分类阶层,具 有相对高的进化水平。巴氏新小绥螨转录组SSR重 复类型中最主要的是三核苷酸(43.75%),与德国小 蠊(Blattella germanica)(王晨等,2015)、二点委夜蛾 (Athetis lepigone)(Li et al.,2013)和云南切梢小蠹(Tomicus yunnanensis)(袁远等,2014)等昆虫一致, 三核苷酸在蛋白质编码区通常较其他重复单元类型 更稳定,很少产生编码框移动突变,且三核苷酸重 复是除单核苷酸重复外编码基因表达序列标签 (Expressed sequence tags,ESTs)中最丰富的微卫星, 可为生物遗传信息研究提供更丰富的基因表达信 息,对研究其代谢、抗逆性等生物过程的基因表达调 控具有更重要价值(Xu et al.,2012)。沙棘木蠹蛾 (Eogystia hippophaecolus)(崔明明等,2017)、意大利 蜜蜂(Apis mellifera ligustica)(郭睿等,2018)、梨小食心虫(Grapholitha molesta)(冷春蒙等,2018)和拟 环纹豹蛛(Pardosa pseudoannulata)(Li et al.,2020) 等具有丰富的二核苷酸重复类型,西花蓟马(Frankli-niella occidentalis)(段惠生等,2012)和家蚕(Bom- byx mori)(王晖等,2022)以单核苷酸为主,蜜蜂 (Apis)(李斌等,2004)以六核苷酸重复类型为主。可见,物种间SSR丰度、碱基组成和优势碱基类型的 差异与物种种类具有一定相关性。
为验证SSR的扩增稳定性,通常会在设计的所有SSR引物中随机挑选数个进行验证。郭睿等 (2018)随机挑选意大利蜜蜂的24对特异性引物对 3个不同来源的意大利蜜蜂幼虫进行SSR位点扩 增,有22对成功扩增出目的片段,证明利用转录组 鉴定SSR位点的方法可行且高效。冷春蒙等(2018) 在随机选择的20对梨小食心虫SSR引物中发现6对 可在2个不同地区的梨小食心虫种群中扩增出符合 预期的特异性片段,证明根据转录组数据开发出的 SSR分子标记可行。本研究从设计的4570对引物 中随机挑选25对引物对巴氏新小绥螨高效氯氟氰 菊酯抗性和敏感品系进行PCR扩增,选取12对引物 对巴氏新小绥螨高效氯氟氰菊酯抗性和敏感品系进 行实时荧光定量PCR验证,结果显示25对引物中有 23对引物能稳定扩增,结果显示8个基因在抗药性品系中表达量高于敏感品系,4个基因在敏感品系中 表达量高于抗性品系,这为巴氏新小绥螨高效氯氟 氰菊酯抗性遗传SSR分子标记筛选提供了更有价值 的信息,证明预测的SSR的实用性,同时也验证了转 录组数据的可靠性。
4结论
巴氏新小绥螨转录组中SSR数据全面充实,位 点数量多,出现频率较高,基元类型丰富,有助于开 发更多适宜的SSR遗传标记。已筛选出的12对在 巴氏新小绥螨不同品系中表达量不同的SSR特异性 引物可应用于对各地巴氏新小绥螨种群对高效氯氟 氰菊酯抗药性的快速检测。
参考文献(References):
常静,李子奇,白琳,刘喆,董晓涵,孟瑞霞.2021.巴氏新小绥 螨溴氰菊酯抗性品系的筛选及其抗性遗传方式研究[J].
应用昆虫学报,58(6):1409-1416.[ChangJ,LiZQ,Bai L, Liu Z, Dong X H, Meng R X. 2021. A deltamethrin resistant strain of Neoseiulus barkeri: Selection for resis- tance and genetic analysis [J]. Chinese Journal of Applied Entomology,58(6):1409-1416.] doi:10.7679/j.issn.2095-1353.2021.142.
崔明明,陶静,宗世祥.2017.基于转录组的沙棘木蠹蛾简单 重复序列特征分析[J].环境昆虫学报,39(3):605-610.
[Cui M M, Tao J, Zong S X. 2017. Feature analysis of simple sequence repeats in Eogystia hippophaecolus transcriptome [J]. Journal of Environmental Entomology, 39
(3):605-610.] doi:10.3969/j.issn. 1674-0858.2017.03.15.
杜炫星,牛敏敏,赵清,蔡波,卢运运,魏久锋,张虎芳.2023.
李白盾蚧转录组分析及SSR位点开发[J].环境昆虫学
报,45(5):1318-1322.[Du XX,Niu M M,Zhao Q,Cai B, Lu Y Y, Wei J F,Zhang H F. 2023. Analysis of the tran- scriptome and development of SSR loci in Pseudaulacas- pis prunicola (Maskell) [J]. Journal of Environmental En- tomology,45(5):1318-1322.] doi:10.969/j/issn.1674-0858.
2023.05.18.
段惠生,张安盛,赵传志,于毅,褚栋.2012.西花蓟马EST-
SSR信息分析、标记筛选及其与Genomic-SSR的多态性
比较[J].昆虫学报,55(6):634-640.[Duan H S,Zhang A S, Zhao C Z, Yu Y, Chu D. 2012. Characterization and molecular marker screening of EST-SSRs and their poly- morphism compared with genomic-SSRs in Frankliniella occidentalis (Thysanoptera: Thripidae) [J]. Acta Entomo- logica Sinica,55(6):634-640.] doi: 10.16380/j.kcxb.2012.06.004.
郭欢,王刚,张树田,黄敏.2018.基于RNA-Seq数据的窄足真
蚋SSR分子标记开发[J].昆虫学报,61(7):815-824.[Guo
H,Wang G,Zhang S T,Huang M. 2018. Development of SSR primers for Simulium (Eusimulium) angustipes (Diptera: Simuliidae) based on RNA-Seq dataset[J]. Acta Entomologica Sinica, 61 (7) : 815-824.] doi: 10.16380/j.kcxb.2018.07.008.
郭睿,陈华枝,庄天艺,熊翠玲,郑燕珍,付中民,陈恒,陈大 福.2018.利用转录组数据开发意大利蜜蜂的SSR分子标记[J].安徽农业大学学报,45(3):404-408.[Guo R, Chen H Z, Zhuang T Y,Xiong C L,Zheng Y Z,Fu Z M, Chen H, Chen D F. 2018. Exploitation of SSR markers for Apis mellifera ligustica based on transcriptome data [J]. Journal of Anhui Agricultural University,45 (3) :404-408.]
doi:10.13610/j.cnki.1672-352x.20180620.015.
侯栋元,丛林,陈飞,杨娟生,周浩楠,成禄艳,于士将,雷双,
刘浩强,冉春.2020.巴氏新小绥螨甲氰菊酯抗性品系生 物学特性及其对常用药剂的交互抗性[J].应用昆虫学
报,57(3):690-699.[Hou D Y,Cong L,Chen F,YangJS, Zhou H N, Cheng L Y, Yu S J, Lei S, Liu H Q, Ran C.
2020. Biological characteristics of a fenpropathrin-resistant strain of Neoseiulus barkeri including cross-resistance to commonly used pesticides in orange orchards [J]. Chinese Journal of Applied Entomology,57(3):690-699.] doi: 10.
7679/j.issn.2095-1353. 2020.070.
柯富士.2013.小菜蛾转录组微卫星序列的分析和多态性位 点的开发[D].福州:福建农林大学.[Ke F S.2013.Chara- cterization and isolation of microsatellites from the Plutella xylostella transcriptome[D]. Fuzhou: Fujian Agri- culture and Forestry University.]
冷春蒙,李引,胡迪,仵均祥,李怡萍.2018.梨小食心虫幼虫 中肠转录组及SSR分子标记分析[J].昆虫学报,61(11):
1272-1283. [Leng C M,Li Y,Hu D,Wu J X, Li Y P. 2018. Analysis of the larval midgut transcriptome and SSR markers in Grapholitha molesta (Lepidoptera: Tortricidae) [J].
Acta Entomologica Sinica, 61 (11) : 1272-1283.] doi: 10.
16380/j.kcxb.2018.11.004.
李斌,夏庆友,鲁成,周泽扬.2004.蜜蜂EST中的微卫星分析
(英文)[J].遗传学报,31(10):1089-1094.[Li B,Xia Q
Y, Lu C, Zhou Z Y. 2004. Analysis of microsatellites derived from bee ESTs[J]. Acta Genetica Sinica,31(10):
1089-1094.]
黎东海,赵萍.2019.基于转录组数据的齿缘刺猎蝽微卫星分 子标记开发[J].昆虫学报,62(6):694-702.[LiDH,Zhao
P. 2019. Development of microsatellite markers based on the transcriptome data of Sclomina erinacea (Heteroptera: Reduviidae) [J]. Acta Entomologica Sinica, 62 (6) : 694702.] doi:10.16380/j.kcxb.2019.06.005.
李冬芝,张美霞,单伊曼,王芳.2022.基于转录组的褐软蜡蚧 微卫星分子标记开发[J].河北师范大学学报(自然科学
版),46(5):502-511.[Li D Z,Zhang M X,Shan Y M, Wang F. 2022. Development of microsatellite markers based on the transcriptome data of Coccus hesperidum (Hemiptera: Coccidae) [J]. Journal of Hebei Normal Uni- versity (Natural Science),46(5):502-511.] doi: 10.13763/ j.cnki.jhebun.nse.202204011.
李敏,张丹丽,李荣荣,雷廷,宋鲜梅,卜文俊.2022.中华大仰 蝽转录组学研究及SSR新标记开发[J].昆虫学报,65
(1):31-43. [Li M,Zhang D L, Li R R, Lei T,Song X M, Bu W J. 2022. Analysis of the transcriptome and development of novel SSR markers in Notonecta chinensis (Hemiptera: Notonectidae)[J]. Acta Entomologica Sinica, 65 (1): 31-43.] doi: 10.16380/j.kcxb.2022.01.004.
李微,张蕾,程云霞,罗礼智,江幸福.2017.应用转录组测序 高通量发掘东方粘虫SSR标记[J].植物保护学报,44
(3):377-384.[Li W,Zhang L,Cheng Y X,Luo L Z,Jiang X F. 2017. High-throughput discovery of microsatellite markers based on transcriptome sequencing in the oriental armyworm, Mythimna separata (Walker) [J]. Journal of Plant Protection, 44 (3) : 377-384.] doi: 10.13802/j. cnki.
zwbhxb.2017.2016058.
刘昭阳,李玉蓉,潘忠玉,郭冰,骆有庆,宗世祥,陶静.2016.
光肩星天牛两型种群表型多样性分析[J].应用昆虫学
报,53(5):1045-1057.[LiuZY,LiYR,Pan ZY,Guo B, Luo Y Q, Zong S X, Tao J. 2016. Phenotypic diversity of
Anoplophora glabripennis(Motschulsky) [J]. Chinese Journal of Applied Entomology, 53 (5) : 1045-1057.] doi: 10.
7679/j.issn.2095-1353.2016.129.
罗梅,张鹤,宾淑英,林进添.2014.基于转录组数据高通量发 掘扶桑绵粉蚧微卫星引物[J].昆虫学报,57(4):395-
400. [Luo M, Zhang H, Bin S Y, Lin J T. 2014. High-
throughput discovery of SSR genetic markers in the mealybug, Phenacoccus solenopsis (Hemiptera: Pseudococcidae) , from its transcriptome database [J]. Acta Entomologica Sinica, 57(4):395-400.] doi: 10.16380/j.kcxb.2014.
04.005.
桑迪,徐叶,王伟亮,向敏,季荣,王晗.2020.基于转录组数据 的意大利蝗微卫星位点分析与分子标记开发[J].应用昆
虫学报,57(3):658-666.[Sang D,Xu Y,Wang W L, Xiang M, Ji R, Wang H. 2020. Analysis of microsatellite loci from Calliptamus italicus (Orthopera: Acrididae) based on a transcriptome dataset [J]. Chinese Journal of Applied Entomology, 57 (3) : 658-666.] doi: 10.7679/j. issn.2095-
1353.2020.066.
唐培安,陶冶心,薛昊,袁明龙.2017.基于转录组数据的印度 谷螟微卫星位点分析[J].植物保护,43(3):43-48.[Tang P A, Tao Y X, Xue H, Yuan M L. 2017. Analysis of micro- satellite loci in Plodia interpunctella based on transcrip- tome dataset[J]. Plant Protection, 43 (3) : 43-48.] doi: 10.
3969/j.issn.0529-1542.2017.03.00.
王晨,杜联明,李鹏,杨茗羽,李午佼,沈咏梅,张修月,岳碧 松.2015.德国小蠊全基因组中微卫星分布规律[J].昆
虫学报,58(10):1037-1045.[Wang C,Du L M,Li P, Yang M Y, Li W J,Shen Y M,Zhang X Y, Yue B S. 2015. Distribution patterns of microsatellites in the genome of the German cockroach (Blattella germanica) [J]. Acta En- tomologica Sinica, 58 (10) : 1037-1045.] doi: 10.16380/j.
kcxb.2015.10.001.
王定锋,李良德,李慧玲,李金玉,吴光远.2021.基于转录组 数据高通量发掘灰茶尺蠖微卫星标记[J].茶叶学报,62
(4):191-197.[Wang D F,Li L D,Li H L,Li J Y,Wu G Y.
2021. High-throughput unveiling of microsatellite markers using Ectropis grisescens transcriptome[J]. Acta Tea Sinica,62(4):191-197.] doi:10.3969/j.issn.1007-4872.2021.
04.006.
王恩东,吴圣勇,吕佳乐,姜晓环,马兆义,刘亚杰,刘振州,赵 宏玉,吴朔,吴霞,徐学农.2020.释放巴氏新小绥螨防治 温室大棚番茄上的烟粉虱[J].植物保护,46(4):234- 238.[Wang E D, Wu S Y, Lü J L,Jiang X H,Ma Z Y,Liu Y J,Liu Z Z,Zhao H Y,Wu S,Wu X,Xu X N. 2020. Con- trol of whitefly Bemisia tabaci on tomato plants in green- house by releasing Neoseiulus barkeri [J]. Plant Protec- tion,46(4):234-238.] doi:10.16688/j.zwbh.2019151.
王晖,王敬,高妍夏,杨帆,马宝骏,古苗,张乘云,薛翠翠,谢 岩.2022.基于家蚕转录组测序的SSR序列分析[J].山 东农业科学,54(1);14-20.[Wang H,WangJ,GaoY X, Yang F, Ma B J,Gu M,Zhang C Y,Xue C C,Xie Y. 2022. SSR sequence analysis based on Bombyx mori transcrip-
tome sequencing [J]. Shandong Agricultural Sciences, 54
(1):14-20.] doi:10.14083/j.issn.1001-4942.2022.01.003.
王蔓,李波,黄婕,门兴元,季洁,刘永杰,尹淑艳.2019.加州 新小绥螨和巴氏新小绥螨对二斑叶螨的捕食能力比较 [J].应用昆虫学报,56(6):1256-1263.[Wang M,Li B, Huang J, Men X Y,Ji J, Liu Y J, Yin S Y. 2019. Compari-son of Neoseiulus californicus and Neoseiulus barkeri as biological controls for Tetranychus urticae [J]. Chinese Journal of Applied Entomology, 56 (6) : 1256-1263.] doi:
10.7679/j.issn.2095-1353.2019.136.
王明明,王思琦,孟威,姜策,江幸福,付晓伟,王小奇,王兴 亚.2021.基于SSR分子标记的辽宁地区甜菜夜蛾遗传
变异与种群遗传结构[J].应用昆虫学报,58(5):1143-
1151.[Wang M M,Wang S Q,Meng W,Jiang C,Jiang X F,Fu X W,Wang X Q,Wang X Y. 2021. Genetic variation and population genetic structure of the beet armyworm,
Spodoptera exigua (Lepidoptera: Noctuidae) , in Liaoning, based on microsatellite marker variation[J]. Chinese Journal of Applied Entomology, 58 (5) : 1143-1151.] doi: 10.
7679/j.issn.2095-1353. 2021.115.
魏丹丹,石俊霞,张夏瑄,陈世春,魏冬,王进军.2014.基于转
录组数据的桔小实蝇微卫星位点信息分析[J].应用生态
学报,25(6):1799-1805.[Wei DD,ShiJX,Zhang XX, Chen S C, Wei D, Wang J J. 2014. Analysis of microsatel- lite loci from Bactrocera dorsalis based on transcriptome dataset [J]. Chinese Journal of Applied Ecology, 25 (6) :
1799-1805.] doi: 10.13287/j.1001-9332.20140409.022.
袁远,张丽芳,吴国星,朱家颖.2014.云南切梢小蠹微卫星的 高通量发掘[J].环境昆虫学报,36(2):166-170.[Yuan
Y, Zhang L F, Wu G X, Zhu J Y. 2014. High-throughput discovery microsatellites in Tomicus yunnanensis (Coleoptera: Scolytinae) [J]. Journal of Environmental Entomology,36(2):166-170.] doi:10.3969/j.issn.1674-0858.2014.02.7.
张丹,林文忠,刘巍巍,孟威,仇贵生,曲智.2021.巴氏新小绥 螨对苹果树苹果全爪螨的生物防治效果[J].中国果树,
(7):62-64. [Zhang D, Lin W Z, Liu W W, Meng W, Qiu G S, Qu Z. 2021. Biocontrol effect of Neoseiulus barkeri on Panonychus ulmi in apple trees [J]. China Fruits, (7) : 62-64.] doi:10.166 26/j.cnki.issn1000-8047.2021.07.013.
张鹏飞,周晓榕,庞保平,谭瑶,常静,高利军.2016.基于转录 组数据高通量发掘沙葱萤叶甲微卫星引物[J].应用昆虫
学报,53(5):1058-1064.[Zhang PF,Zhou XR,Pang B P, Tan Y, Chang J, Gao L J. 2016. High-throughput disco- very of microsatellite markers in Galeruca daurica (Cole- optera: Chrysomelidae) from a transcriptome database[ J].
Chinese Journal of Applied Entomology , 53 (5) : 10581064.] doi: 10.7679/j.issn. 2095-1353.2016.130.
张倩,成立新,温彩霞,常静,孟瑞霞,李海平,王振,戴桂香.
2022.巴氏新小绥螨溴氰菊酯抗药性品系对常见药剂的 交互抗性及其种群参数分析[J].植物保护,48(3):126- 130. [Zhang Q, Cheng L X, Wen C X, Chang J, Meng R X, Li H P, Wang Z, Dai G X. 2022. Cross-resistance to commonly used pesticides and analysis of population parameters in a deltamethrin-resistant strain of Neoseiulus barkeri[J]. Plant Protection, 48 (3) : 126-130.] doi: 10.
16688/j.zwbh.2021495.
Azandeme-Hounmalon G Y, Sikirou R, Onzo A, Fiaboe K K M, Tamo M, Kreiter S, Martin T. 2022. Re-assessing the pest status of Tetranychus evansi (Acari: Tetranychidae) on solanaceous crops and farmers control practices in Benin [J]. Journal of Agriculture and Food Research, 10: 100401. doi: 10.1016/j.jafr.2022.100401.
Chen J Y,Zheng L J, Ye Z P,Wang J Y,Zhang F P,Fu Y G, Zhang C H. 2023. Evaluation of predatory mite Neoseiulus barkeri against spider mites damaging rubber trees [J].
Insects,14(7):648. doi:10.3390/insects 14070648. Deng K P, Deng R J, Fan J X, Chen E F. 2018. Transcriptome analysis and development of simple sequence repeat (SSR) markers in Zingiber striolatum Diels [J]. Physiology and Molecular Biology of Plants, 24 (1) : 125-134. doi: 10.
1007/s12298-017-0485-0.
Jarne P, Lagoda P J L. 1996. Microsatellites, from molecules to populations and back [J]. Trends in Ecology and Evolu-
tion,11(10):424-429. doi:10.1016/0169-5347(96)10049-5.
Kalia R K, Rai M K, Kalia S, Singh R, Dhawan A K. 2011. Microsatellite markers: An overview of the recent progress in plants [J]. Euphytica, 177 (3) : 309-334. doi: 10.1007/
s10681-010-0286-9.
Li C C, Li G Y, Wang Y,Peng Y. 2020. Development of SSR markers based on transcriptome sequences of the Wolf spider Pardosa pseudoannulata (Araneae:Lycosidae) [J]. Entomology News,129:6-15. doi:10.3157/021.129.0102.
Li L T,Zhu Y B,Ma J F, Li Z Y, Dong Z P. 2013. An analysis of the Athetis lepigone transcriptome from four developmental stages[J]. PLoS One, 8 (9) : e73911. doi: 10.1371/ journal.pone.0073911.
Nigam D, Saxena S, Ramakrishna G, Singh A, Singh N K, Gaikwad K. 2017. De novo assembly and characterization of Cajanus scarabaeoides (L.) thouars transcriptome by pairedend sequencing[J]. Frontiers in Molecular Biosciences,4: 00048. doi:10.3389/fmolb.2017.00048.
Ouyang H L,Wang X Y,Zheng X L, Lu W,Qin F P,Chen C.
2021. Full-length SMRT transcriptome sequencing and
SSR analysis of Bactrocera dorsalis (Hendel) [J]. Insetc, 12(10):938-955. doi:10.3390/insects12100938.
Simonato M, Pilati M, Magnoux E, Courtin C, Sauné L, Rous- selet J, Battisti A, Auger R, Marie A, Kerdelhu C. 2019. A population genetic study of the egg parasitoid Baryscapus servadeii reveals large scale automictic parthenogenesis and almost fixed homozygosity[J]. Biological Control, 139:104097. doi:10.1016/j.biocontrol.2019.104097.
Tian C B, Li Y Y, Wang X,Fan W H, Wang G, Liang J Y, Wang Z Y, Liu H. 2019. Effects of UV-B radiation on the survival, egg hatchability and transcript expression of antioxidant enzymes in a high-temperature adapted strain of Neoseiulus barkeri [J]. Experimental and Applied Acaro-
logy,77(4):527-543. doi:10.1007/s10493-019-00361-9.
Tóth G, Gáspári Z, Jurka J. 2000. Microsatellites in different
eukaryotic genomes: Survey and analysis [J]. Genome Research,10(7):967-981. doi:10.1101/gr.10.7.967.
Xu Y,Zhou W W, Zhou Y J, Wu J X, Zhou X P. 2012. Transcriptome and comparative gene expression analysis of Sogatella furcifera (Horváth) in response to southem rice black-streaked dwarf virus [J]. PLoS One, 7(4) : e36238.
doi: 10.1371/journal.pone.0036238.
Yang J,Huang L,Li Z R,Sun H Q,Zhao W X, Yao Y X. 2021. Development and preliminary application of novel genomewide SSR markers for genetic deversity analysis of an economically important bio-control agent Platygaster robiniae (Hymenoptera: Platygastridae) [J]. Journal of Gene-
tics,100(2):67-74. doi:10.1007/s12041-021-01318-x.
