基于转录组测序的关岭牛肌肉生长发育关键基因筛选与鉴定
2024-01-01周迪赵忠海王府杨蓉王燕敖叶谢玲玲陈秋生田兴舟李波







摘要:【目的】基于转录组测序挖掘出贵州关岭牛肌肉生长发育关键基因,为后续培育贵州优质肉牛品种提供理 论依据。【方法】选取3头健康且体重相近的24月龄成年关岭牛,屠宰后采集其心脏、肝脏、脾脏、肺脏、肾脏、背最长肌、 大腿肌及肩峰等组织样品,采用TRIzol法提取各组织总RNA,构建cDNA文库后进行转录组测序,通过生物信息学手段筛选出不同样品间的差异表达基因(DEGs),结合GO功能注释分析和KEGG信号通路富集分析挖掘出关岭牛肌肉生长发育相关候选基因,再经实时荧光定量PCR对候选基因进行验证。【结果】测序样品有效序列(Clean reads)比对 至参考基因组序列的平均比对率为90.76%,检测到26367个新转录本(有编码潜能的转录本19659个,非编码转录本 6708个);在各组织中检测到共表达基因53912个,其中已知基因51688个、预测新基因2224个;GO功能注释分析发现这些共表达基因主要发挥生物调节、代谢过程、生物膜组成、催化活性和转录调节活性等功能。通过DEGseq检测,最 终筛选获得16个与肌肉生长发育相关的DEGs(QKI、CHKB、MYBPCI、MBNLI、MYHII、MYLK2、TPM3、NEXN、EZH2、 TNNT3、PPARGCIA、SGCA、DMPK、FOXPI、FLNB和TNS2),主要参与内吞作用、心肌收缩、癌症、代谢及MAPK信号 通路。实时荧光定量PCR验证结果显示,筛选出的16个DEGs在关岭牛背最长肌或肩峰中高表达,与转录组测序结果一致。【结论】基于转录组测序从关岭牛各组织中筛选出16个与肌肉生长发育相关的DEGs,主要在生物调节、代谢过 程、细胞成分、催化和转录调节等方面发挥功能作用,可作为开展关岭牛肌肉生长发育遗传机制研究的候选基因。
关键词:关岭牛;肌肉生长发育;肌肉性状;差异表达基因(DEGs);转录组测序
中图分类号:S823.81
文献标志码:A
文章编号:2095-1191(2024)03-0611-12
Screening and validation of the key genes for muscle growth and development in Guanling cattle based on transcriptomesequencing
ZHOU Di1, ZHAO Zhong-hai2, WANG Fu1, YANG Rong1, WANG Yan1, AO Ye1, XIE Ling-ling1, CHEN Qiu-sheng2, TIAN Xing-zhou3, LI Bo4*
(1Testing Center for Livestock and Poultry Germplasm, Guiyang, Guizhou 550018, China; 2Animal Husbandry and Fishery Industry Office of Zunyi City, Zunyi, Guizhou 563006, China; 3College of Animal Science, Guizhou Univer-sity/Key Laboratory of Animal Genetics, Breeding and Reproduction of Plateau Mountain Areas, Ministry of Education, Guiyang, Guizhou 550025, China; 4Genetic Resources Management Station of Guizhou,Guiyang, Guizhou 550025, China)
Abstract:【Objective]The purpose of the study was to excavate the key genes related to muscle growth and develop- ment of Guizhou Guanling cattle based on transcriptome sequencing, so as to provide a theoretical basis for the subse-quent breeding of high-quality Guizhou beef cattle breeds. 【Method】 Three healthy 24-month-old adult Guanling cattle with similar body weight were selected, and their heart, liver, spleen, lung, kidney, longissimus dorsi muscle, thigh muscle and acromion tissues were collected after slaughter. TRIzol method was used to extract total RNA from each tis- sue, and transcriptome sequencing was performed after constructing cDNA library. The differentially expressed genes (DEGs) among different samples were screened by bioinformatics, and candidate genes related to muscle growth and de- velopment of Guanling cattle were extracted by combining GO functional annotation analysis and KEGG signaling path- way enrichment analysis, and then the candidate genes were verified by real-time fluorescence quantitative PCR. 【Result] The average mapped rate of clean reads to the reference genome sequence of the sequenced samples was 90.76%, and 26367 new transcripts (19659 with coding potential and 6708 with non-coding transcripts) were detected. A total of 53912 co-expressed genes were detected in each tissue, including 51688 known genes and 2224 predicted new genes. GO func- tional annotation analysis showed that these co-expressed genes mainly played the functions of biological regulation, metabolic process, membrane part, catalytic activity and transcriptional regulation activity. Through the DEGseq test, 16 DEGs related to muscle development were finally screened and obtained (QKI, CHKB, MYBPC1, MBNL1, MYH11,MYLK2, TPM3, NEXN, EZH2, TNNT3, PPARGC1A, SGCA, DMPK, FOXP1, FLNB and TNS2). They were mainlyinvolved in endocytosis, myocardial contraction, cancer, metabolism and MAPK signaling pathway. The results of realtime fluorescence quantitative PCR showed that all the 16 selected DEGs were highly expressed in longissimus dorsi muscle and acromion of Guanling cattle, which was consistent with transcriptome sequencing results. 【Conclusion】Six- teen DEGs related to muscle development are selected from various tissues of Guanling cattle based on transcriptome se- quencing, which mainly play functions in biological regulation, metabolic process, cell components, catalysis and tran- scriptional regulation, and can be used as candidate genes for conducting the study of genetic mechanism of muscle growth and development of Guanling cattle.
Key words: Guanling cattle; muscle growth and development; muscle traits; differentially expressed genes(DEGs); transcriptome sequencing
Foundation items: Guizhou High Level Innovation Talents Cultivation Project (QKHPTRC-GCC[2023)028); Gui-zhou Agricultural Production and Development Project(Qiannongmu2024)010)
0 引言
【研究意义】成肌能力是由动物本身的遗传潜 能所决定,肌肉形成如同其品质的pH、系水力、肌内 脂肪、剪切力、大理石花纹及肌肉纤维等一系列质 量性状,均由多基因共同调控。生长分化因子8(Growth differentiation factor 8,GDF8)负责调控牛 的双肌形成和肉品质(Ceccobelli et al.,2022)。Guo等(2017)研究发现,大多数lncRNAs与共表达基因 一起参与肌肉分化和细胞周期相关的生物学途径, 在调控肌肉分化和肉品质形成过程中发挥作用,因 此通过肉品质判定肌肉生长发育已逐步成为可能。 肉品质是一个综合性状,主要受肌内脂肪和肌纤维 含量的影响。肌纤维含量在胚胎发育过程已决定, 但不同品种牛的肌纤维含量和类型存在一定差异(Wegner et al.,2006;Listrat et al.,2019)。因此,在开 展贵州关岭牛肉品质研究的基础上,筛选并鉴定其 肌肉生长发育相关基因,对推进贵州关岭牛分子遗 传选育具有重要意义。【前人研究进展】随着分子生 物学和遗传学的快速发展,影响畜禽肉品质的相关基因不断被发现,如参与脂肪代谢的H-FABP、 A-FABP和I-FABP基因等(Nechtelberger et al.,2001; Binas et al.,2003;Wang et al.,2017;石鹏飞等,2022,2023),以及参与肌肉生成的生肌决定因子(MyoD)、肌细胞生成素基因(MyoG)、生肌因子5(Mvf5)和生肌因子6(Myf6)(Casas et al.,2004;Robakowska- Hyzorek et al.,2010;Fan et al.,2011)等。Li等(2018) 通过对2种肥尾绵羊的肾周、皮下和尾部脂肪组织 进行转录组测序分析,结果发现FABP4、ADIPOQ、 FABP5和CD36等是脂肪沉积的关键基因;刘娟 等(2021)利用加权基因共表达网络分析(Weighted gene co-expression network analysis,WGCNA)发掘影响猪肌肉发育和性状的基因,结果发现FAM131B、 RIC8B、PLEKHA5、PARP6和CBX7是影响肌肉发育和肌内脂肪沉积的关键基因;Kong等(2022)研究发现,LIPE基因与绵羊肌肉脂肪含量高度相关,且g.4819Agt;G具有潜在的遗传价值;Yu等(2023)通 过代谢组、转录组和蛋白组对我国地方猪种肉质 进行分析,结果发现差异表达基因(Differentiallyexpressed genes,DEGs)、差异丰度蛋白、差异积累代 谢产物等在Wnt、PI3K-Akt、Rap1和Ras等与肉质相 关的信号通路中富集。可见,高通量测序技术的发 展为挖掘地方品种特色优势性状基因提供了技术支 撑。【本研究切入点】关岭牛作为贵州地方黄牛品种, 具有耐粗饲、肉质鲜美、风味独特等优点(孙金魁等, 2022)。贵州关岭牛具有生产优质牛肉的潜能,但至今鲜见针对其肉质形成调控机理及肉质性状关键基 因研究的文献资料。【拟解决的关键问题】以贵州关 岭牛为研究对象,对其心脏、肝脏、脾脏、肺脏、肾脏、 背最长肌、大腿肌和肩峰组织进行转录组测序分析,筛选出组织间的DEGs,同时进行GO功能注释
分析和KEGG信号通路富集分析,旨在筛选出影响 贵州关岭牛肉质性状的关键基因,为后续培育优质 贵州肉牛品种提供理论依据。
1材料与方法
1.1试验材料
在贵州省关岭黄牛原种场选取3头健康且体重 相近的24月龄成年关岭牛,屠宰后采集其心脏、肝 脏、脾脏、肺脏、肾脏、背最长肌、大腿肌及肩峰等组 织样品,0.1%DEPC水冲净表面杂质后迅速投入液 氮中速冻备用。动物试验经贵州大学动物伦理委员 会批准,批准号EAE-GZU-2021-P019。组织总RNA提 取试剂盒购自美国OMEGA公司;SsoFast EvaGreen Supermix和反转录试剂盒购自北京康润诚业生物科 技有限公司;DL10000 DNA Marker购自宝生物工程 (大连)有限公司;质粒提取试剂盒及胶回收试剂盒 购自美国AxyGen公司;DMEM高糖培养基和胎牛 血清购自美国Gibco公司;FuGENE®HD Transfec- tion Reagent购自美国Promega公司。
1.2实时荧光定量PCR扩增引物设计与合成
根据GenBank已公布的基因序列,利用Primer premier5.0设计实时荧光定量PCR扩增引物(表1),以牛β-Actin为内参基因。所有引物委托昆泰锐(武 汉)生物技术有限责任公司合成。
1.3总RNA提取
通过TRIzol法分别提取关岭牛心脏、肝脏、脾脏、肾脏、背最长肌、肩峰和大腿肌组织总RNA,利 用微量紫外分光光度计检测其浓度和纯度,当待 检测样品OD260/OD280介于1.85~2.10,RNA完整值在
8.6~10.0,说明总RNA浓度和完整性较好。
1.4 cDNA文库建立及转录组测序
检测合格的总RNA采用mRNA富集法进行处 理,并以携带Oligo(dT)的磁珠富集有poly(A)尾的mRNA,通过离子打断方式将RNA打断成小片段,利用随机引物N6进行反转录,再合成cDNA第二链以获得双链DNA。将合成的双链DNA末端补平并进行5'端磷酸化,3'端形成突出1个“A”的黏性末端,再连接1个3'端有凸出“T”的鼓泡状接头,连接产物通过特异性引物进行PCR扩增,并将扩增产物热变性成单链,最后以桥式引物将单链DNA环化得到单链环状DNA文库。cDNA文库转录组测序委托深 圳华大基因科技有限公司完成。
1.5实时荧光定量PCR验证
从转录组测序结果中筛选出DEGs,利用实时荧光定量PCR进行验证,以cDNA为模板、β-Actin为 内参基因。每个样品设3次重复,使用2-4c法计算目 的基因相对表达量,并以SPSS18.0对基因相对表达量进行单因素方差分析(One-way ANOVA)。
1.6数据分析
对测序原始数据进行过滤和质控后得到有效序 列(Clean reads),使用HISAT将Clean reads 比对到 参考基因组序列(GCA 000247795.2,Nelore),并进行新转录本预测(Kim et al.,2015);通过StringTie重构每个样品的转录本(Pertea et al.,2015),以Cuff- compare将重构的转录本与参考注释信息进行比对 (Wu et al.,2016),并用CPC对新转录本进行编码潜力预测(Kong et al.,2007),运用GATK检测每个样品的单核苷酸多态性(SNP)(McKenna et al.,2010),使用rMATS检测样品间的差异剪接基因(DSGs) (Shen et al.,2014);利用Bowtie2将Clean reads比对 到参考序列(Campbell et al.,2008),通过RSEM计算基因和转录本的表达水平(Li and Dewey,2011),最后筛选出不同样品间的DEGs,并进行GO功能注释分析和KEGG信号通路富集分析。
2结果与分析
2.1参考基因组比对及新转录本预测结果
将Clean reads比对至参考基因组序列,结果(表2)显示,每个样品的Clean reads平均比对率为 90.76%,样品间均匀的比对率表明转录组测序数 据具有可比性。同时,共检测到26367个新转录本, 其中有编码潜能的转录本19659个、非编码转录本6708个。
2.2DSGs检测结果
与参考基因组序列比对之后,检测不同样品间 的DSGs,若样品间同一基因亚型的相对丰度存在差 异,说明该基因受剪接机制调控。共检测出5种可 变剪接事件调控的DSGs:Skipped exon(SE)、Alter-native 5' splicing site (A5SS)、 Alternative 3' splicing site(A3SS)、Mutually exclusive exons (MXE)和 Re- tained intron(RI)。每个样品的可变剪接事件结果 见图1。
2.3基因及转录本表达统计分析结果
通过RSEM计算基因和转录本的表达水平,结果表明:在心脏中检测到表达基因数平均为31787 个,其中已知基因30142个、预测新基因1645个;在 肩峰中检测到表达基因数平均为30934个,其中已 知基因29384个、预测新基因1550个;在肾脏中检测到表达基因数为34255个,其中已知基因32461个、 预测新基因1794个;在肝脏中检测到表达基因数平 均为30749个,其中已知基因28992个、预测新基因 1757个;在背最长肌中检测到表达基因数平均为30669个,其中已知基因29135个、预测新基因1534个; 在脾脏中检测到表达基因数平均为33899个,其中 已知基因32197个、预测新基因1702个;在大腿肌中 检测到表达基因数平均为30024个,其中已知基因 28524个、预测新基因1500个。对比整合重复基因,检测到共表达基因数53912个,其中已知基因51688个、预测新基因2224个。各样品的基因及转录本数目 统计结果见表3。
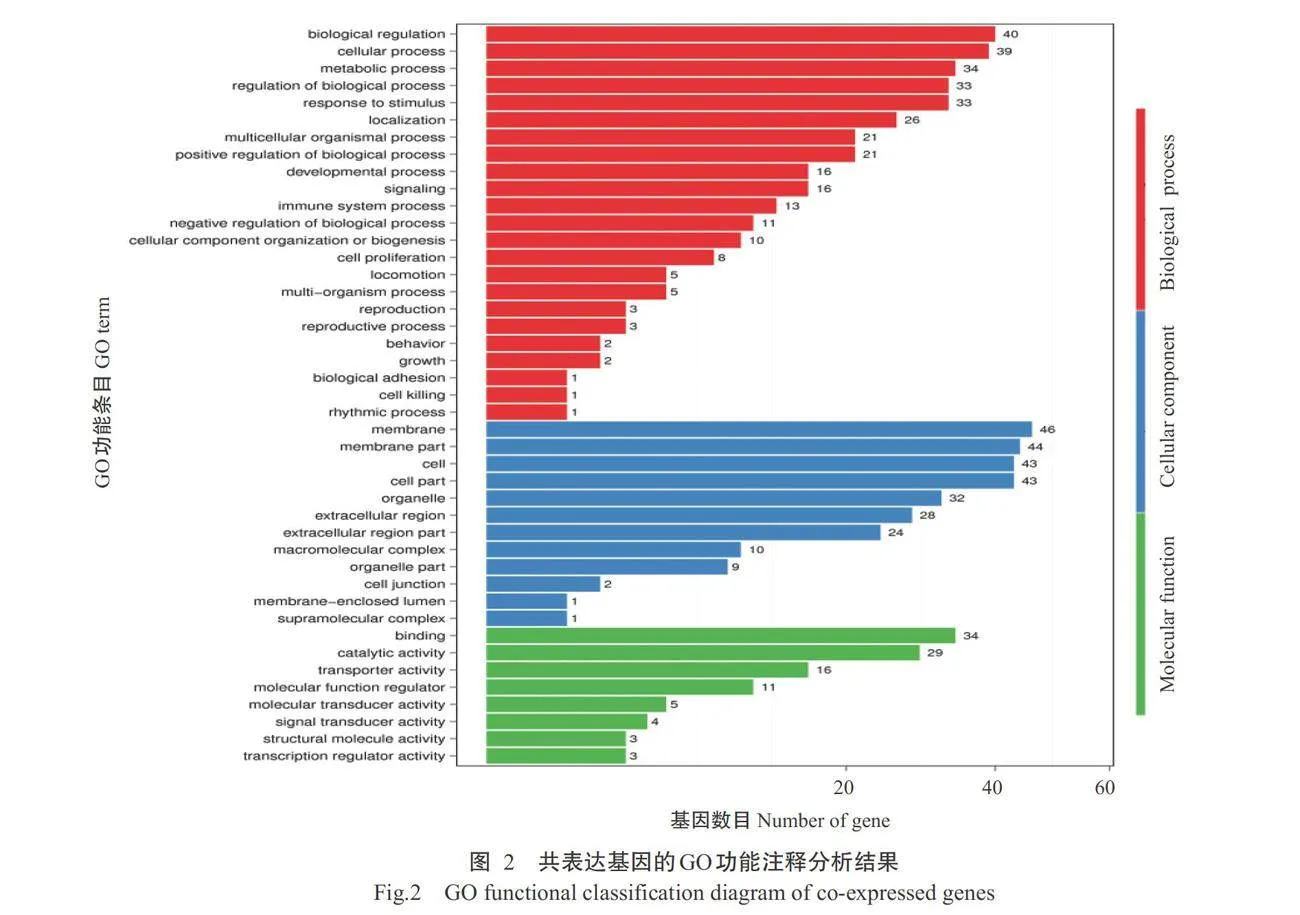
根据共表达基因检测结果,基于GO功能注释 分析进行分类,结果发现这些共表达基因主要发挥 生物调节(Biological regulation)、代谢过程(Meta- bolic process)、生物膜组成(Membrane part)、催化活性(Catalytic activity)及转录调节活性(Transcription regulator activity)等功能。由图2可知,共表达基因 共注释到732条GO功能条目,其中,344条GO功能条目属于生物学过程(Biological process),283条GO 功能条目属于细胞组分(Cellular component),105条 GO功能条目属于分子功能(Molecular function)。
2.4DEGs检测结果
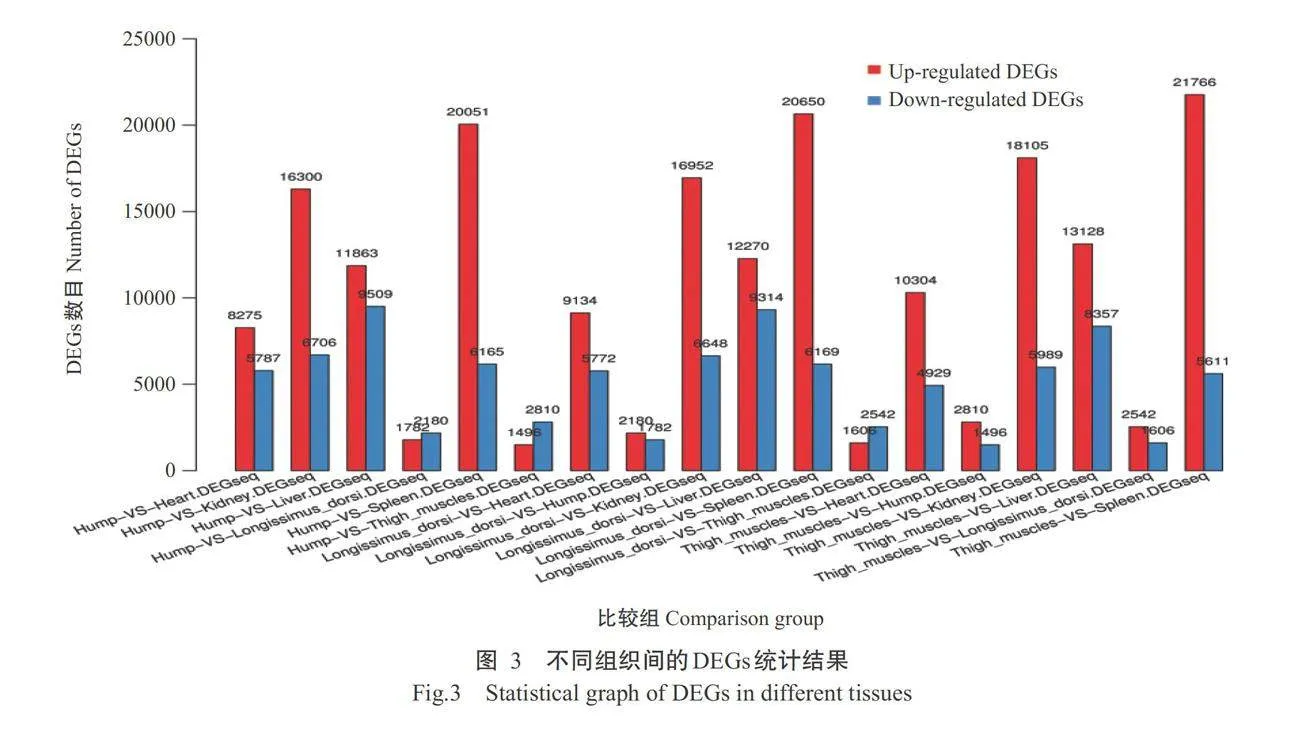
根据各样品基因表达水平,分别以肩峰、背最长 肌和大腿肌为参照,通过DEGseq对各样品DEGs进行检测,筛选出不同组织间的上调DEGs和下调DEGs(图3)。其中,与肌肉发育相关的基因有QKI、 CHKB、MYBPC1、MBNL1、MYH11、MYLK2、TPM3、 NEXN、EZH2、TNNT3、PPARGC1A、 SGCA、DMPK、FOXP1、FLNB和TNS2。
2.5DEGs的GO功能注释分析结果
根据DEGs检测结果进行GO功能注释分析,结 果发现DEGs注释到的GO功能条目也可分为生物 学过程、细胞组分和分子功能三大类。其中,生物学过程以细胞过程(Cellular process)、生物调节(Bio- logical regulation)、代谢过程(Metabolic process)、生 物过程调节(Regulation of biological process)和刺激 反应(Response to stimulus)等GO功能条目为主;细胞 组分主要涉及细胞组成(Cell part)、细胞器(Organe- lle)、膜(Membrane)、细胞器组成(Organelle part)和 大分子复合物(Macromolecular compex)等GO功能 条目;分子功能主要涉及结合(Binding)、催化(Cata- lytic)、转录调节(Transcripyion regulator activity)、信 号转导(Signal transducer activity)和分子功能调节 剂(Molecular function regulator)等GO功能条目。
2.6DEGs的KEGG信号通路富集分析结果
基于KEGG数据库对DEGs进行KEGG信号通 路富集分析,统计各KEGG信号通路不同层级上富集的DEGs数目,确定其主要参与的代谢途径及信号通路,结果(图4)发现主要参与内吞作用(Endocy-tosis)、心肌收缩(Dilated cardiomyopathy,hypertro- phic cardiomyopathy)、癌症(Chronic myeloid leuke- mia)、代谢(Fatty acid degradation,ubiquitin media- ted proteolysis, propanoate metabolism, nitrogen me- tabolism, valine/leucine and isoleucine degradation,primary bile acid biosynthesis)及MAPK信号通路(MAPK signaling pathway)。
2.7实时荧光定量PCR验证结果
对筛选获得的16个与肌肉发育相关的DEGs(QKI、CHKB、MYBPC1、MBNL1、MYH11、MYLK2、TPM3、NEXN、EZH2、TNNT3、PPARGC1A、SGCA、 DMPK、FOXPI、FLNB和TNS2)进行实时荧光定量PCR验证,结果(图5)显示:FOXPI基因在脾脏中的 相对表达量最高,在肝脏中的相对表达量最低; MYBPC1基因在脾脏中的相对表达量最高,在肺脏 中的相对表达量最低;DMPK基因在肾脏中的相对 表达量最高,在肺脏中的相对表达量最低;SGCA基 因在肾脏中的相对表达量最高,在肝脏中的相对表 达量最低;EZH2基因在肾脏中的相对表达量最高, 在心脏中的相对表达量最低;CHKB基因在肾脏中 的相对表达量最高,在脾脏中的相对表达量最低;PPPRGC1A、TNNT3、TNS2、MYLK2和NEXN基因均 表现为背最长肌中的相对表达量最高,脾脏中的相 对表达量最低;TPM3基因在背最长肌中的相对表达 量最高,在肝脏中的相对表达量最低;FLNB在肩峰 中的相对表达量最高,在心脏中的相对表达量最低; QKI和MYH11基因在肩峰中的相对表达量最高,在 肺脏中的相对表达量最低;MBNLI基因在肩峰中的 相对表达量最高,在肝脏中的相对表达量最低。
3讨论
牛肉具有高蛋白、低脂肪、低胆固醇等优点,除 了满足人体的营养物质需求外,已逐渐发展成为消 费者青睐的保健型肉类食品。影响牛肉品质的因素 包括遗传物质和环境因子,如品种、年龄、性别、基因 型及饲养管理等,而衡量牛肉品质的指标有嫩度、大 理石花纹、pH、系水力、肌内脂肪含量、脂肪酸组成和肌纤维粗细等(管鹏宇等,2019;陈浩等,2021;莫 兹源等,2023)。随着现代分子生物技术的快速发 展,与肉质相关的候选基因不断被挖掘,从DNA水平研究控制肉质性状的主效基因已成为可能。Oh等(2012)研究发现,FABP4可与长链脂肪酸及其他 疏水配体结合,在脂肪酸运输和脂质水解过程中起 关键作用,进而影响肉牛IMF含量、脂肪酸组成及胴体重。王丹丹等(2016)研究表明,MSTN作为肌肉 发育的负调控因子,其基因突变会增加肉牛的胴 体重,提高肉牛的屠宰性能和生产效率。此外, PPARY、CEBPA和RXRA作为核转录因子,是脂肪生 成和沉积过程中的重要调控基因,通过调控脂肪代 谢而影响肉品质的嫩度、肌内脂肪含量及脂肪酸组 成等(Goszczynski et al.,2016)。转录组测序在地方 家畜肉质性状相关基因筛选方面已得到广泛应用, 并取得一定成效,为地方畜禽品种在肉质、抗病性和 耐粗饲等优势性状的发掘利用提供了技术支撑,对 地方品种资源保护及遗传改良具有重要指导意义。Wu等(2013)对金华猪和长白猪背最长肌转录谱进行比较,发现FASN、SCD等基因在金华猪群体中参与脂肪合成,Myf6和Foxol基因在长白猪群体中参与调节肌肉生成。Chen等(2015)对69头肉牛肌肉 样本进行转录组测序,结果筛选出749个与大理石 花纹形成相关的DEGs,且涉及多个基因功能和生物 通路,进一步证实转录组测序数据可用于肉牛品种 改良及选育方案计划。为此,本研究基于转录组测 序技术筛选出关岭牛肌肉生长发育关键基因,以期 为后续培育优质贵州肉牛品种提供理论依据。
关岭牛作为贵州地方优良肉牛品种,在肉质性 状上优于安格斯和西门塔尔,但至今关于调控关岭 牛肉质性状的主效基因及信号通路机制尚未明确。 本研究通过对关岭牛各组织进行基因组测序,共检 测到26367个新转录本,其中预测具有编码能力的 转录本19659个,非编码转录本6708个;检测到共表达基因数53912个,其中已知基因51688个、预测新 基因2224个,且这些基因主要在生物调节、代谢过 程、细胞成分、催化和转录调节等方面发挥功能作 用。QKI、NEXN和MYHII基因对平滑肌细胞发育 及其表型具有调控作用(Li et al.,2003;Kuang et al.,2012;Zhu et al.,2018),这3个基因在关岭牛肩峰和 背最长肌中高表达,故推测其参与关岭牛肩峰和背最长肌的发育。MBNL1、TNS2、MYLK2和TPM3基 因与肌肉发育相关(Kanadia et al.,2003;Zhi et al.,2005;Wu et al.,2009;叶保国等,2014;Shan et al.,2019),且TPM3基因在动物发育过程中呈特异性表达,TNS2基因在肾功能、肌肉再生和细胞迁移过程中发挥关键作用。本研究中,MBNL1基因在关岭牛 肩峰中的相对表达量最高;TNS2和TPM3基因在背 最长肌中的相对表达量最高,其次是肩峰;MYLK2 基因在背最长肌中的相对表达量最高。由此可知 MBNL1、TNS2、MYLK2和TPM3基因可能都参与关岭牛的肌肉组织发育。CHKB、MYBPCI、TNNT3、FLNB、FOXPI和EZH2基因在骨骼肌的发育、能量代谢、结构和分化过程中发挥调控作用(Caretti et al.,2004;时伟红等,2012;Tong et al.,2015;Cesario et al.,2016;Turner et al.,2019)。CHKB和EZH2基 因在关岭牛肾脏中的相对表达量最高,FOXP1和MYBPCI基因在脾脏中的相对表达量最高,TNNT3 基因在背最长肌中的相对表达量最高,表明这些基 因在关岭牛的肌肉分化和肝脏代谢过程中起调控作用。SGCA基因(Quijano-Roy et al.,2012)和DMPK 基因(Jauvin et al.,2017)与肌病有关。DMPK基因 在关岭牛肾脏中的相对表达量最高,其次是肩峰; SGCA基因在肾脏中的相对表达量最高,其次是背最长肌。PPARGCIA基因参与多种生物过程,如参与 调节肌肉纤维类型的组成,其3'端非翻译区(3'- UTR)多态性影响肌纤维特性和肉质性状(Lee etal.,2012)。PPARGC1A基因在关岭牛背最长肌中的 相对表达量最高,暗示着其与关岭牛的肉品质有关。 本研究的实时荧光定量PCR验证结果显示,筛选出 的16个DEGs在关岭牛背最长肌或肩峰中高表 达,说明这些基因与关岭牛肌肉组织发育有关,可作 为开展关岭牛肌肉生长发育遗传机制研究的候选 基因。
4结论
基于转录组测序从关岭牛各组织中筛选 出16个与肌肉生长发育相关的DEGs(QKI、CHKB、 MYBPC1、MBNL1、MYH11、MYLK2、TPM3、NEXN、 EZH2、TNNT3、PPARGC1A、SGCA、DMPK、FOXP1、 FLNB和TNS2),主要在生物调节、代谢过程、细胞成 分、催化和转录调节等方面发挥功能作用,可作为开展关岭牛肌肉生长发育遗传机制研究的候选基因。
参考文献(References):
陈浩,王纯洁,斯木吉德,敖日格乐.2021.牛肉品质及其影响 因素研究进展[J].动物营养学报,33(2):669-678.[Chen
H, Wang C J, Smujid, Aogegle. 2021. Research progress on beef quality and its influencing factors[J]. Chinese Journal of Animal Nutrition, 33 (2) : 669-678.] doi: 10.3969/j.
issn.1006-267x.2021.02.008.
管鹏宇,张爱忠,姜宁.2019.牛肉品质影响因素的研究进展 [J].黑龙江畜牧兽医,(11):39-43.[Guan P Y,Zhang A Z, Jiang N. 2019. Research progress on factors influencing beef quality [J]. Heilongjiang Animal Science and Veteri- nary Medicine, (11):39-43.] doi:10.13881/j.cnki.hljxmsy.
2018.08.0134.
刘娟,王舒,左周,路畅,杨阳,蔡春波,赵燕,郭晓红,曹果清, 李步高,高鹏飞.2021.结合WGCNA鉴定与猪肌纤维和
肌内脂肪相关的中枢基因[J].山西农业大学学报(自然 科学版),41(4):109-118.[Liu J,Wang S,Zuo Z,Lu C, Yang Y, Cai C B,Zhao Y, Guo X H, Cao G Q,Li B G, Gao P F. 2021. Identification of hub genes associated with porcine fibers and intramuscular fat by WGCNA[J]. Jour- nal of Shanxi Agricultural University (Natural Science Edition),41(4): 109-118.] doi: 10.13842/j.cnki.issn1671-
8151.202102023.
莫兹源,谭昇,刘文友,李大刚,闵力,张志飞,王翀,童雄.
2023.肌纤维类型影响牛肉品质相关指标的研究进展 [J].中国畜禽种业,19(6):29-37.[Mo ZY,Tan S,Liu W Y, Li D G, Min L,Zhang Z F, Wang C, Tong X. 2023. Research progress on the influence of muscle fiber types on beef quality indexes [J]. The Chinese Livestock and Poultry Breeding, 19(6):29-37.] doi: 10.3969/j.issn.1673-4556.2023.06.007.
石鹏飞,阮涌,刘文娇,许家利,孙金魁,熊讯,许厚强.2022.
关岭牛FABP3和FABP4基因的分子特征及其组织表达 分析[J].南方农业学报,53(8):2281-2293.[Shi PF,Ruan Y, Liu W J, Xu J L, Sun J K, Xiong X, Xu H Q. 2022. Molecular characteristics of FABP3 and FABP4 genes and tissue expression analysis of Guanling cattle[J]. Journal of Southern Agriculture, 53(8) : 2281-2293.] doi: 10.3969/j.
issn.2095-1191.2022.08.021.
石鹏飞,许家利,孙金魁,许厚强.2023.关岭牛FABP1和FABP2基因克隆及其组织表达分析[J].南方农业学报,54(2):598-608. [Shi P F,Xu J L,Sun J K, Xu H Q. 2023. Cloning and tissue expression analysis of Guanling cattle FABP1 and FABP2 genes[J]. Journal of Southern Agriculture,54(2):598-608.] doi:10.3969/j.issn.2095-1191.2023.02.028.
时伟红,李兆阳,杨葳,李翀,刘京津,郑志红.2012.TNNT3
突变转基因小鼠筛选及肌钙蛋白表达的研究[J].现代肿
瘤医学,20(2):254-256.[Shi WH,Li ZY,Yang W,Li C, Liu J J, Zheng Z H. 2012. The screening to study of TNNT3 mutation transgenic mouse and the troponin TNNT3 (R69H) gene expression of fast skeletal muscle in TNNT3 mutation transgenic mouse[J]. Journal of Modern Oncology,20(2):254-256.] doi:10.3969/j.issn.1672-4992.2012.02.11.
孙金魁,许厚强,阮涌,宋林锦,许家利,陈晨,石鹏飞.2022.
关岭牛MEF2A和MEF2B基因克隆及其组织表达特征分 析[J].南方农业学报,53(9):2643-2653.[SunJK,XuH
Q, Ruan Y, Song L J, Xu J L, Chen C, Shi P F. 2022. Cloning and tissue expression analysis of MEF2A and MEF2B genes in Guanling cattle [J]. Journal of Southern Agricul-ture , 53 ( 9) : 2643-2653. ] doi : 10.3969/j.issn.2095-1191.2022.09.027.
王丹丹,刘晓牧,张冉,刘桂芬,万发春,林浴霜.2016.鲁西黄
牛MSTN基因上游序列多态性对转录表达的影响[J].中国畜牧兽医,43(7):1667-1673.[Wang D D,Liu X M, Zhang R, Liu G F, Wan F C, Lin Y S. 2016. Effect of MSTN gene upstream sequence polymorphism on tran- scriptional expression in Luxi Huang cattle [J]. China Ani- mal Husbandry amp; Veterinary Medicine,43(7):1667-1673.]
doi: 10.16431/j.cnki.1671-7236.2016.07.002.
叶保国,张小辉,徐铁山.2014.北京鸭和淮南麻鸭胸肌发育 过程TPM1和TPM3基因的表达变化研究[J].黑龙江畜 牧兽医,(19):67-70.[Ye B G,Zhang XH,Xu T S.2014.
Study on the changes in the TPMl and TPM3 gene expressions during the development of breast muscle in Peking duck and Huainan shelduck[J]. Heilongjiang Animal Science and Veterinary Medicine, (19):67-70.]
Binas B, Han X X, Erol E, Luiken J J F P, Glatz J F C, Dyck D J, Motazavi R, Adihetty P J,Hood D A, Bonen A. 2003. A 1 mutation in H-FABP only partially inhibits skeletal muscle fatty acid metabolism [J]. American Journal of Physiology. Endocrinology and Metabolism,285(3) :E481E489. doi: 10.1152/ajpendo.00060.2003.
Campbell P J, Stephens P J, Pleasance E D, O'Meara S, Li H, Santarius T,Stebbings L A, Leroy C,Edkins S, Hardy C, Teague J W, Menzies A, Goodhead I, Turner D J, Clee C M, Quail M A, Cox A, Brown C, Durbin R, Hurles M E, Edwards P A W, Bignell G R, Stratton M R, Futreal P A.
2008. Identification of somatically acquired rearrangements in cancer using genome-wide massively parallel pairedend sequencing[J]. Nature Genetics, 40: 722-729.
doi:10.1038/ng.128.
Caretti G, Di Padova M, Micales B, Lyons G E, Sartorelli V.
2004. The polycomb EZH2 methyltransferase regulates muscle gene expression and skeletal muscle differentiation [J]. Genes amp; Development, 18 (21) : 2627-2638. doi : 10.
1101/gad.1241904.
Casas E,Keele J W, Shackelford S D, Koohmaraie M, Stone R
T. 2004. Identification of quantitative trait loci for growth and carcass composition in cattle [J]. Animal Genetics, 35(1):2-6. doi:10.1046/j.1365-2052.2003.01067.x.
Ceccobelli S, Perini F, Trombetta M F, Tavoletti S, Lasagna E, Pasquini M. 2022. Effect of myostatin gene mutation on slaughtering performance and meat quality in marchigiana bulls[J]. Animals,12(4):518. doi:10.3390/ani12040518.
Cesario J M, Almaidhan A A, Jeong J. 2016. Expression of forkhead box transcription factor genes Foxpl and Foxp2 during jaw development[J]. Gene Expression Patterns, 20
(2):111-119. doi: 10.1016/j.gep.2016.03.001.
Chen D, Li W F, Du M, Wu M, Cao B H. 2015. Sequencing and characterization of divergent marbling levels in the beef cattle (Longissimus dorsi Muscle) transcriptome [J].
Asian-Australasian Journal of Animal Sciences, 28 (2): 158165. doi: 10.5713/ajas.14.0394.
Fan H T, Cinar M U, Phatsara C, Tesfaye D, Tholen E, Looft C, Schellander K. 2011. Molecular mechanism underlying the differential MYF6 expression in postnatal skeletal muscle of Duroc and Pietrain breeds[J]. Gene, 486 (1-2): 8-14. doi:10.1016/j.gene.2011.06.031.
Goszczynski D E, Mazzucco J P, Ripoli M V, Villarreal E L, Rogberg-Muñoz A, Mezzadra C A, Melucci L M, Giovam- battista G. 2016. Genetic characterisation of PPARG, CEBPA and RXRA, and their influence on meat quality traits in cattle [J]. Journal of Animal Science and Techno-
logy,58(1):14. doi:10.1186/s40781-016-0095-3.
Guo Y W,Wang J N,Zhu M F, Zeng R, Xu Z Y, Li G L, Zuo B. 2017. Identification of myod-responsive transcripts reveals a novel long non-coding RNA (lncrna-ak143003) that negatively regulates myoblast differentiation[J]. Scientific Reports,7(1):2828. doi:10.1038/s41598-017-03071-7.
Jauvin D, Chrétien J, Pandey S K, Martineau L, Revillod L, Bassez G, Lachon A, MacLeod A R, Gourdon G, Wheeler T M, Thornton C A, Bennett C F,Puymirat J. 2017. Targe- ting DMPK with antisense oligonucleotide improves muscle strength in myotonic dystrophy type 1 mice[J].
Molecular Therapy,7:465-474. doi: 10.1016/j.omtn.2017.05.007.
Kanadia R N, Urbinati C R, Crusselle V J, Luo D F, Lee Y J, Harrison J K, Oh S P, Swanson M S. 2003. Developmental expression of mouse muscleblind genes Mbnl1 , Mbnl2 and Mbnl3[J]. Gene Expression Patterns,3 (4) :459-462. doi:
10.1016/S1567-133X(03)00064-4.
Kim D, Langmead B, Salzberg S L. 2015. HISAT: A fast spliced aligner with low memory requirements [J]. Nature Methods,12(4):357-360. doi:10.1038/nmeth.3317.
Kong L, Zhang Y, Ye Z Q, Liu X Q,Zhao S Q, Wei L P, Gao
G. 2007. CPC: Assess the protein-coding potential of transcripts using sequence features and support vector machine [J]. Nucleic Acids Research, 35 (S2) : W345-W349. doi: 10.1093/nar/gkm391.
Kong Y Y, Yuan Z H, Liu X, Li F D, Yue X P. 2022. A novel snp within LIPE gene is highly associated with sheep intramuscular fat content[J]. Small Ruminant Research, 209: 106658. doi: 10.1016/j.smallrumres.2022.106658.
Kuang S Q,Kwartler C S,Byanova K L,Pham J, Gong L M, Prakash S K,Huang J,Kamm K E, Stull J T,Sweeney H L, Milewicz D M. 2012. Rare, nonsynonymous variant in the smooth muscle-specific isoform of myosin heavy chain, MYH11, R247C, alters force generation in the aorta and phenotype of smooth muscle cells [J]. Circulation Research, 110(11):1411-1422. doi:10.1161/CIRCRESAHA.111.261743.
Lee J S, Kim J M, Hong J S, Lim K S,Hong K C, Lee Y S.
2012. Effects of polymorphisms in the 3' untranslated region of the porcine PPARGClA gene on muscle fiber characteristics and meat quality traits [J]. Molecular Biology Reports,39(4):3943-3950. doi: 10.1007/s11033-011-1173-8.
Li B J,Qiao L Y,An L X,Wang W W,Liu J H,Ren Y S,Pan Y Y, Jing J J, Liu W Z. 2018. Transcriptome analysis of adipose tissues from two fat-tailed sheep breeds reveals key genes involved in fat deposition [J]. BMC Genomics, 19
(1):338. doi:10.1186/s12864-018-4747-1.
Li B,Dewey C N. 2011. RSEM: Accurate transcript quantification from RNA-Seq data with or without a reference genome[J]. BMC Bioinformatics, 12(1):323. doi: 10.1186/
1471-2105-12-323.
Li Z H, Takakura N, Oike Y, Imanaka T, Araki K, Suda T, Kaname T, Kondo T, Abe K, Yamamura K I. 2003. Defective smooth muscle development in qkI-deficient mice [J].
Development, Growth amp; Differentiation,45(5-6):449-462.
doi:10.1111/j.1440-169X.2003.00712.x.
Listrat A, Gagaoua M, Picard B. 2019. Study of the chronology of expression of ten extracellular matrix molecules during the myogenesis in cattle to better understand sensory properties of meat [J]. Foods, 8 (3): 97. doi: 10.3390/foods80
30097. McKenna A, Hanna M, Banks E, Sivachenko A, Cibulskis K, Kernytsky A, Garimella K, Altshuler D, Gabriel S, Daly M, DePristo M A. 2010. The genome analysis toolkit: A mapreduce framework for analyzing next-generation DNA sequencing data [J]. Genome Research, 20(9) : 1297-1303.
doi:10.1101/gr.107524.110.
Nechtelberger D, Pires V, Söolknet J, Stur I, Brem G, Mueller M, Mueller S. 2001. Intramuscular fat content and genetic variants at fatty acid-binding protein loci in Austrian pigs
[J]. Journal of Animal Science,79(11):2798-2804.
Oh D Y, Lee Y S, La B M, Yeo J S. 2012. Identification of the
SNP (single nucleotide polymorphism) for fatty acid composition associated with beef flavor-related FABP4 (fatty acid binding protein 4) in Korean cattle [J]. Asian-Australasian Journal of Animal Sciences,25(7) :913-921. doi:
10.5713/ajas.2012.12078.
Pertea M, Pertea G M, Antonescu C M, Chang T C, Mendell J T, Salzberg S L. 2015. StringTie enables improved reconstruction of a transcriptome from RNA-Seq reads[J]. Nature Biotechnology,33(3):290-295. doi:10.1038/nbt.3122.
Quijano-Roy S, Avila-Smirnow D, Carlier R Y. 2012. Whole
body muscle MRI protocol: Pattern recognition in early onset NM disorders[J]. Neuromuscular Disorders,22(S2): S68-S84. doi:10.1016/j.nmd.2012.08.003.
Robakowska-Hyzorek D, Oprzadek J, Zelazowska B, Olbromski R, Zwierzchowski L. 2010. Effect of the g.-723G→T polymorphism in the bovine myogenic factor 5 (Myf5) gene promoter region on gene transcript level in the longis- simus dorsi muscle and on meat traits of Polish Holstein- Friesian cattle[J]. Biochemical Genetics, 48(5) : 450-464.
doi:10.1007/s10528-009-9328-1.
Shan T Z, Liu J Q, Xu Z Y, Wang Y Z. 2019. Roles of phosphatase and tensin homolog in skeletal muscle [J]. Journal of Cellular Physiology,234(4) : 3192-3196. doi: 10.1002/jcp.
26820. Shen S H,Park J W,Lu Z X,Lin L,Henry M D,Wu Y N,Zhou Q, Xing Y. 2014. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data [J]. Proceedings of the National Academy of Sciences of the United States of America, 111 (51) : E5593-E5601.
doi:10.1073/pnas.1419161111.
Tong B, Muramatsu Y, Ohta T, Kose H, Yamashiro H, Sugiyama T, Yamada T. 2015. Association of the expression level of the MYBPCI gene in skeletal muscle with marbling trait in Japanese black beef cattle[J]. Annals of Animal Science, 15(2):349-361. doi: 10.1515/aoas-2015-0014.
Turner D C, Seaborne R A, Sharples A P. 2019. Comparative transcriptome and methylome analysis in human skeletal muscle anabolism, hypertrophy and epigenetic memory [J]. Scientific Reports, 9 (1) : 4251. doi: 10.1038/s41598-019-40787-0.
Wang Y,Chen H W,Han D G, Chen Y,Muhatai G,Kurban T, Xing J M, He J Z. 2017. Correlation of the A-FABP gene polymorphism and mRNA expression with intramuscular fat content in Three-Yellow chicken and Hetian-Black chicken [J]. Animal Biotechnology, 28 (1) :37-43. doi: 10.
1080/10495398.2016.1194288.
Wegner J, Albrecht E, Fiedler I, Teuscher F, Papstein H J, Ender K. 2006. Growth- and breed-related changes of muscle fiber characteristics in cattle[J]. Journal of Animal
Science,78(6):1485-1496. doi:10.2527/2000.7861485x.
Wu G S, Sher R B, Cox G A, Vance D E. 2009. Understanding the muscular dystrophy caused by deletion of choline kinase beta in mice[J]. Biochimica et Biophysica Acta.
Molecular and Cell Biology of Lipids, 1791(5) : 347-356.
doi: 10.1016/j.bbalip.2009.02.006.
Wu S Y,Tong X L,Peng C X,Xiong G,,Lu K P,Hu H,Tan D, Li C L, Han M J, Lu C, Dai F Y. 2016. Comparative analysis of the integument transcriptomes of the black dilute mutant and the wild-type silkworm Bombyx mori[J].
Scientific Reports,6(1):26114. doi:10.1038/srep26114.
Wu T,Zhang Z H,Yuan Z Q,Lo L J,Chen J,Wang Y Z,Peng J R, Wang H M. 2013. Distinctive genes determine different intramuscular fat and muscle fiber ratios of the longissimus dorsi muscles in Jinhua and Landrace pigs [J]. PLoS One,8(1):e53181. doi: 10.1371/journal.pone.0053181.
Yu T Y,Tian X K, Li D,He Y L,Yang P Y,Cheng Y,Zhao X, Sun J C, Yang G S. 2023. Transcriptome, proteome and metabolome analysis provide insights on fat deposition and meat quality in pig [J]. Food Research International, 166:112550. doi: 10.1016/j.foodres.2023.112550.
Zhi G,Ryder J W,Huang J,Ding P G,Chen Y,Zhao Y M, Kamm K E, Stull J T. 2005. Myosin light chain kinase and myosin phosphorylation effect frequency-dependent potentiation of skeletal muscle contraction [J]. Proceedings of the National Academy of Sciences of the United States of America, 102 (48) : 17519-17524. doi: 10.1073/pnas.0506
846102. Zhu B Y,Rippe C,Holmberg J,Zeng S H,Perisic L, Albinsson S,Hedin U,Uvelius B,Swärd K. 2018. Nexilin/NEXN con- trols actin polymerization in smooth muscle and is regu- lated by myocardin family coactivators and YAP[J]. Scien- tific Reports, 8 (1) : 13025. doi: 10.1038/s41598-018-31328-2.
(责任编辑
兰宗宝)
