生物信息学分析筛选肺腺癌靶基因及评估预后的价值
2019-12-27黄琪峰,郑琳琳,张菁
黄琪峰,郑琳琳,张菁

摘要:目的 基于生物信息学分析筛选肺腺癌靶基因及评估预后价值。方法 对三个数据集(GSE118370、GSE32863、TCGA-LUAD)分别使用limma和edgeR包筛选出肺腺癌差异表达基因,对共同差异基因进行功能富集分析,通过String数据库构建蛋白质-蛋白质相互作用(PPI)网络,采用Cytoscape进行可视化分析并用其插件cytoHubba来筛选关键基因,采用Kaplan-Meier曲线进行总体生存分析。结果 共224个共同差异基因,其中上调基因34个,下调基因190个。共同差异基因在血管生成、白细胞调节、免疫反应等生物学过程富集。通过从PPI网络中筛选出8个关键基因,分别为IL6、VWF、PECAM1、SPP1、CDH5、CXCL12、TIMP1、CLDN5。生存分析显示,PECAM1与LUAD的预后有关。肿瘤组织中PECAM1表达高于正常组织,差异有统计学意義(P<0.05)。结论 PECAM1是一种与肺腺癌预后相关的新的生物标志物,有望成为肺腺癌的一个治疗的靶点。
关键词:肺腺癌;差异表达基因;生物标志物;生物信息学分析
中图分类号:R734.2 文献标识码:A DOI:10.3969/j.issn.1006-1959.2019.22.020
文章编号:1006-1959(2019)22-0062-04
Bioinformatics Analysis of Screening Lung Adenocarcinoma Target Genes and
Evaluating Prognostic Value
HUANG Qi-feng1,ZHENG Lin-lin2,ZHANG Jing1
(Department of Pharmacy1,Department of Ultrasound2,Sir Run Run Shaw Hospital,Zhejiang University School of Medicine,
Hangzhou 310000,Zhejiang,China)
Abstract:Objective To screen the target genes of lung adenocarcinoma based on bioinformatics analysis and evaluate the prognostic value. Methods Three data sets (GSE118370, GSE32863, TCGA-LUAD) were used to screen differentially expressed genes in lung adenocarcinoma using limma and edgeR packages, functional enrichment analysis of common differential genes, and protein-protein interactions were constructed by String database. PPI) network, using Cytoscape for visual analysis and using its plug-in cytoHubba to screen key genes, Kaplan-Meier curve for overall survival analysis. Results A total of 224 common differential genes, including 34 up-regulated genes and 190 down-regulated genes. Common differential genes are enriched in biological processes such as angiogenesis, leukocyte regulation, and immune response. Eight key genes were screened from the PPI network, namely IL6, VWF, PECAM1, SPP1, CDH5, CXCL12, TIMP1, and CLDN5. Survival analysis showed that PECAM1 was associated with the prognosis of LUAD. The expression of PECAM1 in tumor tissues was higher than that in normal tissues,the difference was statistically significant (P<0.05). Conclusion PECAM1 is a new biomarker related to the prognosis of lung adenocarcinoma and is expected to be a target for the treatment of lung adenocarcinoma.
Key words:Lung adenocarcinoma;Differentially expressed genes;Biomarkers;Bioinformatics analysis
肺癌(lung cancer)是常见的恶性肿瘤,根据2018年全球癌症统计数据显示,肺癌占癌症总发生病例的11.6%,其死亡率占癌症导致死亡人数的18.4%[1]。肺腺癌是肺癌的一种,属于非小细胞肺癌,一般起源于细小支气管粘膜上皮,少数起源于大支气管的粘液腺,疾病发生的确切机制目前尚不明确[2]。肺腺癌的前期诊断以及预后预测均较困难,一些治疗方案对提高患者生存率的作用并不明显,因此深入了解肺腺癌发生机制对于预防肺腺癌非常重要。而早期肺癌生物标志物的筛选及其在肺癌发生发展中的作用、机制和应用研究对该病的早期诊断、治疗和预后评价具有重要意义[4]。基因芯片是一种高通量和系统性的研究技术,能检测和分析不同组织的差异表達基因。研究发现[5,6],肺癌中存在不同类型的生物标志物,包括编码基因、miRNAs、长非编码RNAs和circRNAs,这些分子的失调均参与了肿瘤的进展或与患者的预后相关。本研究利用TCGA数据库和GEO数据库,筛选出肺腺癌的差异表达基因,利用GO-KEGG富集分析、PPI网络分析等方法探索与肺腺癌进展有关的生物标志物,现报道如下。
1资料与方法
1.1数据下载 从TCGA数据库(https://portal.gdc.cancer.gov/)下载肺腺癌患者癌组织和癌旁组织的基因表达数据;从GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)下载基因表达谱数据(GSE118370、GSE32863),共123例正常样本和599例肺腺癌样本。
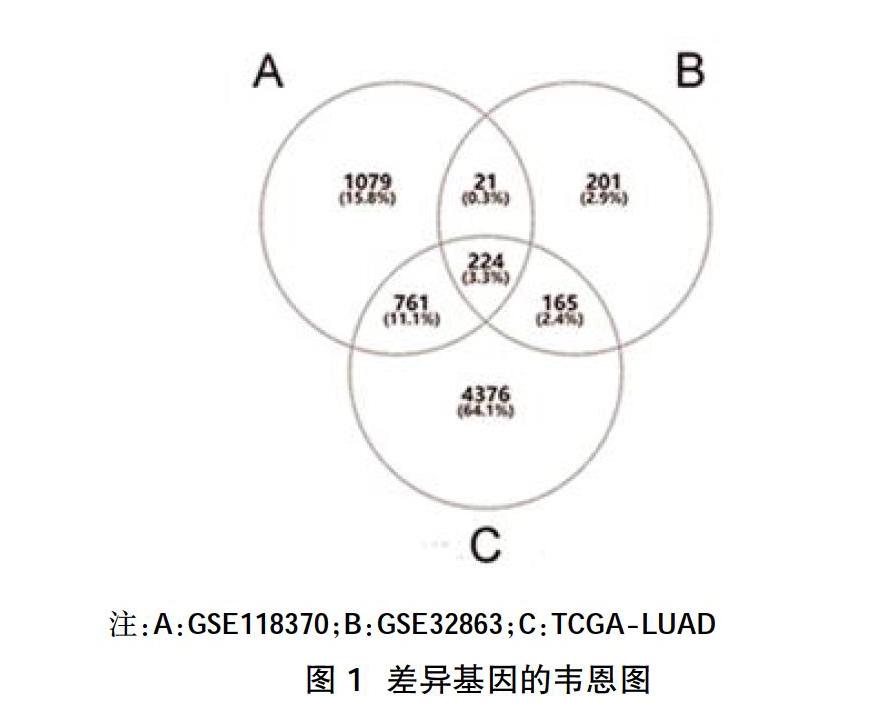
1.2数据处理 利用R语言程序包对下载的数据进行处理,筛选出肺腺癌和癌旁组织表达差异的基因,并对三个数据集筛选出的差异表达基因用韦恩图取交集。筛选标准:Log2∣差异倍数(FC)∣>1,P<0.05。
1.3基因富集分析 利用DAVID(https://david.ncifcrf.gov/)对共同的差异表达基因进行功能注释,包括分子功能、细胞组成与生物过程的GO功能富集分析,FDR<0.05表示差异有统计学意义。
1.4差异基因编码蛋白间相互作用的分析 通过在线分析网站STRING(https://string-db.org/)得到差异表达基因的蛋白互作网络,将所得源文件导入Cytoscape进行可视化分析,用插件cytoHubba进行Hub基因分析,选用其中6种算法,选取前10个关键基因。
1.5生存分析 下载TCGA-LUAD的临床信息,通过Cox比例风险模型调整相对危险度后,采用GraphPad Prism7绘制Kaplan-Meier生存曲线,P<0.05表示差异有统计学意义。
1.6 GEPIA数据库分析 核心基因表达通过GEPIA数据库(http://gepia. cancer-pku.cn/)分析核心基因在泛癌中组织与正常组织的基因表达差异。
2结果
2.1初步筛查 GSE118370中上调基因和下调基因分别为301个和1784个;GSE32863中上调基因和下调基因分别为194个和417个;TCGA-LUAD中上调基因和下调基因分别为3713个和1813个。并根据3个数据集的差异表达基因制作韦恩图,最终获得共同差异表达基因224个,其中上调基因34个,下调基因190个,见图1。
2.2 GO富集分析 DAVID网站对224个差异表达基因的GO富集分析结果显示,差异基因在生物过程(BP)中参与血管生成、对缺氧反应、白细胞调节、免疫反应、受体内化;细胞成分(CC)主要表现为细胞外区和质膜的整体部分;分子功能(MF)包括参与肝素、转化生长因子β、糖胺聚糖的结合过程,见图2。
2.3 PPI网络构建和目的基因的筛选 根据cytoHubba的6种算法,对每种算法的前10个基因进行取交集,最终得到8个目的基因,分别为IL6、VWF、PECAM1、SPP1、CDH5、CXCL12、TIMP1、CLDN5,见表1。
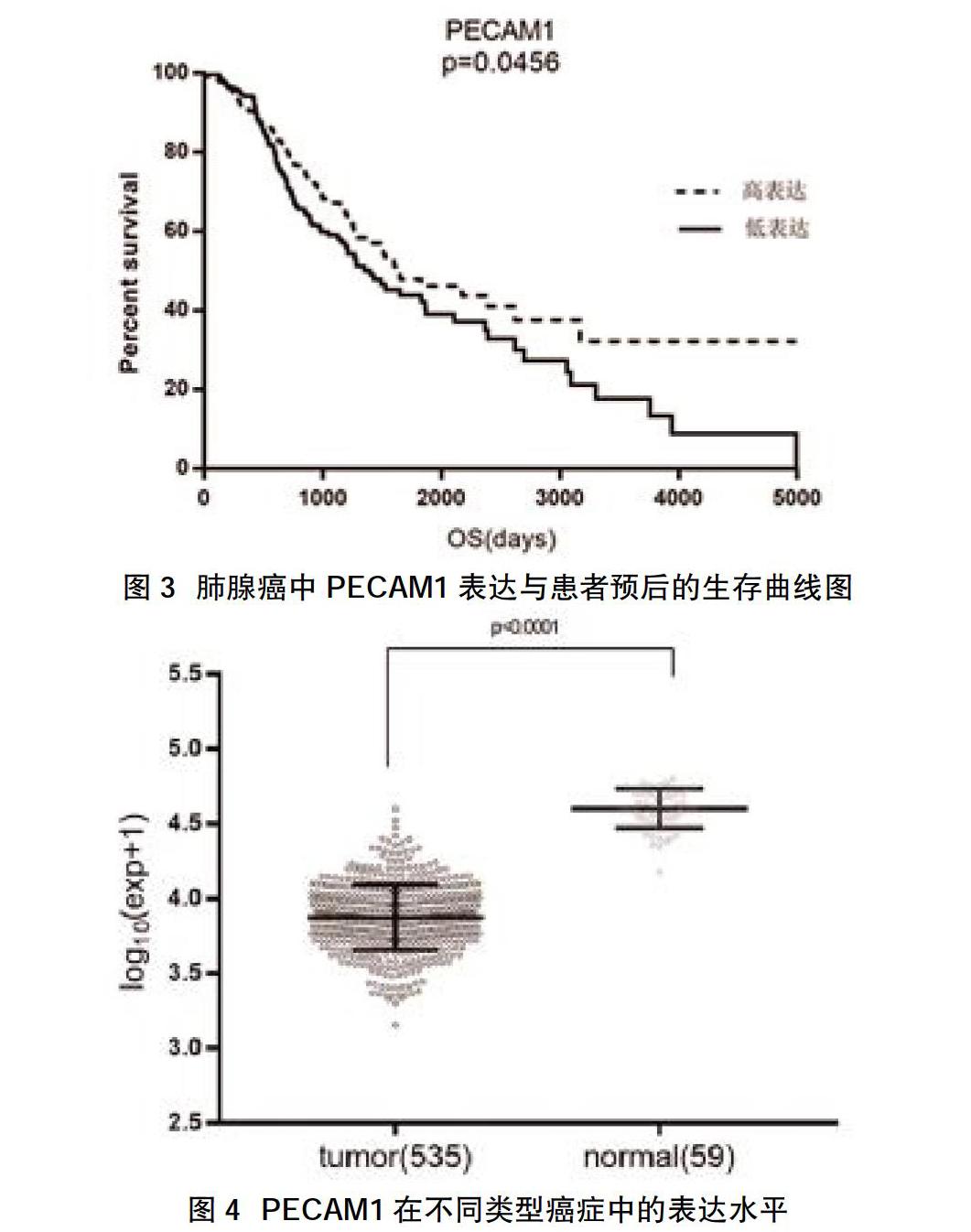
2.4生存分析结果 PECAM1与LUAD的预后有关(P<0.05),低表达的PECAM1患者预后更差,见图3。
2.5肿瘤组织与正常组织中PECAM1表达比较 肿瘤组织中PECAM1表达高于正常组织,差异有统计学意义(P<0.05),见图4。
3讨论
肺腺癌又被称为非鳞状肺癌,是一种非小细胞肺癌,也是最常见的肺癌类型。非小细胞肺癌占肺癌的80%,其中约50%为腺癌。根据肺腺癌的阶段,肺腺癌的治疗包括手术,化疗,放疗,免疫治疗等,取得了较大的进展,但是预后仍然不太理想[7]。肺腺癌与肺部慢性感染有关,其发病机制主要有直接扩散、血行转移、支气管内播散、淋巴转移等,而基因组分析可对肺腺癌发生机制进行全面洞察,还可发现一些新的肺腺癌预后和治疗的潜在生物标志物。
本研究中对3个数据集(GSE118370、GSE32863和TCGA-LUAD)共123例正常样本和599例肺腺癌样本进行分析,最终发现共有224个共同的差异表达基因,其中190个下调基因,34个上调基因。GO富集分析结果显示,差异基因在生物过程(BP)中参与血管生成、对缺氧反应、白细胞调节、免疫反应、受体内化等;细胞成分(CC)主要表现为细胞外区和质膜的整体部分;分子功能(MF)包括参与肝素、转化生长因子β、糖胺聚糖的结合过程。表明肿瘤的发病机制是一个由特定基因和表观遗传变化驱动的复杂生物学过程,多基因的异常调控可通过不同的机制促进肺腺癌的发生和发展。
为了进一步筛选关键基因,本研究利用string构建了一个由224个节点、447条边组成的蛋白质-蛋白质互作网络,利用cytoscape对该网络可视化,并用cytoHubba插来筛选目的基因。根据6种不同算法得到每种算法的前10个关键基因,再对这6种方法得到的关键基因取交集,分别为IL6、VWF、PECAM1、SPP1、CDH5、CXCL12、TIMP1、CLDN5关键基因,其中VWF、SPP1、CDH5已在其他文献中有过报道[8,9]。本研究对PECAM1进行生存曲线分析,结果表明PECAM1的表达与LUAD患者的预后有关(P<0.05),低表达的PECAM1患者的预后更差。另有研究发现,PECAM1在多种肿瘤中与正常组织存在差异表达,如卵巢癌[10]、淋巴瘤[11]、肝癌[12]、胃癌[13]、白血病[14]、结肠癌[15]等。PECAM-1,也称为CD31,是一种130 KDa的糖蛋白,是免疫球蛋白(Ig)超家族的成员,表达于内皮细胞(EC)、白细胞和血小板4,能够与自身以及其他非PECAM分子相互作用,并与粘附和信号传导有关,对于内皮细胞的迁移及血管生成具有重要作用[16,17]。在肿瘤生长过程中,肿瘤血管的生成过程受多种血管生成因子和血管生产抑制物的调控[18],而PECAM1作为常见的黏附蛋白在血管生成过程中起着重要的作用。本研究中,肿瘤组织中PECAM1表达高于正常组织,差异有统计学意义(P<0.05),说明PECAM1可能成为肺腺癌的治疗靶点,值得进一步深入探究。
综上所述,肺腺癌的发生发展受复合基因网络通过不同的生物学途径进行调控,PECAM1是该网络中的核心基因,与肺腺癌的预后相关,有望成为有效抑制肺腺癌的靶点。但本研究尚存在不足之处:①缺乏对关键基因的进一步实验验证;②由于只分析了三个数据集,样本量较小,需要更大的样本研究来证实本次研究;③肺腺癌与诸多因素密切相关,在本次研究中没有分析病因;④生存分析中只对TCGA中594个样本进行分析,还需要进一步的实验证实肺腺癌的具体分子生物学机制。
参考文献:
[1]Bray F,Ferlay J,Soerjomataram I,et al.Global cancer statistics 2018:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J].CA Cancer J Clin,2018,68(6):394-424.
[2]Cho J,Choi SM,Lee J,et al.Proportion and clinical features of never-smokers with non-small cell lung cancer[J].Chin J Cancer,2017,36(1):20.
[3]全斌,喻艳林.肺结核合并肺癌的发生机制研究进展[J].山东医药,2015,55(24):104-106.
[4]Guo T,Ma H,Zhou Y.Bioinformatics analysis of microarray data to identify the candidate biomarkers of lung adenocarcinoma[J].Peer J,2019(7):e7313.
[5]Di X,Jin X,Li R,et al.CircRNAs and lung cancer:Biomarkers and master regulators[J].Life Sci,2019(220):177-185.
[6]Vargas AJ,Harris CC.Biomarker development in the precision medicine era:lung cancer as a case study[J].Nat Rev Cancer,2016,16(8):525-537.
[7]Hanna N,Johnson D,Temin S,et al.Systemic Therapy for Stage IV Non-Small-Cell Lung Cancer:American Society of Clinical Oncology Clinical Practice Guideline Update Summary[J].J Oncol Pract,2017,13(12):832-837.
[8]Piao J,Sun J,Yang Y,et al.Target gene screening and evaluation of prognostic values in non-small cell lung cancers by bioinformatics analysis[J].Gene,2018(647):306-311.
[9]高強,钟英英,丁华杰,等.肺腺癌相关基因的生物信息学分析[J].中国肿瘤生物治疗杂志,2019,26(2):190-195.
[10]Rask L,Hogdall CK,Kjaer SK,et al.Association of CD31 and p53 With Survival of Ovarian Cancer Patients[J].Anticancer Res,2019,39(2):567-576.
[11]Molinsky J,Klánová M,Maswabi B,et al.In vivo growth of mantle cell lymphoma xenografts in immunodeficient mice is positively regulated by VEGF and associated with significant up-regulation of CD31/PECAM1[J].Folia Biol (Praha),2013,59(1):26-31.
[12]Mas VR,Maluf DG,Archer KJ,et al.Genes involved in viral carcinogenesis and tumor initiation in hepatitis C virus-induced hepatocellular carcinoma[J].Mol Med,2009,15(3-4):85-94.
[13]袁梅琴,王增,王海洋,等.血清PECAM-1和IGF-1水平与晚期胃癌临床病理特征及预后的相关性研究[J].中华全科医学,2018(7):1051-1053.
[14]Haslinger C,Schweifer N,Stilgenbauer S,et al.Microarray gene expression profiling of B-cell chronic lymphocytic leukemia subgroups defined by genomic aberrations and VH mutation status[J].J Clin Oncol,2004,22(19):3937-3949.
[15]Skrzypczak M,Goryca K,Rubel T,et al.Modeling oncogenic signaling in colon tumors by multidirectional analyses of microarray data directed for maximization of analytical reliability[J].PLoS One,2010,5(10):e13091.
[16]Lertkiatmongkol P,Liao D,Mei H,et al.Endothelial functions of platelet/endothelial cell adhesion molecule-1 (CD31)[J].Curr Opin Hematol,2016,23(3):253-259.
[17]Umezawa Y,Akiyama H,Okada K,et al.Molecular mechanisms for enhancement of stromal cell-derived factor 1-induced chemotaxis by platelet endothelial cell adhesion molecule 1 (PECAM-1)[J].J Biol Chem,2017,292(48):19639-19655.
[18]Khan I,Bhardwaj M,Shukla S,et al.Carvacrol encapsulated nanocarrier/ nanoemulsion abrogates angiogenesis by downregulating COX-2,VEGF and CD31 in vitro and in vivo in a lung adenocarcinoma model[J].Colloids Surf B Biointerfaces,2019(181):612-622.
收稿日期:2019-9-9;修回日期:2019-9-23
編辑/杜帆
