海洋青铜小单胞菌MCCC 1K05785的生长性质及耐药相关蛋白研究
2024-01-01于东瑾辛蕊马永正牛志广
于东瑾 辛蕊 马永正 牛志广

摘要:青銅小单胞菌MCCC 1K05785分离自海洋近岸沉积物。作为天然产物的来源,它同时表现出了对多种抗生素的耐药性。本研究对该菌株进行了表征,并通过对菌株进行全基因组测序,在基因水平上探索了其潜在含有的耐药相关蛋白,以明确该菌株的耐药机制。首先在实验室环境下培养菌株并测定其生长曲线,再通过对菌株进行药敏实验和全基因组测序,挖掘菌株耐药相关蛋白、潜在合成的次级代谢产物以及其他特性。结果表明,MCCC 1K05785菌落呈橙红色,60~108 h为菌株对数生长期。菌株对氨苄西林、磺胺甲恶唑、利福平、氯霉素、万古霉素、红霉素耐药。菌株基因组全长为6861996 bp,GC含量为72.79%。共预测到6136个基因。预测基因序列总长度为6196530 bp。KEGG数据库注释到336个基因参与次级代谢产物合成,antiSMASH预测到23个基因簇。分析耐药蛋白发现,外排泵是菌株表达丰富耐药性的主要机制。揭示了MCCC 1K05785是具有多重抗生素耐药性的菌株,同时具有合成新颖次级代谢产物的潜力。
关键词:青铜小单胞菌;耐药蛋白;全基因组测序;次级代谢产物;抗生素
中图分类号:R978 文献标志码:A
Study on the growth characteristics and drug resistance related proteins of Micromonospora chalcea MCCC 1K05785
Yu Dongjin1 , Xin Rui1 , Ma Yongzheng1 , and Niu Zhiguang1, 2
(1 School of Marine Science and Technology, Tianjin 300072; 2 International Research Institute of Tianjin University, Fuzhou 350207)
Abstract Micromonospora chalcea MCCC 1K05785 was isolated from offshore sediments. As a source of natural products, it also displayed drug resistance to multiple antibiotics. Armed with whole genome sequencing, this study characterized the strain and explore the potential drug-resistant-related proteins at the genetic level so that to make clear the drug-resistant mechanism of this strain. Firstly, we cultivated the strain in the laboratory environment and determined growth curve. Then, drug sensitivity experiments and whole genome sequencing were conducted to explore the drug-resistant related proteins, potential synthesized secondary metabolites and other characteristics of the strain. Our results show that, the colony of MCCC 1K05785 is orange red. The logarithmic growth period is about 60-108 h. The strain is resistant to ampicillin, sulfamethoxazole, rifampicin, chloramphenicol, vancomycin and erythromycin. The total length of the strain genome is 6861996 bp and the GC content is 72.79%. A total of 6136 genes are predicted. The predicted total length of the gene sequence is 6196530 bp. The KEGG database indicates that 336 genes participate in the synthesis of secondary metabolites and antiSMASH predicts there are 23 gene clusters in this strain. Furthermore, the efflux pumps might play an important the role in the emergence of drug resistance. As a novel strain with multiple antibiotic-resistance, it might be a potential source to discover the new secondary metabolites.
Key words Micromonospora chalcea; Drug-resistant proteins; Whole genome sequencing; Secondary metabolites; Antibiotics
随着世界重大公共卫生事件频发,抗生素滥用导致细菌耐药问题加剧[1],为了应对这一环境问题,从源头出发研究细菌耐药生化机制已迫在眉睫。放线菌是一种高G+C含量的革兰氏阳性菌,是最大的细菌门之一,存在于陆地及海洋沉积物中[2]。海洋环境独特,可使放线菌产生新颖的活性化合物。海洋放线菌包含一系列编码代谢产物的基因,这些产物已被证實具有抗菌、抗肿瘤及特殊的酶活性[3],约70%的天然抗生素由放线菌产生。因此,放线菌也必然表达丰富的抗生素耐药性以保护自己免受自产抗生素的攻击。
小单孢菌属(Micromonospora)在自然界中广泛分布于不同的地理栖息地:土壤、红树林沉积物、海洋沉积物和极端栖息地等。小单孢菌属是革兰阳性菌、由孢子形成的需氧放线菌,具有独特的形态特征,单孢子呈球形或椭圆形,直径为0.7~1.5 ?m[4],好氧,为非酸性和嗜温微生物[5],其菌丝体具有类胡萝卜素,使其菌落呈现黄色、橙色、红色或紫色[6]。长期以来,小单胞菌属是次级代谢产物的重要来源[7]。环境中的微生物通过水平转移的方式使其成为宿主菌表达耐药性,其耐药机制多为基因变化的结果[8]。
随着高通量测序及全基因组测序技术的发展,已经有越来越多的微生物基因组完成测序[9]。纳米孔测序技术(nanopore sequencing(ONT))是一种检测纳米孔单分子的实时电信号测序方法[10]。经过精确匹配,能够准确确定特定的碱基类型,从而实现快速、准确的序列测量。小单胞菌属作为稀有放线菌属,其生产天然产物的潜力仅次于链霉菌属,7%的天然产物来源于小单胞菌属[11]。青铜小单胞菌可以产生雷克西丁衍生物[12]、庆大霉素、西索米星、新型抗真菌剂Turbinmicin[13]等,具有抗细菌、抗肿瘤和抗真菌等功效[14]。有关青铜小单孢菌的全基因组数据研究较少涉及,因此本研究选择青铜小单孢菌MCCC 1K05785,对其进行前期培养观察菌株生长性质并进行全基因组测序,为后续深入研究海洋放线菌的耐药机制提供参考。
1 材料与方法
1.1 材料
1.1.1 实验菌株来源及培养方法
本研究中使用的青铜小单胞菌MCCC 1K05785购买自中国海洋微生物菌种保藏管理中心。该菌株使用2216E培养基进行培养,培养基配方为:蛋白胨:5.0 g、酵母粉1.0 g、柠檬酸铁0.1 g、氯化钠19.45 g、氯化镁5.98 g、硫酸钠3.24 g、氯化钙1.8 g、氯化钾0.55 g、碳酸钠0.16 g、溴化钾0.08 g、氯化锶0.034 g、硼酸0.022 g、硅酸钠0.004 g、氟化钠0.0024 g、硝酸钠0.0016 g、磷酸氢二钠0.008 g。
1.1.2 主要试剂及仪器
主要试剂:2216E培养基、Mueller-Hinton(MH)培养基购买自青岛海博有限公司;胰蛋白胨大豆肉汤(TSB)培养基、马铃薯葡萄糖琼脂(PDA)培养基、酵母粉葡萄糖氯霉素琼脂(YDC)培养基、ISP Medium No.4(ISP4)培养基、ISP Medium No.3(ISP3)培养基、放线菌(改良高氏1号)培养基均购买自天津为科生物技术有限公司。在配置上述培养基时需使用pH值为7.2~7.5的海水,以维持菌株良好生长。药敏纸片购买自常德比克曼生物科技有限公司,纸片所含抗生素种类包括:氨苄西林、阿莫西林、头孢噻吩、庆大霉素、链霉素、卡那霉素、阿米卡星、四环素、环丙沙星、磺胺甲恶唑、利福平、氯霉素、万古霉素、多黏菌素B、红霉素。
主要仪器:尼康TS2倒置生物显微镜、Thermo台式高速冷冻离心机、生物恒温培养箱、场发射扫描电子显微镜(型号:Regulus8100)等。
1.2 方法
1.2.1 MCCC 1K05785菌株的培养与形态观察
划线纯化MCCC 1K05785的单菌落于2216E固体培养基上。固体培养后,用接种环刮取适量菌体于装有100 mL液体2216E培养基的带挡板摇瓶中,150 r/min,
30 ℃摇床培养5~7 d获得液体菌液。分别取200 μL上述菌液涂布于TSB、PDA、YDC、ISP4、ISP3、高氏一号培养基、2216E固体培养基上,30 ℃恒温培养箱培养5~7 d,观察菌落形态、颜色及产孢情况。
将培养5 d的MCCC 1K05785单菌落用接种环从平板中刮下转移至酒精擦拭过干净的无菌载玻片上,将草酸铵结晶紫滴加到细菌涂片上并染色1 min,使用经过高压灭菌的ddH2O将多余的染液洗去。将碘溶液滴加到细菌涂片中,继续染色1 min,使用经过高压灭菌的ddH2O将多余的碘溶液洗去。使用乙醇溶液滴加至涂片脱色30 s,再使用干净的滤纸吸干液体。使用番红染液复染90 s后用高压灭菌的ddH2O洗去多余液体。涂片静置干燥后使用倒置显微镜以40倍物镜观察菌丝体形态结构。
1.2.2 MCCC 1K05785菌株在场发射扫描电子显微镜(SEM)下的微观形态观察
取1 mL菌液接种于2216E固体培养基上培养
5 d。将培养基表面的菌体用接种环刮下,加入2.5%戊二醛溶液,4 ℃固定24 h,后用pH为7.0的0.1 M的PBS溶液漂洗菌体,重复三次,每次15 min去除戊二醛。用梯度浓度乙醇溶液(50%、70%、80%、90%、95%)依次对样品进行脱水处理,每次处理15 min,最后无水乙醇处理两次,每次20 min。用体积比为1:1的乙醇与醋酸异戊酯的混合液处理样品30 min,再用纯醋酸异戊酯处理样品1.5 h,最后将样品冷冻干燥。用镊子夹取少量冷冻干燥后的菌体放置于导电胶上,离子溅射喷金,使用扫描电子显微镜观察菌体形态并拍照。
1.2.3 MCCC 1K05785菌株生长曲线的测定
采用干重法测定菌株生长曲线。将菌株在平板上密集划线后,置于30 ℃恒温培养箱中培养5 d。使用打孔器打下3片菌块,其中两块分别放入两个装有60 mL液体2216E培养基中的100 mL挡板摇瓶中,剩余一块放入100 mL无菌水中,共准备36瓶培养基,统一在30 ℃、150 r/min条件下摇床培养。每隔12 h取出两个实验组及一个空白对照组,最后一组培养时间為144 h。使用直径为50 mm的醋酸纤维滤膜真空抽滤并收集菌丝体,置于80 ℃烘箱中烘干30 min,所得菌丝体重量即为干重。
1.2.4 MCCC 1K05785菌株的药敏实验
接种MCCC 1K05785菌液1 mL至固体2216E培养基,使用玻璃珠涂布至菌落均匀密布。贴药敏纸片使之分布于培养基中央。每种抗生素设置一个对照平板以观察菌株长满平板所需时间。以HB101大肠杆菌作为质控菌株,置恒温培养箱中30 ℃培养7 d后,用游标卡尺测量抑菌圈直径,根据抑菌圈的大小判定菌株对抗生素的敏感性。
1.2.5 MCCC 1K05785菌株的全基因组文库构建与数据信息分析
DNA提取方法:取1 mL培养至对数期的MCCC 1K05785菌液,加入1.5 mL离心管中,室温8000 r/min离心1 min,弃上清,收集菌体。加入180 μL溶菌酶溶液,重悬菌液,37 ℃水浴30~60 min,再加入400 μL的Buffer Digestion,震荡混匀。65 ℃水浴1 h至细胞完全裂解。水浴过程中,每10 min颠倒混匀1次,促进样品裂解。混合液澄清透明证明裂解完全。加入200 μL Buffer PB,充分颠倒混匀,-20 ℃冰箱放置5 min。室温10000 r/min离心5 min,将上清液500 μL转移到新的1.5 mL离心管中。加入等体积的异丙醇,颠倒5-8次使之充分混匀,室温放置2~3 min。室温10000 r/min离心5 min,弃上清。加入1 mL 75%乙醇,颠倒漂洗1~3 min,10000 r/min离心2 min,弃上清。开盖倒置5~10 min至残留的乙醇完全挥发。得到的DNA样本用50~100 μL TE Buffer溶解。在-20 ℃环境下将DNA样本送至北京百迈客生物科技公司进行全基因组测序分析。
测序流程:准备DNA样本2 μg,使用G-TUBE管使核酸片段化,使用NEBNext FFPE DNA Repair Mix和NEBNext Ultra Ⅱ End Repair/dA-Tailing Module完成核酸片段的损伤修复和末端修复加A,使用无扩增条形码扩展试剂盒添加barcode序列并完成测序接头的连接。用测序芯片制备试剂盒(EXP-FLP001 PRO.6)配制Flow cell Priming mix;用连接测序试剂盒(SQK-LSK109)配置上机文库;用PromethION Flow Cells(FLO-PRO002)芯片,在PromethION48测序仪运行MinKnow软件,开始测序。
基因组组分分析包括:重复序列、编码基因、基因岛等分析。利用软件IslandPath-DIMOB v 0.2[15]预测细菌基因组中的基因岛。基因组蛋白功能及耐药相关蛋白注释使用通用或专有数据库,如:GO[16]、KEGG[17]、eggNOG[18]、SwissProt[19]、TCDB[20]等。基因组图谱分析:利用已得基因组信息应用软件Circos v 0.66[21]绘制基因组圈图。使用antiSMASH v5.0.0[22]鉴定和分析细菌基因组序列中生物合成基因簇(Biosynthetic Gene Clusters,BGCs)。
2 结果
2.1 MCCC 1K05785菌株的最适培养基及生长形态
MCCC 1K05785在2216E固体培养基上生长状态最佳,在其余几种培养基下生长状态见表1。其中,+++表示生长状态良好,++表示生长状态较好,+表示生长状态不佳,—表示不生长。
从菌株出现至完全成熟时间约为5~7 d,生长初期菌落呈橙红色,表面干燥不透明,单菌落呈圆形且面积较小,见图1A。菌株在倒置生物显微镜的40倍物镜下的形态见图1B,菌株沿纤细的菌丝体分散生长。
MCCC 1K05785在SEM下的形态见图2。观察到小单胞菌菌株单个孢子直径约为400 μm,孢子表面光滑或有突起,菌丝体细长,细密缠绕形成孢子。
2.2 MCCC 1K05785菌株的生长曲线
MCCC 1K05785的生长曲线见图3。菌株呈典型指数生长特征。菌株的生长迟缓期为0~60 h;菌株的对数生长期为60~108 h,108 h之后菌株进入生长稳定期及衰亡期。
2.3 MCCC 1K05785菌株的药敏实验
采用10类15种抗生素药敏纸片进行实验,结果见表2。MCCC 1K05785对β-内酰胺类药物:氨苄西林,磺胺类药物:磺胺甲恶唑,利福霉素类药物:利福平,氯霉素类药物:氯霉素,糖肽类药物:万古霉素,大环内酯类药物:红霉素表达耐药性;对四环素中度敏感。
2.4 MCCC 1K05785菌株的基因组组分分析
2.4.1 MCCC 1K05785基因组信息
菌株经原始数据处理及组装后,得到菌株基因组相关信息,见表3。全基因组数据NMDC60046431已经存储在国家微生物科学数据中心(NMDC),链接为https://nmdc.cn/resource/genomics/genome/detail/NMDC60046431。
2.4.2 MCCC 1K05785基因岛预测
细菌的基因岛是参与水平转移并具有功能的,长度在10~200 kb左右的基因序列的集合。耐药性基因岛可以使细菌携带耐药蛋白表达耐药性,有助于细菌适应环境。使用软件IslandViewer 4中的IslandPath-DIMOB预测并筛选得到菌株的14个基因岛。注释到3个基因岛上含有N-乙酰转移酶家族(GNAT family N-acetyltransferase),该家族可以调节蛋白质乙酰化导致酶抑制从而使菌株具有耐药性。此外,还注释到1个基因岛上含有TetR/AcrR family transcriptional regulator,该家族过表达外排泵能力赋予菌株多重耐药性[23]。其可视化结果如图4所示。
2.4.3 MCCC 1K05785蛋白编码基因功能注释
利用预测得到的基因序列与GO、KEGG、eggNOG、Swiss-Prot、TrEMBL等通用功能数据库做BLAST v2.2.29比对,得到的基因功能注释统计结果见表4。
GO数据库用于预测生物功能,包括:细胞组分(cellular component,CC)、分子功能(molecular function,MF)、生物过程(biological process,BP)。菌株基因组GO功能注释分类统计图见图5,蓝色部分为CC、红色部分为MF、绿色部分为BP。结果表明,CC类中基因主要负责细胞膜生成,MF类中基因主要负责完成催化反应,BP类中共有9类基因得到注释,占比前三位分别为代谢过程(metabolic process)、细胞过程(cellular process)和单生物代谢过程(single-organism process)。参与代谢过程的基因有2088个,参与细胞过程的基因有1331个,参与单生物代谢过程的基因有1269个。
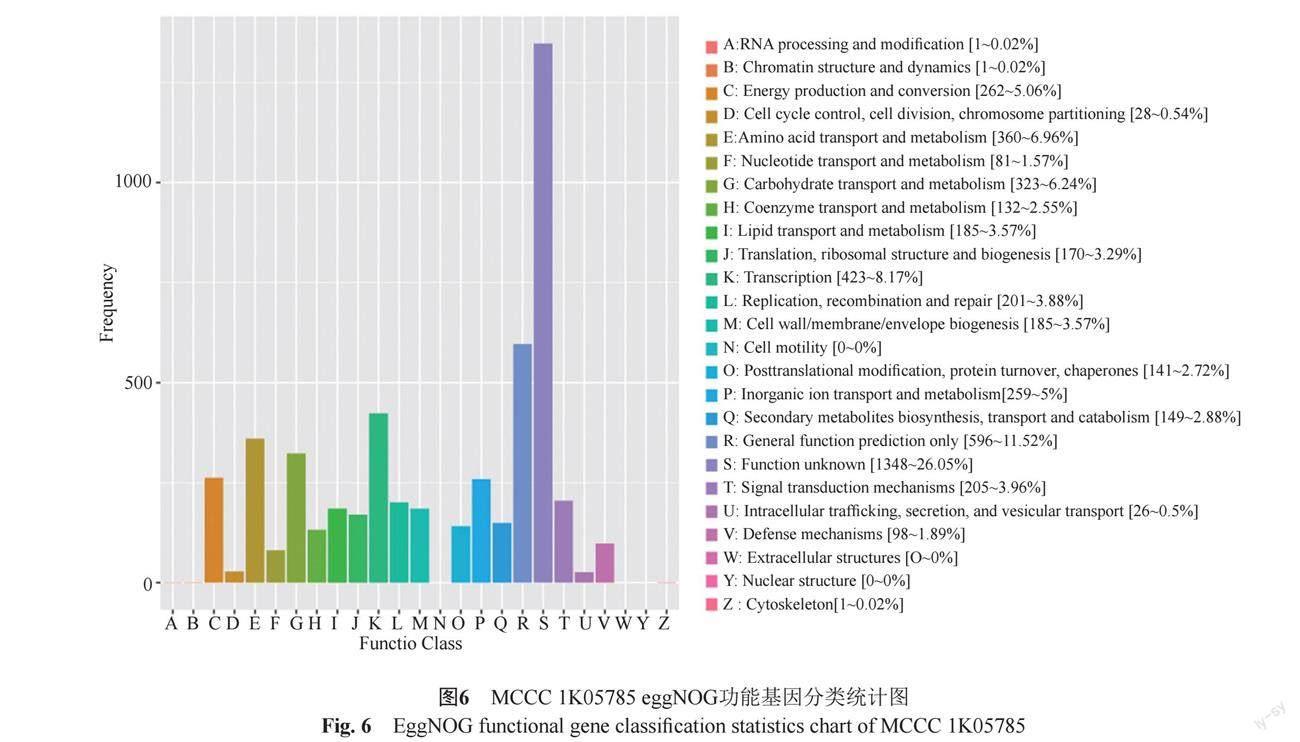
eggNOG数据库注释结果见图6,图中横坐标为eggNOG各分类内容,纵坐标为相应功能基因数目所占相对含量。结果表明,注释到的5089个基因分布于23个类别中。其中1665个功能基因参与物质代谢,包括[C]、[E]、[F]、[G]、[H]、[I]、[P]、[Q],在物质代谢中存在149个基因参与次生代谢产物的生物合成、运输和分解代谢,占比为2.88%。
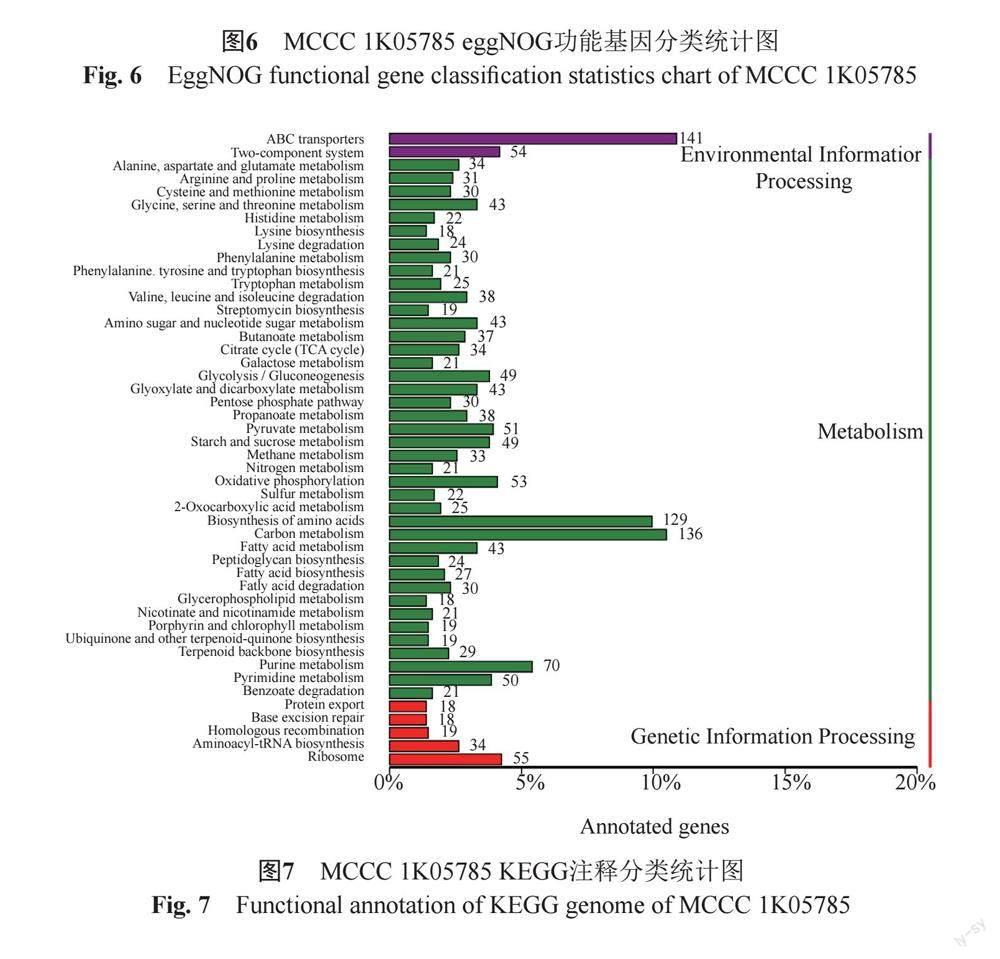
菌株基因组的KEGG注释分类中一级分类共3大类,分别为:紫色代表环境因素(environmental information processing)、绿色代表新陈代谢(metabolism)、红色代表遗传信息处理(genetic information processing);二级分类共48类,具体注释情况见图7。分析发现,菌株含有4个全局型代谢通路,其中共有706个基因参与代谢途径、336个基因参与次生代谢产物的生物合成、224个基因参与不同环境中的微生物代谢、294个基因参与抗生素的生物合成。在环境因素中基因主要负责ABC转运,新陈代谢中基因参与数量占比前三位的是:碳代谢、氨基酸的生物合成和嘌呤代谢,遗传信息处理中基因负责核糖体合成。参与抗生素的生物合成的基因数量占总体注释基因的16.94%,其中有19条代谢通路参与合成链霉素、有12条代谢通路参与合成安莎霉素、有10条代谢通路参与合成万古霉素、有5条代谢通路参与合成四环素、有4条代谢通路参与合成新生霉素、有3条代谢通路参与合成井岗霉素。
对MCCC 1K05785菌株绘制基因组圈图,见图8。图中最外圈表示基因组大小;第2圈和第3圈分别是基因组正链和负链上的基因大小;第4圈是重复序列;第5圈中蓝色是tRNA、紫色是rRNA;第6圈是GC含量;最里圈为GC-skew,深灰色表示G含量大于C,红色表示C含量大于G。
2.4.4 MCCC 1K05785次级代谢产物合成基因簇分析
使用软件antiSMASH v5.0.0鉴定和分析MCCC 1K05785中的BGCs结果见表5。共预测到23个次级代谢产物BGCs,基因簇总长度为1075266 bp,其中合成二氮喹霉素H/J(Diazaquinomycin H/Diazaquinomycin J)的同源性为94%,合成利福霉素(Rifamycin)的同源性为35%。
2.4.5 耐药相关蛋白序列的注释
TCDB数据库共注释到1347个转运蛋白,其一级分类家族包括386个ATP结合盒转运蛋白超家族(ATP binding cassette,ABC)、69个主要协同转运蛋白超家族(major facilitator superfamily,MFS)和3个多重耐药转运VanZ家族。耐药蛋白具体为:氯霉素耐药蛋白、Bcr/CflA耐药转运蛋白、EmrB/QacA耐药转运蛋白、VanZ耐药转运蛋白、柔红霉素抗性ATP结合蛋白DrrA和替瑞霉素C(TcmC)耐药蛋白。注释统计结果见表6,表中耐药蛋白综合比对得分均在280以上。
SwissProt数据库注释到了MCCC 1K05785菌株含有10种耐药蛋白,分別为:多重耐药蛋白:Mmr、EmrB、MdtL、Stp;氯霉素耐药蛋白CmlR、四环素耐药蛋白TetA和万古霉素耐药蛋白VanB。具体结果见表7。
与上述药敏实验结果对比发现,氯霉素耐药蛋白使菌株对氯霉素表达耐药性。VanZ家族和耐药蛋白VanB使菌株对糖肽类抗生素如万古霉素产生耐药性。Bcr/CflA家族耐药转运蛋白的表达使菌株对磺胺类药物产生具有耐药性[24]。Mmr蛋白和TetA蛋白作用于外排泵使菌株对大环内酯类和四环素类抗生素产生耐药性。
3 讨论
小单孢菌属是具有巨大潜力的抗生素产生菌,其产生的化合物多具有抗真菌、抗细菌、抗肿瘤等活性。Iznaga等[25]描述分离自土壤的放线菌具有显著的抗真菌活性,其中的小单孢菌多产生大环内酯类抗生素。Schulze等[26]提到小单孢菌属可以产生利福霉素的异构体:Isorifamycin S,抑制枯草芽孢杆菌的生长。经过全基因组测序和药敏实验发现,MCCC 1K05785菌株不仅具有多种BGCs,可以潜在合成多种抗生素,另外菌株还对6类抗生素产生耐药性。证明该菌株耐药性丰富,其耐药性产生机制值得深入挖掘。
微生物对抗生素的耐药机理复杂且多样,包括形成灭活酶、改变靶位基因、细胞膜渗透性变化或孔蛋白孔径变窄、外排泵机制清空细菌体内药物等[27]。
作为产生抗生素的细菌,它们一般存在着多重耐药机理,以保证自己能够在生成抗菌化合物的环境下继续生存。本研究中药敏实验结果发现MCCC 1K05785菌株对6类抗生素产生耐药性,分别为:β-内酰胺类、大环内酯类、糖肽类、磺胺类、利福霉素类和氯霉素类。大环内酯类抗生素的耐药机制通过两种方式降低药物亲和力而导致药物失效:
①細菌可以修饰核糖体,从而降低药物的亲和力;②由于细菌膜的渗透性发生改变或是表达了外排泵机制,导致大环内酯类抗生素从细菌中流出。细菌对糖肽类抗生素的耐药机制根据菌株的耐药蛋白对细胞壁合成过程中五肽末端修饰结构的不同,主要分为D-Ala-D-Lac和D-Ala-D-Ser两种类型,耐药蛋白包括VanA、VanB、VanN和VanZ等,防止抗生素对细菌细胞壁的合成产生干扰[28]。氯霉素类抗生素的耐药机制有2种,包括通过基因调控合成乙酰化转移酶使氯霉素转化为无抑菌活性的代谢产物或是通过位于细菌内膜的Cml等耐药蛋白主动泵出显著减少氯霉素的含量[29]。药物泵出系统主要由四种家族组成:主要协同转运蛋白超家族(MFS家族)、RND家族、小多重耐药家族(SMR家族)和ATP结合盒转运蛋白超家族(ABC家族)[30]。在MCCC 1K05785菌株中共注释到3个外排泵家族与其耐药性形成有关,分别为ABC家族、MFS家族和SMR家族。耐药蛋白Cml、VanZ和VanB、Mmr和TetA分别使菌株对氯霉素、万古霉素、红霉素和四环素产生耐药性。Bcr/CflA家族和EmrB/QacA亚家族的多重耐药蛋白和调控细胞膜结构的SMR家族同时赋予了MCCC 1K05785菌株丰富耐药性。此外,该菌株还具有博来霉素耐药蛋白、柔红霉素耐药蛋白和替曲霉素C耐药蛋白,推测菌株会对博来霉素、蒽环类抗生素如柔红霉素以及替曲霉素产生耐药性。通过分析MCCC 1K05785菌株的全基因组测序结果,发现该菌株具有多种耐药性的主要原因可能是药物外排泵的过量表达。
研究MCCC 1K05785菌株的代谢产物发现,KEGG数据库中参与合成抗生素的基因占全部注释基因的16.94%,菌株潜在合成的抗生素有:四环素、链霉素、万古霉素、新生霉素、安莎霉素、井岗霉素等。微生物的BGCs包括三类:非核糖体肽合酶(NRPs)、聚酮合酶(PKS)、核糖体合成肽与翻译后修饰肽(RiPP),其中NRPs和PKS主要负责合成抗生素[31]。对菌株的BGCs分析发现,MCCC 1K05785菌株是合成DAQ-H、DAQ-J的潜在来源,它们来源于海洋小单胞菌Micromonospora sp. B006[32],其抗菌作用远超普通抗生素[33];菌株还含有作用于细胞RNA聚合酶的利福霉素BGCs[34]和作用于细胞DNA的卡里霉素BGCs[35]。其他BGCs多与不同种类抗生素合成有关,包括萘啶霉素、阿奇霉素A等[36]。此外,MCCC 1K05785另外存在8个尚未被预测的基因簇,说明其具有合成新颖次级代谢产物的潜力。
4 结论
MCCC 1K05785菌株为革兰阳性菌,分离自海洋近岸沉积物,属于青铜小单胞菌种,菌株基丝纤细呈橙红色,孢子表面光滑或有突起。药敏实验表明菌株对6类抗生素具有耐药性。对该菌株的全基因组进行测序结果表明,基因组总长度为6861996 bp,含有一个Scaffold序列,GC含量为72.79%,共预测到6136个基因。进一步分析发现,MCCC 1K05785菌株含有丰富的耐药蛋白,蛋白编码基因表达菌株耐药性,其耐药机制以外排泵为主;菌株具有合成抗生素或新颖次级代谢产物的潜力,为后续对青铜小单胞菌自抗性机制的研究提供了新方向。
参 考 文 献
Zhang K, Xin R, Zhao Z, et al. Mobile genetic elements are the major driver of high antibiotic resistance genes abundance in the upper reaches of Huaihe river basin[J]. J Hazard Mater, 2021, 401: 123271.
Blunt J W, Copp B R, Keyzers R A, et al. Marine natural products[J]. Nat Prod Rep, 2012, 29(2): 8-53
Siro G, Pipite A, Christi K, et al. Marine actinomycetes associated with stony corals: a potential hotspot for specialized metabolites[J]. Microorganisms, 2022, 10(7): 1349.
Pozo M, Wieme A D, Perez S R, et al. Micromonospora fluminis sp. nov. , isolated from mountain river sediment[J]. Int J Syst Evol Micr, 2020, 70(12): 6428-6436.
Yan S, Zeng M, Wang H, et al. Micromonospora: a prolific source of bioactive secondary metabolites with therapeutic potential[J]. J Med Chem, 2022, 65(13): 8735-8771.
Hifnawy M S, Fouda M M, Sayed A M, et al. The genus Micromonospora as a model microorganism for bioactive natural product discovery[J]. Rsc Adv, 2020, 10(35): 20939-20959.
Hirsch A M, Valdés M. Micromonospora: an important microbe for biomedicine and potentially for biocontrol and biofuels[J]. Soil Biol Biochem, 2010, 42(4): 536-542.
Unsleber S, Wohlleben W, Stegmann E. Diversity of peptidoglycan structure-modifications and their physiological role in resistance in antibiotic producers[J]. Int J Med Microbiol, 2019, 309(6): 151132.
李輝, 方志锴, 郭霞凌. 利普斯他汀高产菌株毒三素链霉菌AP617-N12CA的全基因组测序与分析[J]. 中国抗生素杂志, 2022, 47(01): 28-34.
Kasianowicz J J, Bezrukov S M. On three decades of nanopore sequencing[J]. Nat Biotechnol, 2016, 34(5): 481-482.
Valliappan K, Sun W, Li Z Y. Marine actinobacteria associated with marine organisms and their potentials in producing pharmaceutical natural products[J]. Appl Microbiol Biot, 2014, 98(17): 7365-7377.
Chen L, Xie L, Zhao W, et al. Two new rakicidin derivatives from marine Micromonospora chalcea FIM-R160609[J]. Nat Prod Res, 2022: 1-8.
Zhang F, Zhao M, Braun D R, et al. A marine microbiome antifungal targets urgent-threat drug-resistant fungi[J]. Science, 2020, 370(6519): 974-978.
Komaki H, Ichikawa N, Hosoyama A, et al. In silico analysis of PKS and NRPS gene clusters in arisostatin and kosinostatin-producers and description of micromonospora okii sp. nov. [J]. Antibiotics-Basel, 2021, 10(12): 1447.
Bertelli C, Brinkman F. Improved genomic island predictions with IslandPath-DIMOB[J]. Bioinformatics, 2018, 34(13): 2161-2167.
Ashburner M, Ball C A, Blake J A, et al. Gene ontology: tool for the unification of biology[J]. Nat Genet, 2000, 25(1): 25-29.
Kanehisa M, Goto S, Kawashima S, et al. The KEGG resource for deciphering the genome[J]. Nucleic Acids Res, 2004, 32: D277-D280.
Powell S, Forslund K, Szklarczyk D, et al. EggNOG v4. 0: nested orthology inference across 3686 organisms[J]. Nucleic Acids Res, 2014, 42(D1): D231-D239.
Bairoch A, Apweiler R. The SWISS-PROT protein sequence data bank and its supplement TrEMBL in 1998[J]. Nucleic Acids Res, 1998, 26(1): 38-42.
Saier M H, Tran C V, Barabote R D. TCDB: the transporter classification database for membrane transport protein analyses and information[J]. Nucleic Acids Res, 2006, 34: D181-D186.
Krzywinski M, Schein J, Birol I, et al. Circos: an information aesthetic for comparative genomics[J]. Genome Res, 2009, 19(9): 1639-1645.
Blin K, Shaw S, Steinke K, et al. AntiSMASH 5. 0: updates to the secondary metabolite genome mining pipeline[J]. Nucleic Acids Res, 2019, 47(W1): W81-W87.
Colclough A L, Scadden J, Blair J M A. TetR-family transcription factors in Gram-negative bacteria: conservation, variation and implications for efflux-mediated antimicrobial resistance[J]. Bmc Genomics, 2019, 20(1): 1-12.
Vedantam G, Guay G G, Austria N E, et al. Characterization of mutations contributing to sulfathiazole resistance in Escherichia coli[J]. Antimicrob Agents Chemother, 1998, 42(1): 88-93.
Iznaga Y, Lemus M, Gonzalez L, et al. Antifungal activity of actinomycetes from Cuban soils[J]. Phytother Res, 2004, 18(6): 494-496.
Schulze C J, Donia M S, Siqueira-Neto J L, et al. Genome-directed lead discovery: Biosynthesis, structure elucidation, and biological evaluation of two families of polyene macrolactams against trypanosoma brucei[J]. Acs Chem Biol, 2015, 10(10): 2373-2381.
杜靜怡, 王铖, 郭君慧, 等. 细菌RND外排泵的结构与作用机制研究进展[J]. 中国抗生素杂志, 2022, 47(10): 994-1001.
郑福成, 黄轶群, 潘裕添, 等. 放线菌来源的糖肽类抗生素的药学机制研究进展[J]. 微生物学通报, 2018, 45(01): 197-206.
Zhou J, Chen Y, Li W X, et al. Deciphering the microbial community tolerance mechanism and alteration of antibiotic resistance genes during chloramphenicol wastewater treatment[J]. Int Biodeter Biodegr, 2023, 178: 105546.
廖燕科, 蓝素桂, 苏爱秋, 等. 逆转细菌耐药性研究进展[J]. 中国抗生素杂志, 2023, 48(01): 32-40.
Goel N, Singh R, Sood S, et al. Investigation of Streptomyces sp. strain EMB24 secondary metabolite profile has unraveled its extraordinary antibacterial potency against drug-resistant bacteria[J]. Mar Biotechnol, 2022, 24(6): 1168-1175.
Braesel J, Crnkovic C M, Kunstman K J, et al. Complete genome of Micromonospora sp. strain B006 reveals biosynthetic potential of a lake michigan actinomycete[J]. J Nat Prod, 2018, 81(9): 2057-2068.
Braesel J, Lee J, Arnould B, et al. Diazaquinomycin biosynthetic gene clusters from marine and freshwater actinomycetes[J]. J Nat Prod, 2019, 82(4): 937-946
Kim H, Kim S, Kim M, et al. Bioactive natural products from the genus Salinospora: A review[J]. Arch Pharm Res, 2020, 43(12): 1230-1258.
Vasudevan A, Argiriadi M A, Baranczak A, et al. Covalent binders in drug discovery[J]. Prog Med Chem, 2019, 58: 1-62.
Yang Y, Guo J, Liu Z Z. Progress in the synthesis of analogues of bistetrahdroisoquinoline antitumor alkaloids[J]. Chin J Org Chem, 2019, 39(7): 1913-1922.
