耐喹诺酮类药物革兰阴性菌的适应性研究
2024-01-01王邦娟陈杨黄璐璐刘理慧刘振利程古月
王邦娟 陈杨 黄璐璐 刘理慧 刘振利 程古月


摘要:隨着广谱高效喹诺酮类药物的过度应用,革兰阴性菌对喹诺酮类药物的耐药率逐年上升,已严重威胁到人类健康。革兰阴性菌对喹诺酮类药物的抗性是通过耐药基因突变或者获得外源抗性基因实现的。耐药性的维持通常会对细菌造成一定的适应性代价,表现为细菌生长速率、毒性和传播的减弱,但耐药菌可通过补偿突变缓解适应性代价,甚至增强耐药菌适应性,严重阻遏临床疗效。而且耐药菌株的适应性增强,导致细菌即使在缺乏抗菌剂使用的条件下,其耐药率也增加,导致无法通过减少抗菌剂的使用减弱菌株耐药率。所以本文分别对革兰阴性菌的染色体和PMQR基因介导的耐喹诺酮药物的机制对其适应性变化的影响进行综述,并对耐药菌适应性的研究方法进行总结,以期为耐喹诺酮类药物革兰阴性菌的新型治疗方案、相关的肠道致病菌抑制剂的开发和未来与之相关的研究提供一定参考。
关键词:革兰阴性菌;喹诺酮;耐药性;适应性;适应性代价;补偿突变;适应性测量
中图分类号:R978.1文献标志码:A
Study on the adaptability of quinolone-resistant Gram-negative bacteria
Wang Bangjuan1, Chen Yang1, Huang Lulu1, Liu Lihui1, Liu Zhenli1,2, and Cheng Guyue1,2
(1 MOA Laboratory for Risk Assessment of Quality and Safety of Livestock and Poultry Products Huazhong Agricultural University, Wuhan 430070; 2 National Reference Laboratory of Veterinary Drug Residues (HZAU) and MOA Key Laboratory for the Detection of Veterinary Drug Residues in Foods, Huazhong Agricultural University, Wuhan 430070)
Abstract With the excessive use of broad-spectrum high-efficiency quinolones, the resistance rate of gram-negative bacteria to quinolones has been increasing year by year, which has seriously threatened human health. Resistance to quinolones in gram-negative bacteria is achieved through mutations in resistance genes or acquisition of exogenous resistance genes. The maintenance of drug resistance usually causes a certain adaptive cost to bacteria, manifested as weakening of bacterial growth rate, toxicity and transmission, but drug-resistant bacteria can alleviate the adaptive cost by compensating mutations, and even enhance the adaptability of drug-resistant bacteria, seriously dampening the clinical effect. Moreover, the adaptability of resistant strains is enhanced, resulting in an increase in the resistance rate of bacteria even in the absence of antimicrobial use, resulting in the inability to attenuate the resistance rate of strains by reducing the use of antimicrobials. Therefore, in this paper, the effects of chromosome of gram-negative bacteria and PMQR gene-mediated mechanism of quinolone-resistant drugs on their adaptive changes are reviewed, and the research methods of adaptability of drug-resistant bacteria are summarized in order to provide some reference for new treatment regimens of quinolone-resistant Gram-negative bacteria, the development of related enteropathogen inhibitors and future related research.
Key words Gram-negative bacteria; Quinolone; Drug resistance; Adaptability; Adaptive cost; Compensatory mutation; Adaptability measurement
喹诺酮类(氟喹诺酮类)药物因其抗菌谱广、活性强、安全性高、价格低廉等优点,被广泛用于革兰阳性菌、革兰阴性菌和支原体感染的预防和治疗[1-3]。
喹诺酮类药物通过抑制gyrA和gyrB编码的DNA回旋酶和/或parC、parE编码的DNA拓扑异构酶Ⅳ的活性,阻碍DNA合成来实现抑制细菌的生长[4]。
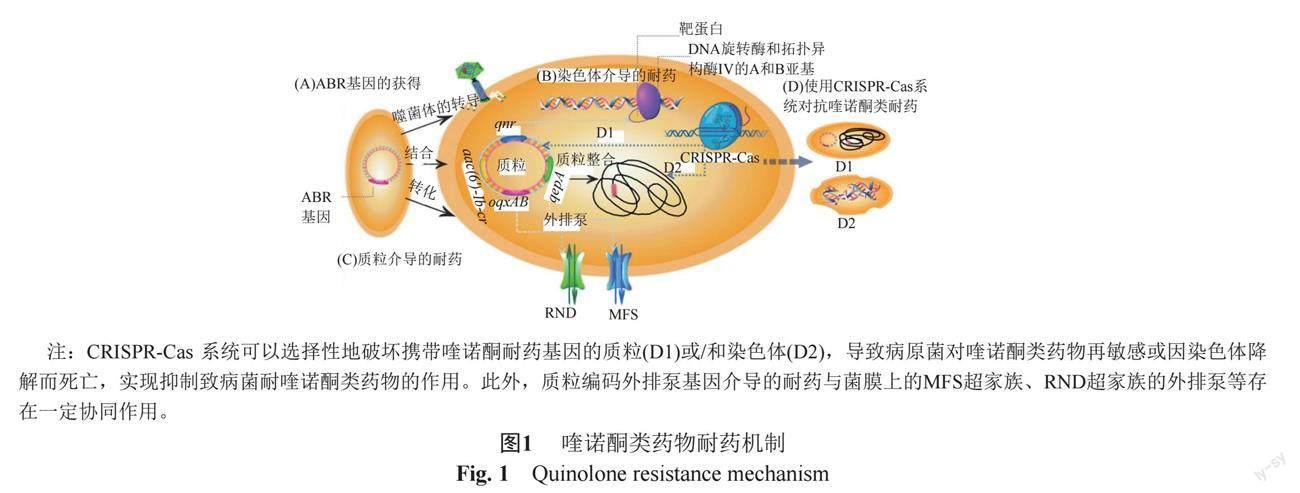
然而,随着临床使用喹诺酮类药物次数和剂量的增加,革兰阴性菌对其耐药率逐年上升。其中耐氟喹诺酮类药物的沙门菌已被WHO划分为急需开发新型抗生素的重点耐药菌之一[5-7]。细菌耐喹诺酮类药物的现象是多因素的结果,主要有:①染色体突变:药物作用靶位的改变、细胞膜通透性的改变和主动外排系统的过度表达;②质粒介导喹诺酮类药物耐药(plasmid-mediated quinolone resistance,PMQR),其中qnr、aac(6')-Ib-cr、qepA和oqxAB为主要的PMQR基因[8]。
细菌的适应性与药物的选择压力都会影响细菌耐药性的流行与传播[9]。细菌的适应性(fitness)一般指细菌在无抗生素的选择压力下,通过调节自身代谢来适应不同环境的能力[10]。而耐药菌适应性的变化已证明存在以下几种情况:①耐药菌产适应性代价。即在无抗生素压力下与敏感菌株相比,发生耐药突变或者获得外源基因的细菌自身适应性降低,表现为耐药菌的传播速度、毒力和竞争能力减弱,最终不敌敏感菌而被消灭。适应性代价也被认为是限制细菌耐药性传播的主要障碍,在一定程度上决定耐药菌能否在宿主体内或体外环境中长久存在[11]。
②耐药菌适应性增强。一方面是因耐药菌的补偿性进化,细菌为了改善自身适应性代价所发生的适应性突变,促进其与敏感菌株竞争。具体表现是产适应性代价的耐药菌在长期进化过程中,通过耐药基因的突变累积来缓解其适应性,促进耐药菌的进化而严重阻遏临床疗效[10,12]。另一方面是在肠杆菌科细菌进化的无数年历史中,虽然大多数耐药突变的出现均会对菌体产生或大或小的适应性代价,但还是存有无适应性代价和适应性增加的耐药突变现象[12]。
耐药菌株的适应性增强,导致细菌即使在缺乏抗菌剂使用的条件下,其耐药率也增加,所以在特定条件下,通过减少抗菌剂的使用也无法达到减弱细菌耐药率的目的。但有学者针对细菌的适应性降低可减弱耐药菌的传播速度和毒力,提出利用此机制有望逆转细菌的耐药性[13]。所以对耐药菌适应性的研究不仅有助于更全面地理解细菌耐药机制和流行性,还可促进人们探索并建立有效的方法来预测和控制耐药菌的流行[10]。革兰阴性菌耐喹诺酮药物日益严峻,本文主要从两方面进行综述:分析革兰阴性菌耐喹诺酮药物的不同机制对其适应性变化的影响和总结耐药菌适应性的研究方法,以期了解革兰阴性菌在不同耐药机制下的适应情况,为指导临床合理用药和帮助设计新型耐药抑制剂提出有针对性的解决思路与方法,从而达到有效控制我国耐药菌的目的。
1 染色体介导的喹诺酮耐药机制对细菌适应性的影响
1.1 靶基因突变对耐药菌适应性的影响
染色体介导细菌耐喹诺酮类药物,是因细菌喹诺酮类药物耐药决定区(plasmid-mediated quinolone resistance,QRDR)的主要和次要靶点(gyrA、gyrB、parC和parE)会发生至少一个氨基酸替换,引起喹诺酮类药物对酶-DNA复合物亲和力[14]降低而减弱药效(图1, B)。而适应性作为影响耐药菌进化的因素[15],已发现耐药菌的染色体突变会影响其适应性和耐药性[16],且不同突变位点会对菌株适应性产生不同影响[17]。
具体来说可提高菌株适应性的染色体突变有(表1):耐FQ的淋病奈瑟菌株中gyrA91/95(S91F和D95N)突变[18];大腸埃希菌中gyrA A119E突变[16];肠炎沙门菌中gyrA(D87N、D87G、D87A、S83Y、S83F)和/
或parC(S80I)存在1~3个突变时[19]可显著增加细菌适应性。降低菌株适应性的染色体突变有(表1):肺炎克雷伯菌中gyrB S431P、parC S80I突变[15]、淋病奈瑟菌株parC86(D86N)突变[18]、大肠埃希菌中的gyrB F458V和/或A468P和/或gyrAΔS83突变[16]均可引起相应耐药菌株的适应性降低,导致其在缺乏抗生素的情况下,竞争力弱于敏感的野生型菌株,无法在细菌种群中持续存在[15,20]。然而发生补偿突变的耐药菌株可通过恢复适应性和毒力重获竞争优势。如适应性降低的耐FQ的淋病奈瑟菌株通过多药外排泵MtrCDE的mtrR-79补偿性突变[18]、耐FQ的大肠埃希菌通过额外突变gyrA(D87Y/D87N/A119E)均可促进其适应性增强[16]。此外,在伤寒沙门菌中发现,在无抗生素压力条件下,gyrA和parC耐药突变菌株比敏感菌株有适应优势,而且由于基因的协同上位性,双突变体菌株比所有单突变体菌株的适应性高,说明抗性基因之间的协同上位性可能是增强菌株适应性、促进耐药菌株进化和流行的一个关键因素[21]。
1.2 外排泵对耐药菌适应性的影响
革兰阴性菌的膜上有许多外排泵,这些泵作为一个三方系统存在,可通过高水平表达输送不止一种类型的抗生素,成为肠杆菌科耐喹诺酮类药物和多药耐药性(MDR)出现的重要机制之一[26-27]。目前,属于RND超家族的AcrAB-TolC,是与喹诺酮耐药有关且研究较为清楚的外排泵。AcrAB-TolC高水平的表达,不仅能将小分子物质(代谢物、染料、洗涤剂、有机溶质和胆盐)从细菌细胞中转运出来,还可在胆汁酸条件下,增强菌株的耐药性和适应性[22-23]。而且在喹诺酮药物选择压力下,含QRDR突变和外排泵过表达的耐药鼠伤寒沙门菌比仅有QRDR突变的耐药肠炎突变菌更具有适应性优势,说明细菌可通过结合外排泵和染色体的靶基因突变增强耐药菌适应性[24](表1)。此外,针对外排泵协同细菌生物膜促进细菌耐药和增强适应性的现象,可通过外排泵抑制剂PAβN,阻断相关QS信号分子跨膜通道的转运,抑制其对菌株耐药的促进作用[28-29]。
1.3 外膜渗透性对耐药菌的适应性影响
细菌外膜渗透率大小主要取决于细菌菌株种类、所用的抗菌剂和生物膜的生长条件[30],其对菌株耐药的促进作用表现为孔道蛋白相关基因的缺失或突变,引起菌体外膜通透性下降,阻碍抗菌药物的进入而实现。如沙门菌通过下调OmpF的表达使膜孔蛋白缺失而对恩诺沙星产生低水平耐药[31]。同时,外膜渗透性的改变会影响细菌适应性[32]。如在Helix-turn-Helix(HTH)家族调节剂(包括AraC/XylS 家族)和双组分系统(TCS)的调节下,通过外排转运蛋白表达增加和/或孔蛋白表达减少,促使外膜渗透性降低,延迟抗生素的摄入而促使革兰阴性菌耐药性、适应性和毒力增强,有效说明外膜渗透性在细菌适应环境压力中起作用[25](表1)。
2 质粒介导的喹诺酮类耐药与细菌适应性之间的关系
PMQR机制主要有:编码DNA结合蛋白对靶点产生保护的qnr基因、引起氟喹诺酮失活的氨基糖苷乙酰转移酶变体aac(6')-Ib-cr和排泄疏水性氟喹诺酮类药物的QepA 和 OqxAB 等主动外排泵(由 qep、oqxA 和 oqxB 基因编码)[33](图1, C),而CRISPR基因编辑技术有助于解决临床菌株的抗性基因对细菌耐药性的促进作用(图1, D)。PMQR基因虽然只引起低水平的喹诺酮耐药,但PMQR基因之间的相互累加效应或与染色体突变位点的共同作用可促进菌株达到临床耐药水平[34-35],并在一定条件下降低或增强宿主菌的适应性,影响包括喹诺酮类药物在内的许多抗菌药物的有效性。此外,Oladeinde等[36]发现沙门菌暴露在酸性pH条件下可促进耐药质粒的水平转移和菌株适应性增强,为今后从环境入手改变细菌适应性来控制喹诺酮耐药性的发展提供新的角度。
2.1 PMQR基因对耐药菌适应性的影响
PMQR耐药机制常见的组合是aac(6')-Ib-cr-oqxAB和qnrB-aac(6')-Ib-cr-oqxAB[35],在PMQR 决定簇中acc(6')-Ib-cr基因的流行率较高,其次是qnrB、oqxAB和qnrS[37]。目前qnr基因介导肠杆菌科适应性研究主要集中在qnrA、qnrB和qnrS基因(表2)。兰芳俊等[38]通过重组质粒方式发现qnrA、qnrB和qnrS的存在会促使耐药菌的生长速率和最大细胞密度增强,而这三种qnr基因增强耐药菌适应性的效果却无明显差别,这也在Kaplan等[39-40]的研究中得到证实(表2)。然而,携带qnr基因的质粒大小不一,那质粒的大小不同又是否会影响菌体适应性呢?对于这个问题,Kaplan与San[40-41]已证明质粒大小不会影响耐药菌的适应性(表2)。此外,在没有喹诺酮类药物的情况下,具有和不具有 aac(6')-Ib-cr 基因的菌株之间的倍增时间和最大生长速率没有显著差异,但在环丙沙星暴露下,携带aac(6')-Ib-cr基因的菌株具有明显的适应性优势[42]。外排泵qepA和oqxAB的过度表达,可在无药物选择压力下,降低菌株适应性[16],
但从临床大肠埃希菌分离株获得的qepA1基因可与无活性I类整合子融合创造的新调节单元,不仅使qepA1的表达受环丙沙星的控制,还可减轻qepA1产生的适应性代价[43]。
2.2 PMQR基因与染色体突变组合对耐药菌适应性的影响
PMQR基因也常结合染色体突变进一步影响耐药菌的适应性。Machuca等[17]通过将qnr基因转化到大肠埃希菌,发现qnr基因与染色体自发突变构成的一到四个抗性突变组合均可增强菌株适应性(表2)。而将外排泵基因qepA2转入到含不同染色体突变的大肠埃希菌中,发现qepA2基因与gyrA(Ser83Leu和/
或Asp87Asn)和/或parC(Ser80Arg)突变菌株结合会产适应性代价,但通过marR基因的缺失可得到补偿,并促使耐药菌的适应性和MPC均增加[13](表2)。同样,在含gyrB F458V和/或A468P和/或gyrAΔS83突变的OqxAB阳性菌株中发现,虽然适应性减弱,但当额外突变gyrA D87Y出现时适应性和耐FQ均增强[16](表2)。此外,无论体内体外,当aac(6')-Ib-cr结合染色体上的4或5个突变位点时不仅促进细菌耐环丙沙星,还可增强细菌适应性[42](表2),进一步说明PMQR基因与染色体突变的一些特定组合会对耐药菌适应性产生重要影响。
2.3 PMQR基因与多重耐药性(MDR)
PMQR决定簇常存于肠杆菌科细菌的多药耐药质粒中,与其他耐药基因关联促进菌株多重耐药性(MDR)的产生。已发现acc(6')-Ib-cr、QRDR突变和qnr基因在多重耐药肺炎克雷伯菌中的共存频率很高[44],而且aac(6')-Ib-cr与blaCTX-M-15、qnr与 SHV或blaCTX-M-9的密切联系均会促进菌株的MDR,而且aac(6')-Ib-cr和blaCTX-M-27基因的共存会促进菌株适应性和生物被膜形成能力增强,对健康和经济造成相当大的负担[45-48]。此外,耐氟喹诺酮类药物的多重耐药菌的适应性变化可能是ST特异性的[20],但对于PMQR基因对多药耐药菌具体的适应性影响的研究还是相对较少,相信这将是未来的一个重要研究方向,并对解决耐喹诺酮类药物的现状提供新的视角。
3 适应性的研究方法
耐藥菌的相对适合性,是决定其在种群和其他环境中进化成功的关键参数。现如今随着分子生物学和生物信息学的发展,对细菌适应性的测量主要是从两方面进行,一方面是从细菌的适应性代价入手,另一方面是从细菌的补偿性突变进行。
3.1 适应性代价的测量
细菌适应性代价的衡量主要有两种方法:①实验性方法;②构建数学模型,具体是在流行病学大数据的基础上,将抗生素的使用量和耐药菌株频率的变化构建一个函数关系来实现对适应性代价的判断[50]。对于前者又分为两种:a:生长曲线,通过比较耐药突变型菌株和缺乏该突变的野生型菌株之间的生长速度差异来评价适应性(图2a);b:在体外(图2b)或小鼠胃肠道定植模型中进行的体内(图2c)竞争性实验。将耐药突变菌株与野生型菌株共培养,通过监测野生型菌株和耐药突变菌株随时间推移的菌数变化来衡量适应性,菌株计数高的则代表适应性强[50]。对于携带耐药质粒细菌的适应性代价测量公式[51]为1-WB/P,WB/P=ln(Nf,B/P/Ni,B/P) / ln(Nf,B/Ni,B),其中WB/P表示质粒携带菌的相对适应性,Ni,B/P和Nf,B/P 表示竞争前后质粒携带菌的密度,Ni,B和Nf,B表示竞争前后野生型菌的密度。然而,体外测量的结果可能无法完全反映临床环境中的情况,基于此杨焰学者已提出可结合临床研究和流行病学的综合数据来评价细菌的适应性代价,并且对于小的适应性效应,可通过数学模型和计算机预测相结合的竞争分析方法来测量。
3.2 适应性补偿突变的分析
耐药菌可通过代偿突变快速适应和进化,导致转移速率、毒力、生存能力增强。目前已发现存在两种补偿突变:染色体突变和质粒突变。染色体突变是通过细菌分裂垂直遗传,但质粒突变既可垂直也可水平遗传,而且能降低质粒到达新宿主细胞的初始成本。因此对细菌进行适应性补偿突变分析时,不仅要先判断适应性补偿突变是否发生,还要分析补偿突变是来源质粒/染色体或两者。
DelaFuente[51]等已描述了一个进化实验判断是否存在适应性代偿突变。具体是将携带进化质粒的进化菌株(Be/Pe)和携带原始质粒的野生型菌株(B/P)在LB培养基中繁殖10 d后,第一步测量Be/Pe相对于B/P的相对适合度:若WBe/Pe/B>WB/P/B,则说明由质粒产生的成本可能被补偿或菌株对实验条件产生了普遍适应,后者与适应性补偿无关。因此第二步判断是否存在适应性代价补偿:通过将Be/Pe中的质粒去除,将Be/Pe菌株与无质粒的对应物Be进行竞争实验后,计算Be/Pe菌株的适应性代价:1-WBe/Pe/Be,如果最终Be/Pe的适应性代价小于B/P菌株的,则说明菌株发生适应性补偿突变。一旦适应性补偿而且得到确认,就可调查其来源。DelaFuente等[51]是构建两株菌株(将进化质粒(Pe)结合到野生型细菌(B)得到新菌株B/Pe和将原始质粒(P)结合到进化的无质粒细菌(Be)得到新菌株Be/P)进行竞争实验,通过判断不同菌株的适应性代价大小判断补偿的起源。其中B/Pe的适应性代价为:1-WB/Pe/B、Be/P的适应性代价为:1-WBe/P/Be,若前者小于后者则说明适应性代价的补偿突变来源于质粒,反之则来源于细菌染色体,但要注意也可能是二者同时产生[51]。此外,一旦对补偿的起源进行了表型研究,下一步就可通过基因组测序和转录组学分析从分子层面进一步确定补偿突变和相关适应性代价的来源。
4 总结与展望
综上所述,目前国内外不少学者对耐喹诺酮类药物革兰阴性菌的适应性变化进行分析,发现在革兰阴性菌耐喹诺酮类药物肆虐的今天,与耐药菌适应性相关的适应性代价和补偿性进化的变化,主要取决于与染色体和/或可移动元件携带的与喹诺酮类药物相关的特定耐药基因的变化。本综述发现耐喹诺酮类药物革兰阴性菌的不同耐药机制造成耐药形式更加复杂的情况下,对耐药菌适应性变化的影响主要表现为:增强或减弱。同时,针对细菌适应性的降低可减弱耐药菌的传播速度和毒力,已有学者说明利用此机制有望逆转细菌的耐药性[13],所以本综述的分析不仅有助于补充细菌耐药监测和细菌适应性有关的喹诺酮耐药机制研究,还促进我们依据适应性降低有关的耐药机制来设计新型喹诺酮类药物。
此外,上述分析也为以后学者的研究提供以下借鉴:①耐喹诺酮类药物革兰阴性菌的适应性代价和补偿与耐药菌或抗生素的种类有无直接关系,假如存在,其作用的机理如何?②细菌耐喹诺酮类药物的机制和传播受地理区域差异的影响[52],那耐喹诺酮类药物革兰阴性菌的适应性变化是否也受特定区域的影响?③耐喹诺酮类药物基因的普遍存在与临床日益严重的MDR存在一定联系,但目前PMQR基因对多重耐药革兰阴性菌适应性影响的研究相对较少。我们坚信随着科学技术尤其是与分子生物学相关技术的不断进步,这些疑问在被解决的同时,一定会探索出更高效的策略去阻遏或控制与喹诺酮类药物相关的耐药菌的发展。
参 考 文 献
Redgrave L S, Sutton S B, Webber M A, et al. Fluoroquinolone resistance: Mechanisms, impact on bacteria, and role in evolutionary success[J]. Trends Microbiol, 2014, 22(8): 438-445.
陈杨, 刘理慧, 吴翠蓉, 等. 喹诺酮类耐药因qnr的研究进展[J]. 国外医药(抗生素分册), 2021, 42(4): 193-203.
牟豪, 王豪举, 丁红雷. 喹诺酮外排泵耐药基因oqxAB的耐药机制及其流行情况[J]. 微生物学通报, 2019, 46(6): 1510-1519.
Correia S, Poeta P, Hebraud M, et al. Mechanisms of quinolone action and resistance: Where do we stand?[J]. J Med Microbiol, 2017, 66(5): 551-559.
呂红玲, 刘春林, 邓德耀, 等. 临床常见病原菌对喹诺酮类抗菌药物的耐药性分析[J]. 检验医学与临床, 2021, 18(21): 3148-3152.
Cuypers W L, Jacobs J, Wong V, et al. Fluoroquinolone resistance in Salmonella: Insights by whole-genome sequencing[J]. Microb Genom, 2018, 4(7): e000195.
Tacconelli E, Carrara E, Savoldi A, et al. Discovery, research, and development of new antibiotics: The WHO priority list of antibiotic-resistant bacteria and tuberculosis[J]. 2018, 18(3): 318-327.
周珊, 刘家云, 曲芬, 等. 喹诺酮类药物对2018—2020年多中心临床分离菌的耐药性分析[J]. 中国抗生素杂志, 2021, 46(11): 1050-1053.
刘德俊. 携带ermS基因畜禽源弯曲菌的流行及适应性机制的研究[D]. 北京: 中国农业大学, 2017: 133.
陈品仙. CTX-M-64在养殖场中的流行分布及传播分子特征[D]. 广州: 华南农业大学, 2018.
Sherrard L J, Wee B A, Duplancic C, et al. Emergence and impact of oprD mutations in Pseudomonas aeruginosa strains in cystic fibrosis[J]. J Cyst Fibros, 2022, 21(1): e35-e43.
刘鹏, 王少林. 肠杆菌科细菌耐药性的遗传稳定性[J]. 中国兽医杂志, 2022, 58(2): 90-94.
Machuca J, Briales A, Diaz-de-Alba P, et al. Effect of the efflux pump QepA2 combined with chromosomally mediated mechanisms on quinolone resistance and bacterial fitness in Escherichia coli[J]. J Antimicrob Chemother, 2015, 70(9): 2524-2527.
Hooper D C, Jacoby G A.Topoisomerase inhibitors: Fluoroquinolone mechanisms of action and resistance[J].Cold Spring Harb Perspect Med, 2016, 6(9): a025320.
Knopp M, Andersson D I. Predictable phenotypes of antibiotic resistance mutations[J]. mBio, 2018, 9(3): e00770-18.
Wang J, Guo Z W, Zhi C P, et al. Impact of plasmid-borne oqxAB on the development of fluoroquinolone resistance and bacterial fitness in Escherichia coli[J]. J Antimicrob Chemother, 2017, 72(5): 1293-1302.
Machuca J, Briales A, Labrador G, et al. Interplay between plasmid-mediated and chromosomal-mediated fluoroquinolone resistance and bacterial fitness in Escherichia coli[J]. J Antimicrob Chemother, 2014, 69(12): 3203-3215.
Dillon J A, Parti R P. Fluoroquinolone resistance in Neisseria gonorrhoeae: Fitness cost or benefit?[J]. J Infect Dis, 2012, 205(12): 1775-1777.
Burgess D J.Microbial genetics: Fitness from drug resistance[J]. Nat Rev Genet, 2014, 15(2): 64.
Toth A, Kocsis B, Damjanova I, et al. Fitness cost associated with resistance to fluoroquinolones is diverse across clones of Klebsiella pneumoniae and may select for CTX-M-15 type extended-spectrum beta-lactamase[J]. Eur J Clin Microbiol Infect Dis, 2014, 33(5): 837-843.
Baker S, Duy P T, Nga T V, et al. Fitness benefits in fluoroquinolone-resistant Salmonella typhi in the absence of antimicrobial pressure[J]. ELIFE, 2013, 2: e1229.
Schaffner S H, Lee A V, Pham M, et al. Extreme acid modulates fitness trade-offs of multidrug efflux pumps MdtEF-TolC and AcrAB-TolC in Escherichia coli K-12[J]. Appl Environ Microbiol, 2021, 87(16): e72421.
徐紅红. 沙门菌的分子流行病学和耐药性状研究[D]. 扬州: 扬州大学, 2021: 66.
任思琪. 环丙沙星压力下鼠伤寒和肠炎沙门菌的适应性研究[D]. 广州: 华南农业大学, 2016: 48.
Ferrand A, Vergalli J, Pages J M, et al. An intertwined network of regulation controls membrane permeability including drug influx and efflux in Enterobacteriaceae[J].Microorganisms, 2020, 8(6): 833.
Blair J M, Richmond G E, Piddock L J. Multidrug efflux pumps in Gram-negative bacteria and their role in antibiotic resistance[J]. Future Microbiol, 2014, 9(10): 1165-1177.
Nabilou M, Babaeekhou L, Ghane M. Correction to: Fluoroquinolone resistance contributing mechanisms and genotypes of ciprofloxacin unsusceptible Pseudomonas aeruginosa strains in Iran: Emergence of isolates carrying qnr/aac(6)Ib genes[J]. Int Microbiol, 2022, 25(1): 223.
马光强. 贵州省畜禽主要病原菌耐药性及耐药基因研究[D]. 贵阳: 贵州大学, 2016: 135.
Dawan J, Li Y, Lu F, et al. Role of efflux pump-mediated antibiotic resistance in quorum sensing-regulated biofilm formation by Salmonella typhimurium[J]. Pathogens, 2022, 11(2): 147.
邱潇. 生物膜群感效应调控对细菌耐药性转移和毒力因子表达影响机制研究[D]. 济南: 山东大学, 2020: 78.
谷宇锋. 亚抑菌浓度恩诺沙星选择压力下肠炎沙门菌的耐药发生条件及机制研究[D]. 武汉: 华中农业大学, 2020: 162.
Nowak A, Zur-Pinska J, Pinski A, et al. Adaptation of phenol-degrading Pseudomonas putida KB3 to suboptimal growth condition: A focus on degradative rate, membrane properties and expression of xylE and cfaB genes[J].Ecotoxicol Environ Saf, 2021, 221: 112431.
Rodríguez-Martínez J M, Machuca J, Cano M E, et al. Plasmid-mediated quinolone resistance: Two decades on[J]. Drug Resist Updat, 2016, 29: 13-29.
Lin D, Chen K, Wai-Chi C E, et al. Increasing prevalence of ciprofloxacin-resistant food-borne Salmonella strains harboring multiple PMQR elements but not target gene mutations[J]. Sci Rep, 2015, 5: 14754.
Kim J O, Yoo I Y, Yu J K, et al. Predominance and clonal spread of CTX-M-15 in cefotaxime-resistant Klebsiella pneumoniae in Korea and their association with plasmid-mediated quinolone resistance determinants[J]. J Infect Chemother, 2021, 27(8): 1186-1192.
Oladeinde A, Abdo Z, Press M O, et al. Horizontal gene transfer is the main driver of antimicrobial resistance in broiler chicks infected with Salmonella enterica serovar heidelberg[J]. Msystems, 2021, 6(4): e72921.
Geetha P V, Aishwarya K, Mariappan S, et al. Fluoroquinolone resistance in clinical isolates of Klebsiella pneumoniae[J]. J Lab Physicians, 2020, 12(2): 121-125.
蘭芳俊. qnr基因对细菌喹诺酮耐药突变选择窗及其适应性代价影响的研究[D]. 福州: 福建医科大学, 2018: 60.
Michon A, Allou N, Chau F, et al. Plasmidic qnrA3 enhances Escherichia coli fitness in absence of antibiotic exposure[J].Plos One, 2011, 6(9): e24552.
Kaplan E, Marano R, Jurkevitch E, et al. Enhanced bacterial fitness under residual fluoroquinolone concentrations is associated with increased gene expression in wastewater-derived qnr plasmid-harboring strains[J]. Front Microbiol, 2018, 9: 1176.
San M A, Heilbron K, MacLean R C. Positive epistasis between co-infecting plasmids promotes plasmid survival in bacterial populations[J]. ISME J, 2014, 8(3): 601-612.
Machuca J, Ortiz M, Recacha E, et al. Impact of AAC(6')-Ib-cr in combination with chromosomal-mediated mechanisms on clinical quinolone resistance in Escherichia coli[J]. J Antimicrob Chemother, 2016, 71(11): 3066-3071.
Brandis G, Gockel J, Garoff L, et al. Expression of the qepA1 gene is induced under antibiotic exposure[J]. J Antimicrob Chemother, 2021, 76(6): 1433-1440.
Araujo B F, Campos P A, Royer S, et al. High frequency of the combined presence of QRDR mutations and PMQR determinants in multidrug-resistant Klebsiella pneumoniae and Escherichia coli isolates from nosocomial and community-acquired infections[J]. J Med Microbiol, 2017, 66(8): 1144-1150.
Higashino M, Murata M, Morinaga Y, et al. Fluoroquinolone resistance in extended-spectrum β-lactamase-producing Klebsiella pneumoniae in a Japanese tertiary hospital: Silent shifting to CTX-M-15-producing K. pneumoniae[J]. J Med Microbiol, 2017, 66(10): 1476-1482.
Park K S, Kim M H, Park T S, et al. Suh prevalence of the plasmid-mediated quinolone resistance genes, aac(6′)-Ib-cr, qepA, and oqxAB in clinical isolates of extended-spectrum beta-lactamase (ESBL)-producing Escherichia coli and Klebsiella pneumoniae in Korea Ann[J]. 2012, 42(2): 191-197.
Zeng L, Zhang J, Li C, et al. The determination of gyrA and parC mutations and the prevalence of plasmid-mediated quinolone resistance genes in carbapenem resistant Klebsiella pneumoniae ST11 and ST76 strains isolated from patients in Heilongjiang Province, China[J]. Infect Genet Evol, 2020, 82: 104319.
Jacoby G A. Study of Plasmid-mediated quinolone resistance in bacteria[J]. Methods Mol Biol, 2018, 1703: 317-325.
劉艳艳. 携带aac(6')-Ib-cr及aac(6')-Ib-cr+blaCTX-M-27基因菌株的适应性研究[D]. 广州: 华南农业大学, 2020.
杨焰, 游雪甫, 李聪然. 耐药细菌的适应性代价及补偿性进化[J]. 中国抗生素杂志, 2019, 44(7): 784-792.
DelaFuente J, Rodriguez-Beltran J, San M A. Methods to study fitness and compensatory adaptation in plasmid-carrying bacteria[J]. Methods Mol Biol, 2020, 2075: 371-382.
Abdel-Rahim M H, El-Badawy O, Hadiya S, et al. Patterns of fluoroquinolone resistance in Enterobacteriaceae isolated from the Assiut University Hospitals, Egypt: A comparative study[J]. Microb Drug Resist, 2019, 25(4): 509-519.
