基于转录组数据的芦笋EST-SSR标记的开发及通用性分析
2023-12-29仪泽会赵婧毛丽萍
仪泽会,赵婧,毛丽萍
基于转录组数据的芦笋EST-SSR标记的开发及通用性分析
仪泽会,赵婧,毛丽萍
山西农业大学园艺学院/蔬菜花卉种质资源创新与利用山西省重点实验室,太原 030031
【目的】明确芦笋转录组中SSR位点的分布规律,开发高信息量的EST-SSR标记并分析通用性,为天门冬属植物系统进化分析、功能基因挖掘及分子标记辅助育种提供工具。【方法】以笔者课题组前期获得的15个芦笋根系RNA-seq数据为基础,采用MISA软件检索SSR位点、Primer3.0软件批量设计引物及TB-tools软件批量e-PCR与Primer-blast程序逐一e-PCR相结合的方法剔除非有效引物。通过与基因组序列比对,获取标记所属基因id、物理位置、潜在功能等信息。随机合成引物50对,以9份芦笋、7份天门冬、5份文竹和3份蓬莱松种质为材料,检测引物有效性、多态性和通用性。【结果】共检索出SSR位点36 590个,分布于30 229条Unigene,发生频率为4.78%,平均间距为9.17 kb。SSR位点非均匀分布于10条染色体,7号染色体数量最多(4 642个),3号染色体密度最高(37.86 SSR/Mb)。SRR位点从单核苷酸至六核苷酸均有分布,以三核苷酸(46.92%)类型最丰富,AG/CT(16.58%)基序最具优势。成功设计EST-SSR引物19 695对,经e-PCR检测共剔除非有效引物15 147对,其中,3 085对无扩增产物、10 102对扩增产物条带大小与预期严重不符、1 289对物理位置未知、402对存在与目的条带大小相似的其他扩增产物、269对扩增产物无SSR序列。以位于基因区域的2 517个EST-SSR标记为基础,构建染色体密度分布图,总覆盖长度1 125.51 Mb,平均图距447.16 kb,潜在功能涉及产量、品质及抗逆性等多方面。随机合成的50对引物均可扩增出清晰目标条带,其中36对具有多态性,平均多态性信息含量为0.330,在天门冬、文竹和蓬莱松中的通用性分别达到100.00%、92.00%和88.00%。基于EST-SSR鉴定结果的聚类分析,可将24份材料分为芦笋、天门冬、文竹和蓬莱松4类,与植物学分类完全一致。【结论】成功开发出高信息量的芦笋EST-SSR标记2 517个,有效扩增率100%,总覆盖长度1 125.51 Mb,平均图距447.16 kb,可用于芦笋及其近缘物种的系统进化分析。同时,可为其他物种的EST-SSR标记开发提供借鉴。
转录组;芦笋;EST-SSR;染色体密度分布图;功能注释
0 引言
【研究意义】芦笋(L.)是一种重要的蔬菜作物,天门冬科天门冬属,富含氨基酸、维生素、矿物质等人体必需营养元素,以及皂苷、芦丁、黄酮等生物活性成分,具有较高的营养、保健和药用价值,深受消费者喜爱[1-2]。芦笋是典型的雌雄异株、多年生植物,基于表型选择的传统育种手段存在周期长、难度大等缺陷,导致品种更新换代速度难以满足生产需求,尤其是缺少抗病、高品质和设施专用品种[1]。分子标记辅助选择(MAS)是对基因型进行直接选择,不受生长条件、取材部位和株龄影响,可实现对目标基因的加速聚合,缩短育种周期[3]。目前,由于缺乏高质量的分子标记,致使芦笋分子辅助选择育种进展缓慢,远落后于大白菜、辣椒等蔬菜作物。EST-SSR分子标记的开发可为MAS提供分子工具,对实现芦笋育种早期选择、提高育种效率具有重要意义。【前人研究进展】简单重复序列(simple sequence repeats,SSR)是由1—6个核苷酸组成的基序,经多次重复形成的DNA片段,广泛分布于基因组[4]。SSR基序重复次数高度变化、侧翼序列相对保守,导致PCR产物长度多态性,SSR分子标记应运而生。与RAPD、SRAP等分子标记相比,SSR标记具有分布广泛、多态性高、重复性好、共显性和操作简便等优点[5]。EST-SSR来源于基因内部,保守性更高,基序重复次数变化即可能对基因功能或表达产生影响[5]。因此,与g-SSR相比,EST-SSR往往具有更高的种间通用性、标记辅助选择效率和功能标记潜在性,利用价值更高[5]。目前,利用转录组数据开发EST-SSR标记已广泛应用于牡丹[6]、开心果[7]、巢蕨[8]、象草[9]等众多植物中,极大促进了植物的遗传多样性分析[10]、遗传图谱构建[11]、重要农艺性状定位[12-13]及分子标记辅助选择育种[14-15]等研究。Li等[16]基于芦笋基因组序列开发了41 917个SSR标记,但引物有效扩增率、标记物理位置、所属基因id及潜在的生物学功能等信息未知,影响使用效率;盛文涛等[17]基于EST序列开发了424个EST-SSR标记,但引物有效扩增率仅为84%,类似的情况也存在于牡丹(51.50%)[6]、紫楠(43.62%)[18]苏丹草(76.7%)[19]苔草(74.61%)[20]和芒草(75.30%)[21]等。【本研究切入点】目前,芦笋中可供利用的分子标记数量仍非常有限,尤其是缺乏高质量的EST-SSR分子标记。大量非有效引物的存在,不仅增加了鉴定成本,而且影响基因型鉴定的准确性。【拟解决的关键问题】以笔者课题组前期获得的芦笋根系转录组数据为基础,挖掘SSR位点并分析其在转录组中的分布规律;以芦笋基因组序列为模板,采用TB-tools批量e-PCR与Primer-blast逐一e-PCR相结合的方法剔除非有效扩增引物;通过与基因组序列比对,完善EST-SSR标记的所属基因id、物理位置及潜在生物学功能等信息,以丰富芦笋EST-SSR分子标记数量,为天门冬属植物系统进化分析、分子标记辅助育种及优异功能基因挖掘等奠定基础。
1 材料与方法
试验于2021—2022年在山西农业大学东阳试验示范实习实训基地和蔬菜花卉种质资源创新与利用山西省重点实验室进行。
1.1 试验材料
以24份天门冬属植物为材料,其中,芦笋9份、天门冬7份、文竹5份、蓬莱松3份(表1)。转录组数据来源于对15个芦笋根系盐胁迫转录组的Illumina高通量测序,共128.42 Gb数据,856 152 362个原始读长。
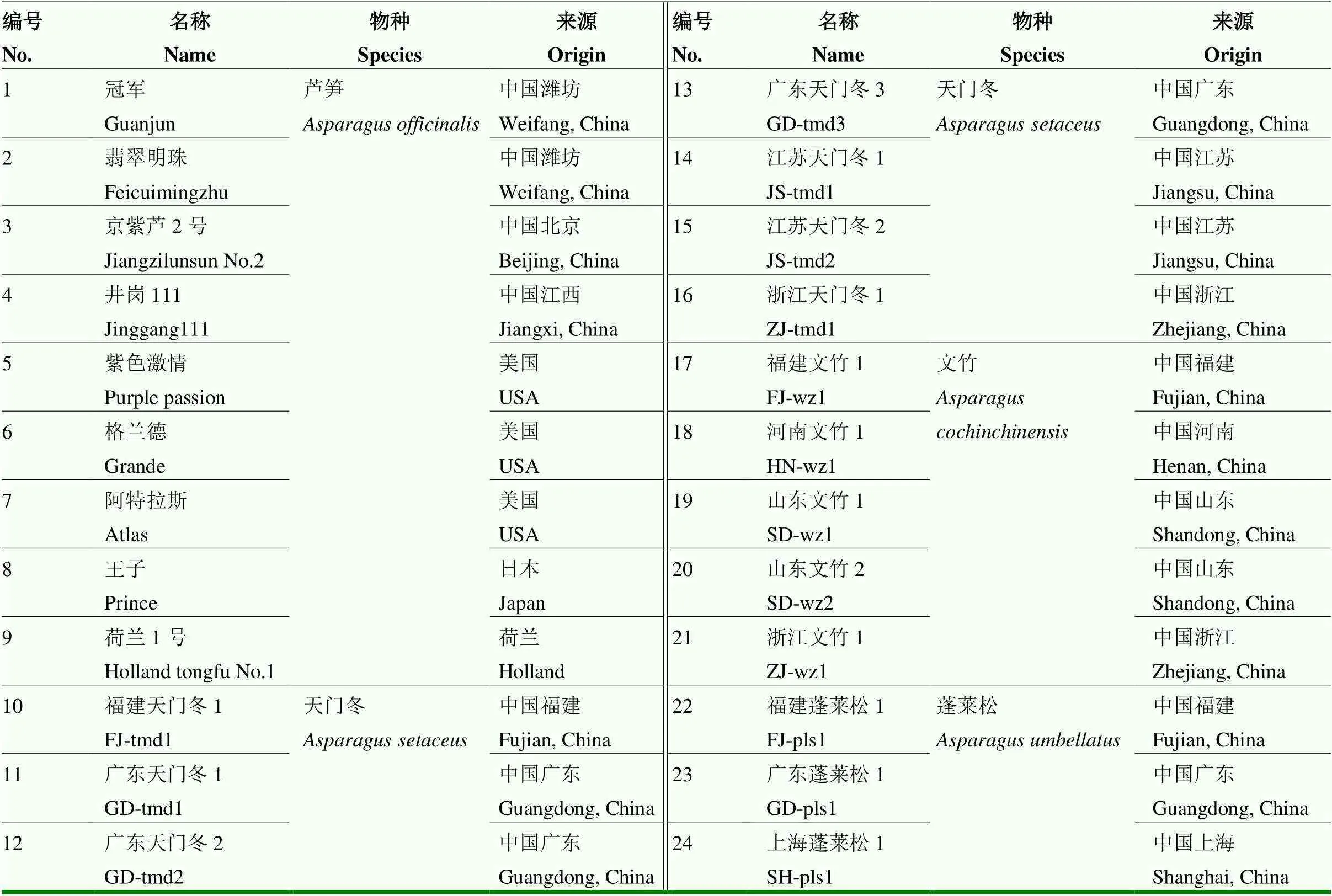
表1 24份天门冬属种质信息
1.2 测序数据的处理、SSR位点搜索及引物设计
测序数据经过滤,剔除带接头、N比例大于10%和低质量的序列,获得828 661 100个去杂读长。随后,用De Novo拼接和去冗余获得Unigene。采用MISA(http://pgrc.ipk-gater sleben.de/misa/)软件检索SSR位点,检索条件为1—6核苷酸重复次数分别为12、6、5、5、4和4。若两个SSR间的距离≤50 bp,则认定为复合SSR位点。SSR位点发生频率(%)=SSR位点数量×100/Unigene数量;SSR位点平均距离=Unigene总碱基数量/SSR位点数量。采用Primer 3.0软件批量设计EST-SSR引物,参数为:引物长度18—25 bp,扩增长度为100—300 bp,退火温度为50—65 ℃,GC含量维持在45%—65%,无发卡和引物二聚体等二级结构。每个位点设计3对引物,择优选用。
1.3 引物质量的e-PCR检测、物理图谱构建及功能注释
以芦笋基因组为模板,采用TB-tools软件对引物质量进行批量e-PCR检测,检测条件为:距3'端5 bp范围内无错配碱基,总错配碱基数≤2。据检测结果,择优选取SSR引物,剔除无扩增(Ⅰ类)、扩增产物长度<100 bp或>300 bp(Ⅱ类)和物理位置不确定的引物(Ⅲ类)。初步筛选出的引物,再经NCBI的Primer-blast逐一e-PCR质量检测,剔除扩增产物存在2个及以上与目的条带大小相似的引物(Ⅳ类)及无SSR位点的引物(Ⅴ类)。通过与芦笋基因组序列比对,完善SSR标记的物理位置、所属基因及基因区域等信息,进一步剔除所属基因不明确的引物(Ⅵ类)。据标记物理位置,采用Phython软件绘制染色体密度分布图。据标记所属基因序列,利用GO(gene ontology)数据库和KEGG(kyoto encyclopedia of genes and genomes)数据库进行功能注释和代谢通路分析。
1.4 芦笋DNA提取及PCR检测
从各材料中选择最具代表性的5株,取幼叶、等量混合后,采用植物DNA提取试剂盒(上海生工生物工程有限公司)提取DNA,采用NanoDrop 1000检测DNA质量和浓度,于-80 ℃保存备用。随机选取均匀分布于10条染色体的50对SSR引物用于PCR检测,由上海生工生物工程有限公司合成。PCR反应总体系为10 µL,包括50 ng·µL-1DNA模板0.2 µL、10 µmol·L-1引物0.5 µL、2×Taq PCR Mix 3 µL、灭菌ddH2O 5.8 µL。PCR扩增程序为:94 ℃预变性4 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸35 s,循环30次;72 ℃延伸10 min。PCR扩增产物采用8.0%的非变性聚丙烯酰胺凝胶电泳分离,分别采用0.2% AgNO3和1.5% NaOH染色和显色。产物回收方法:从PAGE胶中割取目标片段,用100 µL ddH2O洗涤2次后,加入1×PCR Buffer 100 µL并将凝胶碾碎,65 ℃下水浴10 min,离心,以1 µL上清液为模板,进行PCR扩增。扩增产物经琼脂糖凝胶电泳检测,若为目标条带,则直接进行产物测序。
1.5 数据分析
利用Popgen32软件计算多态性信息含量(PIC),采用Microsoft Excel 2010软件进行数据整理,用Origin 2019和Phython软件绘图。
2 结果
2.1 转录组测序及组装
从15个芦笋根系转录组中共测得856 152 362个原始读长,经过滤获得828 661 100个去杂读长,Q30达90.81%,GC含量为48.45%。经De Novo组装和去冗余,共获得631 883条Unigene,总长度为335 428 792 bp,其中,176 961条Unigene长度≥500 bp,平均长度为530 bp,N50为701 bp,可用于后续分析。
2.2 SSR位点的分布
共检索到SSR位点36 590个,分布于30 229条Unigene,发生频率为4.78%,平均密度为109.1 SSR/Mb,即每9.17 kb出现一个SSR位点。其中,4 974条Unigene含2个及以上SSR位点,2 233条Unigene以复合SSR位点形式存在。SSR位点非均匀分布于10条染色体,其中,7号染色体数量最多(4 642个),3号染色体密度最高(37.86 SSR/Mb)(图1)。
SSR重复类型从单核苷酸到六核苷酸,数量差异明显(表2)。其中,三核苷酸(46.92%)占比最高,其次为二核苷酸(27.18%)和单核苷酸(19.02%)(表2)。SSR重复次数分布范围为4—65次,其中4—14次重复占比达88.23%,主要集中在5次(34.10%)、6次(20.23%)、7次(10.77%)、8次(7.06%)和12次(6.19%)重复(表2)。单核苷酸至六核苷酸的优势重复次数分别为12、6、5、5、4和4次,分别占各自重复类型的24.31%、33.09%、52.92%、64.02%、77.73%和85.56%。
共发现337种SSR基序,单核苷酸至六核苷酸分别为2、4、10、29、91和201种。A/T、AG/CT、AGC/GCT、AAAT/ATTT分别为单核苷酸至四核苷酸的优势重复基序,分别占各自重复类型的86.62%、61.01%、28.22%和19.86%;AAAAT/ATTTT和AAAAG/CTTTT是五核苷酸重复的优势基序,占比分别为14.06%和13.54%;六核苷酸重复中,基序AAAAAG/CTTTTT和AACCCT/ AGGGTT分布具有一定优势,占比分别为7.38%和6.31%(图2)。SSR长度分布范围为12—190 bp,以12—25 bp为主,占总数的87.69%。其中,长度15 bp占比最高(24.03%),其次为18 bp(12.20%)和12 bp(11.92%)(图3)。
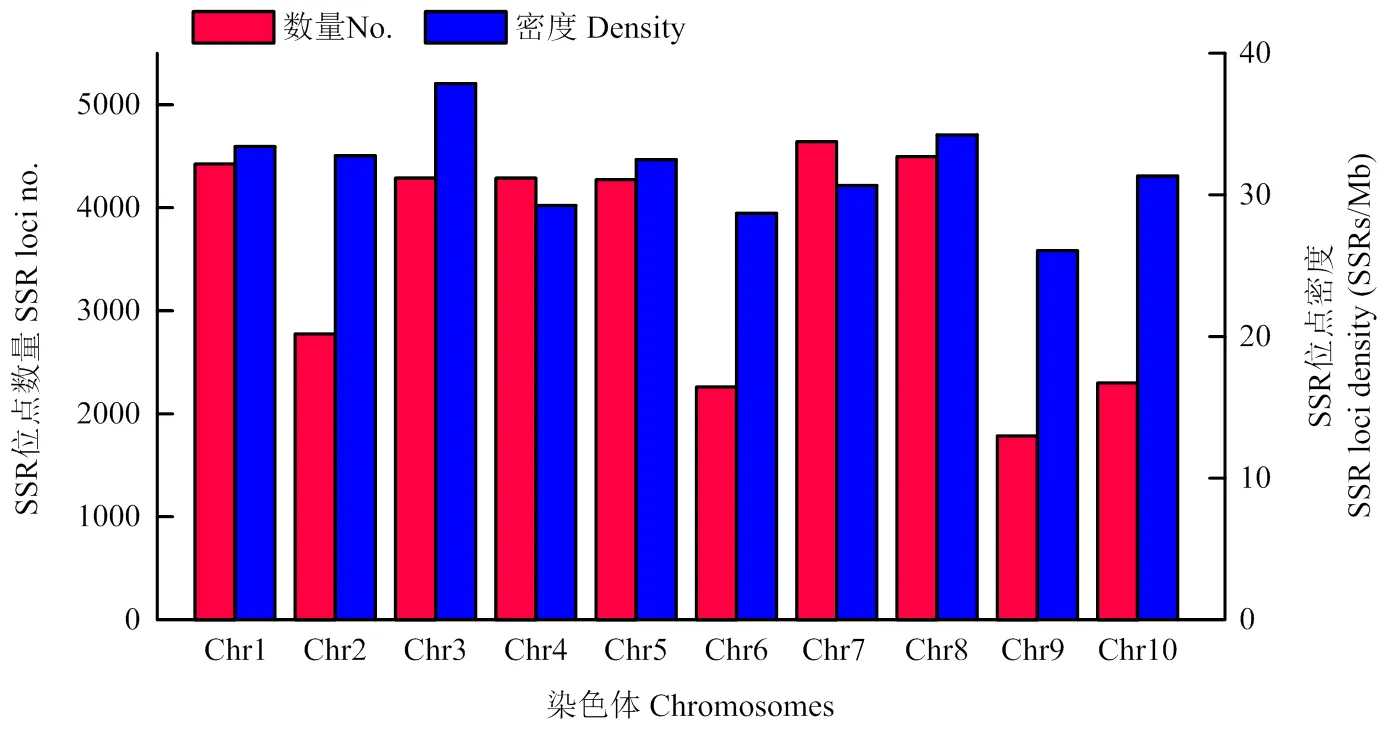
图1 芦笋转录组SSR位点在染色体中的分布特征

表2 SSR位点在芦笋转录组中的分布特征
2.3 SSR标记的开发、e-PCR质量检测及图谱构建
36 590个SSR位点中,共有19 695个位点成功设计出EST-SSR引物。经TB-tools软件批量e-PCR质量检测,共剔除SSR引物14 476对,其中,Ⅰ类3 085对、Ⅱ类10 102对及Ⅲ类1 289对。采用NCBI中的Primer-blast对筛选出的5 219对SSR引物逐一e-PCR质量检测,共剔除SSR引物671对,其中,Ⅳ类402对、Ⅴ类269对。随机选择Ⅰ—Ⅴ类引物各10对进行PCR验证(附表1,https://pan.baidu.com/s/1wgSxI9ru830awx1FSeyMTg? pwd=8888)。结果表明,Ⅰ类引物中,均无扩增产物;Ⅱ类引物中,10对引物的扩增产物大小均与预期严重不符,经测序,6对引物的扩增产物含内含子,4对引物的扩增产物为非目标条带;Ⅲ类引物中,10对均可扩增出预期条带,产物序列经与芦笋基因组比对,均无法获得具体物理位置;Ⅳ类引物中,10对引物均存在2个或以上的扩增条带,经测序发现存在与目标序列大小相似的非目标序列,对基因型鉴定造成严重干扰;Ⅴ类引物中,10对引物的扩增产物大小与预期相似,但序列均不含SSR位点。进一步对4 548个有效扩增的EST-SSR序列进行基因区域分析,发现2 031个位点位于基因间区(Ⅵ类),2 517个位点位于基因区域,共涉及2 208个基因。以2 517个SSR标记的物理位置为基础,构建染色体密度分布图,整体呈现两端密集、中间稀疏的特点(图4),总覆盖长度1 125.51 Mb,平均密度为2.24个/Mb,平均图距447.16 kb。其中,7号染色体SSR标记数量最多(340个),3号染色体密度最高(2.70个/Mb)。SSR标记的引物序列、产物大小、物理位置、所属基因、基因区域、潜在功能及可能涉及的农艺性状等信息详见附表2(https://pan.baidu.com/s/1aVnuo3uI13QeXN9HbLSr3g? pwd=8888)。
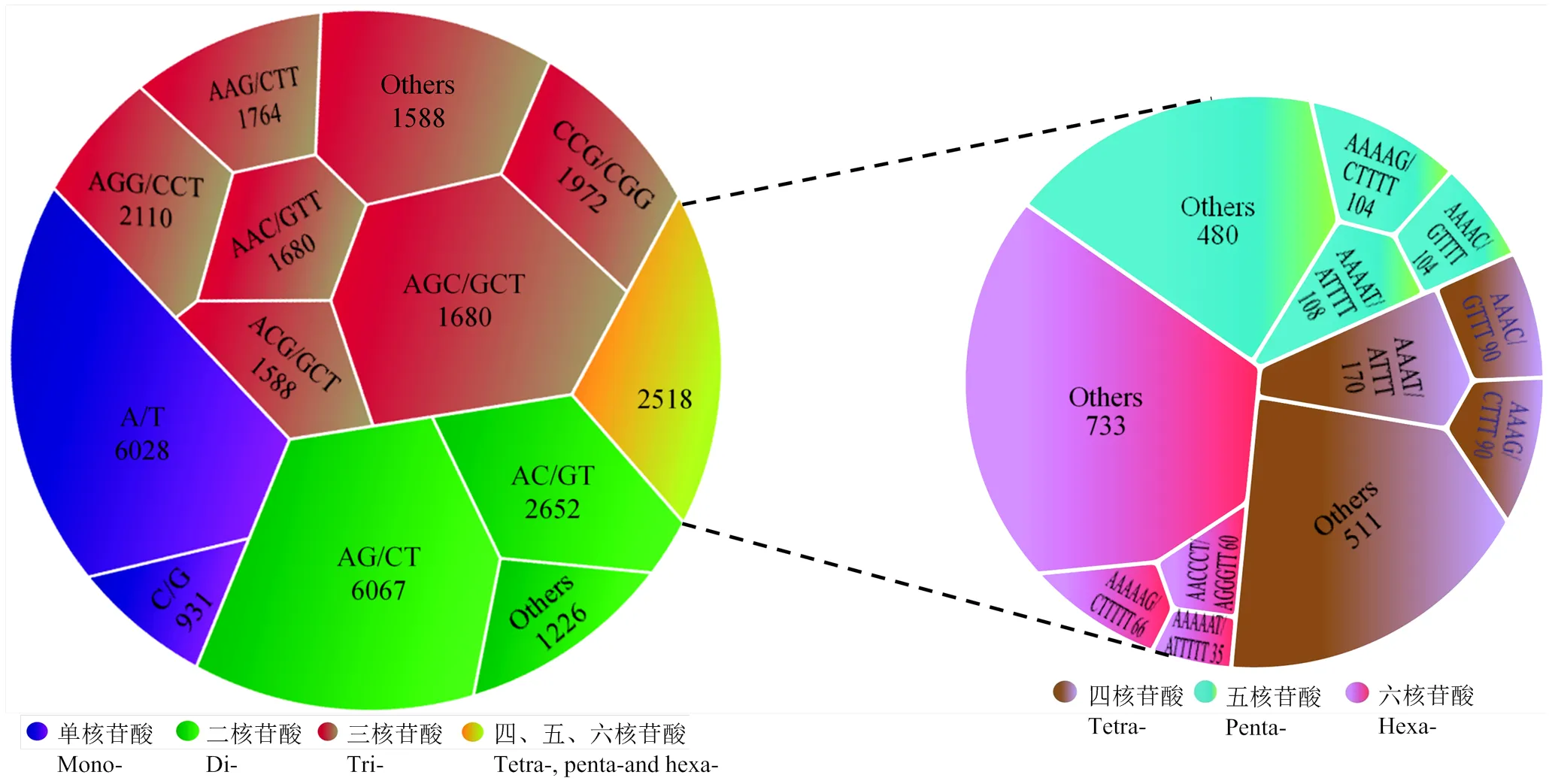
图2 芦笋转录组中的SSR基序及分布
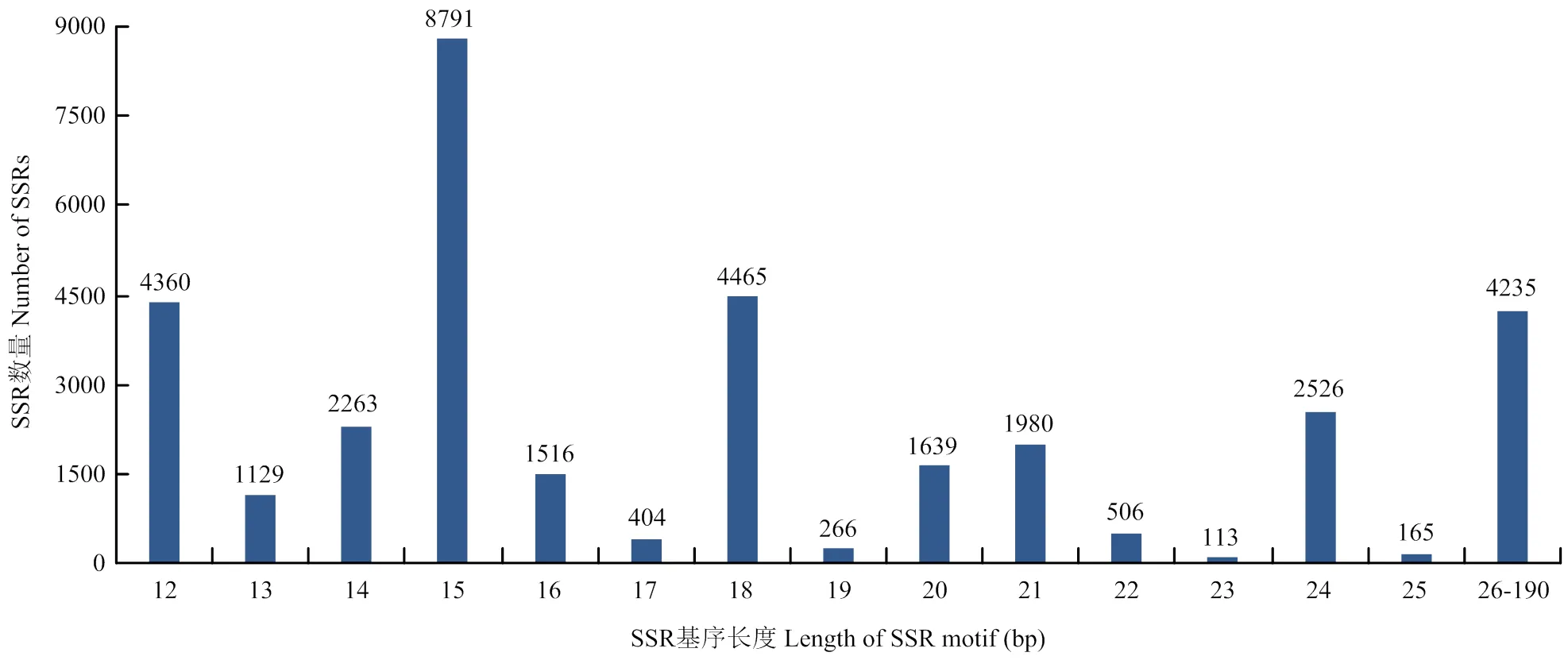
图3 芦笋转录组中的SSR基序长度分布
2.4 SSR标记所属基因的功能注释
将包含2 517个EST-SSR标记的2 208个基因与GO数据库比对,共有1 902个基因被注释,分别有1 459、1 501和1 676个基因被注释到生物学过程、细胞组分与分子功能,共涵盖5 143个子集。三大GO功能分类中,占比最高的分别为代谢过程、细胞膜和蛋白结合,分别为21.26%、46.19%和48.56%。SSR位于编码区的基因中,最丰富的GO条目为蛋白结合(12.56%),其次为细胞过程(11.53%)和细胞器(10.99%);SSR位于3'-UTR的基因中,蛋白结合(11.79%)和细胞器(11.79%)条目最丰富,其次为细胞过程(11.23%);SSR位于5'-UTR的基因中,细胞器(10.95%)和细胞过程(10.95%)条目最丰富,其次为蛋白结合(9.94%);SSR位于内含子的基因中,细胞过程(12.16%)条目占比最高,其次为细胞器(11.45%)和代谢过程(9.24%)(图5)。
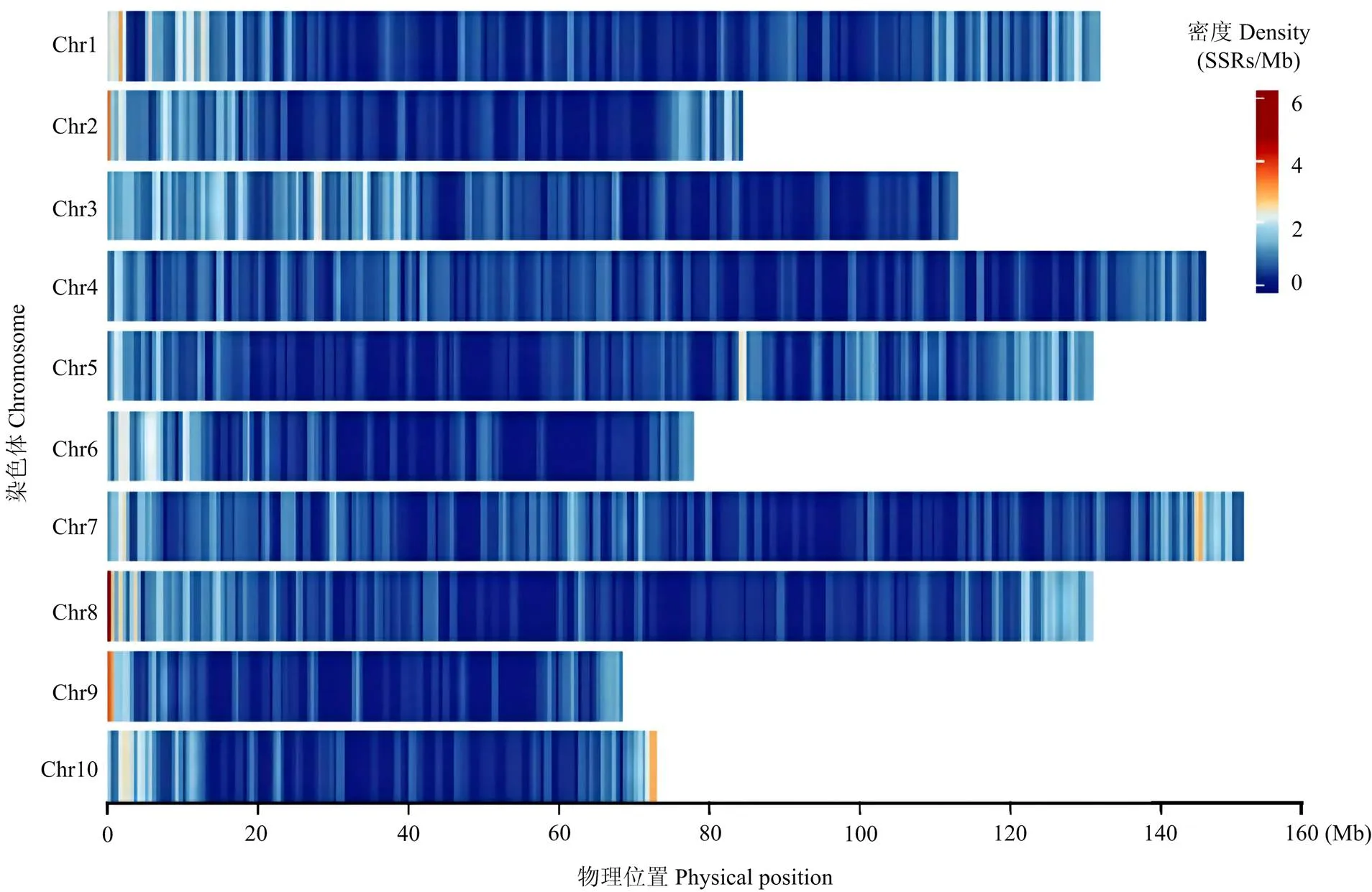
图4 2517个EST-SSR标记在染色体中的分布
KEGG分析发现,2 208个基因中有471个基因被注释到222条代谢通路中,归为细胞过程、环境信息处理、遗传信息处理、新陈代谢和生物系统5个类别,分别包含43、54、110、214和50个基因,优势通路分别为运输与代谢、信号传导、翻译、碳水化合物代谢和发育,占比分别为54.95%、98.78%、54.35%、16.76%和39.51%。SSR位于编码区的基因中,最丰富的通路为信号转导(14.16%),其次为翻译(12.33)和碳水化合物代谢(7.76%);SSR位于3'-UTR的基因中,翻译(12.20%)最为丰富,其次为次级代谢产物合成(11.23%);SSR位于5'-UTR的基因中,信号转导(11.57%)最为丰富,其次为碳水化合物代谢(8.26%)和翻译(7.44%);SSR位于内含子的基因中,翻译(14.64%)通路占比最高,其次为信号转导(8.19%)和氨基酸代谢(7.69%)(图6)。
2.5 50对SSR标记的多态性检测
随机合成的50对EST-SSR引物均可扩增出清晰的目标条带(图7),有效扩增率为100%(表3)。其中,36对引物存在多态性扩增,比例为72.00%(图7)。50对SSR引物共扩增出条带100种,平均每对引物具有2个等位基因;多态性信息含量(PIC)分布于0—0.691,均值为0.330(表3)。位于基因不同区域的SSR多态性存在明显差异。SSR位于CDS、3'-UTR、5'-UTR和Intron的引物中,多态性引物比例分别为64.29%、100.00%、85.71%和76.92%;平均每对引物分别可扩增1.89、2.5、2.14和2.15种条带,平均PIC值分别为0.253、0.544、0.374和0.319;14对非多态性引物中,SSR位于CDS的引物占据10对,比例达71.43%。
2.6 EST-SSR标记的通用性分析
50对随机EST-SSR引物中,分别有50对、46对和44对在天门冬、文竹和蓬莱松中成功扩增出清晰目标条带(图7),通用性分别达100.00%、92.00%和88.00%。其中,具有多态性的引物分别有25、10和11对,多态性比例分别为50.00%、21.74%和25.00%。说明本研究开发的芦笋EST-SSR标记在近缘物种天门冬、文竹、蓬莱松中均具有较高的通用性。聚类分析表明,24份天门冬属材料被分为4类,分类结果与植物学分类完全一致(图8),表明本研究开发的EST-SSR标记可用于天门冬属植物的系统进化分析。
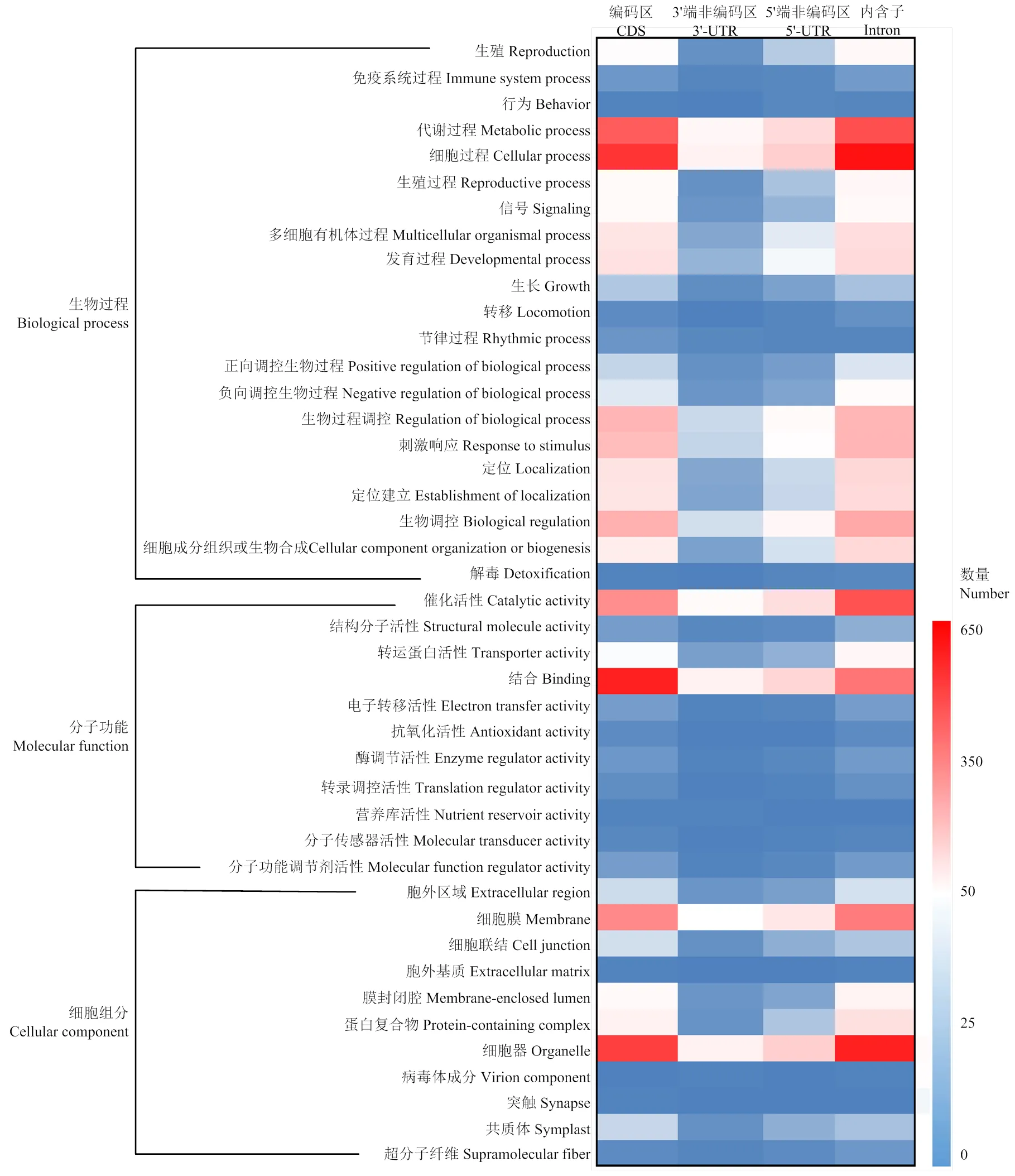
图5 EST-SSR标记所属基因的GO功能注释
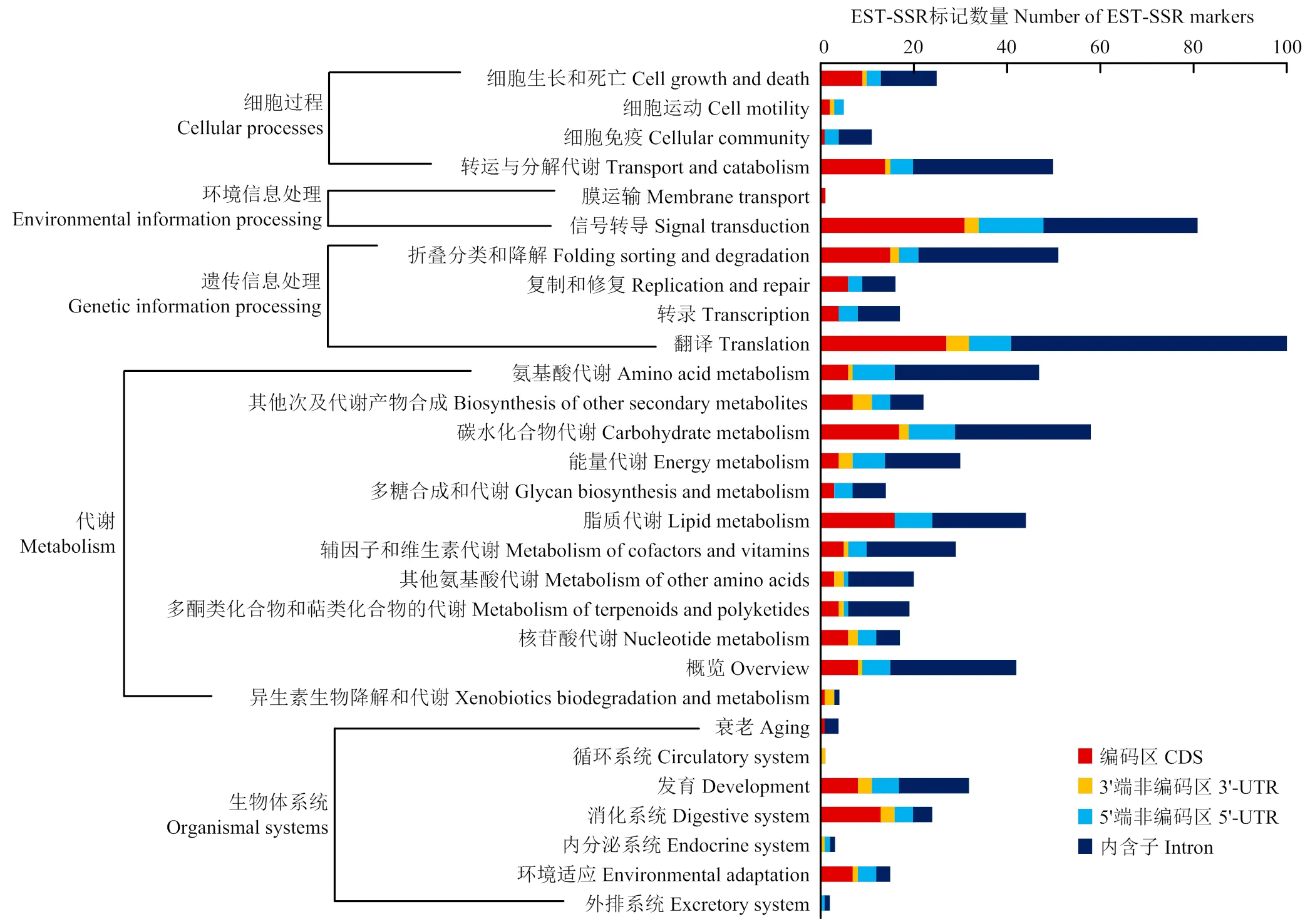
图6 EST-SSR标记所属基因的KEGG分析
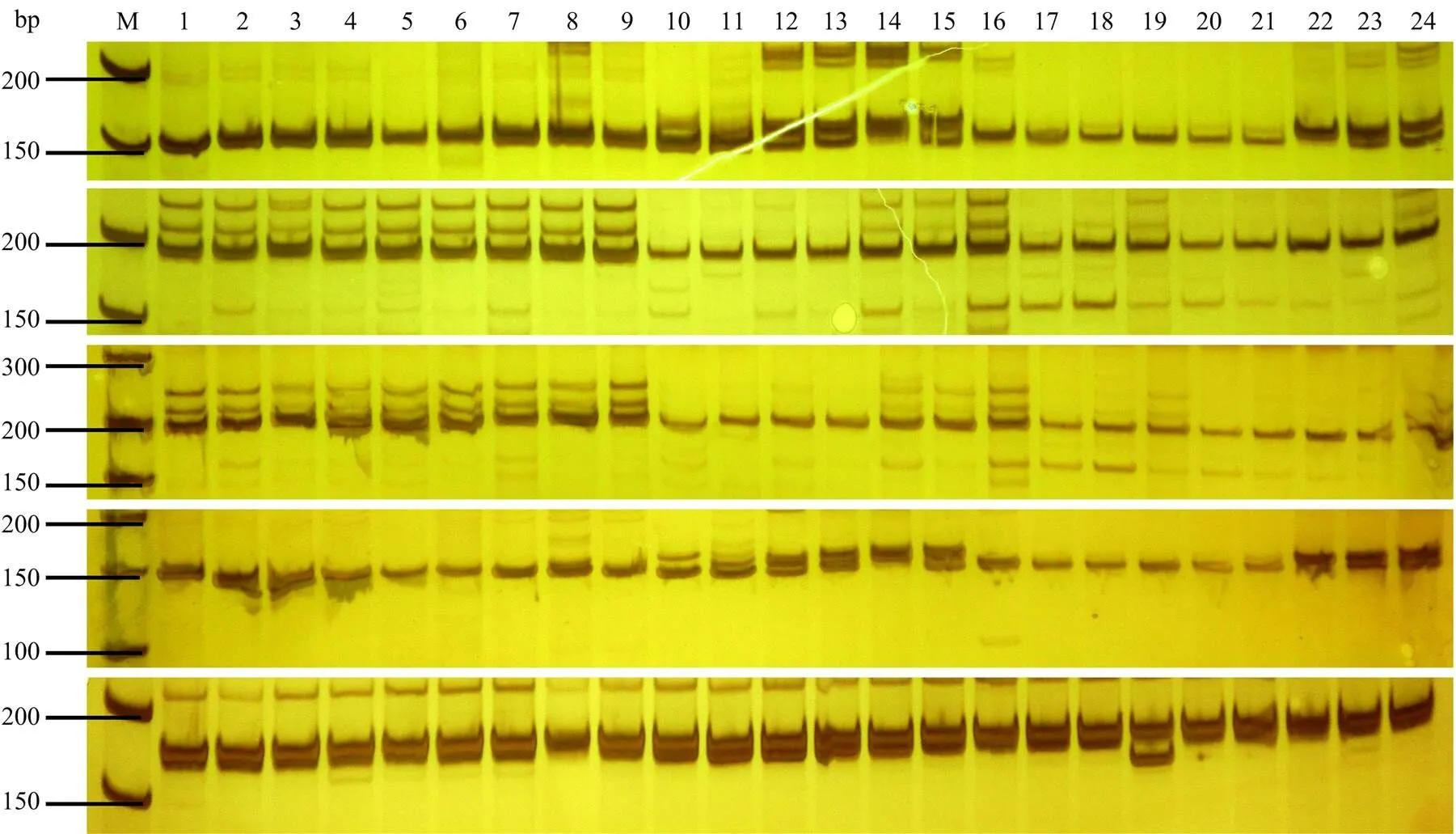
图7 随机EST-SSR引物在24份天门冬属种质中的扩增(部分)
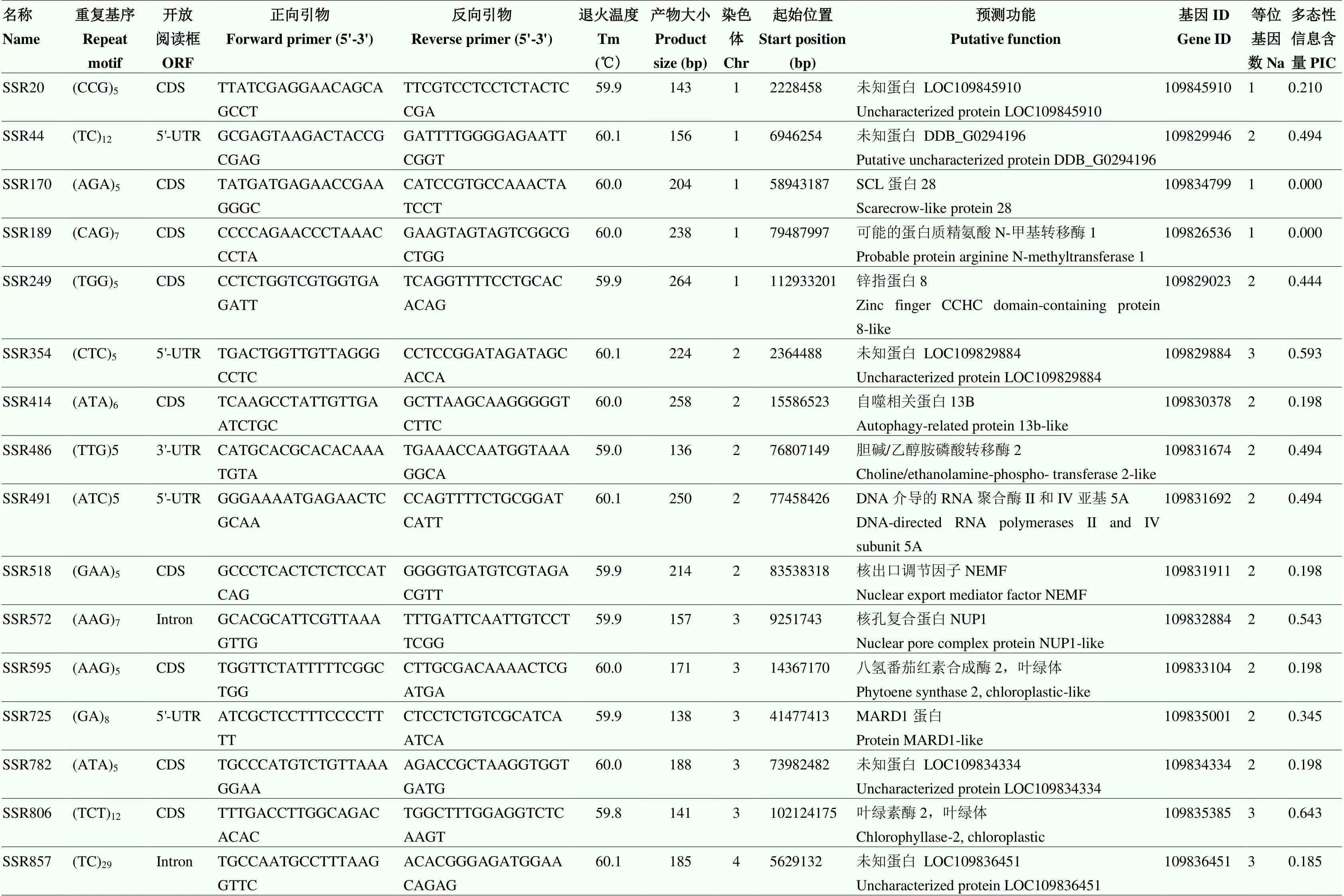
表3 50对随机合成的芦笋EST-SSR引物信息
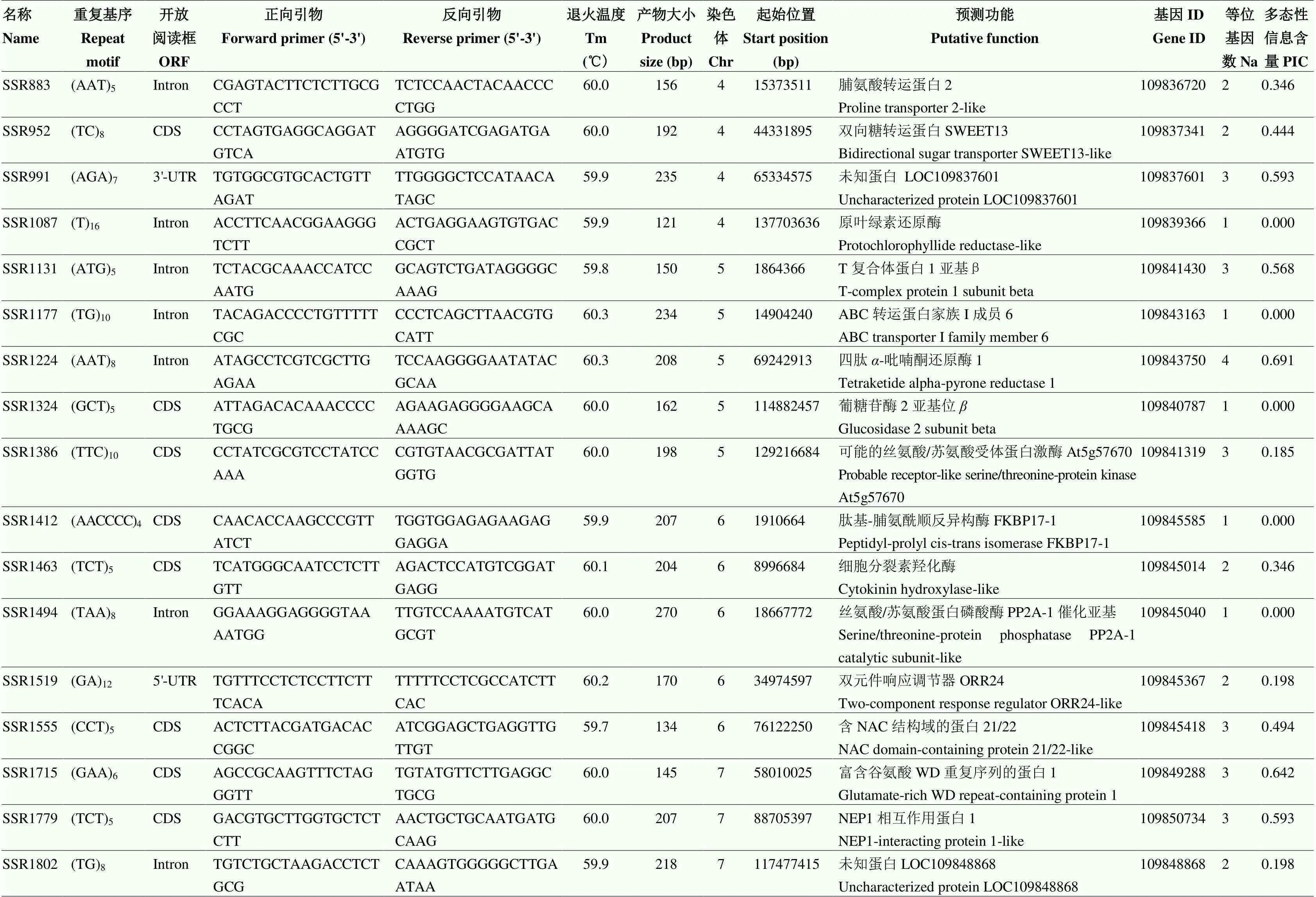
续表3 Continued table 3
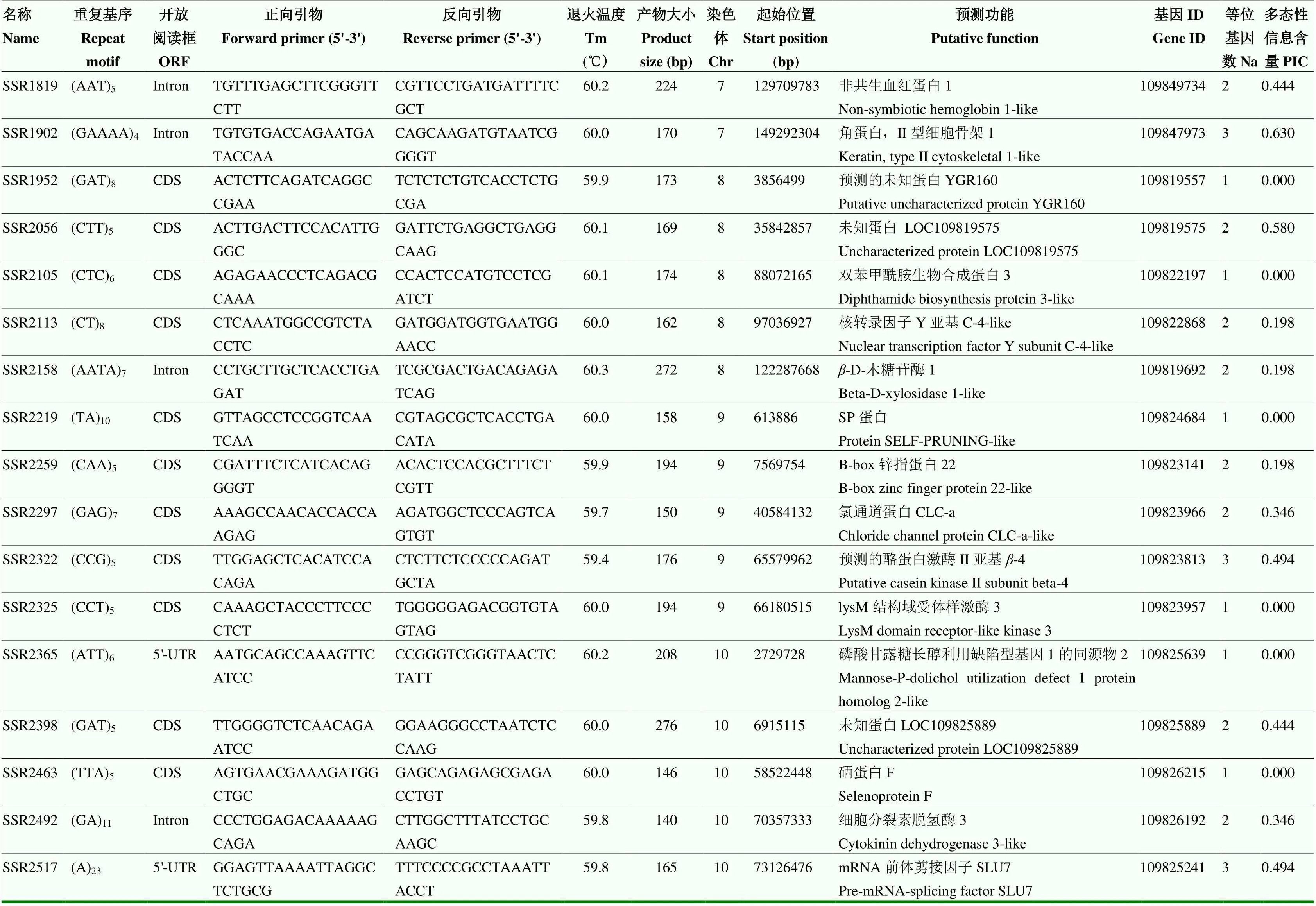
续表3 Continued table 3
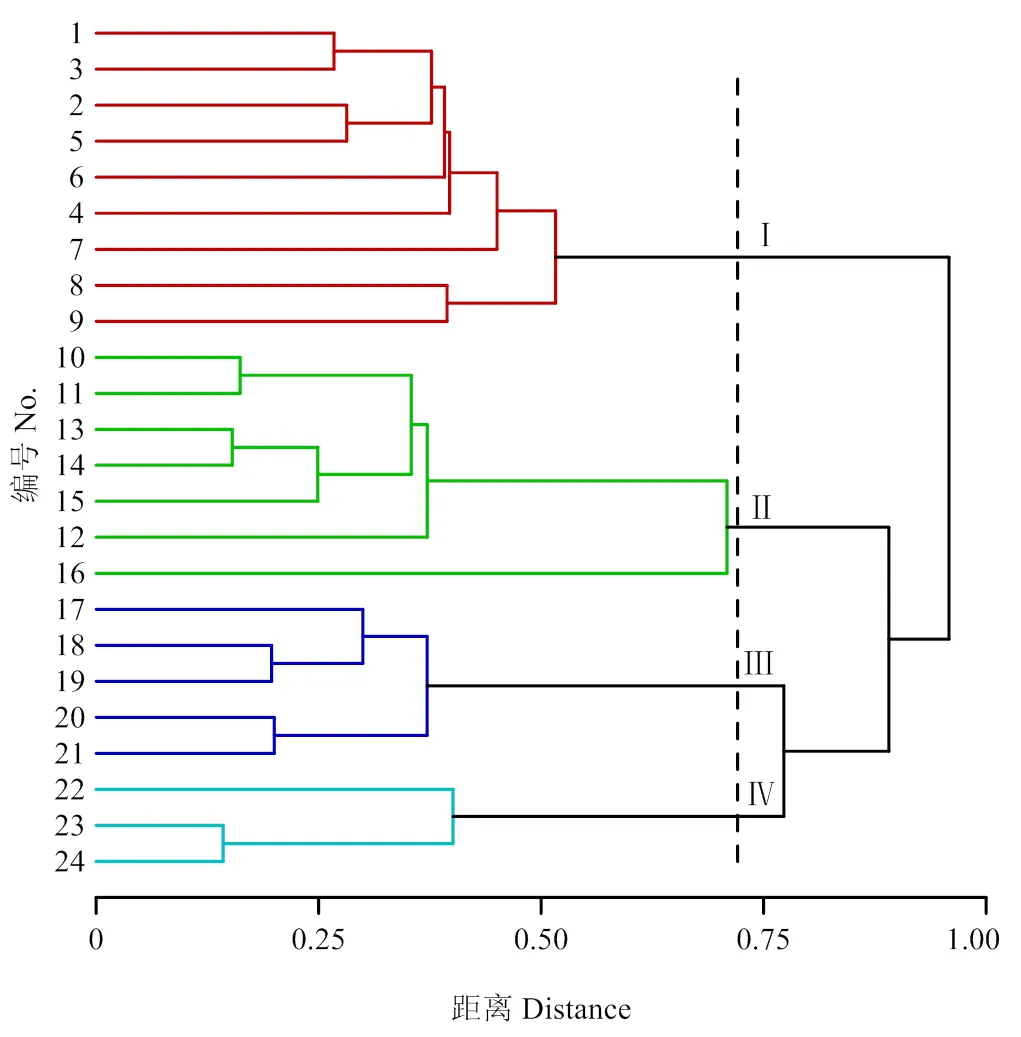
图8 24份天门冬属材料基于EST-SSR鉴定数据的聚类分析
3 讨论
3.1 芦笋转录组SSR位点结构及分布
微卫星序列是基因组的重要组成部分,常因发生滑动链错配和非正常重组交换而导致基序重复次数变化,是物种进化、生物多样性形成及部分遗传疾病发生的主要原因之一[4,22-24]。前人研究表明,SSR分布因物种、染色体和染色体区段不同而存在较大差异[4,25-26]。本研究中,从631 883条Unigene中共检索出位点36 590个,出现频率为4.78%,低于牡丹(5.21%)[6]、巢蕨(11.71%)[8]和象草(9.41%)[9],高于湿地松(4.24%)[27]和拟轮枝镰孢菌(1.09%)[28],可能由物种转录组大小、测序质量和SSR位点检索参数设置等多个方面差异共同造成。从重复类型来看,芦笋转录组三核苷酸(46.92%)占比最高,其次为二核苷酸(27.18%)和单核苷酸(19.02%),而四、五、六核苷酸类型仅占SSR总量的6.88%,这与开心果[7]、四棱豆[10]和甘蔗[29]等结果一致。自身长度过长是导致高级基序发生频率过低的主要原因;转录组序列包含编码区,三核苷酸重复次数变异不会引起移码突变,具有较好的容受性,这是三核苷酸重复占比最高的主要原因。单核苷酸重复中,基序A/T(86.62%)最为常见,与花生[25]结果一致;AG/CT(61.01%)是二核苷酸重复中的优势基序,与开心果[7]和花生[25]等一致,而与牡丹(5.21%)[6]等存在一定出入;三核苷酸重复类型中,AGC/GCT(28.22%)、AGG/CCT(12.29%)和CCG/CGG(11.49%)是最常见基序,与象草[9]情况类似,而甘蔗[29]则以ACA/TGT、CCG/GGC和CGG/GCC为优势基序,说明SSR优势基序因物种而异,具有一定的偏向性。
3.2 芦笋EST-SSR标记的开发、图谱构建及功能注释
基于RNA序列开发的EST-SSR分子标记中往往存在大量的非有效扩增引物,不仅给鉴定造成一定浪费,而且影响基因型鉴定的准确性[6,18-21]。本研究中,基于转录组数据成功设计出EST-SSR引物19 695对,采用TB-tools批量e-PCR与Primer-blast逐一e-PCR相结合的方法剔除非有效扩增引物14 476对,具体包括无扩增产物、扩增产物条带大小与预期严重不符,物理位置未知、存在其他与目的条带大小相似的扩增产物及扩增产物无SSR位点等5类。非有效引物的产生原因主要与转录组组装错误、引物位于剪接位点、扩增产物含有大的内含子及引物在基因组中存在非唯一结合位点等有关[6]。随机选择的50对EST-SSR引物均可扩增出清晰目标条带,有效扩增率达100%,远高于牡丹(51.50%)[6]、紫楠(43.62%)[18]苏丹草(76.7%)[19]苔草(74.61%)[20]、芒草(75.30%)[21]等物种;同时,随机选择的非有效引物的PCR验证结果与ePCR完全一致。说明采用本方法剔除非有效扩增引物高效可行,可为其他物种的EST-SSR分子标记开发提供一定借鉴。
转录组SSR变异与基因功能密切相关[23-24,30]。CDS区SSR变异影响蛋白质翻译,直接影响基因功能;5'-UTR为转录和翻译起始调控区,SSR变异间接影响基因活性;3'-UTR区SSR变异容易引起转录滑移,导致转录延伸或提前终止,间接影响基因功能;Intron区SSR变异可影响mRNA的剪接和外输,从而影响基因活性和功能[24]。本研究开发的2 517个EST-SSR标记中,SSR序列位于CDS、5'-UTR、3'-UTR和Intron的分别有1 003、328、169和1 017个。GO分类发现2 170个SSR标记所属的1 902个基因被成功注释,主要涉及细胞过程和代谢过程等生物过程,细胞器及细胞膜等细胞组分,结合及催化活性等分子功能。KEGG注释发现包含于471个基因的539个SSR标记,存在大量与植物优良农艺性状形成密切相关的基因,如海藻糖磷酸合酶(trehalose phosphate synthases,)[31]、焦磷酸盐﹕果糖-6-磷酸1磷酸转移酶(pyrophosphate: fructose-6- phosphate 1 phosphotransferase,)[32]等高产相关基因;-呋喃果糖苷酶(-fructofuranosi- dase,)[33]、蔗糖磷酸合酶(sucrose-phosphate synthase,)[34]等品质相关基因;L-抗坏血酸过氧化物酶6(L-ascorbate peroxidase 6,)[35]、多胺氧化酶(polyamine oxidases,)[36]等抗逆相关基因,是开发功能标记的优良资源。
3.3 芦笋EST-SSR标记的通用性
SSR标记通常在近缘物种中具有较高的通用性,转移近缘物种的SSR标记已成为遗传信息缺乏物种的一种便捷、高效的分子标记开发方法[6-7,28]。本研究新开发的芦笋EST-SSR标记在同属天门冬、文竹和蓬莱松中的通用性分别达到100.00%、92.00%和88.00%,高于基因组SSR标记[16],类似结果也在牡丹[6]、开心果[7]和拟轮枝镰孢菌[28]被发现。源自基因区域,保守性高于非编码区,是EST-SSR分子标记通用性显著高于基因组SSR标记的重要原因[5]。同样,本研究开发的EST-SSR标记可用于芦笋及近缘物种的系统发育、物种进化及亲缘关系等研究。
4 结论
以芦笋转录组数据为基础,鉴定出36 590个SSR位点,非均匀分布于10条染色体,以三核苷酸(46.92%)、二核苷酸(27.18%)和单核苷酸(19.02%)为主要重复类型,以AG/CT(16.58%)、A/T(16.47%)和AGC/GCT(13.24%)为主要重复基序。开发出高信息量的EST-SSR标记2 517个,总覆盖长度1 125.51 Mb,平均密度为2.24个/Mb,有效扩增率达100%,平均多态性信息含量为0.330。EST-SSR标记在天门冬、文竹和蓬莱松中的通用性分别达到100.00%、92.00%和88.00%,潜在功能涉及产量、品质及抗性形成等多个方面,是开发功能标记的优良资源。
[1] 厉广辉, 于继庆, 李书华, 李保华, 李霞, 李芳. 包艳存, 牟萌. 我国芦笋育种研究进展及展望. 核农学报, 2016, 30(10): 1934-1940.
Li G H, Yu J Q, Li S H, LI B H, Li X, Li F, Bao Y C, Mu M. Research progress and perspective in asparagus breeding in China. Journal of Nuclear Agricultural Sciences, 2016, 30(10): 1934-1940. (in Chinese)
[2] GUO Q B, WANG N F, LIU H H, LI Z J, LU L F, WANG C L. The bioactive compounds and biological functions ofL.–A review. Journal of Functional Foods, 2020, 65: 103727.
[3] ABOUZARI A, SOLOUKI M, GOLEIN B, FAKHERI B A, SABOURI A, DADRAS A R. Screening of molecular markers associated to cold tolerance-related traits in. Scientia Horticulturae, 2020, 263: 109145.
[4] SAEED A F, WANG R Z, WANG S H. Microsatellites in pursuit of microbial genome evolution. Frontiers in Microbiology, 2016, 6: 1462.
[5] TAHERI S, ABDULLAH T L, YUSOP M R, HANAFI M M, SAHEBI M, AZIZI P, SHAMSHIRI R R. Mining and development of novel SSR markers using next generation sequencing (NGS) data in plants. Molecules, 2018, 23(2): 399.
[6] LUO L M, YANG Y Y, ZHAO H W, LENG P S, HU Z H, WU J, ZHANG K Z. Development of EST-SSR markers and association analysis of floral scent in tree peony. Scientia Horticulturae, 2021, 289: 110409.
[7] KARCΙ H, PAIZILA A, TOPÇU H, ILIKÇIOĞLU E, KAFKAS S. Transcriptome sequencing and development of novel genic SSR markers fromL. Frontiers in Genetics, 2020, 11: 1021.
[8] JIA X P, DENG Y M, SUN X B, LIANG L J, SU J L.assembly of the transcriptome ofusing Illumina paired-end sequencing and development of EST-SSR markers. Molecular Breeding, 2016, 36(7): 1-12.
[9] ZHOU S F, WANG C R, FRAZIER T P, YAN H D, CHEN P L, CHEN Z H, HUANG L K, ZHANG X Q, PENG Y, MA X, YAN Y H. The first Illumina-basedtranscriptome analysis and molecular marker development in Napier grass (). Molecular Breeding, 2018, 38(7): 1-14.
[10] WONG Q, TANZI A, HO W, MALLA S, BLYTHE M, KARUNARATNE A, MASSAWE F, MAYES S. WONG Q N, TANZI A S, HO W K, MALLA S, BLYTHE M, KARUNARATNE A, MASSAWE F, MAYES S. Development of gene-based SSR markers in winged bean ((L.) DC.) for diversity assessment. Genes, 2017, 8(3): 100.
[11] XIANG C G, DUAN Y, LI H B, MA W, HUANG S W, SUI X L, ZHANG Z H, WANG C L. A high-density EST-SSR-based genetic map and QTL analysis of dwarf trait inL. International Journal of Molecular Sciences, 2018, 19(10): 3140.
[12] WU M, LIU Y N, ZHANG C, LIU X T, LIU C C, GUO R, NIU K X, ZHU A Q, YANG J Y, CHEN J Q, WANG B. Molecular mapping of the gene(s) conferring resistance to Soybean mosaic virus and Bean common mosaic virus in the soybean cultivar Raiden. Theoretical and Applied Genetics, 2019, 132(11): 3101-3114.
[13] RONG F X, CHEN F F, HUANG L, ZHANG J Y, ZHANG C W, HOU D, CHENG Z H, WENG Y Q, CHEN P, LI Y H. A mutation in class III homeodomain-leucine zipper (HD-ZIP III) transcription factor results in curly leaf (cul) in cucumber (L.). Theoretical and Applied Genetics, 2019, 132(1): 113-123.
[14] GAUTAM T, DHILLON G S, SARIPALLI G, RAKHI, SINGH V P, PRASAD P, KAUR S, CHHUNEJA P, SHARMA P K, BALYAN H S, GUPTA P K. Marker-assisted pyramiding of genes/QTL for grain quality and rust resistance in wheat (L.). Molecular Breeding, 2020, 40(5): 49-62.
[15] ZHU X, ZHAO J F, ABBAS H M K, LIU Y J, CHENG M L, HUANG J, CHENG W J, WANG B B, BAI C Y, WANG G Y, DONG W B. Pyramiding of nine transgenes in maize generates high-level resistance against necrotrophic maize pathogens. Theoretical and Applied Genetics, 2018, 131(10): 2145-2156.
[16] LI S F, ZHANG G J, LI X, WANG L J, YUAN J H, DENG C L, GAO W J. Genome-wide identification and validation of simple sequence repeats (SSRs) from. Molecular and Cellular Probes, 2016, 30(3): 153-160.
[17] 盛文涛, 邓建兰, 饶友生, 柴学文, 刘建坤. 基于NCBI数据库芦笋EST-SSR标记的开发. 分子植物育种, 2019, 17(13): 4307-4313.
SHENG W T, DENG J L, RAO Y S, CHAI X W, LIU J K. Development of EST-SSR markers inbased on NCBI database. Molecular Plant Breeding, 2019, 17(13): 4307-4313. (in Chinese)
[18] 陆云峰, 杨安娜, 张俊红, 楼炉焕, 黄华宏, 童再康. 紫楠转录组EST-SSR标记开发及通用性分析. 农业生物技术学报, 2018, 26(6): 1014-1024.
LU Y F, YANG A N, ZHANG J H, LOU L H, HUANG H H, TONG Z K. Development and transferability evaluation of EST-SSR markers based on transcriptome data of. Journal of Agricultural Biotechnology, 2018, 26(6): 1014-1024. (in Chinese)
[19] ZHU Y Q, WANG X, HUANG L K, LIN C W, ZHANG X Q, XU W Z, PENG J H, LI Z, YAN H D, LUO F X, WANG X, YAO L, PENG D D. Transcriptomic identification of drought-related genes and SSR markers in Sudan grass based on RNA-seq. Frontiers in Plant Science, 2017, 8: 687.
[20] LI M N, LONG R C, FENG Z R, LIU F Q, SUN Y, ZHANG K, KANG J M, WANG Z, CAO S H. Transcriptome analysis of salt-responsive genes and SSR marker exploration inusing RNA-seq. Journal of Integrative Agriculture, 2018, 17(1): 184-196.
[21] NIE G, TANG L, ZHANG Y J, HUANG L K, MA X, CAO X, PAN L, ZHANG X, ZHANG X Q. Development of SSR markers based on transcriptome sequencing and association analysis with drought tolerance in perennial grassfrom China. Frontiers in Plant Science, 2017, 8: 801.
[22] RANATHUNGE C, CHIMAHUSKY M E, WELCH M E. A comparative study of population genetic structure reveals patterns consistent with selection at functional microsatellites in common sunflower. Molecular Genetics and Genomics, 2022, 297(5): 1329-1342.
[23] HAMWIEH A, IMTIAZ M, HOBSON K, AHMED KEMAL S. Genetic diversity of microsatellite alleles located at quantitative resistance loci forblight resistance in a global collection of chickpea germplasm. Phytopathologia Mediterranea, 2013, 52(1): 183-191.
[24] HAUSE R J, PRITCHARD C C, SHENDURE J, SALIPANTE S J. Classification and characterization of microsatellite instability across 18 cancer types. Nature Medicine, 2016, 22(11): 1342-1350.
[25] 徐志军, 赵胜, 徐磊, 胡小文, 安东升, 刘洋. 基于RNA-seq数据的栽培种花生SSR位点鉴定和标记开发. 中国农业科学, 2020, 53(4): 695-706. doi: 10.3864/j.issn.0578-1752.2020.04.003.
XU Z J, ZHAO S, XU L, HU X W, AN D S, LIU Y. Discovery of microsatellite markers from RNA-seq data in cultivated peanut (). Scientia Agricultura Sinica, 2020, 53(4): 695-706. doi: 10.3864/j.issn.0578-1752.2020.04.003. (in Chinese)
[26] BHATTARAI G, SHI A N, KANDEL D R, SOLÍS-GRACIA N, DA SILVA J A, AVILA C A. Genome-wide simple sequence repeats (SSR) markers discovered from whole-genome sequence comparisons of multiple spinach accessions. Scientific Reports, 2021, 11(1): 14381.
[27] 易敏, 张露, 雷蕾, 程子珊, 孙世武, 赖猛. 湿地松转录组SSR分析及EST-SSR标记开发. 南京林业大学学报(自然科学版), 2020, 44(2): 75-83.
YI M, ZHANG L, LEI L, CHENG Z S, SUN S W, LAI M. Analysis of SSR information in transcriptome and development of EST-SSR molecular markers inEngelm. Journal of Nanjing Forestry University (Natural Science Edition), 2020, 44(2): 75-83. (in Chinese)
[28] 李新凤, 王建明, 姜晓东, 郝晓娟, 张祖维, 田宏先. 拟轮枝镰孢菌EST-SSR信息分析与标记开发. 植物保护学报, 2018, 45(4): 819-826.
LI X F, WANG J M, JIANG X D, HAO X J, ZHANG Z W, TIAN H X. EST-SSR information analysis and marker development for genetic diversity analysis of. Journal of Plant Protection, 2018, 45(4): 819-826. (in Chinese)
[29] XIAO N Y, WANG H B, YAO W, ZHANG M Q, MING R, ZHANG J S. Development and evaluation of SSR markers based on large scale full-length transcriptome sequencing in sugarcane. Tropical Plant Biology, 2020, 13(4): 343-352.
[30] YAMASHITA K, KAWASAKI A, MATSUSHITA T, FURUKAWA H, KONDO Y, OKIYAMA N, NAGAOKA S, SHIMADA K, SUGII S, KATAYAMA M, HIROHATA S, OKAMOTO A, CHIBA N, SUEMATSU E, SETOGUCHI K, MIGITA K, SUMIDA T, TOHMA S, HAMAGUCHI Y, HASEGAWA M, SATO S, KAWAGUCHI Y, TAKEHARA K, TSUCHIYA N. Association of functional (GA)n microsatellite polymorphism in thegene with susceptibility to human systemic sclerosis. Rheumatology, 2020, 59: 3553-3562.
[31] PAUL M J, WATSON A, GRIFFITHS C A. Trehalose 6-phosphate signalling and impact on crop yield. Biochemical Society Transactions, 2020, 48(5): 2127-2137.
[32] DUAN E C, WANG Y H, LIU L L, ZHU J P, ZHONG M S, ZHANG H, LI S F, DING B X, ZHANG X, GUO X P, JIANG L, WAN J M. Pyrophosphate: Fructose-6-phosphate 1-phosphotransferase (PFP) regulates carbon metabolism during grain filling in rice. Plant Cell Reports, 2016, 35(6): 1321-1331.
[33] MALEK J A, MATHEW S, MATHEW L S, YOUNUSKUNJU S, MOHAMOUD Y A, SUHRE K. Deletion of beta-fructofuranosidase (invertase) genes is associated with sucrose content in Date Palm fruit. Plant Direct, 2020, 4(5).
[34] GROF C P L, ALBERTSON P L, BURSLE J, PERROUX J M, BONNETT G D, MANNERS J M. Sucrose-phosphate synthase, a biochemical marker of high sucrose accumulation in sugarcane. Crop Science, 2007, 47(4): 1530-1539.
[35] LIU F, HUANG N, WANG L, LING H, SUN T T, AHMAD W, MUHAMMAD K, GUO J X, XU L P, GAO S W, QUE Y X, SU Y C. A novel L-ascorbate peroxidase 6 gene,, plays an important role in the regulation of response to biotic and abiotic stresses in sugarcane. Frontiers in Plant Science, 2018, 8: 2262.
[36] YU Z, JIA D Y, LIU T B. Polyamine oxidases play various roles in plant development and abiotic stress tolerance. Plants, 2019, 8: 184.
Development and Transferability of EST-SSR Markers Based on Transcriptome Data from
Yi ZeHui, Zhao Jing, Mao LiPing
College of Agriculture, Shanxi Agricultural University/Shanxi Provincial Key Laboratory of Innovation and Utilization of Vegetable and Flower Germplasm Resources, Taiyuan 030031
【Objective】The aim of this study was to clarify the distribution law of SSR loci across the transcriptome of, to develop highly informativeEST-SSR markers and to analyze their transferability, so as to provide the tools for phylogenetic analysis, functional gene mining and molecular marker-assisted breeding of asparagus plants.【Method】Based on the RNA-seq data of 15 asparagus roots obtained from the previous stageby our research group, MISA software was used to retrieve SSR loci,and Primer 3.0 software was employed to design primers in batches. Then, the ineffective primers were eliminated by performing batch e-PCR with TB-tools software and one-to-one e-PCR with the Primer-blast programme.The information of EST-SSR markers (such as gene id, physical location, and potential function) was obtained by comparison with the genome of.The DNA of 9varieties, 7s varieties, 5varieties, and 3varieties were used as templates, and 50 pairs of randomly synthesized primers were used as primers to detect the effectiveness, polymorphism and transferability of the primers developed.【Result】A total of 36 590 simple sequence repeats (SSRs) loci distributed in 30 229 unigenes with a frequency of 4.78% and an average distance of 9.17 kb were identified based on data from 15 root transcriptomes of.The SSRs were unevenly distributed in the 10 chromosomes, with the highest number in chromosome 7 (4 642) and the highest density in chromosome 3 (37.86 SSRs/Mb).The SRRs were distributed from di- to hexa-, with tri- (46.92%) and AG/CT (16.58%) as the most abundant repeat type and predominant repeat motif, respectively. A total of 19 695 pairs of EST-SSR primers weresuccessfullydesigned, and 15 147 pairs ineffective primers were eliminated by e-PCR.Among them, 3 085 pairs ineffective primers didn’t produce any amplification products, 10 102 pairs produced severely inconsistent amplification productsin terms of fragment size, 1 289 pairshad unknown physical positions in the genome, 402 pairs gave other amplification products of similar size to the target fragments, and 269 pairs generated amplification products without SSRs.Based on 2517 EST-SSR markers located in the gene region developed in this study, the chromosome density distribution map was constructed, with a total coverage length of 1125.51 Mb and an average distance of 447.16 kb.The potential functions of these markers were involved in many aspects, such as yield, quality, stress resistance, and so on.All 50 pairs of randomly synthesized primers could amplify target bands clearly, of which 36 pairs were polymorphic, and the average polymorphic information content was 0.330. These markers could be used in three other species of: the transferability to,, andwere 100%, 92%, and 88%, respectively.Cluster analysis based on the EST-SSR alleles grouped the 24 accessions into four clusters that corresponded to the species of,,, and.【Conclusion】In this study, 2517 highly informative EST-SSR markers of asparaguswere successfully developed, and the effective amplification rate was 100%. The total coverage length of the physical map was 1125.51 Mb, and the average distance was 447.16 kb, which could be used for phylogenetic analysis of asparagus and related species. Moreover, it provided a reference for the development of EST-SSR markers in other species.
transcriptome; asparagus; EST-SSR; chromosome density distribution map; functional annotation

10.3864/j.issn.0578-1752.2023.22.011
2023-04-10;
2023-08-04
山西省高等学校科技创新项目(2022L104)、山西农业大学农业科技创新研究课题(YCX2020YQ07)、山西农业大学园艺学院“双一流”学科建设科研补助项目
通信作者仪泽会,E-mail:yizehui2008bj@163.com
(责任编辑 赵伶俐)
