棉花EXO70基因家族全基因组的鉴定及种间比较
2023-12-28董琰钰徐碧玉董泽宇汪露瑶陈锦文方磊
董琰钰,徐碧玉,董泽宇,汪露瑶,陈锦文,方磊
棉花EXO70基因家族全基因组的鉴定及种间比较
董琰钰,徐碧玉,董泽宇,汪露瑶,陈锦文,方磊
浙江大学农业与生物技术学院,杭州 310058
【目的】当前四倍体棉花的主栽品种是陆地棉与海岛棉,陆地棉产量高且适应性好,海岛棉纤维品质优异但产量一般。EXO70是胞外分泌体(exocyst complex)中的关键亚基,在植物生长发育、胁迫响应等方面发挥重要作用。通过全基因组水平鉴定和分析陆地棉与海岛棉EXO70基因家族成员,并研究其在纤维发育、环境适应等方面的功能,有助于揭示陆地棉与海岛棉种间性状差异形成的分子基础。【方法】从拟南芥数据库(TAIR)获取EXO70家族蛋白序列作为参考序列,采用HMMER、ExPASy、MEME、TBtools等分析工具对陆地棉TM-1和海岛棉Hai7124基因组中EXO70家族成员进行鉴定与分析,系统比较该家族在基因表达模式、重要农艺性状的相关性、逆境响应等方面的异同点。【结果】通过对陆地棉和海岛棉的基因组水平分析,均鉴定出54个EXO70家族成员,根据系统进化发育可将其分为8个分支。陆地棉和海岛棉同源基因可一一对应,分布在陆地棉与海岛棉的20条染色体上,绝大多数成员为单外显子,但是两者间有12对同源基因在阅读框序列上存在较大差异。陆地棉与海岛棉大部分同源基因表达模式类似,但在同一纤维发育时期表达量存在差异,如分支A中的与同源基因。重要农艺性状关联分析中发现,海岛棉有16个EXO70基因与纤维品质性状显著相关,多于陆地棉;陆地棉有13个基因与产量性状衣分显著相关,多于海岛棉。发现部分性状关联的差异可能由海陆种间的基因结构差异造成,如和。在不同逆境胁迫下,陆地棉中有25个基因受诱导,显著多于海岛棉中10个受诱导基因。【结论】在棉花四倍体形成、分化与驯化过程中,陆地棉与海岛棉EXO70家族基因序列结构相对保守,基因表达模式也保持较高的一致性。海岛棉有更多的EXO70基因与纤维品质性状相关,陆地棉有更多的基因与产量性状相关且对环境逆境更为敏感。
陆地棉;海岛棉;EXO70基因家族;表达模式;性状关联分析;逆境响应
0 引言
【研究意义】棉花是世界上最重要的经济作物之一,陆地棉()与海岛棉()是目前最主要的2个棉花栽培种。陆地棉产量高、适应性强,而海岛棉产量低但纤维品质较好,因此,研究陆地棉适应性强和海岛棉纤维品质好的相关基因对于培育高抗优质的棉花新品种具有重要意义。EXO70(exocyst component of 70 kDa)是控制囊泡运输的重要基因[1],参与细胞壁形成、细胞分泌和抗逆应答等重要细胞学过程。鉴定陆地棉和海岛棉的EXO70家族基因,可为揭示EXO70在棉花生长发育尤其是纤维发育中的作用提供基础数据,进行该类基因与重要农艺性状的关联分析为挖掘关键EXO70基因找到切入点,分析和对比该类基因在陆地棉、海岛棉中的表达模式,将为深入探究EXO70调控机制奠定参考基础。【前人研究进展】囊泡运输是真核细胞中一种关键的跨膜运输和物质交流方式,用于转运大分子等无法直接穿越细胞膜和质膜的物质[2],为细胞的分裂、生长、形态建成和极性建立等基本的生理活动运送原料或传递信号[3]。经典的囊泡运输可划分为4个过程:出芽(小泡形成)、转运(沿细胞骨架运输)、拴系(两膜最初接触)、膜融合(运输完成),由拴系因子介导的拴系过程是膜融合发生的必要条件[4]。Exocyst复合体是重要的拴系因子之一,是由SEC3(Secretory3)、SEC5、SEC6、SEC8、SEC10、SEC15、EXO70和EXO84(exocyst component of 84 kDa)组成的八亚基复合体[5-8]。EXO70是Exocyst复合体的关键亚基[9],决定胞吐时的靶标膜位点。近年来,越来越多植物中EXO70基因家族成员的功能研究被报道,主要集中在调控花粉管伸长和极化、根毛生长、细胞壁物质沉积、细胞板激活和成熟、防御和自噬方面[10-15],广泛参与植物生长发育和免疫反应。在植物生长方面,拟南芥影响细胞板的组装、导管组织分化及运输、凯氏带及次生壁的形成[11, 16-17];和参与调节花粉管的极性生长与成熟[1, 18];突变会影响水稻对矿质营养素的吸收及维管束的分化[19]。在免疫调节方面,拟南芥EXO70B1和EXO70B2蛋白可以相互作用调节鞭毛受体蛋白(FLS2)向质膜的运输,从而参与植物的免疫调节[12]。水稻与AVR-Pii互作,进而参与调节免疫过程[20];沉默烟草中的2个EXO70B基因可以增加植株对晚疫病菌的感病性[21]。在不同逆境胁迫条件下,葡萄中14个EXO70基因的表达有显著差异,这些基因在抗逆调控中起重要作用[22]。目前,对于棉花EXO70家族的报道主要集中在抗逆与免疫调节方面。在棉花应对盐胁迫过程中发挥重要作用,并且还与自噬相关[23]。2021年,Zhu等[24]在亚洲棉、雷蒙德氏棉、陆地棉和海岛棉共鉴定到165个EXO70基因,系统发育分析表明拟南芥与棉花这两种双子叶植物中EXO70基因相似。通过VIGS技术沉默陆地棉验证基因功能,揭示了棉花EXO70基因可能广泛参与代谢,也可能影响植物激素的生物合成和分布[24]。【本研究切入点】虽然关于棉花EXO70基因家族及部分棉花EXO70基因的功能已有相关报道,但还未见针对陆地棉与海岛棉EXO70家族成员的组织表达及关联重要农艺性状的海陆种间比较研究,且EXO70家族成员对棉花纤维发育的影响尚不明确。【拟解决的关键问题】本研究基于陆地棉和海岛棉基因组数据和二者不同组织及逆境胁迫的转录组数据,利用生物信息学方法进行海岛棉和陆地棉的EXO70基因家族全基因组鉴定和种间比较分析等工作,旨在揭示EXO70基因家族在生长发育过程和抗逆境胁迫过程中的作用,结合陆地棉与海岛棉EXO70基因各自优势可进一步应用于棉花选育与改良方面。
1 材料与方法
1.1 棉花EXO70基因家族成员的全基因组鉴定
为了鉴定EXO70基因家族成员,从Cottonomics数据库(http://cotton.zju.edu.cn/)[25]中下载异源四倍体陆地棉TM-1(v2.1)、异源四倍体海岛棉Hai7124(v1.1)基因组序列及基因注释文件(general feature format,GFF),从Pfam[26]数据库(http://pfam.xfam.org/)中下载EXO70蛋白结构域的隐马尔可夫模型(编号PF3081),从拟南芥基因组网站(https://www.arabidopsis. org/)下载已报道的拟南芥EXO70蛋白的氨基酸序列。利用HMMER 3.0软件的hmmsearch搜索程序鉴定棉花全基因组中含有EXO70保守结构域的蛋白序列,同时,利用BLAST+v2.6.0[27]搜索棉花全基因组中与已报道的拟南芥EXO70蛋白的氨基酸序列最相近的蛋白序列(值<1×10-5),将所获得的EXO70蛋白序列提交至NCBI网站的保守结构域数据库(conserved domain database,CDD)进行保守结构域验证。
1.2 棉花EXO70基因家族成员蛋白序列的理化性质分析
根据陆地棉与海岛棉注释文件,通过ExPASy在线网站(http://cn.expasy.org/tools)对GhEXO70和GbEXO70蛋白序列的等电点(isoelectric point,)、相对分子质量(relative molecular mass,Mr)、亲水性平均系数(grand average of hydropathicity,GRAVY)等理化性质进行预测。
1.3 棉花EXO70基因系统发育树的构建及保守基序分析
利用MEGA 11.0软件中的MUSCLE对拟南芥、海岛棉、陆地棉的EXO70家族成员的氨基酸序列进行多重序列比对,采用最大似然法自动选择最佳替代模型,自展值(bootstrap value)设为1 000构建系统进化树,利用iTOL(https://itol.embl.de/)对结果进行可视化分析。通过MEME程序[28]对家族成员进行基序分析(最大发现数设为20)。通过陆地棉与海岛棉基因组注释文件,利用TBtools[29]软件对EXO70基因的基因结构及保守基序进行数据可视化处理。
1.4 棉花EXO70基因家族成员种内共线性分析
通过MCScanX[30]软件对海岛棉和陆地棉中EXO70基因进行共线性分析,鉴定基因复制事件,用Circos[31]对共线性分析结果进行可视化,参数均使用软件默认参数。利用TBTools软件分别计算陆地棉和海岛棉复制基因对的非同义突变率(non-synonymous mutation rate,Ka)、同义突变率(synonymous mutation rate,Ks)和二者的比值(Ka/Ks)。
1.5 棉花EXO70基因家族不同组织中的表达模式分析
从Cottonomics数据库和NCBI数据库中下载海岛棉与陆地棉不同组织、不同纤维发育时期的转录组原始数据,数据编号为PRJNA490626。使用软件fastp[32]过滤以获得高质量读长,使用HISAT2(v2.1)[33]将高质量读长比对到TM-1参考基因组或Hai7124参考基因组上,使用featureCounts 2.0.0[34]对基因表达进行定量,计算EXO70基因的表达分度FPKM值以评估基因的表达水平。取陆地棉和海岛棉叶、根、茎、花瓣及以开花后天数(day post-anthesis,DPA)计算的纤维发育时期(0、1、3、5、10、20和25 DPA)的EXO70家族基因FPKM数据,利用R语言做组织表达模式可视化分析。
1.6 棉花EXO70基因家族主要纤维发育时期基因表达分析
采用陌凡生物公司EASYspin PLUS植物RNA快速提取试剂盒,提取2021年种植在杭州市永安基地的陆地棉TM-1的0 DPA、3 DPA、5 DPA和10 DPA的RNA,分别取3 μl总RNA用诺唯赞公司的HiScript Ⅱ 1st Strand cDNA Synthesis Kit(+gDNA wiper)进行逆转录,将反转后的cDNA稀释为100—200 ng·μl-1。选择作为内参基因,随机挑选4个基因(、、和)进行qRT-PCR。反应体系选用ChamQ SYBR qPCR Master Mix(诺唯赞)体系。每个样品进行3次重复试验,用2-ΔΔCt方法计算该基因的相对表达量。
1.7 棉花EXO70基因家族成员关于重要农艺性状的关联分析
下载陆地棉群体和海岛棉群体遗传变异信息和对应的表型数据[35-37],利用ANNOVAR软件[38]分别对遗传变异位点进行注释,随后基于非同义突变的分型对EXO70基因进行单倍型分析。EXO70基因的不同单倍型的纤维品质和产量相关的重要农艺性状(即纤维长度、纤维强度和衣分)进行差异分析(<0.05),利用R语言可视化。
1.8 棉花EXO70基因家族逆境胁迫下表达模式分析
从Cottonomics数据库和NCBI数据库中下载海岛棉与陆地棉不同逆境胁迫处理的转录组原始数据(编号PRJNA490626)。原始数据的处理和基因表达量的计算方法同1.5,以FPKM=1作为条件进行过滤,即FPKM>1的基因视为表达,若所有组织中FPKM<1的基因视为不表达,不表达的基因过滤不进行差异基因分析。基于基因转移格式(gene transfer format,GTF)的注释文件和DESeq2软件包对陆地棉和海岛棉应对逆境胁迫差异表达基因(differentially expressed genes,DEGs)进行鉴定并筛选,差异基因的筛选标准为取伪发现率(false discovery rate,FDR)矫正后的<0.05。对应逆境胁迫条件下筛选到的差异基因也即受到胁迫诱导的基因记为“1”,其他记为“0”,并用R语言可视化标记EXO70家族基因是否受到胁迫诱导。
2 结果
2.1 棉花EXO70基因家族成员的鉴定和分布
通过序列相似性与保守结构域搜索在陆地棉TM-1和海岛棉Hai7124基因组中均鉴定到了54个EXO70家族基因。以拟南芥、陆地棉和海岛棉EXO70氨基酸序列构建棉花EXO70家族成员系统发育树(图1),所有EXO70可分为8个分支,每个分支的成员数量不等但各分支内海岛棉和陆地棉EXO70基因数量相同,分支A、B、C、D、E、F、G和H中的陆地棉成员数量分别为4、2、9、4、9、4、7和15个。从鉴定家族成员与系统进化发育来看,陆地棉与海岛棉在EXO70家族的每个分支数量保持一致,并且陆地棉与海岛棉同源基因能够一一对应,表明EXO70基因家族在海岛棉与陆地棉分化过程中相对保守。
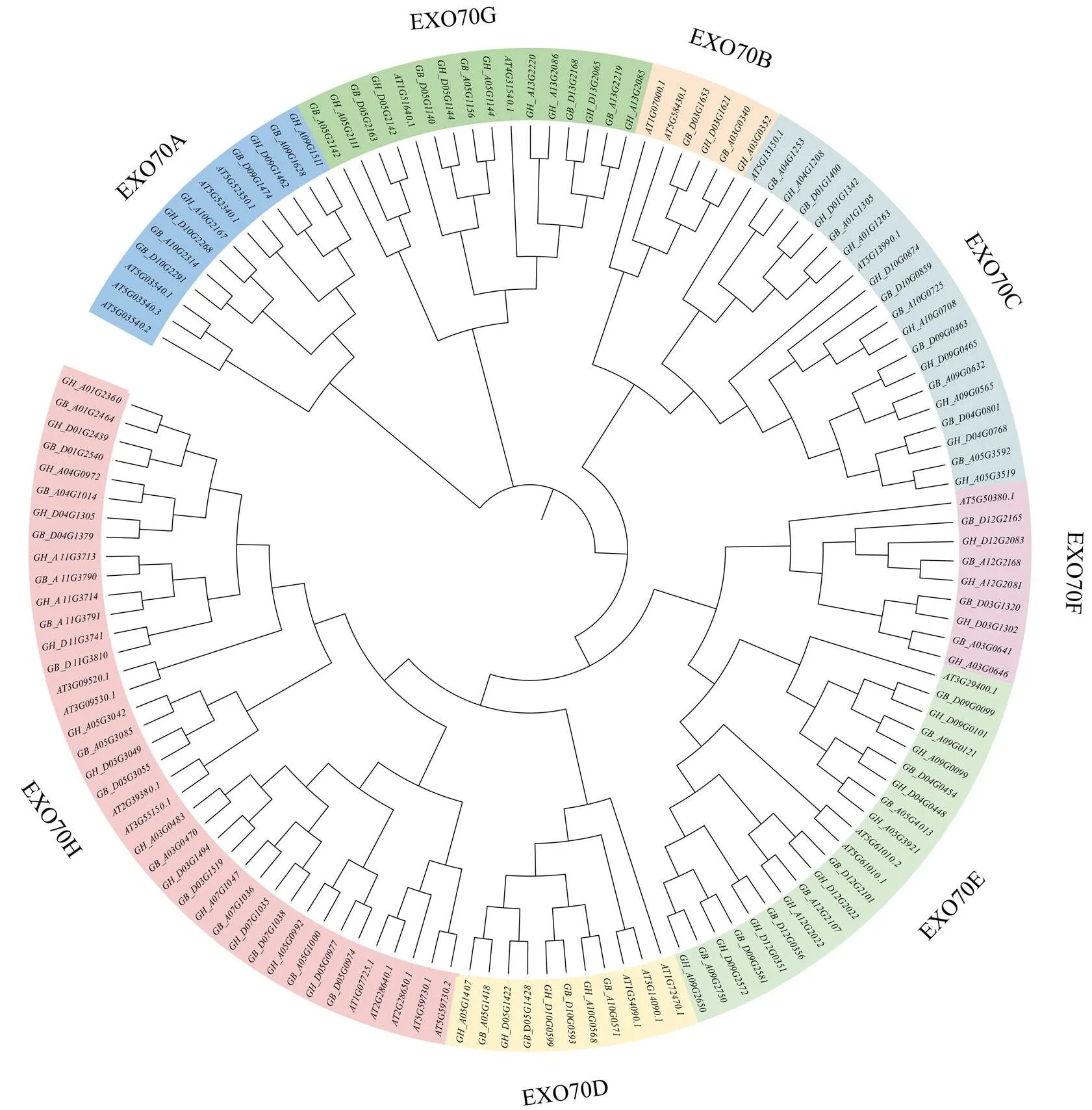
图1 拟南芥(At)、陆地棉(Gh)和海岛棉(Gb)EXO70基因家族的系统发育树
2.2 棉花EXO70家族成员的蛋白质理化性质和结构特征
通过对陆地棉EXO70家族成员编码的蛋白质进行理化性质分析(电子附表1)。EXO70家族成员编码的肽链由310—743个氨基酸组成,相对分子质量介于34 871.98—86 021.92 Da,平均值为71 029.997 Da;理论等电点介于4.84—8.94,平均值为6.22,多数偏酸性;蛋白不稳定指数介于37.42—61.76,平均值为46.32,其中8个EXO70成员为稳定蛋白(蛋白不稳定指数<40),其余的46个基因为不稳定蛋白(蛋白不稳定指数>40)。亚细胞定位预测显示,EXO70家族基因主要定位于叶绿体、细胞核、细胞质以及线粒体中,其中,有48个基因定位在细胞核中,22个定位在叶绿体上,9个定位在细胞质中,4个定位在线粒体上,以细胞核中分布最广泛。海岛棉家族成员理化性质(电子附表2)与陆地棉类似,无明显差异。
2.3 棉花EXO70基因家族保守基序和基因结构分析及比较
通过对陆地棉与海岛棉EXO70家族的保守基序、基因结构进行查询和比较后发现,同一分支中的海陆同源基因含有相似的保守基序和外显子结构。保守基序分析(图2)将预测到的20种基序依次命名为motif 1—motif 20。同一分支的蛋白序列呈现相似的保守基序排列方式,不同EXO70蛋白所包含的保守基序数目及种类存在差异。陆地棉与海岛棉EXO70蛋白中,基序种类最少的蛋白仅含8种,而最多的含有19种motif,说明家族中的一些成员在保守基序上存在差异。陆地棉和海岛棉除了分支A的全部成员与分支G的个别成员,其他成员都为单外显子基因。从基因长度来看,不同的基因具有相似长度的编码区序列,说明EXO70基因在进化过程中内含子长度存在较大的变异,这可能是导致其功能多样化的原因之一。
通过对比陆地棉和海岛棉同源基因全长序列与编码序列,发现有12对陆地棉海岛棉同源基因编码序列有差异(电子附表3)。其中,编码序列差异较大的有6对同源基因集中在分支H中,如的长度小于陆地棉同源基因,且氨基酸数目比后者少83个。此外,分支A中的海岛棉的编码序列比它在陆地棉中的同源基因长1 035 bp,相应氨基酸数目多345个。分析对比外显子个数可知,在54对陆地棉海岛棉同源基因中,45对同源基因为单外显子,6对多外显子,其余3对同源基因外显子个数存在差异。如有2个外显子,而海岛棉同源基因为单外显子基因。
2.4 棉花EXO70基因家族成员的共线性分析
对陆地棉和海岛棉中EXO70基因分别进行种内共线性分析,对该家族成员进行复制基因比对。在陆地棉中共检测出108个同源基因对(图3),其中,A、D亚组内分别有23对、20对直系同源基因,A、D亚组间有65对旁系同源基因,这些同源基因对的Ka/Ks值范围在0.04—1.05(电子附表4)。而同源基因对中只有与的Ka/Ks值大于1,为1.043。有6个同源基因对发生同义突变,有22个基因没有找到复制的同源基因对。在海岛棉中共检测出104个同源基因对(图3),其中,A亚组内有24对同源基因,D亚组内有18对,A、D亚组间有62对旁系同源基因。这些同源基因对的Ka/Ks值范围在0.03—1.05(电子附表5),也只有与基因对的Ka/Ks值>1,为1.047。其中,有2个同源基因对发生同义突变,有21个基因没有找到复制的同源基因对。从图3可知,陆地棉除了A亚组的第2、6和8染色体与D亚组的第2、6和8染色体没有EXO70家族成员外,其他每条染色体上都至少含有一个EXO70基因,其中,A亚组第5染色体上最多,包含7个EXO70基因,海岛棉EXO70基因在染色体上的分布情况与陆地棉一致。
但这个趋势越来越明显。像教育、家装、交通,医疗,都在发生很大的变化,都出现了有一定代表性的网络协同或智能商业的企业。
2.5 棉花EXO70家族基因组织表达模式分析及比较
根据已发表的陆地棉和海岛棉不同组织和不同发育阶段的转录组数据(电子附表5和电子附表6),比较分析陆地棉和海岛棉EXO70基因的表达模式(图4)。总体来看,不同EXO70家族分支呈现不同的表达规律,陆地棉和海岛棉在同一分支中的表达趋势又大致相似。在分支A中,陆地棉与海岛棉存在4对同源基因在纤维发育时期较其他时期呈现优势表达,分支F中的4对同源基因与分支A情况一致。分支B中的2对同源基因都呈现组成型表达,在主要组织和纤维发育时期表达量都较高。陆地棉与海岛棉在分支C与分支G中的成员在组织和胚珠纤维发育中表达量极低。由图4可知,存在多数基因呈组成型表达,陆地棉中共有18个,海岛棉中共有17个。陆地棉在纤维发育时期表达基因有34个,海岛棉中有36个,多于陆地棉。
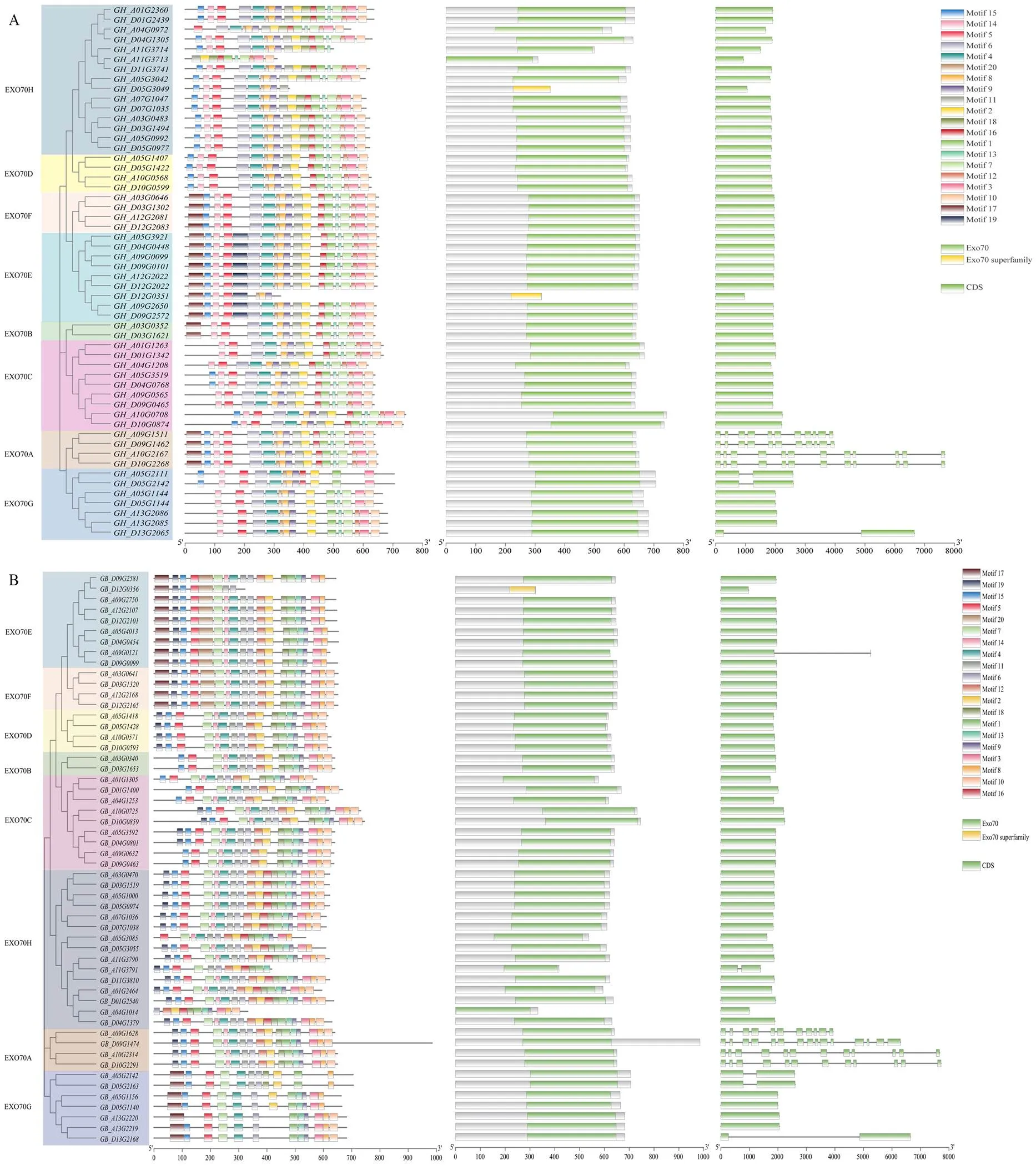
图2 陆地棉(A)和海岛棉(B)EXO70基因家族系统进化树、蛋白基序、超家族和基因结构
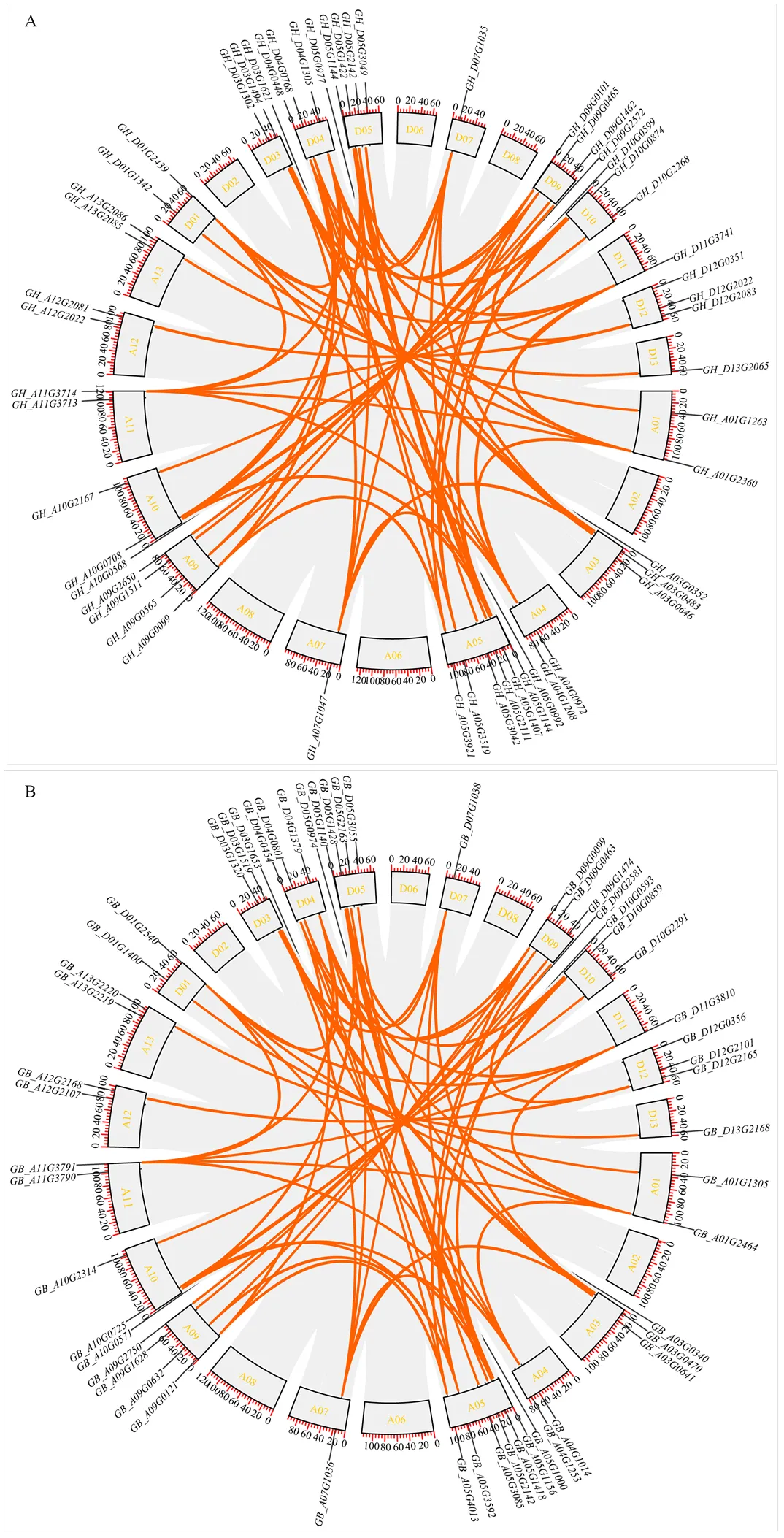
图3 陆地棉(A)与海岛棉(B)EXO70家族种内共线性分析
海岛棉和陆地棉同源基因表达模式大致相似,但在纤维发育时期中,海岛棉整体表达量高于陆地棉。如,分支C中的海岛棉在纤维发育时期20 DPA时表达量高于同源基因(图4)。从纤维整体发育时期来看,陆地棉与海岛棉同源基因的表达峰值时期也有所差异。分支A中陆地棉与同源基因都在纤维发育后期表达较高(图4),前者集中表达在10 DPA,而海岛棉在20 DPA表达达到高峰并高于陆地棉,表达高峰较陆地棉延后。为了进一步验证转录组数据的准确性,随机选取4个基因设计引物(电子附表8)对其在纤维发育时期中的0、3、5和10 DPA进行定量表达分析验证,发现表达趋势均与转录组数据一致(图4和图5)。
2.6 棉花EXO70家族与重要农艺性状的关联分析
通过对EXO70家族基因的不同单倍型的表型特征进行差异分析,发现15个陆地棉和18个海岛棉的EXO70基因的不同单倍型存在显著性差异(<0.05)(电子附表9)。经过分析对比可知,海岛棉EXO70基因成员大部分与纤维长度和纤维强度这两个纤维品质性状相关,陆地棉EXO70基因成员更多是与产量性状衣分相关(电子附表9)。如图6-A可知,与纤维长度性状呈显著相关的海岛棉基因个数有15个,而陆地棉只有7个,与纤维强度性状显著相关的海岛棉基因有16个,而陆地棉只有7个。在产量相关性状——衣分方面,陆地棉与之显著相关基因个数多于海岛棉,分别为13和8个。分析比对同源基因可知,陆地棉与海岛棉只有2对基因与纤维长度性状相关联,有3对同源基因与纤维强度相关联,有2对同源基因与衣分相关联。其中,只有分支B中的陆地棉和海岛棉同源基因可找到与3个性状同时关联的位点。
结合EXO70家族成员基因结构分析与基因关联分析可知,基因结构存在差异的陆地棉与海岛棉同源基因,在关联性状时也有差异。如,在分支H中的陆地棉与同源基因海岛棉编码序列存在较大差异(图6-B),前者相对于后者存在提前终止情况,而海岛棉在A11:115905472位点处可显著关联纤维长度、纤维强度性状但陆地棉同源基因在对应位点无关联性状。分支A中存在编码序列差异的与(电子附表3),海岛棉在D09:42925528与D09:42925541 2个位点与纤维长度、纤维强度和衣分3个性状显著关联(电子附表8),而在陆地棉同源基因对应位点处无性状关联。
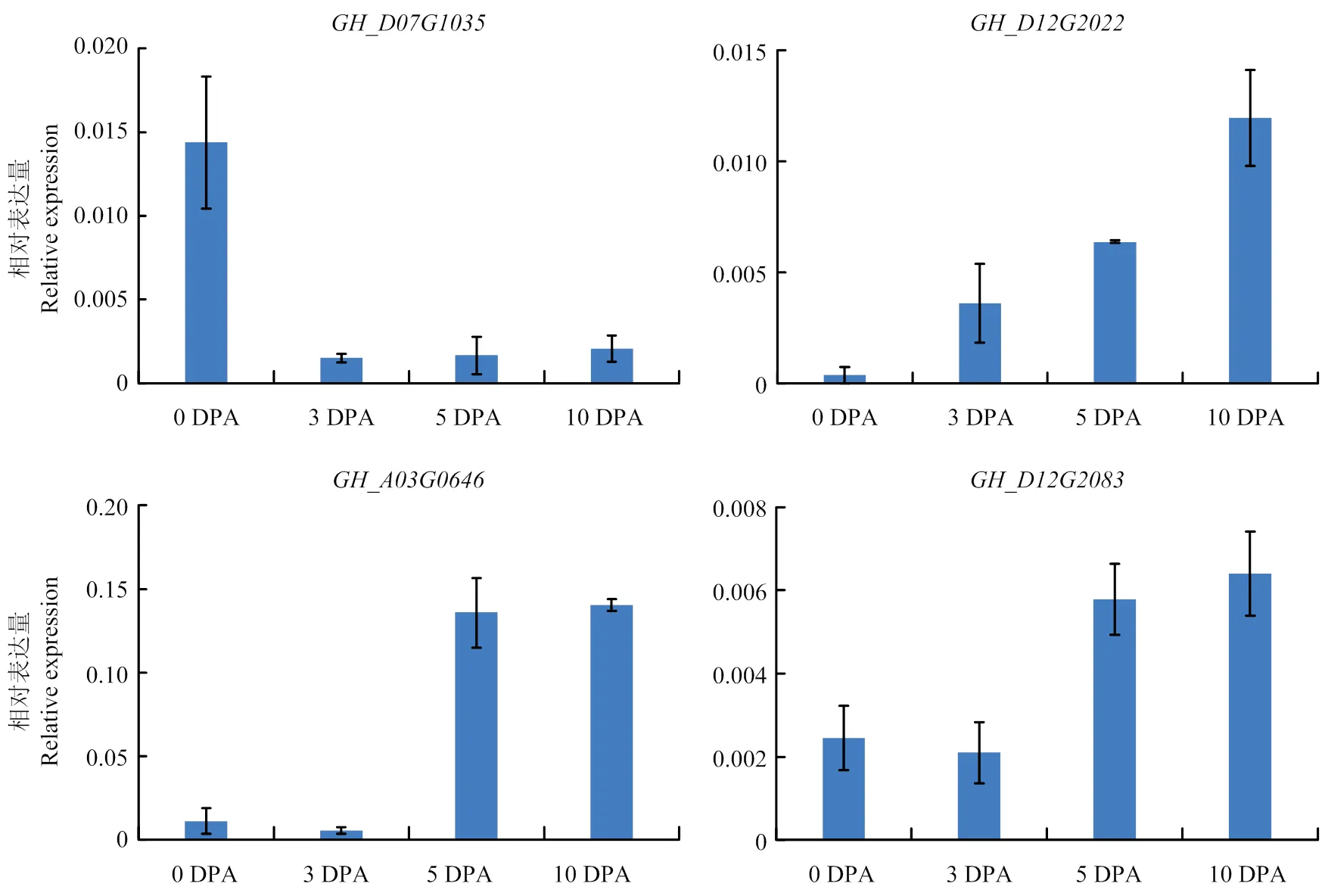
图5 4个基因在纤维发育时期的定量表达分析
结合组织表达模式与关联分析结果可知,纤维发育时期的表达差异与基因关联表型的差异相互印证。如,陆地棉在纤维发育时期基本不表达,而海岛棉同源基因有表达,并且在20 DPA时达到表达高峰(图4),群体数据中显示部分海岛棉的 EXO基因可关联纤维长度、纤维强度等纤维品质性状,而陆地棉并无EXO70基因与此类纤维相关性状关联,且只能关联到衣分这种产量性状(电子附表9)。
2.7 棉花EXO70家族基因胁迫表达模式分析及比较
如图7所示,在相同算法条件下,将胁迫处理中是否受诱导基因区分,陆地棉和海岛棉的同源基因在应对低温(4 ℃)、高温(37 ℃)、盐胁迫(NaCl)、干旱胁迫(PEG)逆境条件下响应表达有所不同。4种非生物胁迫条件下,陆地棉受逆境胁迫诱导的EXO70基因有25个,而海岛棉只存在10个。整体数量上看,陆地棉要多于海岛棉:在低温4 ℃胁迫条件下,陆地棉受诱导基因有11个,海岛棉中只有1个;在高温37 ℃胁迫条件下,受诱导陆地棉基因个数为11,而海岛棉为6个;在盐(NaCl)胁迫条件下,陆地棉有14个基因受诱导,而海岛棉中有2个;在干旱(PEG)胁迫条件下,陆地棉有4个基因受诱导,海岛棉中也有4个。
不同进化分支中,陆地棉与海岛棉在4种非生物胁迫条件下受诱导情况各自都具有一定的偏好性。陆地棉受诱导基因分布在分支C、D、E、F、G和H中,集中分布于E和H这2个分支中。而海岛棉受诱导基因存在于A、C和H中,在分支H中受诱导基因个数最多,为5个,占总海岛棉EXO70整体受诱导基因个数的50%,其余分支不存在海岛棉基因受诱导的情况。
对于同一分支中,陆地棉与海岛棉同源基因在不同胁迫条件下受诱导情况也不同。例如,在分支H中的陆地棉在4 ℃与NaCl条件下都会受到诱导,而海岛棉同源基因却只在干旱条件(PEG)下受诱导。存在于分支C的陆地棉在高温、低温条件下都会受诱导,而同源基因只在高温中存在诱导情况。
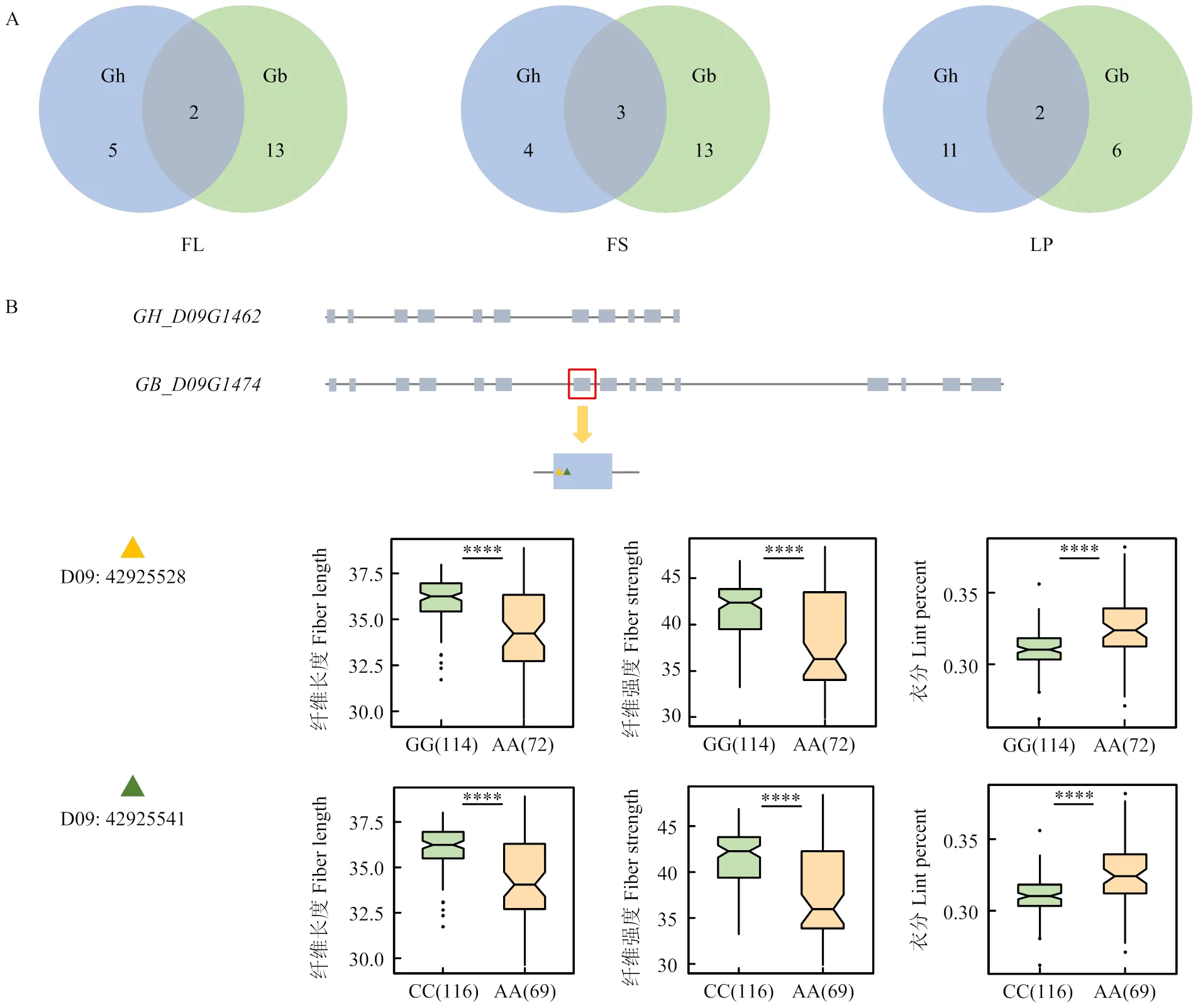
A:陆地棉(Gh)和海岛棉(Gb)与纤维长度(FL)、纤维强度(FS)、衣分(LP)性状呈显著相关基因个数;B:GH_D09G1462与同源基因GB_D09G1473的基因结构,和位于外显子的非同义SNP(上部);GB_D09G1473的2个非同义SNP的不同单倍型在纤维长度、纤维强度和衣分的差异;****表示在P<0.05时单倍型间差异极显著(t检验)
3 讨论
3.1 棉花EXO70基因家族的鉴定和表达分析为后续深入研究提供借鉴
陆地棉和海岛棉是目前世界上最主要的2个棉花栽培种,且均已完成全基因组序列的测序和组装工作[39-41],这为本研究进行海岛棉和陆地棉中EXO70基因家族的鉴定和分析提供极有利的先决条件。本文从陆地棉(TM-1)、海岛棉(Hai7124)2个四倍体栽培棉种中都分别鉴定出54个EXO70基因,说明EXO70这类多拷贝基因在四倍体棉花的分别进化阶段在数量上较为保守。二倍体拟南芥EXO70基因家族成员数是23个,本文鉴定出异源四倍体棉花EXO70家族成员数量是拟南芥的2.35倍,可能是因为棉花进化过程中发生了基因组的加倍和复制事件。近几年,随着测序技术的不断更新和改进,基因组测序和组装的水平得到了大幅提高。这样的技术进展导致不同的基因组版本中基因注释等信息存在一些差异。未来,整合多个版本的基因组信息和分子试验,可以更加有效和准确地鉴定EXO70基因家族成员。酵母与大多数动物的EXO70基因为单拷贝,而大部分植物尤其是陆生植物中EXO70基因为多拷贝,这可能是因为植物具有多种多样的细胞壁及复杂的液泡结构,需要多种胞泌复合体满足其适应环境,进而演化出不同基因特有的调控机制[42-43],相关报道也证明不同的EXO70蛋白会参与细胞壁形成、细胞分泌和细胞免疫等不同的重要细胞学过程[44]。本研究鉴定的EXO70家族成员中既有组织特异型基因,也有少数组成型基因,这说明不同的棉花EXO70基因通过组织特异的表达模式参与不同组织的生长发育进程。已有报道证明,在非生物胁迫下,囊泡运输相关蛋白可通过参与质膜修复来增加植物对温度等非生物胁迫的耐受性[28],而在本研究的另一个表达模式——逆境胁迫诱导表达模式研究中,不同分支的EXO70成员存在胁迫诱导多样化、不同基因特异逆境诱导的情况,说明EXO70基因在棉花抗逆境胁迫过程中具有一定的功能。

热图表示基因在应对非生物胁迫过程中,是否显著差异表达(红色,差异表达;白色,未差异表达)
3.2 EXO70家族基因在棉花进化过程中具有保守性
陆地棉与海岛棉EXO70家族同源基因结构有所差异,并且外显子的个数也有所不同,而内含子-外显子的基因结构通常被认为是进化的印记[45]。陆地棉与海岛棉同源基因存在的基因结构差异,可能会导致二者基因表达的差异从而引起基因功能的不同。从种内共线性分析来看,陆地棉与海岛棉中存在发生同义突变的基因对,说明这些序列分歧度太大,进化距离很远。此外,陆地棉与海岛棉都只有1个基因对的Ka/Ks值大于1,其余都小于1,说明这些基因是受到正向选择,EXO70家族基因在进化过程中经历了较为强烈的净化选择[46],暗示大部分EXO70基因进化保守,功能稳定。
3.3 EXO70家族可能参与棉花纤维发育过程
棉花纤维的各个发育时期对纤维品质与产量都至关重要。棉花纤维是从棉花胚珠表皮单个细胞突起进而发育形成,与拟南芥表皮毛形成过程类似,并且当今学者普遍认为二者表皮毛发育相关基因的调控网络类似[47]。在拟南芥表皮毛无胼胝质的突变体中,功能缺失可导致表皮毛中胼胝质合成酶PMR4分泌受阻,在毛状体成熟阶段细胞壁成分积累产生缺陷,而野生型毛状体细胞壁可发生大量二次增厚[42, 48]。还有研究表明是在拟南芥气管元件(tracheary element)发育过程中表达的基因,并在TE的囊泡运输中起作用从而调节细胞壁增厚[16]。本研究通过对比陆地棉与海岛棉EXO70基因家族中同源基因在编码序列、组织表达及重要农艺性状的基因关联分析,可为后续棉花选育与改良方面结合陆地棉与海岛棉优势基因,提供基础。
4 结论
陆地棉和海岛棉均存在54个EXO70家族基因,在进化过程中基因家族数目相对保守,但基因结构序列存在部分差异,同源基因的分子功能也发生了分化。海岛棉存在更多EXO70基因与纤维长度、强度等品质性状显著关联,可能与其优异的纤维品质特性相关;陆地棉存在更多EXO70基因与衣分、籽指等产量性状显著关联,并且具有更强的非生物胁迫响应能力,可能与其高产、广适性等特性相关。
[1] LI S P, VAN OS G M A, REN S C, YU D L, KETELAAR T, EMONS A M C, LIU C M. Expression and functional analyses ofgenes inimplicate their roles in regulating cell type-specific exocytosis. Plant Physiology, 2010, 154(4): 1819-1830.
[2] WU L G, HAMID E, SHIN W, CHIANG H C. Exocytosis and endocytosis: modes, functions, and coupling mechanisms. Annual Review of Physiology, 2014, 76: 301-331.
[3] ZHANG L, XING J J, LIN J X. At the intersection of exocytosis and endocytosis in plants. The New Phytologist, 2019, 224(4): 1479-1489.
[4] BONIFACINO J S, GLICK B S. The mechanisms of vesicle budding and fusion. Cell, 2004, 116(2): 153-166.
[5] PLESKOT R, CWIKLIK L, JUNGWIRTH P, ŽÁRSKÝ V, POTOCKÝ M. Membrane targeting of the yeast exocyst complex. Biochimica et Biophysica Acta, 2015, 1848(7): 1481-1489.
[6] YU I M, HUGHSON F M. Tethering factors as organizers of intracellular vesicular traffic. Annual Review of Cell and Developmental Biology, 2010, 26: 137-156.
[7] TERBUSH D R, MAURICE T, ROTH D, NOVICK P. The Exocyst is a multiprotein complex required for exocytosis in. The EMBO Journal, 1996, 15(23): 6483-6494.
[8] TERBUSH D R, NOVICK P. Sec6, Sec8, and Sec15 are components of a multisubunit complex which localizes to small bud tips in. The Journal of Cell Biology, 1995, 130(2): 299-312.
[9] HE B, GUO W. The exocyst complex in polarized exocytosis. Current Opinion in Cell Biology, 2009, 21(4): 537-542.
[10] SYNEK L, SCHLAGER N, ELIáS M, QUENTIN M, HAUSER M-T, ZáRSKý V. AtEXO70A1, a member of a family of putative exocyst subunits specifically expanded in land plants, is important for polar growth and plant development. The Plant Journal, 2006, 48(1): 54-72.
[11] FENDRYCH M, SYNEK L, PEČENKOVá T, TOUPALOVá H, COLE R, DRDOVá E, NEBESÁŘOVÁ J, ŠEDINOVÁ M, HÁLA M, FOWLER J E, ŽÁRSKÝ V. Theexocyst complex is involved in cytokinesis and cell plate maturation. The Plant Cell, 2010, 22(9): 3053-3065.
[12] WANG W, LIU N, GAO C Y, CAI H R, ROMEIS T, TANG D Z. Theexocyst subunits EXO70B1 and EXO70B2 regulate FLS2 homeostasis at the plasma membrane. The New Phytologist, 2020, 227(2): 529-544.
[13] KULICH I, PEČENKOVá T, SEKEREŠ J, SMETANA O, FENDRYCH M, FOISSNER I, HöFTBERGER M, ZáRSKý V.exocyst sub complex containing subunit EXO70B1 is involved in autophagy-related transport to the vacuole. Traffic (Copenhagen, Denmark), 2013, 14(11): 1155-1165.
[14] ZáRSKý V, KULICH I, FENDRYCH M, PEČENKOVá T. Exocyst complexes multiple functions in plant cells secretory pathways. Current Opinion in Plant Biology, 2013, 16(6): 726-733.
[15] LIU N, HAKE K, WANG W, ZHAO T, ROMEIS T, TANG D Z. Calcium-dependent protein kinase5 associates with the truncated NLR protein TIR-NBS2 to contribute to-mediated immunity. The Plant Cell, 2017, 29(4): 746-759.
[16] LI S P, CHEN M, YU D L, REN S C, SUN S F, LIU L D, KETELAAR T, EMONS A M C, LIU C M. EXO70A1-mediated vesicle trafficking is critical for tracheary element development in. The Plant Cell, 2013, 25(5): 1774-1786.
[17] KALMBACH L, HÉMATY K, DE BELLIS D, BARBERON M, FUJITA S, URSACHE R, DARASPE J, GELDNER N. Transient cell-specific EXO70A1 activity in the CASP domain and Casparian strip localization. Nature Plants, 2017, 3: 17058.
[18] SYNEK L, VUKA¡INOVIĆ N, KULICH I, HÁLA M, ALDORFOVÁ K, FENDRYCH M, ŽÁRSKÝ V. EXO70C2 is a key regulatory factor for optimal tip growth of pollen. Plant Physiology, 2017, 174(1): 223-240.
[19] TU B, HU L, CHEN W L, LI T, HU B H, ZHENG L, LV Z, YOU S J, WANG Y P, MA B T, CHEN X W, QIN P, LI S G. Disruption ofcauses irregular vascular bundles and perturbs mineral nutrient assimilation in rice. Scientific Reports, 2015, 5: 18609.
[20] FUJISAKI K, ABE Y, ITO A, SAITOH H, YOSHIDA K, KANZAKI H, KANZAKI E, UTSUSHI H, YAMASHITA T, KAMOUN S, TERAUCHI R. Rice Exo70 interacts with a fungal effector, AVR-Pii, and is required for AVR-Pii-triggered immunity. The Plant Journal, 2015, 83(5): 875-887.
[21] DU Y, OVERDIJK E J R, BERG J A, GOVERS F, BOUWMEESTER K. Solanaceous exocyst subunits are involved in immunity to diverse plant pathogens. Journal of Experimental Botany, 2018, 69(3): 655-666.
[22] WANG H, MA Z H, MAO J, CHEN B H. Genome-wide identification and expression analysis of the EXO70 gene family in grape (l.). PeerJ, 2021, 9: e11176.
[23] 丁颜朋, 葛晓阳, 王鹏, 吴洁, 王省芬, 李付广. 棉花盐胁迫应答基因功能分析. 棉花学报, 2018, 30(6): 423-434.
DING Y P, GE X Y, WANG P, WU J, WANG X F, LI F G. Functional analysis of a salt stress response gene,, in upland cotton (L.). Cotton Science, 2018, 30(6): 423-434. (in Chinese)
[24] ZHU Y Q, QIU L, LIU L L, LUO L, HAN X P, ZHAI Y H, WANG W J, REN M Z, XING Y D. Identification and comprehensive structural and functional analyses of thegene family in cotton. Genes, 2021, 12(10): 1594.
[25] DAI F, CHEN J D, ZHANG Z Q, LIU F J, LI J, ZHAO T, HU Y, ZHANG T Z, FANG L. COTTONOMICS: A comprehensive cotton multi-omics database. Database, 2022, 2022: baac080.
[26] EL-GEBALI S, MISTRY J, BATEMAN A, EDDY S R, LUCIANI A, POTTER S C, QURESHI M, RICHARDSON L J, SALAZAR G A, SMART A, SONNHAMMER E L, HIRSH L, PALADIN L, PIOVESAN D, TOSATTO S C, FINN R D. The Pfam protein families database in 2019. Nucleic Acids Research, 2019, 47(D1): D427-D432.
[27] CAMACHO C, COULOURIS G, AVAGYAN V, MA N, PAPADOPOULOSJ, BEALER K, MADDEN T L. BLAST+: architecture and applications. BMC Bioinformatics, 2009, 10: 421.
[28] BAILEY T L, BODEN M, BUSKE F A, FRITH M, GRANT C E, CLEMENTI L, REN J Y, LI W W, NOBLE W S. MEME Suite: tools for motif discovery and searching. Nucleic Acids Research, 2009, 37(supp1_2): W202- W208.
[29] CHEN C J, CHEN H, ZHANG Y, THOMAS H R, FRANK M H, HE Y H, XIA R. TBtools: an integrative toolkit developed for interactive analyses of big biological data. Molecular Plant, 2020, 13(8): 1194-1202.
[30] WANG Y P, TANG H B, DEBARRY J D, TAN X, LI J P, WANG X Y, LEE T H, JIN H Z, MARLER B, GUO H, KISSINGER J C, PATERSON A H.: a toolkit for detection and evolutionary analysis of gene synteny and collinearity. Nucleic Acids Research, 2012, 40(7): e49.
[31] KRZYWINSKI M, SCHEIN J, BIROL I, CONNORS J, GASCOYNE R, HORSMAN D, JONES S J, MARRA M A. Circos: An information aesthetic for comparative genomics. Genome Research, 2009, 19(9): 1639-1645.
[32] CHEN S F, ZHOU Y Q, CHEN Y R, GU J. Fastp: an ultra-fast all-in- one FASTQ preprocessor. Bioinformatics, 2018, 34(17): i884-i890.
[33] KIM D, LANGMEAD B, SALZBERG S L. HISAT: a fast spliced aligner with low memory requirements. Nature Methods, 2015, 12(4): 357-360.
[34] LIAO Y, SMYTH G K, SHI W. FeatureCounts: an efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics, 2014, 30(7): 923-930.
[35] HU Y, CHEN J D, FANG L, ZHANG Z Y, MA W, NIU Y C, JU L Z, DENG J Q, ZHAO T, LIAN J M, BARUCH K, FANG D, LIU X, RUAN Y L, RAHMAN M U, HAN J L, WANG K, WANG Q, WU H T, MEI G F, ZANG Y H, HAN Z G, XU C Y, SHEN W J, YANG D F, SI Z F, DAI F, ZOU L F, HUANG F, BAI Y L, ZHANG Y G, BRODT A, BEN-HAMO H, ZHU X F, ZHOU B L, GUAN X Y, ZHU S J, CHEN X Y, ZHANG T Z.andgenomes provide insights into the origin and evolution of allotetraploid cotton. Nature Genetics, 2019, 51(4): 739-748.
[36] FANG L, GONG H, HU Y, LIU C X, ZHOU B L, HUANG T, WANG Y K, CHEN S Q, FANG D D, DU X M, CHEN H, CHEN J D, WANG S, WANG Q, WAN Q, LIU B L, PAN M Q, CHANG L J, WU H T, MEI G F, XIANG D, LI X H, CAI C P, ZHU X F, CHEN Z J, HAN B, CHEN X Y, GUO W Z, ZHANG T Z, HUANG X H. Genomic insights into divergence and dual domestication of cultivated allotetraploid cottons. Genome Biology, 2017, 18(1): 33.
[37] FANG L, ZHAO T, HU Y, SI Z F, ZHU X F, HAN Z G, LIU G Z, WANG S, JU L Z, GUO M L, MEI H, WANG L Y, QI B W, WANG H, GUAN X Y, ZHANG T Z. Divergent improvement of two cultivated allotetraploid cotton species. Plant Biotechnology Journal, 2021, 19(7): 1325-1336.
[38] WANG K, LI M Y, HAKONARSON H. Annovar: Functional annotation of genetic variants from high-throughput sequencing data. Nucleic Acids Research, 2010, 38(16): e164.
[39] LI F G, FAN G Y, LU C R, XIAO G H, ZOU C S, KOHEL R J, MA Z Y, SHANG H H, MA X F, WU J Y, LIANG X M, HUANG G, PERCY R G, LIU K, YANG W H, CHEN W B, DU X M, SHI C C, YUAN Y L, YE W W, LIU X, ZHANG X Y, LIU W Q, WEI H L, WEI S J, HUANG G D, ZHANG X L, ZHU S J, ZHANG H, SUN F M, WANG X F, LIANG J, WANG J H, HE Q, HUANG L H, WANG J, CUI J J, SONG G L, WANG K B, XU X, YU J Z, ZHU Y X, YU S X. Genome sequence of cultivated Upland cotton (TM-1) provides insights into genome evolution. Nature Biotechnology, 2015, 33(5): 524-530.
[40] ZHU T, LIANG C Z, MENG Z G, SUN G Q, MENG Z, GUO S D, ZHANG R. CottonFGD: an integrated functional genomics database for cotton. BMC Plant Biology, 2017, 17(1): 101.
[41] WANG M J, TU L L, YUAN D J, ZHU D, SHEN C, LI J Y, LIU F Y, PEI L L, WANG P C, ZHAO G N, YE Z X, HUANG H, YAN F L, MA Y Z, ZHANG L, LIU M, YOU J Q, YANG Y C, LIU Z P, HUANG F, LI B Q, QIU P, ZHANG Q H, ZHU L F, JIN S X, YANG X Y, MIN L, LI G L, CHEN L L, ZHENG H K, LINDSEY K, LIN Z X, UDALL J A, ZHANG X L. Reference genome sequences of two cultivated allotetraploid cottons,and. Nature Genetics, 2019, 51(2): 224-229.
[42] KULICH I, VOJTÍKOVÁ Z, SABOL P, ORTMANNOVÁ J, NEDĚLA V, TIHLAŘÍKOVÁ E, ŽÁRSKÝ V. Exocyst subunit EXO70H4 has a specific role in callose synthase secretion and silica accumulation. Plant Physiology, 2018, 176(3): 2040-2051.
[43] YAMAZAKI T, KAWAMURA Y, MINAMI A, UEMURA M. Calcium-dependent freezing tolerance ininvolves membrane resealing via synaptotagmin SYT1. The Plant Cell, 2009, 20(12): 3389-3404.
[44] SEKEREŠ J, PEJCHAR P, ŠANTRŮČEK J, VUKAŠINOVIĆ N, ŽáRSKý V, POTOCKý M. Analysis of exocyst subunit EXO70 family reveals distinct membrane polar domains in tobacco pollen tubes. Plant Physiology, 2017, 173(3): 1659-1675.
[45] YU H H, LI M, SANDHU J, SUN G C, SCHNABLE J C, WALIA H, XIE W B, YU B, MOWER J P, ZHANG C. Pervasive misannotation of microexons that are evolutionarily conserved and crucial for gene function in plants. Nature Communications, 2022, 13: 820.
[46] HURST L D. Theratio: Diagnosing the form of sequence evolution. Trends in Genetics, 2002, 18(9): 486.
[47] GUAN X Y, YU N, SHANGGUAN X X, WANG S, LU S, WANG L J, CHEN X Y.trichome research sheds light on cotton fiber development mechanisms. Chinese Science Bulletin, 2007, 52(13): 1734-1741.
[48] KULICH I, VOJTíKOVá Z, GLANC M, ORTMANNOVá J, RASMANN S, ŽáRSKý V. Cell wall maturation oftrichomes is dependent on exocyst subunit EXO70H4 and involves callose deposition. Plant Physiology, 2015, 168(1): 120-131.
Genome-wide identification and interspecific comparative analysis of the EXO70 gene family in cotton
DONG YanYu, XU BiYu, DONG ZeYu, WANG LuYao, CHEN JinWen, FANG Lei
College of Agriculture & Biotechnology, Zhejiang University, Hangzhou 310058
【Objective】The two main types of allotetraploid cotton that are currently cultivated are upland cotton, known for its high yield and good adaptability, and island cotton, which boasts excellent fiber quality but lower yield. EXO70, a vital subunit of the exocyst complex, plays a significant role in plant growth, development, and stress response. By identifying and analyzing members of the EXO70 gene family in upland and island cotton at the whole-genome level, and studying their functions in fiber development and environmental adaptation, we can shed light on the molecular basis for the differences in traits between these two varieties. 【Method】The reference sequences of EXO70 protein inwere obtained from the TAIR database. HMMER, ExPASy, MEME, TBtools, and other analysis tools were used to identify and analyze the members of EXO70 gene family in the genomes of upland cotton TM-1 and island cotton Hai7124. The similarities and differences in gene expression patterns, correlations with crucial agronomic traits, and stress responses of this family were systematically compared. 【Result】Through genome-level analysis of upland cotton and sea-island cotton, 54 EXO70 family members were identified in both upland and sea-island cotton, which could be divided into eight subgroups based on phylogenetic analysis. Orthologous genes between upland cotton and sea-island cotton can be paired one-to-one and are distributed across the 20 chromosomes of both species. The majority of the members have single exons, while 12 pairs of homologous genes displayed significant differences in the reading frame sequences. Most orthologous genes inanddisplay similar expression patterns, but differences in expression levels are observed during the same fiber development stage, such asand its orthologous geneand. Under different stresses, upland cotton showed a significantly higher number of induced genes compared to sea-island cotton. 【Conclusion】The sequence structure and gene expression patterns of the EXO70 family were found to be relatively conserved in both upland and island cotton during the formation, differentiation, and domestication of tetraploid cotton. However, in terms of EXO70 family members, island cotton had more genes related to fiber quality traits, while upland cotton had more genes related to yield traits and exhibited greater sensitivity to environmental stress.
;; EXO70 gene family; expression pattern; trait association analysis; abiotic stress response

10.3864/j.issn.0578-1752.2023.23.005
2023-02-14;
2023-05-04
国家自然科学基金面上基金(32172008)、中央高校基本科研业务费专项资金(226-2022-00153)
董琰钰,E-mail:dongyy@zju.edu.cn。通信作者方磊,E-mail:fangl@zju.edu.cn
(责任编辑 李莉)
