棉花功能基因图位克隆的研究进展
2023-12-28臧新山王康文张先亮王雪平王军梁雨裴小雨任翔吕宇龙高宇王星星彭云玲马雄风
臧新山,王康文,张先亮,王雪平,王军,梁雨,裴小雨,任翔,吕宇龙,高宇,王星星,彭云玲,马雄风,,4
棉花功能基因图位克隆的研究进展
臧新山1,2,4,王康文1,3,张先亮1,2,王雪平1,王军1,梁雨1,裴小雨1,任翔1,2,吕宇龙1,2,高宇1,王星星1,彭云玲3,马雄风1
1中国农业科学院棉花研究所/棉花生物学国家重点实验室/农业农村部棉花生物学与遗传育种重点实验室,河南安阳 455000;2中国农业科学院西部农业研究中心,新疆昌吉 831100;3甘肃农业大学农学院/甘肃省干旱生境作物学重点实验室/甘肃省作物遗传改良与种质创新重点实验室,兰州 730070;4郑州大学农学院,郑州 450001
图位克隆是鉴定特定表型变异遗传基础的经典有效策略。棉花功能基因图位克隆,对育种工作者创新利用种质资源、培育和定向设计新品种、提高育种效率有重要指导作用。近年来,随着雷蒙德氏棉、亚洲棉、陆地棉和海岛棉等基因组测序的完成和不断完善,基因的物理位置信息已知,省去了筛选基因组文库和构建候选区段物理图谱的过程,棉花功能基因图位克隆研究进入快速发展期。2016年,利用正向遗传学方法首次图位克隆了陆地棉显性无腺体Gl(),目前已有20个质量性状基因和5个数量性状基因通过图位克隆策略鉴定。本文从基因符号、名称、染色体定位、候选基因等方面系统综述棉花纤维、腺体、蜜腺、叶型、株型、植株颜色、育性等性状相关图位克隆基因;并从图位克隆作图群体和集团分离分析法测序(bulked segregate analysis-sequencing,BSA-seq)应用等方面系统综述图位克隆策略。随着基因组测序技术的升级、测序成本的降低、BSA-seq等新方法的应用,图位克隆发展更加快速准确。利用转基因和基因组编辑技术对基因功能开展全面系统的鉴定评价,将为棉花分子设计育种提供理论基础和基因资源,加快棉花遗传改良进程。
棉花;图位克隆;分子标记;作图群体;BSA-seq
0 引言
棉花是重要的天然纤维作物,也是重要的油料作物,在国民经济中占有重要地位。图位克隆是鉴定特定表型变异遗传基础的经典有效策略,从表型变异入手,通过组配遗传群体、精细定位引起表型变异的染色体片段,并克隆候选基因,属于正向遗传学研究。棉花功能基因定位和图位克隆研究为棉花分子设计育种提供了基因资源。
棉花基因组学的快速发展为棉花功能基因图位克隆奠定了坚实基础。2012年,棉花雷蒙德氏棉D基因组全基因组测序完成[1-2]。2014年,亚洲棉全基因组测序完成[3]。2015年,异源四倍体陆地棉品种TM-1[4-5]、海岛棉品种3-79和Xinhai21全基因组测序完成[6-7]。近年来,随着三代测序技术的不断发展完善,亚洲棉、陆地棉和海岛棉基因组不断完善,并且多个陆地棉栽培品种的基因组也相继组装完成[8-11]。随着棉花参考基因组测序数据的陆续公布,棉花图位克隆研究取得新突破,并发展迅速。
棉花功能基因图位克隆研究相对于主粮作物研究滞后,直至2016年,陆地棉腺体Gl首次被图位克隆鉴定[12]。因为陆地棉和海岛棉基因组是复杂的异源四倍体(AADD,包括2个亚基因组(At和Dt)),所以高质量参考基因组发布较晚。本文从棉花质量性状和数量性状基因的图位克隆及策略方面综述纤维、腺体、蜜腺、叶型、株型、植株颜色、育性等性状的研究进展。
1 棉花基因的图位克隆与功能分析
1.1 质量性状基因的图位克隆与功能分析
质量性状一般受1—2个主效基因控制,遗传基础简单,杂交F2群体分离,呈明显的不连续变异。迄今,棉花中已有20个质量性状基因被图位克隆(表1)。
1.1.1 纤维 棉花纤维是纺织工业最重要的天然原料,分为长绒(Lint)和短绒(Fuzz)。轧花时,从种子上轧下来的是长绒,留在种子上的是短绒[13]。目前,已有光籽(N)、李氏无纤维(Li和Li)和纤维颜色(Lc)等纤维性状相关基因被克隆。
光籽:2016年,Wan等[14]克隆了陆地棉显性光籽N——,为MYB类转录因子家族成员。在突变体N中极低表达,与天然反义转录本(natural antisense transcripts,NAT)产生相关,的mRNA可能与NAT形成dsRNA,进而影响纤维发育,这是棉花中第一个通过图位克隆方法鉴定到的纤维发育调控基因。
李氏无纤维:2017年,Thyssen等[15]克隆了显性李氏无纤维基因Li——,其第65个氨基酸由甘氨酸(Glycine,Gly)突变为缬氨酸(Valine,Val),影响了棉花细胞骨架和极性生长,导致无纤维表型。2018年,Wu等[16]克隆了调控纤维起始的隐性基因Li——,为MIXTA类家族成员,在726 bp处由碱基胞嘧啶(Cytosine,C)突变为腺嘌呤(Adenine,A),导致无纤维表型。
纤维颜色:2018年,Yan等[17]克隆到显性棕色纤维基因Lc——,为TRANSPARENT TESTA 2(TT2)家族成员,调控磷脂酸的生物合成与纤维着色。
1.1.2 腺体 腺体是棉属植物特有的形态特征之一,在棉花的茎、叶、花、铃等组织上均有分布。棉花腺体主要包括茎秆无腺体(gl、Gl和Gl)、有无腺体植株(Gl、Gl、Gl、Gl和gl)、无腺体植株修饰基因(gl和gl)和渗漏腺体基因()[13]。腺体中的棉酚有利于棉花抵抗病虫害及不良环境的胁迫,但对人畜具有毒害作用。目前,腺体相关基因Gl和gl已被图位克隆。
2016年,Ma等[12]克隆了控制腺体有无的显性基因Gl——,为bHLH转录因子家族成员,是Gl和Gl的复等位基因;Gl存在时,能够消除棉花植株的所有腺体,导致棉酚含量降低,这是第一个利用正向遗传学手段图位克隆的棉花基因。同年,Cheng等[18]也精细定位到该基因。2021年,Zang等[19]克隆了隐性茎秆无腺体基因gl——,为GRAS家族成员,主要调控棉花茎和叶柄上腺体的形成;在突变体T582中,编码区2 009 bp处碱基胞嘧啶(C)突变为腺嘌呤(A),翻译提前终止,导致茎上无腺体表型。
1.1.3 蜜腺 蜜腺是植物体上一种分泌糖液的外部分泌结构。棉花蜜腺较多,蜜腺期长,是棉花害虫的重要食物来源。陆地棉有叶蜜腺、花蜜腺和花外蜜腺。叶蜜腺通常位于棉花叶片背面中脉上,离叶基约1 cm处,呈窝状凹陷,窝内有许多乳头状突起;以花萼为分界,花萼内侧基部的蜜腺称为花蜜腺;而花萼外侧的蜜腺称为花外蜜腺。花外蜜腺()、无蜜腺(ne和ne)基因已被图位克隆。
2020年,HU等[20]克隆了亚洲棉隐性花外蜜腺基因,编码含有PB1结构域的蛋白,在无蜜腺突变体中,由于缺失5个氨基酸而引起蛋白质三维结构的改变,导致蛋白功能发生改变。2021年,PEI等[21]克隆了隐性无蜜腺基因GhNe和GhNe,属于AD亚基因组上的同源基因,编码AP2/ERF转录因子家族成员,调控花外蜜腺和花蜜腺发育。
1.1.4 叶型 植物叶片是进行光合作用的主要场所,根据叶裂的深浅,棉属的叶型可以分为阔叶和鸡脚叶,鸡脚叶又可分为超鸡脚叶和亚鸡脚叶。陆地棉叶型主要为阔叶,少数品种为鸡脚叶或超鸡脚叶;海岛棉叶片一般为三至五裂,叶型多数为海岛型鸡脚叶,也存在少数阔叶。鸡脚叶基因位点上有多个不完全显性等位基因,主要包括鸡脚叶L、超鸡脚叶L、亚鸡脚叶L、海岛棉叶L等。
2016年,ZHU等[22]克隆了调控叶型的复等位基因L——,为同源异型-亮氨酸拉链(HD-Zip)转录因子家族成员;与鸡脚叶材料相比,阔叶材料有8 bp的缺失,翻译提前终止,出现阔叶表型。同年,CHANG等[23]也克隆了L。2017年,ANDRES等[24]克隆了调控叶型的基因-D——,发现鸡脚叶叶型棉花的启动子存在133 bp的串联重复,表达量上升;阔叶叶型棉花的第3个外显子存在8 bp的缺失,翻译提前终止。
1.1.5 株型 棉花株型结构比较复杂,具有无限生长、营养生长和生殖生长重叠等特点。棉花分枝分为营养枝和果枝,营养枝在主茎的下部着生,果枝在主茎的上部着生。
2018年,SI等[25]克隆了隐性丛生铃基因Cl——,与番茄()同源,其811 bp处的胞嘧啶(C)突变为胸腺嘧啶(Thymine,T),导致第113位氨基酸从脯氨酸(Proline,Pro)突变为丝氨酸(Serine,Ser),出现零式果枝表型。同年,LIU等[26]克隆了隐性短果枝基因——,与棉花生长习性和丛生铃形成相关。2020年,CHEN等[27]克隆了隐性柱头外露ob——,在陆地棉中,它存在1 783 bp的缺失,导致第3个外显子丢失;在海岛棉(3-79)中,它的第3个外显子存在8 bp缺失,导致出现柱头外露表型。
1.1.6 植株颜色 棉花植株的颜色主要包括叶绿素缺失或改变导致的持久性的颜色变化和芽黄。棉花植株一般为绿色,也包括红株R、亚红株R和矮红株R。长期以来,红色植株一直作为形态学标记应用于棉花遗传研究,而且红色植株相比绿色植株的抗虫性更好。芽黄是优良的指示性状,在棉花杂种优势利用中被棉花育种工作者重视;多数芽黄突变体在苗期表现明显,子叶或真叶呈不同程度的黄色,后期逐渐转为绿色。
2017年,ZHU等[28]克隆了隐性芽黄基因v——,编码镁离子螯合酶,在AAA+保守区域存在1个位点突变,即第360位氨基酸由精氨酸(Arginine,Arg)突变为赖氨酸(Lysine,Lys),进而导致芽黄表型。2018年,MAO等[29]也克隆了该基因。2019年,LI等[30]克隆了显性红株基因R——,为R2R3-MYB转录因子家族成员,通过调控花青素合成途径相关基因的表达,促进花青素合成。
1.1.7 育性 杂种优势利用是提高作物产量、改善品质、增强抗性的重要途径。棉花具有明显的杂种优势,雄性不育是杂种优势利用的有效途径。棉花雄性不育系主要有核雄性不育系、质核互作型雄性不育系和光温敏感型不育系3种类型。
2019年,DENG等[31]克隆了Le——,是造成海岛棉和陆地棉杂交种致死的关键基因。2022年,MA等[32]克隆了双隐性核不育系基因——/,基因编码区突变导致酶催化活性丧失,当2个基因都突变时,出现不育表型。同年,WU等[33]克隆了隐性核不育恢复基因,在耐高温棉花花药中受高温诱导提前表达,而在高温敏感材料花药中不受高温诱导,影响花粉外壁和花粉刺突的正常形成。
1.1.8 其他性状 类病变突变体是一种在没有受到生物和非生物胁迫下,植物产生坏死病斑的突变材料。2017年,CHAI等[34]克隆了单隐性类病变突变体基因,编码5-氨基乙酰丙酸脱水酶(ALAD蛋白),可以增强对黄萎病的抗性。
花冠颜色是棉花重要的显性形态标记,将红色花冠作为性状标记为转基因抗虫杂交棉带上天然防伪标签。2022年,CHAI等[35]克隆了显性红色花瓣基因R——,编码谷胱甘肽-S-转移酶(glutathione S-transferase,GST),bZIP转录因子基因调控,且特异性结合陆地棉红色花冠近等基因系启动子,诱导花青素的积累,形成红色花瓣。
花瓣的斑点、条纹可以提高传粉昆虫的到访率,提升棉花三系杂交种产量。2022年,ABID等[36]克隆了海岛棉控制花瓣基斑形成的关键基因,在陆地棉中,该基因的突变导致花瓣基斑消失;它可以直接与黄酮类物质合成基因(、、和)启动子结合,调控花瓣基斑的形成。

表1 棉花质量性状基因的图位克隆
1.2 数量性状基因的图位克隆与功能分析
数量性状属于主基因和多基因的混合遗传模式,受环境影响较大,其表型和基因型之间无明确对应关系,需用度量数值来表示其表型。棉花大多数农艺性状(如品质、产量、抗逆性和生育期等)均属于数量性状(表2)。
棉花纤维品质直接关系纺织产品的质量,决定了其在市场上的竞争力。2021年,ZANG等[37]克隆了控制纤维强度的主效QTL位点——,通过泛素26s蛋白酶体途径降低纤维中GhSPL1蛋白含量来调节纤维螺旋生长,揭示了螺旋纤维和纤维强度之间的内在关系。2021年,ZHANG等[38]克隆了纤维长度主效QTL位点——,其启动子区域缺失214 bp,导致不能与NF-YA转录因子相互作用,进而不能调控棉纤维的伸长。
铃重是棉花重要的产量构成因素,受环境因素、品种遗传等因素影响而存在差异。2020年,AHMED等[39]克隆了调控棉花铃重的,编码油菜素内酯反应环H2蛋白,调控棉铃发育。
黄萎病(,VW)是一种土传真菌病害,严重影响棉花生产。2018年,ZHAO等[40]克隆了海岛棉黄萎病抗性基因和。

表2 棉花数量性状基因的图位克隆
2 棉花基因图位克隆的策略
图位克隆首先需要构建作图群体,通过杂交、回交和自交等方式产生群体后代(如F2、BC1、RIL、NIL等),并进行遗传图谱构建。基于遗传图谱,利用多态性分子标记进行目的基因初步定位,再开发多态性分子标记对其进行精细定位,进而图位克隆到候选基因。本文对图位克隆作图群体(表3)和BSA-seq应用进行了综述。
2.1 棉花图位克隆作图群体
利用F2群体图位克隆到的基因主要包括Gl、1、、、v、ob、、、L、、、、和,定位群体大小分别为2 197[12]/4 530[18]、2 012[14]、763[34]、2 481[20]、2 576[28]/4 232[29]、6 137[27]、1 678[35]、1 962[32]、2 843[23]、5 780[36]、1 864[37]、1 081[38]、176[40]和176个[40],平均群体大小为2 529个。如DONG等[41]利用BC1群体将基因Gl初步定位在分子标记NAU3778和NAU2251b之间1 Mb的物理区间内;在此基础上,MA等[12]利用2 197个F2群体,将Gl精细定位在分子标记w7954和w5383之间43 kb的物理区间内,区间内存在7个基因,进而克隆了显性无腺体基因Gl——。如ZANG等[37]利用F2群体将纤维强度主效QTL位点初步定位在D03染色体23.5 cM的遗传区间内,进一步开发SSR、SNP和InDel分子标记,将精细定位在K5219和K5221之间1.14 cM的遗传区间内,对应0.93 Mb的物理区间,区间内存在23个基因,进而克隆了——。
利用F2群体结合其他群体图位克隆到的基因主要包括Li、/、Cl、Lc和,群体大小分别为9 330[15]、6 477[21]、2 079[25]、1 968[17]和1 904个[33],平均群体大小为4 351个。如SI等[25]前期利用F2群体将Cl初步定位到D07染色体,进一步利用852个F2/BC1群体将Cl精细定位在分子标记K4918和K5833之间0.39 Mb的物理区间内,区间内存在13个基因,进而克隆了隐性丛生铃基因Cl——。YAN等[17]前期利用270个RIL群体对Lc进行初步定位,利用1 698个F2群体进一步将Lc精细定位到分子标记TT2-1A和TT2-3A之间67 kb的物理区间内,区间内存在3个基因,进而克隆了显性棕色纤维基因Lc——。WU等[33]前期利用BSA-seq方法,将初步定位在630 kb的物理区间内,进一步利用NIL群体将精细定位到103 kb的物理区间内,区间内存在9个基因,进而克隆了单隐性败育恢复基因。

表3 棉花图位克隆的作图群体
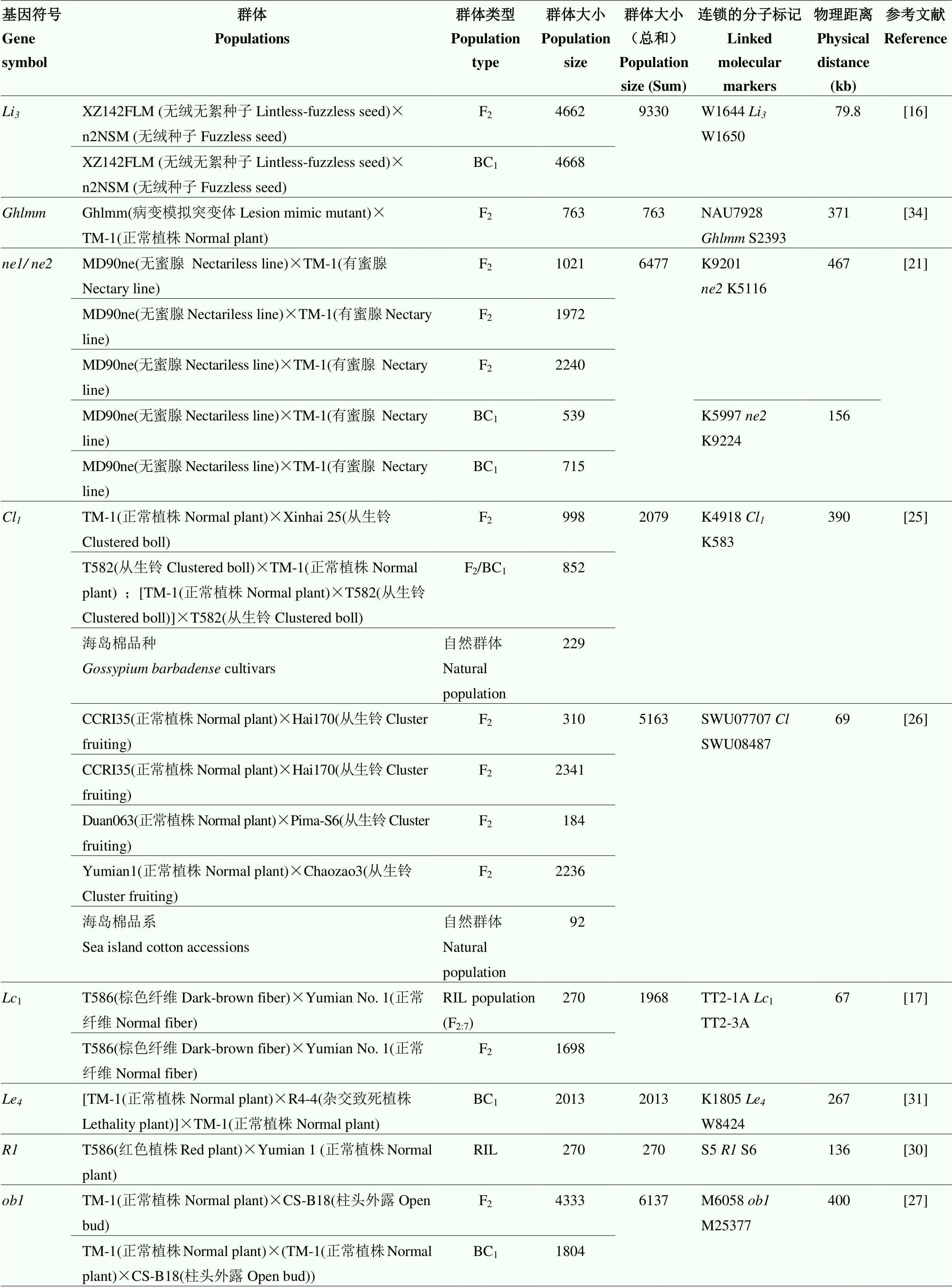
续表3 Continued table 3

续表3 Continued table 3
SONG等[42]前期利用F2群体将Le初步定位在分子标记BNL3279和BNL1154之间9.4 cM的遗传区间内。DENG等[31]进一步利用BC1群体(群体大小为2 013个)将Le精细定位到分子标记K1805和W8424之间267 kb的物理区间内,区间内存在3个基因,进而克隆了基因Le——。
LIU等[43]将R初步定位在D07染色体上,在此基础上,LI等[30]进一步开发了分子标记,利用RIL群体(群体大小为270个)将基因R精细定位到分子标记S5和S6之间136 kb的物理区间内,区间内存在3个基因,进而克隆了红色植株基因R——。AHMED等[39]首先利用BSA-seq在第12染色体上检测到QTL位点,然后利用F3群体将其定位在1.8 cM的遗传区间内,进一步利用F4群体将其精细定位在0.89 cM的遗传区间内,最终将其定位到分子标记AD-A12_07和AD-FM_44之间180 kb的物理区间内,区间内存在11个基因,进而克隆了调控棉花铃重的主效QTL位点——。
2.2 BSA-seq的应用
集团分离分析法(bulked segregant analysis,BSA)是一种快速定位控制目标性状基因的方法,由MICHELMORE等[44]于1991年首次提出,并成功应用于莴苣霜霉病抗性基因的遗传定位和连锁分子标记开发。
BSA-seq是由BSA和高通量测序技术结合而成简单、高效、准确的遗传定位策略。从作图群体中挑选极端个体,构成2个DNA池;对亲本和2个DNA池进行高通量测序,鉴定亲本和2个DNA池中的共有SNP,计算2个DNA池中相同变异位点的基因型频率及其差值;以差值体现标记在2个DNA池间的多态性,进而对候选基因进行定位。高通量测序技术的快速迭代与发展,极大地推进了BSA-seq技术的应用,先后衍生出了Mutmap、Mutmap+、QTL-seq、mQTL-seq、RAD-seq和SLAF-seq等技术。此外,BSR-seq技术也应用于一些未获得参考基因组的物种进行基因定位。BSA-seq技术具有快速、成本低、适用于多种作图群体等特点,极大促进棉花功能基因图位克隆的研究进程[28]。
BSA-seq已被大量用于水稻、大豆、黄瓜、番茄等植物基因定位中,成功定位了水稻耐盐[45]、稻瘟病[46]、大豆疫霉病[47]、黄瓜早花[48]、番茄果实重量[49]等基因。在棉花中,通过BSA-seq定位的基因主要包括gl[19]、Cl[25]、v[28]、Li[50]和[51]。如ZHU等[28]利用BSA-seq将v初步定位在D10染色体1.5 cM的遗传区间内,进一步将其精细定位到分子标记K5499和K5846之间44 kb的物理区间内,区间内存在8个基因,进而克隆了芽黄基因v——。
3 总结与展望
棉花参考基因组发布相对滞后,导致棉花功能基因图位克隆研究进展缓慢。2012年以来,随着棉花基因组测序数据的相继公布和不断完善,棉花功能基因图位克隆研究进入快速发展期。
1986年,COULSON等[52]首次提出图位克隆的概念,是随着分子标记开发、遗传连锁图谱构建逐步发展起来的一种经典的基因克隆策略。在图位克隆提出的早期阶段,由于缺乏基因组序列信息,只能根据候选基因在染色体上精细定位的遗传信息,筛选与候选基因紧密连锁的基因组文库,构建候选基因区域的染色体片段重叠群,再通过染色体步移缩小定位区间,最终克隆候选基因并鉴定其生物学功能;随着基因组序列信息的不断完善,分子标记的不断开发,基因物理位置信息已知,省去了筛选基因组文库和构建候选区段物理图谱的过程,图位克隆技术越来越成为高效可行的鉴定候选基因的方法。
目前,除少数存在重组交换异常的染色体区段,通过图位克隆策略挖掘棉花功能基因已基本不存在瓶颈。随着基因组测序技术的升级,测序成本的降低,BSA-seq等[45]一些新方法的出现,使定位克隆更加快速准确,越来越多棉花功能基因相继被图位克隆。全基因组关联分析(genome-wide association study,GWAS)也逐渐成为筛选功能基因位点的重要手段[53-54],但GWAS在一般情况下较难直接克隆到目的基因。利用转基因[55]和基因组编辑技术[56]对基因功能开展全面系统的鉴定评价,为棉花分子设计育种提供理论基础和基因资源,必将加快棉花遗传改良进程。
[1] WANG K B, WANG Z W, LI F G, YE W W, WANG J Y, SONG G L, YUE Z, CONG L, SHANG H H, ZHU S L, ZOU C S, LI Q, YUAN Y L, LU C R, WEI H L, GOU C Y, ZHENG Z Q, YIN Y, ZHANG X Y, LIU K, WANG B, SONG C, SHI N, KOHEL R J, PERCY R G, YU J Z, ZHU Y X, WANG J, YU S X. The draft genome of a diploid cotton. Nature Genetics, 2012, 44(10): 1098-1103.
[2] PATERSON A H, WENDEL J F, GUNDLACH H, GUO H, JENKINS J, JIN D C, LLEWELLYN D, SHOWMAKER K C, SHU S Q, UDALL J, YOO M J, BYERS R, CHEN W, DORON-FAIGENBOIM A, DUKE M V, GONG L, GRIMWOOD J, GROVER C, GRUPP K, HU G J, LEE T H, LI J P, LIN L F, LIU T, MARLER B S, PAGE J T, ROBERTS A W, ROMANEL E, SANDERS W S, SZADKOWSKI E, TAN X, TANG H B, XU C M, WANG J P, WANG Z N, ZHANG D, ZHANG L, ASHRAFI H, BEDON F, BOWERS J E, BRUBAKER C L, CHEE P W, DAS S, GINGLE A R, HAIGLER C H, HARKER D, HOFFMANN L V, HOVAV R, JONES D C, LEMKE C, MANSOOR S, RAHMAN M U, RAINVILLE L N, RAMBANI A, REDDY U K, RONG J-K, SARANGA Y, SCHEFFLER B E, SCHEFFLER J A, STELLY D M, TRIPLETT B A, VAN DEYNZE A, VASLIN M F S, WAGHMARE V N, WALFORD S A, WRIGHT R J, ZAKI E A, ZHANG T D, DENNIS E S, MAYER K F X, PETERSON D G, ROKHSAR D S, WANG X Y, SCHMUTZ J. Repeated polyploidization ofgenomes and the evolution of spinnable cotton fibres. Nature, 2012, 492(7429): 423-427.
[3] LI F G, FAN G Y, WANG K B, SUN F M, YUAN Y L, SONG G L, LI Q, MA Z Y, LU C R, ZOU C S, CHEN W B, LIANG X M, SHANG H H, LIU W Q, SHI C C, XIAO G H, GOU C Y, YE W W, XU X, ZHANG X Y, WEI H L, LI Z F, ZHANG G Y, WANG J Y, LIU K, KOHEL R J, PERCY R G, YU J Z, ZHU Y X, WANG J, YU S X. Genome sequence of the cultivated cotton. Nature Genetics, 2014, 46(6): 567-572.
[4] LI F G, FAN G Y, LU C R, XIAO G H, ZOU C S, KOHEL R J, MA Z Y, SHANG H H, MA X F, WU J Y, LIANG X M, HUANG G, PERCY R G, LIU K, YANG W H, CHEN W B, DU X M, SHI C C, YUAN Y L, YE W W, LIU X, ZHANG X Y, LIU W Q, WEI H L, WEI S J, HUANG G D, ZHANG X L, ZHU S J, ZHANG H, SUN F M, WANG X F, LIANG J, WANG J H, HE Q, HUANG L H, WANG J, CUI J J, SONG G L, WANG K B, XU X, YU J Z, ZHU Y X, YU S X. Genome sequence of cultivated Upland cotton (TM-1) provides insights into genome evolution. Nature Biotechnology, 2015, 33(5): 524-530.
[5] ZHANG T Z, HU Y, JIANG W K, FANG L, GUAN X Y, CHEN J D, ZHANG J B, SASKI C A, SCHEFFLER B E, STELLY D M, HULSE-KEMP A M, WAN Q, LIU B L, LIU C X, WANG S, PAN M Q, WANG Y K, WANG D W, YE W X, CHANG L J, ZHANG W P, SONG Q X, KIRKBRIDE R C, CHEN X Y, DENNIS E, LLEWELLYN D J, PETERSON D G, THAXTON P, JONES D C, WANG Q, XU X Y, ZHANG H, WU H T, ZHOU L, MEI G F, CHEN S Q, TIAN Y, XIANG D, LI X H, DING J, ZUO Q Y, TAO L N, LIU Y C, LI J, LIN Y, HUI Y Y, CAO Z S, CAI C P, ZHU X F, JIANG Z, ZHOU B L, GUO W Z, LI R Q, CHEN Z J. Sequencing of allotetraploid cotton (L. acc. TM-1) provides a resource for fiber improvement. Nature Biotechnology, 2015, 33(5): 531-537.
[6] YUAN D J, TANG Z H, WANG M J, GAO W H, TU L L, JIN X, CHEN L L, HE Y H, ZHANG L, ZHU L F, LI Y, LIANG Q Q, LIN Z X, YANG X Y, LIU N, JIN S X, LEI Y, DING Y H, LI G L, RUAN X A, RUAN Y J, ZHANG X L. The genome sequence of Sea-Island cotton () provides insights into the allopolyploidization and development of superior spinnable fibres. Scientific Reports, 2016, 5(1): 17662.
[7] LIU X, ZHAO B, ZHENG H J, HU Y, LU G, YANG C Q, CHEN J D, CHEN J J, CHEN D Y, ZHANG L, ZHOU Y, WANG L J, GUO W Z, BAI Y L, RUAN J X, SHANGGUAN X X, MAO Y B, SHAN C M, JIANG J P, ZHU Y Q, JIN L, KANG H, CHEN S T, HE X L, WANG R, WANG Y Z, CHEN J, WANG L J, YU S T, WANG B Y, WEI J, SONG S C, LU X Y, GAO Z C, GU W Y, DENG X, MA D, WANG S, LIANG W H, FANG L, CAI C P, ZHU X F, ZHOU B L, JEFFREY CHEN Z, XU S H, ZHANG Y G, WANG S Y, ZHANG T Z, ZHAO G P, CHEN X Y.genome sequence provides insight into the evolution of extra-long staple fiber and specialized metabolites. Scientific Reports, 2015, 5(1): 14139.
[8] MA Z Y, ZHANG Y, WU L Q, ZHANG G Y, SUN Z W, LI Z K, JIANG Y F, KE H F, CHEN B, LIU Z W, GU Q S, WANG Z C, WANG G N, YANG J, WU J H, YAN Y Y, MENG C S, LI L H, LI X X, MO S J, WU N, MA L M, CHEN L T, ZHANG M, SI A J, YANG Z W, WANG N, WU L Z, ZHANG D M, CUI Y R, CUI J, LV X, LI Y, SHI R K, DUAN Y H, TIAN S L, WANG X F. High-quality genome assembly and resequencing of modern cotton cultivars provide resources for crop improvement. Nature Genetics, 2021, 53(9): 1385-1391.
[9] HE S P, SUN G F, GENG X L, GONG W F, DAI P H, JIA Y H, SHI W J, PAN Z E, WANG J D, WANG L Y, XIAO S H, CHEN B J, CUI S F, YOU C Y, XIE Z M, WANG F, SUN J, FU G Y, PENG Z, HU D W, WANG L R, PANG B Y, DU X M. The genomic basis of geographic differentiation and fiber improvement in cultivated cotton. Nature Genetics, 2021, 53(6): 916-924.
[10] HUANG G, WU Z G, PERCY R G, BAI M Z, LI Y, FRELICHOWSKI J E, HU J, WANG K, YU J Z, ZHU Y X. Genome sequence ofand genome updates ofandprovide insights into cotton A-genome evolution. Nature Genetics, 2020, 52(5): 516-524.
[11] YANG Z E, GE X Y, YANG Z R, QIN W Q, SUN G F, WANG Z, LI Z, LIU J, WU J, WANG Y, LU L L, WANG P, MO H J, ZHANG X Y, LI F G. Extensive intraspecific gene order and gene structural variations in upland cotton cultivars. Nature Communications, 2019, 10(1): 2989.
[12] MA D, HU Y, YANG C Q, LIU B L, FANG L, WAN Q, LIANG W H, MEI G F, WANG L J, WANG H P, DING L Y, DONG C G, PAN M Q, CHEN J D, WANG S, CHEN S Q, CAI C P, ZHU X F, GUAN X Y, ZHOU B L, ZHU S J, WANG J W, GUO W Z, CHEN X Y, ZHANG T Z. Genetic basis for glandular trichome formation in cotton. Nature Communications, 2016, 7: 10456.
[13] 臧新山, 耿延会, 裴文锋, 吴嫚, 李兴丽, 张金发, 于霁雯. 棉花形态性状质量遗传分析与基因定位研究进展. 棉花学报, 2018, 30(6): 473-485.
ZANG X S, GENG Y H, PEI W F, WU M, LI X L, ZHANG J F, YU J W. Research progress on the mendelian genetic analysis and molecular mapping of morphological qualitative traits in cotton. Cotton Science, 2018, 30(6): 473-485. (in Chinese)
[14] WAN Q, GUAN X Y, YANG N N, WU H T, PAN M Q, LIU B L, FANG L, YANG S P, HU Y, YE W X, ZHANG H, MA P Y, CHEN J D, WANG Q, MEI G F, CAI C P, YANG D L, WANG J W, GUO W Z, ZHANG W H, CHEN X Y, ZHANG T Z. Small interfering RNAs from bidirectional transcripts ofregulate cotton fiber development. The New Phytologist, 2016, 210(4): 1298-1310.
[15] THYSSEN G N, FANG D D, TURLEY R B, FLORANE C B, LI P, MATTISON C P, NAOUMKINA M. A Gly65Val substitution in an actin,, disrupts cell polarity and F-actin organization resulting in dwarf, lintless cotton plants. The Plant Journal, 2017, 90(1): 111-121.
[16] WU H T, TIAN Y, WAN Q, FANG L, GUAN X Y, CHEN J D, HU Y, YE W X, ZHANG H, GUO W Z, CHEN X Y, ZHANG T Z. Genetics and evolution ofgenes regulating cotton lint fiber development. The New Phytologist, 2018, 217(2): 883-895.
[17] YAN Q, WANG Y, LI Q, ZHANG Z S, DING H, ZHANG Y, LIU H S, LUO M, LIU D X, SONG W, LIU H F, YAO D, OUYANG X F, LI Y H, LI X, PEI Y, XIAO Y H. Up-regulation ofin cotton fibres during secondary wall thickening results in brown fibres with improved quality. Plant Biotechnology Journal, 2018, 16(10): 1735-1747.
[18] CHENG H L, LU C R, YU J Z, ZOU C S, ZHANG Y P, WANG Q L, HUANG J, FENG X X, JIANG P F, YANG W C, SONG G L. Fine mapping and candidate gene analysis of the dominant glandless geneGlin cotton (spp.). Theoretical and Applied Genetics, 2016, 129(7): 1347-1355.
[19] ZANG Y H, XU C Y, XUAN L S, DING L Y, ZHU J K, SI Z F, ZHANG T Z, HU Y. Identification and characteristics of a novel gland-forming gene in cotton. The Plant Journal, 2021, 108(3): 781-792.
[20] HU W, QIN W Q, JIN Y Y, WANG P, YAN Q D, LI F G, YANG Z E. Genetic and evolution analysis of extrafloral nectary in cotton. Plant Biotechnology Journal, 2020, 18(10): 2081-2095.
[21] PEI Y F, ZHANG J, WU P, YE L, YANG D F, CHEN J D, LI J, HU Y, ZHU X F, GUO X P, ZHANG T Z.encoding a class VIIIb AP2/ERF is required for both extrafloral and floral nectary development in. The Plant Journal, 2021, 106(4): 1116-1127.
[22] ZHU Q H, ZHANG J, LIU D X, STILLER W, LIU D J, ZHANG Z S, LLEWELLYN D, WILSON I. Integrated mapping and characterization of the gene underlying the okra leaf trait inL. Journal of Experimental Botany, 2016, 67(3): 763-774.
[23] CHANG L J, FANG L, ZHU Y J, WU H T, ZHANG Z Y, LIU C X, LI X H, ZHANG T Z. Insights into interspecific hybridization events in allotetraploid cotton formation from characterization of a gene- regulating leaf shape. Genetics, 2016, 204(2): 799-806.
[24] ANDRES R J, CONEVA V, FRANK M H, TUTTLE J R, SAMAYOA L F, HAN S-W, KAUR B, ZHU L, FANG H, BOWMAN D T, ROJAS-PIERCE M, HAIGLER C H, JONES D C, HOLLAND J B, CHITWOOD D H, KURAPARTHY V. Modifications to agene are responsible for the major leaf shapes of Upland cotton (L.). Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(1): E57-E66.
[25] SI Z F, LIU H, ZHU J K, CHEN J D, WANG Q, FANG L, GAO F K, TIAN Y, CHEN Y L, CHANG L J, LIU B L, HAN Z G, ZHOU B L, HU Y, HUANG X Z, ZHANG T Z. Mutation ofhomologs in cotton promotes short-branching plant architecture. Journal of Experimental Botany, 2018, 69(10): 2543-2553.
[26] LIU D X, TENG Z H, KONG J, LIU X Y, WANG W W, ZHANG X, ZHAI T F, DENG X P, WANG J X, ZENG J Y, XIAO Y H, GUO K, ZHANG J, LIU D J, WANG W R, ZHANG Z S. Natural variation in a CENTRORADIALIS homolog contributed to cluster fruiting and early maturity in cotton. BMC Plant Biology, 2018, 18(1): 286.
[27] CHEN W, YAO J B, LI Y, ZHU S H, GUO Y, FANG S T, ZHAO L J, WANG J Y, YUAN L, LU Y J, ZHANG Y S. Open-bud duplicate loci are identified as, orthologs of MIXTA-like genes on homologous chromosomes of allotetraploid cotton. Frontiers in Plant Science, 2020, 11: 81.
[28] ZHU J K, CHEN J D, GAO F K, XU C Y, WU H T, CHEN K, SI Z F, YAN H, ZHANG T Z. Rapid mapping and cloning of the virescent-1 gene in cotton by bulked segregant analysis-next generation sequencing and virus-induced gene silencing strategies. Journal of Experimental Botany, 2017, 68(15): 4125-4135.
[29] MAO G Z, MA Q, WEI H L, SU J J, WANG H T, MA Q F, FAN S L, SONG M Z, ZHANG X L, YU S X. Fine mapping and candidate gene analysis of the virescent gene v in Upland cotton (Gossypium). Molecular Genetics and Genomics, 2018, 293(1): 249-264.
[30] LI X, OUYANG X F, ZHANG Z S, HE L, WANG Y, LI Y H, ZHAO J, CHEN Z, WANG C N, DING L L, PEI Y, XIAO Y H. Over- expression of the red plant geneRenhances anthocyanin production and resistance to bollworm and spider mite in cotton. Molecular Genetics and Genomics, 2019, 294(2): 469-478.
[31] DENG J Q, FANG L, ZHU X F, ZHOU B L, ZHANG T Z. Agene induces hybrid lethality in cotton. Journal of Experimental Botany, 2019, 70(19): 5145-5156.
[32] MA H H, WU Y L, LV R L, CHI H B, ZHAO Y L, LI Y L, LIU H B, MA Y Z, ZHU L F, GUO X P, KONG J, WU J Y, XING C Z, ZHANG X L, MIN L. Cytochrome P450 mono-oxygenase CYP703A2 plays a central role in sporopollenin formation andfertility in cotton. Journal of Integrative Plant Biology, 2022, 64(10): 2009-2025
[33] WU Y L, LI X, LI Y L, MA H H, CHI H B, MA Y Z, YANG J, XIE S, ZHANG R, LIU L Y, SU X J, LV R J, KHAN A H, KONG J, GUO X P, LINDSEY K, MIN L, ZHANG X L. Degradation of de-esterified pctin/homogalacturonan by the polygalacturonaseis necessary for pollen exine formation and male fertility in cotton. Plant Biotechnology Journal, 2022, 20(6): 1054-1068.
[34] CHAI Q C, SHANG X G, WU S, ZHU G Z, CHENG C Z, CAI C P, WANG X Y, GUO W Z. 5-aminolevulinic acid dehydratase gene dosage affects programmed cell death and immunity. Plant Physiology, 2017, 175(1): 511-528.
[35] CHAI Q C, WANG X L, GAO M, ZHAO X C, CHEN Y, ZHANG C, JIANG H, WANG J B, WANG Y C, ZHENG M N, BALTAEVICH A M, ZHAO J, ZHAO J S. A glutathione S‐transferasedetermines flower petal pigmentation via regulating anthocyanin accumulation in cotton. Plant Biotechnology Journal, 2022, 21: 433-448.
[36] ABID M A, WEI Y X, MENG Z G, WANG Y, YE Y L, WANG Y N, HE H Y, ZHOU Q, LI Y Y, WANG P L, LI X G, YAN L H, MALIK W, GUO S D, CHU C C, ZHANG R, LIANG C Z. Increasing floral visitation and hybrid seed production mediated by beauty mark in. Plant Biotechnology Journal, 2022, 20(7): 1274-1284.
[37] ZANG Y H, HU Y, XU C Y, WU S J, WANG Y K, NING Z Y, HAN Z G, SI Z F, SHEN W J, ZHANG Y Y, FANG L, ZHANG T Z.controlling helical growth results in production of stronger cotton fiber. iScience, 2021, 24(8): 102930.
[38] ZHANG R T, SHEN C, ZHU D, LE Y, WANG N, LI Y X, ZHANG X L, LIN Z X. Fine-mapping and candidate gene analysis ofcontrolling fiber length in upland cotton (L.). Theoretical and Applied Genetics, 2022, 135(12): 4483-4494.
[39] AHMED M M, HUANG C, SHEN C, KHAN A Q, LIN Z X. Map-based cloning ofdiscovered brassinosteroid-mediated control of organ size in cotton. Plant Science, 2020, 291: 110315.
[40] ZHAO J, LIU J G, XU J W, ZHAO L, WU Q J, XIAO S H. Quantitative trait locus mapping and candidate gene analysis forwilt resistance usingchromosomal segment introgressed line. Frontiers in Plant Science, 2018, 9: 682.
[41] DONG C G, DING Y Z, GUO W Z, ZHANG T Z. Fine mapping of the dominant glandless GeneGlin Sea-island cotton (L.). Chinese Science Bulletin, 2007, 52(22): 3105-3109.
[42] SONG L, GUO W Z, ZHANG T Z. Interaction of novel Dobzhansky–Muller type genes for the induction of hybrid lethality betweenandcv. Coastland R4-4. Theoretical and Applied Genetics, 2009, 119(1): 33-41.
[43] LIU D X, LIU F, SHAN X R, ZHANG J, TANG S Y, FANG X M, LIU X Y, WANG W W, TAN Z Y, TENG Z H, ZHANG Z S, LIU D J. Construction of a high-density genetic map and lint percentage and cottonseed nutrient trait QTL identification in upland cotton (L.). Molecular Genetics and Genomics, 2015, 290(5): 1683-1700.
[44] MICHELMORE R W, PARAN I, KESSELI R V. Identification of markers linked to disease-resistance genes by bulked segregant analysis: a rapid method to detect markers in specific genomic regions by using segregating populations. Proceedings of the National Academy of Sciences of the United States of America, 1991, 88(21): 9828-9832.
[45] TAKAGI H, TAMIRU M, ABE A, YOSHIDA K, UEMURA A, YAEGASHI H, OBARA T, OIKAWA K, UTSUSHI H, KANZAKI E, MITSUOKA C, NATSUME S, KOSUGI S, KANZAKI H, MATSUMURA H, URASAKI N, KAMOUN S, TERAUCHI R. MutMap accelerates breeding of a salt-tolerant rice cultivar. Nature Biotechnology, 2015, 33(5): 445-449.
[46] TAKAGI H, ABE A, YOSHIDA K, KOSUGI S, NATSUME S, MITSUOKA C, UEMURA A, UTSUSHI H, TAMIRU M, TAKUNO S, INNAN H, CANO L M, KAMOUN S, TERAUCHI R. QTL-seq: rapid mapping of quantitative trait loci in rice by whole genome resequencing of DNA from two bulked populations. The Plant Journal, 2013, 74(1): 174-183.
[47] ZHONG C, SUN S L, LI Y P, DUAN C X, ZHU Z D. Next-generation sequencing to identify candidate genes and develop diagnostic markers for a novel Phytophthora resistance gene,, in soybean. Theoretical and Applied Genetics, 2018, 131(3): 525-538.
[48] LU H F, LIN T, KLEIN J, WANG S H, QI J J, ZHOU Q, SUN J J, ZHANG Z H, WENG Y Q, HUANG S W. QTL-seq identifies an early flowering QTL located near Flowering Locus T in cucumber. Theoretical and applied genetics, 2014, 127(7): 1491-1499.
[49] ILLA-BERENGUER E, VAN HOUTEN J, HUANG Z J, VAN DER KNAAP E. Rapid and reliable identification of tomato fruit weight and locule number loci by QTL-seq. Theoretical and Applied Genetics, 2015, 128(7): 1329-1342.
[50] NAOUMKINA M, THYSSEN G N, FANG D D, FLORANE C B, LI P. A deletion/duplication in the Ligon lintless-2 locus induces siRNAs that inhibit cotton fiber cell elongation. Plant Physiology, 2022, 190(3): 1792-1805.
[51] WANG X Y, ZHANG X W, FAN D R, GONG J W, LI S Q, GAO Y J, LIU A Y, LIU L J, DENG X Y, SHI Y Z, SHANG H H, ZHANG Y M, YUAN Y L. AAQSP increases mapping resolution of stable QTLs through applying NGS-BSA in multiple genetic backgrounds. Theoretical and Applied Genetics, 2022, 135(9): 3223-3235.
[52] COULSON A, SULSTON J, BRENNER S, KARN J. Toward a physical map of the genome of the nematode Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America, 1986, 83(20): 7821-7825.
[53] LI T G, MA X F, LI N Y, ZHOU L, LIU Z, HAN H Y, GUI Y J, BAO Y M, CHEN J Y, DAI X F. Genome-wide association study discovered candidate genes ofwilt resistance in upland cotton (L.). Plant Biotechnology Journal, 2017, 15(12): 1520-1532.
[54] LIU S M, ZHANG X J, XIAO S H, MA J, SHI W J, QIN T,XI H, NIE X H, YOU C Y, XU Z, WANG T Y, WANG Y J, ZHANG Z N, LI J Y, KONG J, AIERXI A, YU Y, LINDSEY K, KLOSTERMAN S J, ZHANG X L, ZHU L F. A Single-nucleotide mutation in a GLUTAMATE RECEPTOR-LIKE gene confers resistance towilt in. Advanced Science, 2021, 8(7): 2002723.
[55]GE X Y, XU J T, YANG Z E, YANG X F, WANG Y, CHEN Y L, WANG P, LI F G. Efficient genotype‐independent cotton genetic transformation and genome editing. Journal of Integrative Plant Biology, 2023, 65(4): 907-917.
[56] WANG P C, ZHANG J, SUN L, MA Y Z,XU J, LIANG S J, DENG J W, TAN J F, ZHANG Q H, TU L L, DANIELL H, JIN S X, ZHANG X L. High efficient multisites genome editing in allotetraploid cotton () using CRISPR/Cas9 system. Plant biotechnology journal, 2018, 16(1): 137-150.
Research advances of map-based cloning genes in cotton
ZANG XinShan1,2,4, WANG KangWen1,3, ZHANG XianLiang1,2, WANG XuePing1, WANG Jun1, LIANG Yu1, PEI XiaoYu1, REN Xiang1,2, Lü YuLong1,2, GAO Yu1, WANG XingXing1, PENG YunLing3, MA XiongFeng1,2,3,4
1Institute of Cotton Research of Chinese Academy of Agricultural Sciences/State Key Laboratory of Cotton Biology/Key Laboratory of Biological and Genetic Breeding of Cotton, Ministry of Agriculture and Rural Affairs, Anyang 455000, Henan;2Western Agricultural Research Center, Chinese Academy of Agricultural Sciences, Changji 831100, Xinjiang;3College of Agronomy, Gansu Agricultural University/Gansu Provincial Key Lab of Arid Land Crop Science/Gansu Key Lab of Crop Improvement and Germplasm Enhancement, Lanzhou 730070;4School of Agricultural Sciences, Zhengzhou University, Zhengzhou 450001
Map-based cloning is a classical and effective method to identify candidate genes for specific phenotypic variants. Map-based cloning of functional genes plays important roles in the innovative utilization of germplasm resources, molecular design breeding and improving breeding efficiency. In recent years, the whole-genome sequencing of,,,andhas been completed and improved. map-based cloning has entered into a crucial period. In 2016, the dominant glandless geneGl() was the first map-based cloning gene in cotton. So far, 20 qualitative traits genes and 5 quantitative traits genes have been identified by map-based cloning technology. In this paper, research progress was systematically reviewed in fiber, gland, nectary, leaf type, plant architecture, plant color, and fertility in terms of gene symbols, names, chromosomal positioning, and candidate genes. Moreover, map-based cloning strategies were systematically reviewed in mapping populations and bulked segregate analysis-sequencing (BSA-seq). With the reduction of sequencing cost and utilization of BSA-seq, it is believed that more and more genes will be cloned by map-based cloning technology. In addition, transformation and genome editing have been successfully used to evaluate the function of the candidate gene in the target interval. It is believed that map-based cloning could provide a theoretical basis and genetic resources for molecular design breeding in cotton.
cotton; map-based cloning; molecular marker; mapping population; BSA-seq

10.3864/j.issn.0578-1752.2023.23.006
2023-02-08;
2023-04-17
国家棉花产业技术体系(CARS-15-07)、新疆维吾尔自治区重点研发任务专项(2022B02052-2)、新疆维吾尔自治区天山英才计划(2021)、昌吉回族自治州科技重大专项(2021Z01-01)
臧新山,E-mail:zangxinshan@163.com。王康文,E-mail:wangkangwen@126.com。臧新山和王康文为同等贡献作者。通信作者彭云玲,E-mail:pengyunlingpyl@163.com。通信作者马雄风,E-mail:maxf_caas@163.com
(责任编辑 李莉)
