棉花FLA基因家族的全基因组鉴定及GhFLA05在棉纤维发育中的功能分析
2023-12-28唐丽媛蔡肖王海涛李兴河张素君刘存敬张建宏
唐丽媛,蔡肖,王海涛,李兴河,张素君,刘存敬,张建宏
棉花FLA基因家族的全基因组鉴定及在棉纤维发育中的功能分析
唐丽媛,蔡肖,王海涛,李兴河,张素君,刘存敬,张建宏
河北省农林科学院棉花研究所/农业农村部黄淮海半干旱区棉花生物学与遗传育种重点实验室/国家棉花改良中心河北分中心,石家庄 050051
【背景】伴随着棉纺织工艺水平的提升和人们对高品质纺织品的追求,提升棉花纤维品质日益重要。类成束阿拉伯半乳糖蛋白(fasciclin-like arabinogalactan proteins,FLAs)在棉纤维起始发育、次生壁合成等过程中可能具有重要作用。【目的】通过对棉花FLA基因家族进行全面鉴定与分析,研究该家族成员的共性特征及特异性表达模式,为FLA在棉纤维发育中的功能研究提供参考。【方法】根据棉花全基因组数据,使用HMMER 3.0对棉花FLA基因家族成员进行鉴定,并通过Pfam、Smart等软件进一步确认。使用ExPASy、TMHMM分析蛋白理化性质及跨膜结构域,应用MEGA、MCScanX、GSDS、MEME、TBtools、Jalview等工具进行进化树构建、染色体定位、共线性分析和蛋白保守结构域序列比对等。通过转录组数据分析陆地棉FLA基因在不同组织中的表达情况。利用实时荧光定量聚合酶链式反应(quantitative real-time polymerase chain reaction,qRT-PCR)检测在不同纤维品质材料的胚珠及不同发育时期纤维中的表达差异。利用病毒诱导的基因沉默(virus induced gene silencing,VIGS)技术验证的功能。【结果】在陆地棉、海岛棉、亚洲棉和雷蒙德氏棉全基因组中分别鉴定出41、40、20和21个FLA家族成员,系统进化树显示,棉花FLA蛋白可以分为4个群组。进一步对陆地棉FLA家族蛋白进行分析,41个成员均具有1—2个AGP-like糖基化区域和1—2个类成束蛋白结构域(fasciclin-like domain,FAS),其中,37个含有信号肽(signal peptide,SP),25个含有糖基化磷脂酰肌醇(glycosylphosphatidy linositol anchored protein,GPI)锚定信号,基因结构和基序组成在各组中相对保守。亚细胞定位显示,GhFLA05_D可能定位在细胞质的内质网,呈聚集状颗粒,GhFLA18_A和GhFLA22在细胞膜/壁、细胞质和细胞核中均有表达。转录组测序结果表明,Group A和Group B中的FLA蛋白主要在纤维中高表达,可能参与了棉纤维发育伸长和次生壁加厚等过程。在纤维品质差异显著的2个材料中,Group A和Group B成员具有相似的表达模式,并主要在纤维次生壁发育阶段、尤其是20—25 DPA时期优势表达;其中,在次生壁增厚期表现出特异性表达,两材料间存在显著差异,在高比强的RIL229的次生壁阶段更早达到最大值,推测可能在调控纤维比强度差异形成中发挥作用。利用VIGS技术沉默后,使棉纤维断裂比强度降低。【结论】在陆地棉、海岛棉、亚洲棉和雷蒙德氏棉中鉴定出122个FLA家族成员,可分为4个群组,不同群组成员间具有较高的结构和功能相似性,并从中鉴定了Group A和Group B成员,可能是主要与棉纤维发育相关的基因。明确家族中是次生壁合成阶段优势表达基因,并与陆地棉不同材料纤维比强度差异形成密切相关。
棉花;FLA;纤维发育;基因家族;表达分析
0 引言
【研究意义】棉花是世界上最重要的天然纤维作物,棉纤维是纺织工业的重要原料[1]。因棉花产量和纤维品质呈负相关,传统育种选择上产量优于品质,育成品种的纤维品质很难适应当前日益提高的棉纺织工艺的需求和人们对高品质纺织品的追求[2],提升棉花纤维品质已成为当务之急。近年来,随着棉花基因组学研究的迅猛发展[1],分子育种为提升棉花纤维品质、加速育种进程提供了有效的策略。部分研究表明,类成束阿拉伯半乳糖蛋白(fasciclin-like arabinogalactan proteins,FLAs)在棉纤维形成中发挥重要作用[3-8],因此,从全基因组水平鉴定分析棉花FLA基因家族,有助于快速锁定基因家族内纤维发育相关基因,丰富棉花纤维发育研究的基因网络,为优质棉精准育种提供基因资源和理论基础。【前人研究进展】阿拉伯半乳聚糖蛋白(arabinogalactan-proteins,AGPs)是一类富含羟脯氨酸的细胞壁糖蛋白,影响植物生长、发育、繁殖和适应环境等多种生物进程[9]。根据核心蛋白骨架差异AGPs可分为6个亚类[10-11]。FLA蛋白是其中的一个亚类[12],不仅具有1—2个典型的AGP-like糖基化区域,还含有1—2个类成束蛋白结构域(fasciclin-like domain,FAS)[13],且大多数FLA基因具有N端信号肽(signal peptide,SP)和C末端糖基化磷脂酰肌醇(glycosylphosphatidy linositol anchored protein,GPI)锚定信号[3, 14]。其中,AGP-like糖基化区域主要是由脯氨酸(Pro)、丙氨酸(Ala)、丝氨酸(Ser)及苏氨酸(Thr)形成的(Ser/Thr/Ala)-Hyp-(Ser/Thr/Ala)-Hyp和(Ser/Thr/Ala)-Hyp-Hyp重复序列区域(proline-alanine-serine-threonine,PAST),经典AGPs中PAST序列占比超过50%[15],而在FLA基因中PAST序列比例普遍低于35%[11]。FAS结构域一般由110—150个氨基酸组成,序列相似性较低,但所有的FAS都含有2个高度保守的H1、H2区域,且中间有1个[Phe/Tyr]-His Motif[16]。蛋白具有AGP-like糖基化区域可能参与细胞间相互作用、细胞黏附和细胞壁生物合成,具有FAS结构域通常在细胞外基质间起到黏连作用[17],SP可能促使分子进入内质网,而GPI锚定信号可以将分子锚定到细胞膜上[14]。FLA是一个复杂的蛋白聚糖多基因家族,在植物生长发育过程中可能发挥重要作用,受到发育和应激的共同调控[9]。利用生物信息学方法,目前,已经在拟南芥[18]、水稻[19]、小麦[20]、杨树[21]、桉树[22]、梨树[23]、香蕉[24]、黄麻[25]、纺织大麻[14]和甘蓝[26]等作物中分别鉴定出21、24、34、35、18、38、30、19、23和33个,He等[16]从藻类、苔类、苔藓、石松类、裸子植物、双子叶植物和单子叶植物的13种植物中鉴定出246个。FLA基因家族的鉴定加速了植物功能的解析。研究发现,拟南芥、杨树、桉树、大麻、棉花等多种作物中在纤维或其他组织细胞的起始及伸长发育、次生壁(secondary cell wall,SCW)合成方面发挥作用。拟南芥与根伸长基因共表达[27],和能够在厚壁组织中高强度表达,在增加茎秆强度和弹性方面发挥作用[28],的H2区域密码子突变会导致细胞壁变薄[29]。杨树在成熟茎的木质部中特异性表达,并主要积累在茎木质部纤维中[30]。桉树和与纤维素微纤丝角和茎秆强度相关[22]。大麻、和参与韧皮纤维细胞的起始和伸长,、、、和参与次生细胞壁的生物合成[14]。棉花中多个编码FLA蛋白的基因在棉花纤维中优势表达[3-4]。[5]和[6]与纤维伸长相关,会影响棉花细胞的延伸及次生细胞壁发育时期的胞间交流[4]。海岛棉在棉纤维SCW沉积阶段的表达量显著高于陆地棉,最终影响纤维强度[31]。除上述功能外,还可能参与维管束的形成与发育[32]、细胞通讯及分子信号传导[26]、聚糖稳定[33]和花粉育性[34-35]等过程,并可以响应各种生物和非生物胁迫,如盐胁迫、冷胁迫、干旱胁迫、热胁迫及外源激素脱落酸和氟啶酮等[20, 29, 36-38]。【本研究切入点】Huang等[3]利用棉花纤维cDNA库鉴定和分析了19个陆地棉FLA基因,但是,自棉花参考基因组公布后,对棉花FLA基因家族全面、系统地鉴定分析仍鲜见报道。【拟解决的关键问题】本研究拟通过生物信息学手段鉴定异源四倍体陆地棉、海岛棉和二倍体亚洲棉、雷蒙德氏棉的FLA基因家族成员,并重点对陆地棉FLA基因家族进行特征分析,通过研究FLA基因在陆地棉不同组织中的表达特异性,挖掘该家族与纤维发育相关的基因;以前期利用优质杂交棉冀1518的亲本构建的高代重组近交系(recombinant inbred lines,RILs)群体[39]中纤维品质性状稳定分离的RIL131和RIL229为研究对象,检测在不同纤维品质材料的胚珠及不同发育时期纤维中的表达差异,并对其中的进行克隆分析和基因沉默验证,为研究棉花FLA基因的功能和在棉花纤维发育中的重要作用奠定基础。
1 材料与方法
1.1 棉花FLA基因家族成员的鉴定
从Cottongen数据库(https://www.cottongen.org/)下载异源四倍体陆地棉(,AD1)[40]、海岛棉(,AD2)[40]和二倍体亚洲棉(,A2)[41]、雷蒙德氏棉(,D5)[41]的全基因组数据。从拟南芥TAIR数据库(https:// www.arabidopsis.org/)下载拟南芥AtFLA蛋白序列信息。从Pfam数据库(http://pfam.xfam.org/)[42]下载FLA蛋白的种子文件PF02469,并利用HMMER 3.0软件的hmmsearch搜索程序鉴定含有FLA保守结构域的陆地棉氨基酸序列(value<0.0001),去除无AGP-like糖基化区域的基因,将所有获得的棉花FLA氨基酸序列通过Pfam(https://pfam.xfam.org/)、Smart(https://smart.embl-heidelberg.de/)及NCBI网站CDD(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)数据库[42-45]进一步进行保守结构域确认,最后,将同时含有FAS结构域和AGP-like糖基化区域的序列鉴定为FLA蛋白。利用在线网页工具Softberry(https://www.softberry.com/berry.phtml?topic =fgenesh&group=programs&subgroup=gfind)对具有明显结构缺失基因重新预测基因模型,并对结构域进行校验推测。
1.2 FLA家族蛋白的理化性质
通过ExPASy(https://web.expasy.org/protparam/)在线软件[46]分析FLA家族成员编码蛋白的相对分子质量、理论等电点等。使用TMHMM Server v.2.0(https://services.healthtech.dtu.dk/service.php?TMHMM -2.0)[47]进行跨膜结构域预测。
1.3 FLA家族蛋白进化树构建
利用MEGA7软件[48]中的Muscle对陆地棉、海岛棉、亚洲棉、雷蒙德氏棉、拟南芥FLA基因家族成员的蛋白序列进行比对分析。采用邻接法(neighbor joining,NJ)构建系统发育进化树,Bootstrap值为1 000。使用在线工具iTOL(https://itol.embl.de/)[49]对进化树进行可视化。
1.4 陆地棉FLA基因染色体分布及共线性分析
从陆地棉参考基因组中获取各染色体FLA基因位置等信息,利用软件MCScanX[50]检测陆地棉全基因组复制基因对,并利用TBtools绘制染色体定位和共线性分析的circos图[51]。利用KaKs_CalculatorL2.0软件计算陆地棉复制基因对的非同义突变率(non- synonymous mutation rate,Ka)、同义突变率(synonymous mutation rate,Ks)及Ka/Ks值。
1.5 基因结构分析及保守结构域分析
利用基因组数据提取FLA基因内含子、外显子位置信息,采用MEME(https://meme-suite.org/meme/ tools/meme)[52]在线分析保守基序,Motif数量设置为10,利用TBtools v1.105可视化FLA家族的基因结构和Motif分布[51]。利用GSDS2.0(https://gsds.gao- lab.org/)绘制保守结构域位置图[53]。根据前人提出的“Hyp连续性假说”[54],提取陆地棉FLA家族成员中的FAS结构域,使用Clustal X进行多序列比对,并使用Jalview2.10.3对比对的结果进行可视化。
1.6 GhFLAs的克隆和亚细胞定位
以前期利用优质杂交棉冀1518的亲本构建的RIL群体[39]中RIL229家系的纤维、根、茎及叶的cDNA为模板,使用Ⅰ酶切,运用同源重组的方法,将、、、编码区(coding sequence,CDS)区段构建到带有35S CaMV启动子和GFP(green fluorescent protein)标签并改造过的pCAMBIA1302载体中(电子附图1),载体购自武汉伯远科技有限公司,在GFP标签和目的基因C末端连有linker,减小直接连接GFP对GPI锚定序列定位可能存在的影响。引物设计相关信息见电子附表1,由金唯智生物科技有限公司合成。将构建好的载体转化GV3101农杆菌,注射本氏烟叶片背面瞬时过表达,以空载体为对照。注射后弱光培养48 h,用激光共聚焦显微镜(Leica TCS SP8)观察叶片GFP荧光分布。GFP荧光激发波长488 nm,叶绿体激发波长640 nm。
1.7 转录组数据分析基因表达特性
从NCBI的SRA数据库(http://www.ncbi.nlm.nih. gov/sra/)下载陆地棉TM-1根、茎、叶、苞叶、花托、萼片、花瓣、雄蕊、花丝、花药等10个组织和不同发育时期胚珠、纤维的转录组测序数据,序列号为PRJNA4906267,对FPKM(fragments per kilobase of transcript per million fragments mapped)数据进行log2(1+FPKM)标准化处理,使用TBtools v1.105生成热图。
1.8 GhFLAs在不同材料不同发育时期纤维的表达
以前期构建的RIL群体中纤维品质性状稳定分离的RIL131和RIL229为研究对象,2017—2021年连续5年种植在河北省农林科学院棉花研究所石家庄小安舍试验站进行鉴定。取两材料开花后0天(day post-anthesis,DPA)胚珠及5、10、15、20和25 DPA纤维,提取样品总RNA,反转录合成第一链cDNA,以第一链cDNA为模板进行实时荧光定量聚合酶链式反应(quantitative real-time polymerase chain reaction,qRT-PCR)鉴定基因在各组织中的表达情况。对Group A和Group B中的设计特异引物(电子附表1),其中,和未设计出特异性引物。以陆地棉组蛋白基因为内参,在CFX96定量PCR仪(Bio-Rad)上进行qRT-PCR。按照TB Green® Premix Ex Taq™ II(Tli RNaseH Plus)推荐的反应体系进行PCR扩增,每个样品进行3次生物学重复,采用2-△△CT法对基因相对表达量进行分析。
1.9 GhFLA05的克隆和VIGS技术验证功能
以RIL131和RIL229的20DPA纤维cDNA为模板,利用Primer Premier 5.0设计基因克隆引物(电子附表1),采用TaKaRa公司的PrimeSTAR HS高保真酶扩增、的CDS区段,连接到®-Blunt(全式金)克隆载体并转入1-T1感受态细胞,筛选阳性克隆并送测序。
通过病毒诱导的基因沉默(virus induced gene silencing,VIGS)技术验证的功能。利用SGN VIGS Tool(https://vigs.solgenomics.net/# opennewwindow)设计沉默片段,通过双酶切(Ⅰ和Ⅰ)方法将中的目标片段构建到棉花叶皱缩病毒(cotton leaf crumple virus,CLCrV)载体[55]上,该体系经多项研究证明病毒活力能够持续到棉花纤维发育的伸长和次生壁加厚等阶段[56-57]。试验所用引物见电子附表1。将构建好的载体转化LBA4404农杆菌。试验涉及到的菌液主要有辅助质粒CLCrVB、空载体CLCrVA、阳性对照载体CLCrVA-和目的基因载体CLCrVA-。
在河北省农林科学院温室内,以RIL229种子为材料,播种7 d后,取子叶刚刚完全平展的棉花幼苗,分别用含pCLCrVB:CLCrVA-、pCLCrVB: CLCrVA-重组载体的菌液和含有pCLCrVB: CLCrVA空载体的菌液按1﹕1的比例混合均匀后侵染整个子叶,注射面积保证在98%以上。注射后的植株避光生长12 h后转入正常光温条件(光照/黑暗=14 h/10 h,28 ℃)下培养。以阳性对照出现白化现象作为侵染成功的参考。在棉花植株现蕾初期对主茎叶腋处进行二次注射以延长基因沉默时间、确保后期基因沉默效果。取试验植株20 DPA的棉铃进行qRT-PCR分析,计算沉默效率。待棉铃成熟、自然吐絮后,单株收获轧取棉纤维,因收获单株纤维样品量可能达不到测定重量,所以选取沉默效率高的6株,随机将2株纤维样品作为一组进行混合,使用USTER HVI1000 M700大容量棉花检测仪测定棉花纤维品质指标,包括纤维的长度、断裂比强度、马克隆值、整齐度、伸长率等。
2 结果
2.1 棉花FLA基因家族成员的鉴定
通过棉花全基因组蛋白序列信息分析,分别在陆地棉、海岛棉、亚洲棉和雷蒙德氏棉中鉴定出41、40、20和21个FLA基因(表1,电子附表2—4)。按照其在染色体上的位置(图1)排序进行命名,对于四倍体棉,先以A亚组染色体上位置排序,A、D亚组根据基因同源性以A、D区分命名,D亚组未命名到的基因再按染色体顺序继续编号。其中,陆地棉中有文献命名报道过的有21个(电子附表2),其他3个棉种FLA基因报道较少,因此,未做统计。理化性质分析表明,棉花中FLA蛋白的氨基酸数量为239—515 aa,分子质量为25.42—57.29 kDa,等电点变化为5.11—9.41。稳定蛋白有65个(24.91—39.89),占53.28%,在陆地棉和海岛棉中较二倍体棉比例高,分别为56.10%和55.00%。亲水性平均系数表明,家族中疏水性蛋白75个(0.009—0.280),占61.48%。家族中跨膜结构域数量为0—3个,59.84%的FLA不存在跨膜结构域,37.70%有1个,仅GrFLA08有2个,同为A亚组的GhFLA13和GaFLA13存在3个跨膜结构域。
2.2 棉花FLA系统进化分析
以122个棉花FLAs和21个拟南芥FLAs的氨基酸序列构建FLA家族成员的系统发育树(图2)。所有FLA家族成员可以分为Group A—Group D 4个亚群,其中,Group A成员最多,有59个成员,6个来源于拟南芥,陆地棉和海岛棉各有18个,8个来自于亚洲棉,9个来自于雷蒙德氏棉;Group D成员最少,包含有9个成员,3个来自于拟南芥,陆地棉和海岛棉各有2个,亚洲棉和雷蒙德氏棉各有1个;Group C中包含53个成员,内部分枝较多,暗示其功能可能具有多样性。棉花与拟南芥亲缘关系相对较远,分析发现只有2组直系同源关系:和、、直系同源,和、、、直系同源。

表1 陆地棉FLA基因家族成员的特性分析

陆地棉A亚组、陆地棉D亚组、亚洲棉、雷蒙德氏棉、海岛棉A亚组、海岛棉D亚组的染色体分别用绿色、深蓝色、黄色、红色、天蓝色、粉色表示
2.3 棉花FLA家族基因的染色体定位和共线性分析
染色体定位(图3-A)结果表明,二倍体亚洲棉和雷蒙德氏棉的FLA基因分别分布在9、10条染色体上,四倍体陆地棉和海岛棉均分布在19条染色体上。每条染色体上有1—6个FLA基因,不同基因(亚)组中均为第7染色体上最多,均有6个FLA基因。在四倍体的陆地棉和海岛棉中,A、D亚组均有19对对应拷贝基因,陆地棉在D亚组没有拷贝,和在A亚组没有拷贝,海岛棉和在A亚组中没有对应拷贝。
对4个棉种基因组内的FLA基因共线性分析(图3-A)显示,所有来自全基因组复制或片段复制,无串联重复,亚洲棉、雷蒙德氏棉、陆地棉和海岛棉种内分别有6、10、32和33对基因具有共线性关系。各个棉种内D基因组的共线性关系基因对数量多于A基因组。对4个棉种基因组间的FLA基因共线性分析(图3-B),陆地棉A亚组与亚洲棉的共线性基因对为31对,比海岛棉的多4对,D亚组与雷蒙德氏棉的共线性基因对有44对,比海岛棉多7对,说明与同样多倍化后的海岛棉相比,陆地棉FLA基因与二倍体棉具有更高的同源性。亚洲棉在雷蒙德氏棉和2个四倍体棉中均无共线性基因对,推测该基因可能在3个棉种的进化中丢失。所有共线性关系中,的Ka/Ks=1,进化中主要经历了中性选择,、、、、的Ka/Ks>1,经历了正向选择,其余基因对Ka/Ks<1,说明棉花多数FLA基因经历了纯化选择,进化较保守,暗示功能相对稳定。
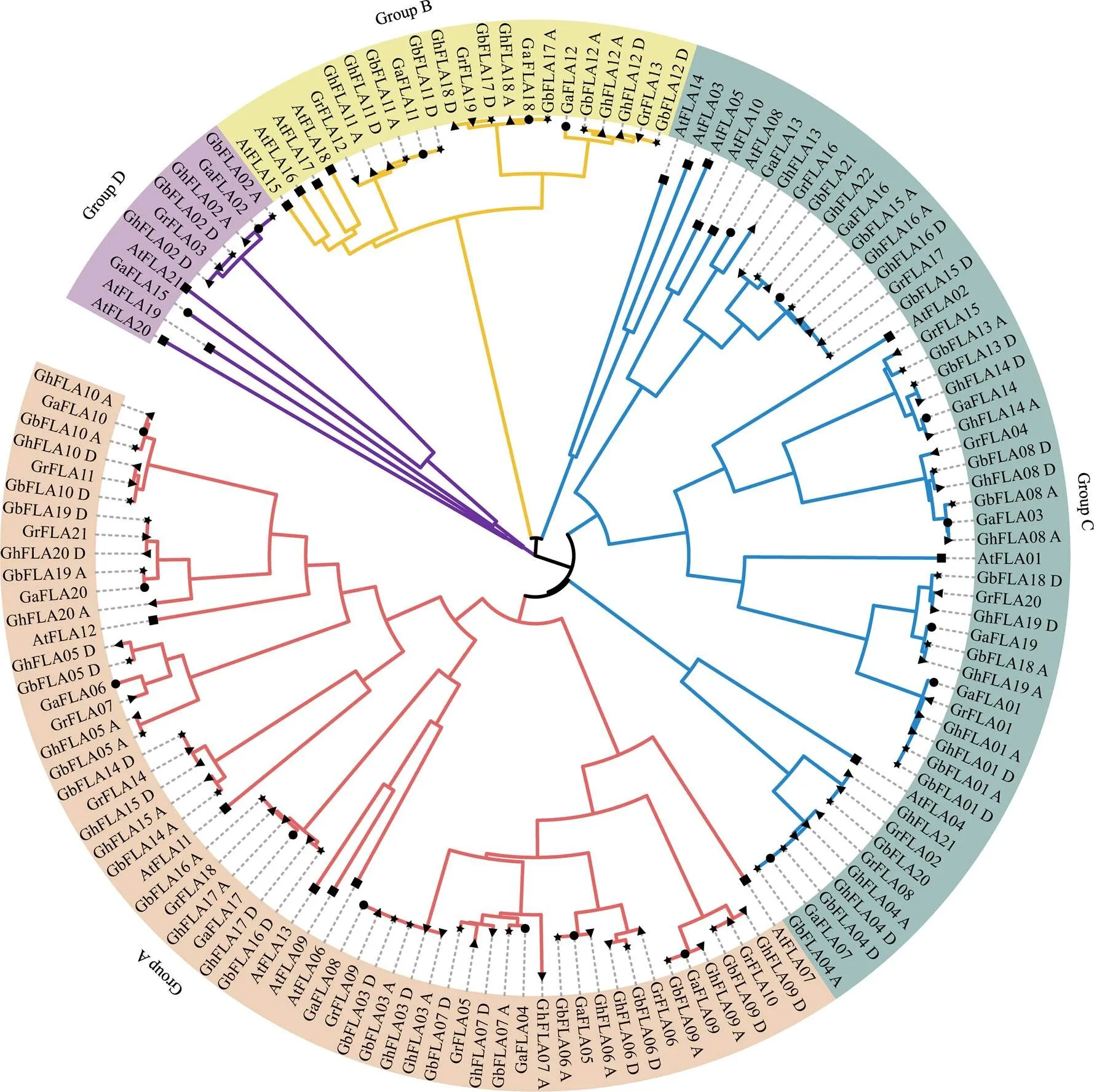
图2 拟南芥和棉花FLA蛋白的系统进化树
2.4 陆地棉FLA基因的基因结构分析
外显子-内含子排列方式可能在基因家族的多样化过程中发挥重要作用。通过分析陆地棉FLA基因外显子和内含子的排布情况(图4-A)。发现与很多作物FLA基因相似,主要分为2种序列分布方式:一是仅有1个外显子;二是有2个外显子和1个内含子。Group A中1个基因、Group B中7个基因、Group C中4个基因的外显子被中间一段内含子分隔,其他多数成员均只有1个外显子。针对Group A中只有存在内含子,通过与Cottongen数据库中TM-1标准系其他版本参考基因组(WHU v1、HAU v1)比对,发现该基因在这两个参考基因组中的ID分别为和,与D组对应基因长度相同,不包含内含子,编码263 aa,推测基因结构中前210 bp的CDS序列及内含子区域不属于该基因编码序列(图4),需要矫正去掉。
进一步寻找陆地棉FLA蛋白的特异性区域,使用在线工具MEME预测其保守基序,鉴定了10个不同的基序,每个基序长度从21到50 aa不等,GhFLAs的基序数目从4到9不等(图4-B)。不同群组包含的保守基序具有特异性,其中,Motif 2隶属于FAS结构域,在所有GhFLAs中都存在。除此之外,Group A还包含Motif 1、Motif 3、Motif 4、Motif 5和Motif 9;Group B还包含Motif 1、Motif 4、Motif 6、Motif 7和Motif 9,其中,Motif 7为该组特有且保守性极高;Group C包含9个Motif,数量最多,Motif 10是该组中特有;Group D包含的Motif最少,仅有4个。以上结果表明,同组蛋白组成具有相同或相似的结构特征,特别是Group B、Group D成员具有完全相同的基序,Group A、Group C只在个别Motif上有差异,推测同一群组中的GhFLA蛋白可能在依赖于结构域/基序的生化等功能上具有保守性。
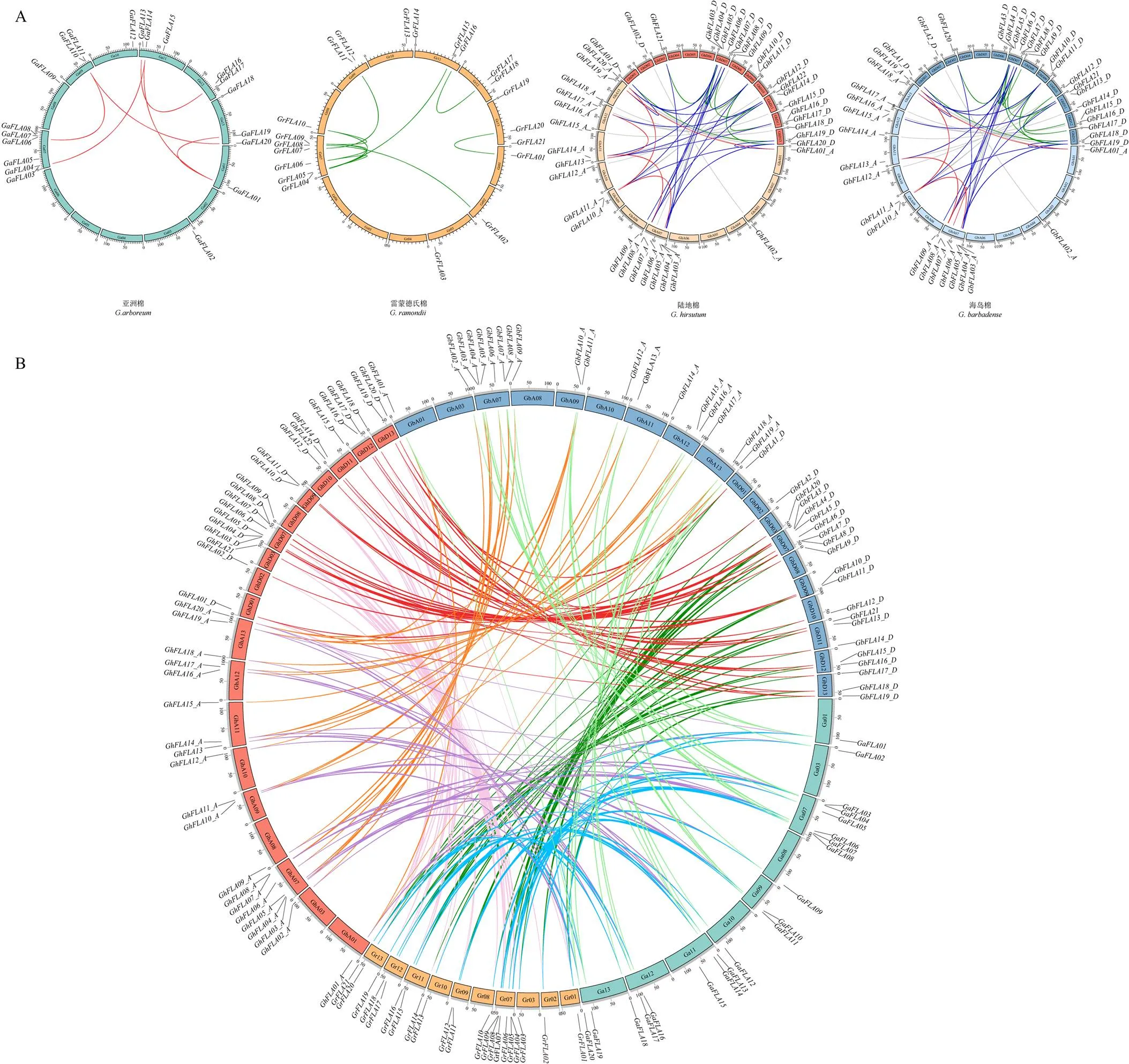
A:4个棉种基因组内FLA基因定位及共线性关系;B:不同棉种棉花基因组之间的FLA共线性关系
此外,通过保守基序分析,发现GhFLA13(GH_A11G0846)的氨基酸长度为234 aa,Motif结构与同组其他序列不同(图4-B),缺少Motif 3、Motif 4、Motif 6和Motif 10。因此,调取该基因前后各5 000 bp序列,利用Softberry软件进行预测矫正,预测该蛋白全长应为366 aa(表1),补充该基因序列信息后,基因全长由704 bp增加为1 959 bp,外显子区域由1个变为2个(图4-B),CDS长度由704 bp变为1 101 bp。但与该组其他基因相比,GhFLA13在矫正预测后仍然有Motif 3和Motif 4的缺失。
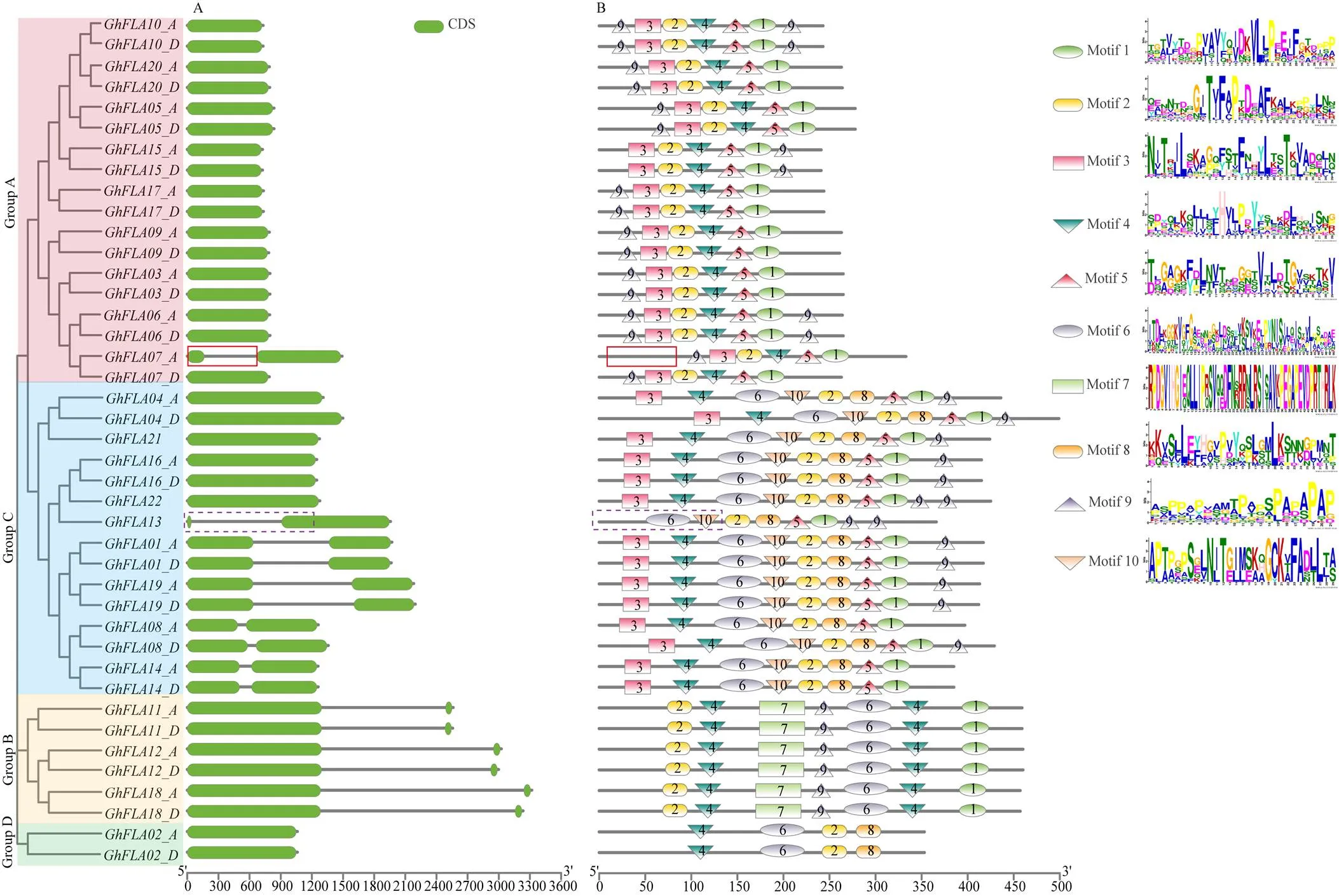
实线矩形框选部分为基因矫正后删除的序列结构,虚线矩形框选部分为基因矫正后添加的序列结构;CDS:编码区序列
2.5 陆地棉FLA蛋白结构域分析
FLA蛋白通常具有典型的AGP-like糖基化区域、FAS结构域,且多数FLA基因具有N端SP和(或)C末端GPI锚定信号。AGP-like和FAS结构域与细胞黏附、胞间作用和细胞壁生物合成有关,SP和GPI可分别促使FLA分子进入内质网腔和锚定到细胞膜。将陆地棉FLA蛋白序列按群组分析其典型结构域(图5),挖掘不同群组的差异性特征。Group A成员均含有1个FAS结构域,两侧各有1个AGP-like糖基化区域,且至少在N端有SP或在C端有GPI锚定信号;Group B的FLA蛋白有2个FAS结构域,中间有1个AGP-like糖基化区域,N端均有SP,C端无GPI锚定信号;Group C为2个AGP-like糖基化区域中间有1个FAS结构域或2个FAS结构域的一侧有一个AGP-like糖基化区域,Group A和Group C具有较相似的结构,但蛋白总长度和结构域的位置具有较大差异,因此,未归为一类;Group D的FLA蛋白仅包含1个AGP-like糖基化区域和一个FAS结构域,且距离较远,N端无SP,C端也无GPI锚定信号。FAS结构域长度分为2种:一种是较短的92—107 aa;另一种为较长的131—139 aa,其中,包含2个FAS结构域的8个蛋白均包含了这两种类型。AGP-like糖基化区域则是5—49 aa长度不等,在Group B和Group D中比较一致,而在另外2组无明显规律。37个(90.24%)GhFLAs蛋白含有SP,25个(60.98%)GhFLAs蛋白含有C端GPI锚定信号,5个(12.20%)GhFLAs蛋白信号肽SP和GPI锚定信号均不含。
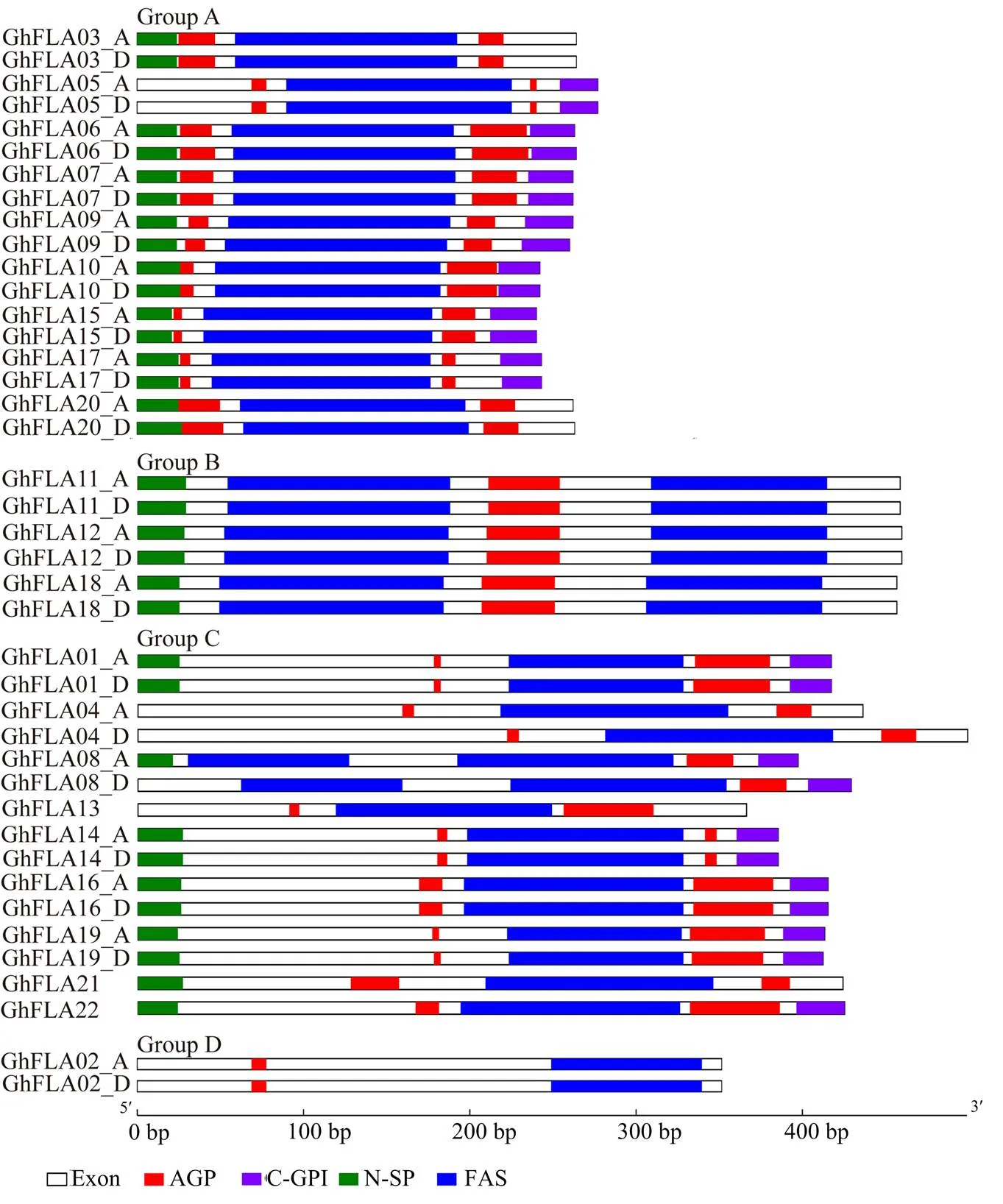
Exon:外显子,AGP:AGP-like糖基化区域,C-GPI:C末端糖基化磷脂酰肌醇锚定信号,N-SP:N端信号肽,FAS:类成束蛋白结构域
提取GhFLAs的FAS结构域序列进行比对(图6),结果显示,该结构域具有2个高度保守的区域H1和H2,并在两区域间存在[Tyr-Phe]His([Y/F]H)基序。与拟南芥、杨树、黄麻等多种作物一样,GhFLA的H1区域Thr(五角星标注)是绝对保守的,Thr后的第6个氨基酸残基大部分为Asp(D),少部分为Asn(N),H1区域其余氨基酸残基主要由Ala(A)、Ile(I)、Phe(F)、Pro(P)和Val(V)组成。H2区域含有较多保守的疏水性氨基酸,如Val(V)、Ala(A)、Leu(L)和Ile(I),其他部分具有相对保守的特点。在H1和H2区域之间,还有一个绝对保守的氨基酸残基Leu(L)(五角星标注),该位点在上述提到的其他物种中不存在如此高的保守性。陆地棉FLA的[Y/F]H基序保守性也较高,只有GhFLA01_ A/D、GhFLA08_A/D、GhFLA19_A/D这三组蛋白在此基序中His(H)变异为Phe(F)。GhFLA蛋白在FAS结构域的这三个区域均具有相对较高的保守性,同时在其他氨基酸位点,不同群组间具有明显的特异性,暗示不同群组FLA蛋白可能从相同的祖先基因进化而来,随着在不同物种中的分化功能可能不同。
2.6 GhFLAs的亚细胞定位分析
为进一步研究陆地棉FLA家族蛋白的功能,在每个群组随机选择一个基因,进行烟草叶片瞬时表达,初步分析目的蛋白表达位置(图7)。结果表明,GhFLA05_D为Group A成员,融合蛋白荧光分析其可能定位在细胞质内的内质网,并呈现聚集状颗粒;GhFLA18_A为Group B成员,可能位于细胞核和细胞膜/壁,同时在细胞质中可能也有表达;GhFLA22为Group C成员,较广泛的表达于细胞膜/壁、细胞质与细胞核;GhFLA02A/D是Group D的唯一成员,但由于其在各组织中表达量较低(图7-A),利用棉花各组织cDNA均未克隆出该基因,因此,未能进行亚细胞定位。

图6 陆地棉FLAs蛋白FAS结构域的多序列比对
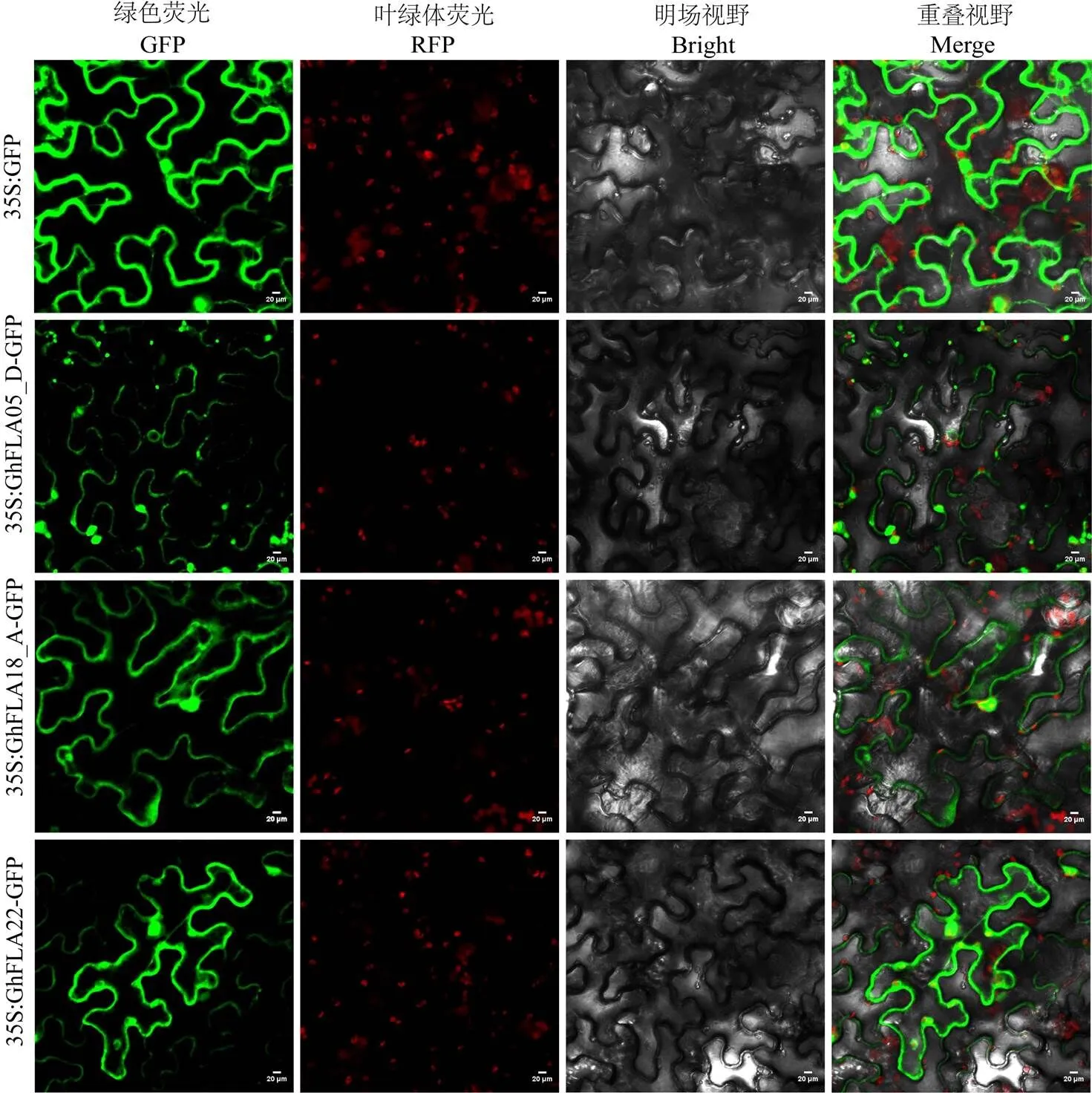
图7 陆地棉不同群组FLAs蛋白的亚细胞定位
2.7 GhFLAs的组织特异性表达分析
为进一步分析陆地棉FLA基因在棉花纤维发育进程中的作用,利用转录组数据对41个在陆地棉TM-1根、茎、叶、苞叶、花托、萼片、花瓣、雄蕊、花丝、花药等10个组织和不同发育时期胚珠、纤维的表达进行分析(图8)。在4个群组中,大部分Group A、Group B的基因在纤维发育期较高或高表达,其中,、、、自纤维发育伸长期(3 DPA)已开始高表达,而、、、、、、主要在次生壁增厚期(15—25 DPA)高表达,在纤维发育起始阶段(-3—1 DPA)相对较高表达,只有在纤维发育时期无特异表达;其他2组中,除在纤维起始、伸长时期(-3—10 DPA)相对高表达,其他基因在纤维发育时期无明显特异性表达。
2.8 GhFLAs在不同材料纤维中的表达分析
在家族的4个亚群中,Group A、Group B的基因主要在纤维发育期较高或高表达。因此,利用纤维比强度和马克隆值性状稳定分离的2个家系RIL131和RIL229(图9-A),重点分析这两个亚群成员在不同材料纤维中的基因表达情况(图9-B)。因在A、D亚组对应拷贝CDS区碱基差异较少,在qRT-PCR表达量检测种中无法严格区分,所以不再标注A、D区分。结果表明,两亚群成员在不同品质的RIL131和RIL229纤维中总体具有相似的表达模式,并与TM-1转录组测序结果(图8)一致。除在10—15 DPA相对较高表达,其他基因主要在SCW发育阶段、尤其是20—25 DPA优势表达;2个材料的20 DPA和25 DPA纤维中,、均极显著差异表达,均显著差异表达,且3个基因中,在SCW发育阶段有较RIL229的20 DPA纤高的优势表达,并在高比强材料纤维中的表达量最高,且极显著高于同时期低比强材料RIL131,推测可能在调控纤维比强度中发挥作用。

图8 陆地棉FLA基因在不同组织、不同发育时期胚珠和纤维中的表达分析

RIL131和RIL229:前期构建的RIL群体中纤维品质性状稳定分离的2个家系,*:差异显著(p<0.05),**:差异极显著(p<0.01),n.s.:无显著差异。下同
2.9 GhFLA05的克隆和VIGS技术功能验证
在RIL131和RIL229中分别克隆和,经比对,2个基因在2个材料中的序列一致,与参考基因组序列相同,无SNP差异,表明该组基因相对保守。和的CDS全长均为837 bp,2个基因间有21个SNP,序列相似性97.49%,造成14个氨基酸差异(电子附图2)。
利用VIGS技术对功能进行验证。以RIL229为材料,以为基因沉默对象,通过阳性对照组棉株的白化症状初步判断沉默效果,取阴性对照和:植株20 DPA纤维,利用qRT-PCR检测基因沉默效率。选择家族中与其同源性最高的、(CDS比对相似性为55.02%—60.98%)(电子附图2),分析基因沉默的特异性。结果(图10-A)表明,阳性对照出现的白化症状会持续到棉铃发育时期,使棉铃苞叶、萼片、铃壳等均出现白化现象,说明该病毒在棉铃发育时期仍具有基因沉默作用。和植株在注射后的生长外部形态上与野生型(WT)无明显差异。qRT-PCR结果(图10-B)显示,部分株系沉默效率为35.82%—68.60%,平均沉默效率53.19%,而对同源性最高的家族成员、的表达无显著影响。纤维品质检测结果(图10-C)中,与阴性对照相比,棉纤维断裂比强度显著降低,马克隆值有所降低但差异不显著,其他3个性状差异均不显著。上述结果表明,沉默使棉花纤维断裂比强度下降,同时可能对马克隆值也有一定影响,暗示可能主要在棉花纤维发育中后期尤其是SCW加厚期具有调控作用。

图10 GhFLA05沉默后植株表型(A)、在纤维中基因表达分析(B)及纤维品质性状测定(C)
3 讨论
3.1 棉花FLA基因家族的鉴定
近年来,棉花基因组学研究飞速发展,基因组数据不断完善[58],大大拓宽了育种者利用基因调控网络提高育种效率的思路,而基于基因家族分析快速挖掘功能基因是丰富基因调控网络的有效手段。FLA蛋白是一类广泛分布于植物体内的富含羟脯氨酸的糖蛋白,在植物生长、形态构建等发育过程发挥着重要作用[59]。通过在全基因组水平系统的鉴定分析FLA蛋白,为更好地利用FLA蛋白奠定基础。前人在拟南芥中鉴定出21个FLA蛋白[18],随后陆续从小麦、水稻、杨树等多种植物鉴定得到了FLA基因家族[19-26]。本研究在陆地棉、海岛棉、亚洲棉和雷蒙德氏棉中共鉴定出122个FLA家族成员,其中,陆地棉41个。Huang等[3]曾基于棉花纤维cDNA库鉴定和分析了19个陆地棉FLA基因,经比对,发现17个与本研究相同,而其鉴定的()和()在本研究中未纳入该家族,主要因为该研究中的检测评分较低,且在Smart验证中未检测出FAS结构域;的检测阈值未达到本研究基因家族要求。
通过进化树分析,棉花FLA蛋白与拟南芥[18]及杨树[21]、黄麻[25]、纺织大麻[14]、甘蓝[26]等其他作物相同,均可分为4组,每组的FLA具有相似的基因结构、基序和保守域。共线性分析显示陆地棉的FLA基因比海岛棉与二倍体棉种具有更多的同源基因对,推测在进化过程中海岛棉的一些FLA基因序列结构可能产生更多的变异,并可能在形成优异纤维品质方面发挥作用。对陆地棉FLA蛋白的结构域分析,41个成员均具有1—2个AGP-like糖基化区域和1—2个FAS结构域,多数含有SP和(或)GPI锚定信号。在陆地棉FAS结构域的研究中发现,除了2个高度保守的区域H1、H2和两区域之间的[Y/F]H基序这三段相对保守序列,与其他作物不同的是,在H1和[Y/F]H基序之间,有一个绝对保守的氨基酸残基Leu,这在拟南芥[18]、杨树[13]、香蕉[24]和黄麻[25]等物种FAS结构域序列比对中并不存在,是棉花的特异保守位点。由于FLA特殊保守结构域及SP信号肽、GPI锚定信号的存在,FLA经常被报道定位在内质网或细胞膜。Huang等[2]研究表明(本研究中的基因名称为,下同)主要定位在细胞壁上,且在2个细胞连接处信号很强,可能这个蛋白对细胞间信息的交流有作用,而()可能定位于细胞壁和细胞质膜上,并可能分泌到细胞外。烟草和均只定位于细胞膜中[58]。本研究通过对进化树不同组中3个蛋白的定位发现,FLA蛋白的表达范围可能较广,不止在细胞膜部位,相似的结果也在其他一些研究中有过报道,如杨树、、、广泛分布在细胞质和细胞膜,其中,、在细胞质也呈颗粒状亮点分布[13, 30],烟草存在于细胞膜和细胞核[13, 58]。
此外,通过同一组内蛋白共性特征分析,可以发现结构异常的基因,并对可能存在问题的基因进行矫正。本研究通过对陆地棉FLA基因的结构分析,发现本研究参考基因组中多了210 bp CDS序列及490 bp内含子区域,经过与其他版本参考基因组比对分析进行矫正。通过对陆地棉FLA蛋白的保守基序分析发现,() Motif结构与同组其他序列相比,缺少部分Motif基序,因此,调取该基因上下游序列,进行预测矫正,但其在矫正后仍有Motif 3和Motif 4的缺失,同时发现,在陆地棉中D亚组无对应拷贝,在整个基因组内无同源基因,说明与家族中通常成对出现的其他成员相比,该基因在序列和结构上可能存在特异性,其具体序列结构仍需进一步验证完善。通过对家族成员基因结构的分析,发现异常的序列进行预测矫正,对深入完善基因组信息具有一定意义。
3.2 对FLA基因家族中纤维发育相关基因的鉴定
棉花FLA的保守结构域与拟南芥[18]、杨树[21]及黄麻[25]等物种FLA蛋白相同,推测生理功能可能相似。据研究,可能参与拟南芥根伸长[27],杨树调控木质部纤维生长[30],桉树和与纤维素微纤丝角和茎秆强度相关[22],大麻中多个FLA基因参与韧皮纤维细胞的起始、伸长和SCW的生物合成[14]。以上不同作物的研究均表明在植物木质部特异表达或与细胞壁发育相关。本研究通过NCBI的SRA数据库获得转录组数据,分析陆地棉在不同组织、不同发育时期胚珠和纤维中的表达情况,发现与纤维伸长、SCW加厚紧密关联的FLA基因主要分布在Group A和Group B,暗示这两组的FLA基因可能在陆地棉纤维发育中起到重要作用。在棉花中,该家族基因鉴定出、和(、和)在10 DPA棉纤维中优势表达[3],()对纤维伸长具有正调控作用[5];()和()在棉花5—20 DPA纤维中大量积累,()在纤维中大量积累[4],以上鉴定的FLA基因与纤维起始、伸长及SCW成熟密切相关,与本研究转录组及基因定量表达分析鉴定结果一致,验证了本研究对FLA基因家族中棉纤维发育相关基因表达分析的准确性。
3.3 GhFLA05在纤维SCW合成期具有重要作用
前人分离克隆获得海岛棉,该基因CDS区为720 bp,编码239个氨基酸,经比对,与本研究(CDS全长837 bp)为同一基因,研究发现此基因在海岛棉纤维SCW沉积阶段表达量显著高于陆地棉[29],对海岛棉形成高强纤维起重要作用。有研究分析(和)和(和)分别是拟南芥和在棉花中的直系同源基因[60],敲除和导致茎秆强度和弹性变弱[28]。本研究构建的进化树显示与为直系同源关系,与不是直系同源关系但在较近的分枝上,表明可能在陆地棉中也具有类似的功能。因此,本研究确定以陆地棉中未鉴定报道过的为对象,以陆地棉中纤维断裂比强度和马克隆值性状差异显著的RIL131和RIL229为试验材料,克隆并分析其在不同发育时期棉纤维中的表达情况。和分别在2个材料中序列一致,与参考基因组序列相同,无SNP差异,表明该组基因相对保守,而在SCW合成期(15—25 DPA)均优势表达并表现显著差异,利用VIGS技术沉默后,棉纤维断裂比强度显著降低。根据以上研究结果分析是一个纤维SCW合成期相关的基因,尤其与陆地棉纤维品质中纤维强度密切相关。陆地棉与海岛棉[29]、陆地棉不同材料间该基因具有差异表达,并调控纤维强度,但其编码区序列在不同材料间保守,推测基因差异表达的主要原因不是由基因编码序列本身造成,而主要受上游基因调控影响,进一步推测在海岛棉和高比强的陆地棉RIL229中可能有相似的基因网络调控模式,使所在调控通路通过提前SCW加厚的起始时间,延长了SCW加厚期的累积时长,有效促进纤维素合成,从而形成较高的棉纤维比强度。但该基因具体参与的调控途径、作用方式和调控机制还有待进一步研究,本研究下步计划:一方面通过对不同材料中基因启动子调控区域分析,明确上游互作蛋白,分析差异表达原因;另一方面通过过表达和基因敲除进一步明确功能,解析其在棉纤维发育中所处的基因调控网络及调控模式。
4 结论
在四倍体陆地棉、海岛棉和二倍体亚洲棉、雷蒙德氏棉中分别鉴定出了41、40、20和21个FLA家族成员,可分为4个群组,不同群组成员间具有较高的结构和功能相似性,并从中筛选了在纤维发育特定时期发挥作用的基因,明确家族中是SCW合成阶段特异性表达基因,并与陆地棉不同材料纤维比强度差异形成密切相关。
[1] WANG M J, LI J Y, QI Z Y, LONG Y X, PEI L L, HUANG X H, GROVER C E, DU X M, XIA C J, WANG P C, LIU Z P, YOU J Q, TIAN X H, MA Y Z, WANG R P, CHEN X Y, HE X, FANG D D, Sun Y Q, Tu L L, JIN S X, ZHU L F, WENDEL J F, ZHANG X L. Genomic innovation and regulatory rewiring during evolution of the cotton genus. Nature Genetics, 2022, 54(12): 1959-1971.
[2] CHEN Q, WANG W, WANG C X, ZHANG M, YU J W, ZHANG Y F, YUAN B T, DING Y Y, JONES D C, PATERSON A H, CHEE P W, WANG B H. Validation of QTLs for fiber quality introgressed fromby selective genotyping. G3 Genes |Genomes|Genetics, 2020, 10(7): 2377-2384.
[3] HUANG G Q, XU W L, GONG S Y, LI B, WANG X L, XU D, LI X B. Characterization of 19 novel cottongenes and their expression profiling in fiber development and in response to phytohormones and salt stress. Physiologia Plantarum, 2008, 134(2): 348-359.
[4] LIU D Q, TU L L, LI Y J, WANG L, ZHU L F, ZHANG X L. Genes encoding fasciclin-like arabinogalactan proteins are specifically expressed during cotton fiber development. Plant Molecular Biology Reporter, 2008, 26(2): 98-113.
[5] HUANG G Q, GONG S Y, XU W L, LI W, LI P, ZHANG C J, LI D D, ZHENG Y, LI F G, LI X B. A fasciclin-like arabinogalactan protein,, is involved in fiber initiation and elongation of cotton. Plant Physiology, 2013, 161(3): 1278-1290.
[6] MA J J, JIANG Y F, PEI W F, WU M, MA Q F, LIU J, SONG J K, JIA B, LIU S, WU J Y, ZHANG J F, YU J W. Expressed genes and their new alleles identification during fibre elongation reveal the genetic factors underlying improvements of fibre length in cotton. Plant Biotechnology Journal, 2022, 20(10): 1940-1955.
[7] 王雅琴,李艳军, 张新宇, 刘永昌, 石淼, 孙杰. 棉花基因的克隆及表达分析. 新疆农业科学, 2013, 50(5): 785-793.
WANG Y Q, LI Y J, ZHANG X Y, LIU Y C, SHI M, SUN J. Cloning and expression analysis of cottongene. Xinjiang Agricultural Sciences, 2013, 50(5): 785-793. (in Chinese)
[8] 胡海燕, 刘迪秋, 李允静, 李阳, 涂礼莉. 一个棉花纤维伸长期优势表达启动子pGhFLA1的克隆与鉴定. 作物学报, 2017, 43(6): 849-854.
Hu H Y, LIU D Q, LI Y J, LI Y, TU L L. Identification of promoter GhFLA1 preferentially expressed during cotton fiber elongation. Acta Agronomica Sinica, 2017, 43(6): 849-854. (in Chinese)
[9] TAN L, SHOWALTER A M, EGELUND J, HERNANDEZ- SANCHEZ A, DOBLIN M S, BACIC A. Arabinogalactan-proteins and the research challenges for these enigmatic plant cell surface proteoglycans. Frontiers in Plant Science, 2012, 3: 140.
[10] SEIFERT G J, ROBERTS K. The biology of arabinogalactan proteins. Annual Review of Plant Biology, 2007, 58: 137-161.
[11] SHOWALTER A M, KEPPLER B, LICHTENBERG J, GU D Z, WELCH L R. A bioinformatics approach to the identification, classification, and analysis of hydroxyproline-rich glycoproteins. Plant Physiology, 2010, 153(2): 485-513.
[12] XU F, CHEN Q, HUANG L, LUO M. Advances about the roles of membranes in cotton fiber development. Membranes, 2021, 11(7): 471.
[13] ZANG L N, ZHENG T C, CHU Y G, DING C J, ZHANG W X, HUANG Q J, SU X H. Genome-wide analysis of the fasciclin-like arabinogalactan protein gene family reveals differential expression patterns, localization, and salt stress response in. Frontiers in Plant Science, 2015, 6: 1140.
[14] GUERRIERO G, MANGEOT-PETER L, LEGAY S, BEHR M, LUTTS S, SIDDIQUI K S, HAUSMAN J F. Identification of fasciclin-like arabinogalactan proteins in textile hemp (L.): in silico analyses and gene expression patterns in different tissues. BMC Genomics, 2017, 18(1): 741.
[15] TAN L, LEYKAM J F, KIELISZEWSKI M J. Glycosylation motifs that direct arabinogalactan addition to arabinogalactan-proteins. Plant Physiology, 2003, 132(3): 1362-1369.
[16] HE J D, ZHAO H, CHENG Z L, KE Y W, LIU J X, MA H L. Evolution analysis of the fasciclin-like arabinogalactan proteins in plants shows variable fasciclin-AGP domain constitutions. International Journal of Molecular Sciences, 2019, 20(8): 1945.
[17] KIM D, LANGMEAD B, SALZBERG S L. HISAT: a fast spliced aligner with low memory requirements. Nature Methods, 2015, 12(4): 357-360.
[18] JOHNSON K L, JONES B J, BACIC A, SCHULTZ C J. The fasciclin-like arabinogalactan proteins of. A multigene family of putative cell adhesion molecules. Plant Physiology, 2003, 133(4): 1911-1925.
[19] MA H L, ZHAO J. Genome-wide identification, classification, and expression analysis of the arabinogalactan protein gene family in rice (L.). Journal of Experimental Botany, 2010, 61(10): 2647-2668.
[20] FAIK A, ABOUZOUHAIR J, SARHAN F. Putative fasciclin-like arabinogalactan-proteins (FLA) in wheat () and rice (): identification and bioinformatic analyses. Molecular Genetics and Genomics, 2007, 277(1): 97.
[21] SHOWALTER A M, KEPPLER B D, LIU X, LICHTENBERG J, WELCH L R. Bioinformatic identification and analysis of hydroxyproline-rich glycoproteins in. BMC Plant Biology, 2016, 16(1): 229.
[22] MACMILLAN C P, TAYLOR L, BI Y D, SOUTHERTON S G, EVANS R, SPOKEVICIUS A. The fasciclin-like arabinogalactan protein family ofcontains members that impact wood biology and biomechanics. The New Phytologist, 2015, 206(4): 1314-1327.
[23] LI X Q, CHENG M Y, TANG C R, ZHU X X, QI K, ZHANG S L, WU J Y, WANG P. Identification and function analysis of fasciclin-like arabinogalactan protein family genes in pear (). Plant Systematics and Evolution, 2021, 307: 48.
[24] MENG J, HU B, YI G J, LI X Q, CHEN H B, WANG Y Y, YUAN W N, XING Y Q, SHENG Q M, SU Z X, XU C X. Genome-wide analyses of banana fasciclin-likegenes and their differential expression under low-temperature stress in chilling sensitive and tolerant cultivars. Plant Cell Reports, 2020, 39(6): 693-708.
[25] HOSSAIN M S, AHMED B, ULLAH M W, AKTAR N, HAQUE M S, ISLAM M S. Genome-wide identification of fasciclin-like arabinogalactan proteins in jute and their expression pattern during fiber formation. Molecular Biology Reports, 2020, 47(10): 7815-7829.
[26] LI J, WU X M. Genome-wide identification, classification and expression analysis of genes encoding putative fasciclin-like arabinogalactan proteins in chinese cabbage (L.). Molecular Biology Reports, 2012, 39(12): 10541-10555.
[27] AALLELIGN SHAGRE H, ZALTZMAN D, IDAN-MOLAKANDOV A, ROMANO H, TZFADIA O, HARPAZ-SAAD S. FASCICLIN-LIKE 18 is a new player regulating root elongation in. Frontiers in Plant Science, 2021, 12: 645286.
[28] MACMILLAN C P, MANSFIELD S D, STACHURSKI Z H, EVANS R, SOUTHERTON S G. Fasciclin-like arabinogalactan proteins: specialization for stem biomechanics and cell wall architecture in Arabidopsis and. The Plant Journal, 2010, 62(4): 689-703.
[29] SHI H Z, KIM Y, GUO Y, STEVENSON B, ZHU J K. Thelocus encodes a putative cell surface adhesion protein and is required for normal cell expansion. The Plant Cell, 2003, 15(1): 19-32.
[30] WANG H H, JIANG C M, WANG C T, YANG Y, YANG L, GAO X Y, ZHANG H X. Antisense expression of the fasciclin-like arabinogalactan proteingene ininhibits expression of its homologous genes and alters stem biomechanics and cell wall composition in transgenic trees. Journal of Experimental Botany, 2015, 66(5): 1291-1302.
[31] LIU H W, SHI R F, WANG X F, PAN Y X, LI Z K, YANG X L, ZHANG G Y, MA Z Y. Characterization and expression analysis of a fiber differentially expressed fasciclin-like arabinogalactan protein gene in sea island cotton fibers. PloS ONE, 2013, 8(7): e70185.
[32] MAJEWSKA-SAWKA A, NOTHNAGEL E A. The multiple roles of arabinogalactan proteins in plant development. Plant Physiology, 2000, 122(1): 3-10.
[33] XUE H, VEIT C, ABAS L, TRYFONA T, MARESCH D, RICARDI M M, ESTEVEZ J M, STRASSER R, SEIFERT G J.functions as a glycan-stabilized soluble factor via its carboxy-proximal Fasciclin 1 domain. The Plant Journal, 2017, 91(4): 613-630.
[34] ZHANG M, WEI H L, LIU J, BIAN Y J, MA Q, MAO G Z, WANG H T, WU A M, ZHANG J J, CHEN P Y, MA L, FU X K, YU S X. Non-functionalare responsible for the male sterility caused by hybrid breakdown in cotton (spp.). The Plant Journal, 2021, 107(4): 1198-1212.
[35] LI J, YU M, GENG L L, ZHAO J. The fasciclin-like arabinogalactan protein gene,, is involved in microspore development of. The Plant Journal, 2010, 64(3): 482-497.
[36] ZHANG Z Y, XIN W W, WANG S F, ZHANG X, DAI H F, SUN R R, FRAZIER T, ZHANG B H, WANG Q L. Xylem sap in cotton contains proteins that contribute to environmental stress response and cell wall development. Functional & Integrative Genomics, 2015, 15(1): 17-26.
[37] TAKAHASHI D, KAWAMURA Y, UEMURA M. Cold acclimation is accompanied by complex responses of glycosylphosphatidylinositol (GPI)-anchored proteins in. Journal of Experimental Botany, 2016, 67(17): 5203-5215.
[38] SEIFERT G J, XUE H, ACET T. Thefasciclin like arabinogalactan protein 4gene acts synergistically with abscisic acid signalling to control root growth. Annals of Botany, 2014, 114(6): 1125-1133.
[39] 张素君, 周晓栋, 唐丽媛, 李兴河, 王海涛, 刘存敬, 蔡肖, 张香云, 张建宏. 杂交棉‘冀1518’纤维品质性状的QTL定位及遗传分析. 分子植物育种, 2021, 19(11): 3627-3637.
ZHANG S J, ZHOU X D, TANG L Y, LI X H, WANG H T, LIU C J, CAI X, ZHANG X Y, ZHANG J H. QTL Mapping and genetic analysis of fiber quality traits in hybrid cotton ‘Ji1518’. Molecular Plant Breeding, 2021, 19(11): 3627-3637. (in Chinese)
[40] HU Y, CHEN J D, FANG L, ZHANG Z Y, MA W, NIU Y C, JU L Z, DENG J Q, ZHAO T, LIAN J M, BARUCH K, FANG D, LIU X, RUAN Y L, RAHMAN M U, HAN J L, WANG K, WANG Q, WU H T, MEI G F, ZANG Y H, HAN Z G, XU C Y, SHEN W J, YANG D F, SI Z F, DAI F, ZOU L F, HUANG F, BAI Y L, ZHANG Y G, BRODT A, BEN-HAMO H, ZHU X F, ZHOU B L, GUAN X Y, ZHU S J, CHEN X Y, ZHANG T Z.andgenomes provide insights into the origin and evolution of allotetraploid cotton. Nature Genetics, 2019, 51(4): 739-748.
[41] WANG M J, LI J Y, WANG P C, LIU F, LIU Z P, ZHAO G N, XU Z P, PEI L L, GROVER C E, WENDEL J F, WANG K B, ZHANG X L. Comparative genome analyses highlight transposon-mediated genome expansion and the evolutionary architecture of 3D genomic folding in cotton. Molecular biology and evolution, 2021, 38(9): 3621-3636.
[42] FINN R D. Pfam: clans, web tools and services. Nucleic Acids Research, 2006, 34(90001): D247-D251.
[43] FINN R D, CLEMENTS J, EDDY S R. HMMER web server: interactive sequence similarity searching. Nucleic Acids Research, 2011, 39(suppl_2): W29-W37.
[44] LETUNIC I, DOERKS T, BORK P. SMART 7: recent updates to the protein domain annotation resource. Nucleic Acids Research, 2012, 40(D1): D302-D305.
[45] LU S N, WANG J Y, CHITSAZ F, DERBYSHIRE M K, GEER R C, GONZALES N R, GWADZ M, HURWITZ D I, MARCHLER G H, SONG J S, THANKI N, YAMASHITA R A, YANG M Z, ZHANG D C, ZHENG C J, LANCZYCKI C J, MARCHLER-BAUER A. CDD/SPARCLE: the conserved domain database in 2020. Nucleic Acids Research, 2020, 48(D1): D265-D268.
[46] GASTEIGER E, HOOGLAND C, GATTIKER A, DUVAUD S, WILKINS M R, APPEL R D, BAIROCH A. Protein Identification and Analysis Tools on the Expasy Server. The Proteomics Protocols Handbook, Humana Press, 2005: 571-607.
[47] EDDY S R. Profile hidden Markov models. Bioinformatics, 1998, 14(9): 755-763.
[48] KUMAR S, STECHER G, TAMURA K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution, 2016, 33(7): 1870-1874.
[49] LETUNIC I, BORK P. Interactive Tree Of Life (iTOL) v5: an online tool for phylogenetic tree display and annotation. Nucleic Acids Research, 2021, 49(W1): W293-W296.
[50] WANG Y P, TANG H B, DEBARRY J D, TAN X, LI J P, WANG X Y, LEE T H, JIN H Z, MARLER B, GUO H, KISSINGER J C, PATERSON A H. MCScanX: a toolkit for detection and evolutionary analysis of gene synteny and collinearity. Nucleic Acids Research, 2012, 40(7): e49,
[51] CHEN C J, CHEN H, ZHANG Y, THOMAS H R, FRANK M H, HE Y H, XIA R. TBtools: an integrative toolkit developed for interactive analyses of big biological data. Molecular Plant, 2020, 13(8): 1194-1202.
[52] BAILEY T L, JOHNSON J, GRANT C E, NOBLE W S. The MEME suite. Nucleic Acids Research, 2015, 43(W1): W39-W49.
[53] HU B, JIN J P, GUO A Y, ZHANG H, LUO J C, GAO G. GSDS 2.0: an upgraded gene feature visualization server. Bioinformatics, 2015, 31(8): 1296-1297.
[54] SCHULTZ C J, RUMSEWICZ M P, JOHNSON K L, JONES B J, GASPAR Y M, BACIC A. Using genomic resources to guide research directions. the arabinogalactan protein gene family as a test case. Plant Physiology, 2002, 129(4): 1448-1463.
[55] GU Z H, HUANG C J, LI F F, ZHOU X P. A versatile system for functional analysis of genes and microRNAs in cotton. Plant Biotechnology Journal, 2014, 12(5): 638-649.
[56] TIAN Z L, ZHANG Y Z, ZHU L P, JIANG B, WANG H Q, GAO R X, FRIML J, XIAO G H. Strigolactones act downstream of gibberellins to regulate fiber cell elongation and cell wall thickness in cotton ()., 2022, 34(12): 4816-4839.
[57] LIU G Y, LIU J, PEI W F, LI X H, WANG N H, MA J J, ZANG X S, ZHANG J F, YU S X, WU M, YU J W. Analysis of thegene family and the role ofPlanta, 2019, 250(6): 2147-2158.
[58] YANG Z E, GAO C X, ZHANG Y H, YAN Q D, HU W, YANG L, WANG Z, LI F G. Recent progression and future perspectives in cotton genomic breeding. Journal of Integrative Plant Biology, 2023, 65(2): 548-569.
[59] WU X Y, LAI Y C, LV L Q, JI M F, HAN K L, YAN D K, LU Y W, PENG J J, RAO S F, YAN F, ZHENG H Y, CHEN J P. Fasciclin-like arabinogalactan gene family in: genome-wide identification, classification and expression in response to pathogens. BMC Plant Biology, 2020, 20(1): 305.
[60] WANG C, LV Y D, XU W, ZHANG T Z, GUO W Z. Aberrant phenotype and transcriptome expression during fiber cell wall thickening caused by the mutation of the Im gene in immature fiber (im) mutant inL.. BMC genomics, 2014, 15: 94.
Genome-Wide Identification of Cotton FLA Gene Family and Functional Analysis of
TANG LiYuan, CAI Xiao, WANG HaiTao, LI XingHe, ZHANG SuJun, LIU CunJing, ZHANG JianHong
Institute of Cotton, Hebei Academy of Agriculture and Forestry Sciences/Key Laboratory of Cotton Biology and Genetic Breeding in Huanghuaihai Semiarid Area, Ministry of Agriculture and Rural Affairs/National Cotton Improvement Center Hebei Branch, Shijiazhuang 050051
【Background】It is of great importance to improve the quality of cotton fiber to meet the improvement of cotton textile production and the pursue of people for high quality cotton. Fasciclin-like arabinogalactan proteins (FLAs) play an important role in the initial development of cotton fibers and secondary wall synthesis. 【Objective】Comprehensive identification and analysis of cotton FLA gene family members to reveal their common characteristics and specific expression patterns, provided a reference for the function study of FLAs in cotton fiber development.【Method】According to the whole genome data of cotton, members of FLA gene family were identified by HMMER3.0 and further verified by online softwares of Pfam and Smart. Physical and chemical properties and transmembrane domains of these proteins were analyzed by ExPASy and TMHMM. Phylogenetic tree construction, chromosome localization, collinearity analysis and protein conserved domain sequence alignment were conducted and displayed using GSDS, MCScanX, MEGA, MEME, TBtools and Jalview. Expression of FLA genes in different tissues were analyzed by cotton transcriptome data. Expression differences ofin different developmental stages of ovules and fibers between different fiber quality materials was analyzed by quantitative real-time polymerase chain reaction (qRT-PCR). Function ofwas verified by virus induced gene silencing (VIGS). 【Result】A total of 41, 40, 20 and 21 FLA family members were identified in,,and, respectively. The phylogenetic tree showed that cotton FLA proteins could be divided into four groups. Gene structure and motif composition were relatively conserved in each group. Further analysis of FLA proteins inshowed that all 41 FLA members had 1-2 AGP-like glycosylation regions and 1-2 fasciclin-like domains (FAS), 37 of which contained signal peptide (SP) and 25 contained glycosylphosphatidylinositol anchored protein (GPI) anchoring signals. Subcellular localization showed that GhFLA05_D showing aggregated granules in the cytoplasm was probably localized in endoplasmic reticulum, and GhFLA18_A and GhFLA22 were expressed in cell membrane/wall, cytoplasm and nucleus. Transcriptome sequencing results showed that FLA proteins in Group A and B were mainly highly expressed in fibers, which may be involved in the process of cotton fiber elongation development and secondary wall thickening. In general, group A and B members had a similar expression pattern in two materials with significant differences in fiber quality and expressed mainly in the secondary wall development stage, especially in 20-25 DPA period.exhibited specific expression at the secondary wall thickening stage with significant differences between two materials, which expressed with a high maximum value in earlier stage of secondary wall thickening stage in high specific strength material RIL229, suggestingmay take a part in the regulation of cotton fiber strength difference formation. The fiber strength and micronaire value decreased ingene-silenced cotton plants by VIGS.【Conclusion】A sum of 122 FLA family members were identified in,,and, which could be divided into four groups. Members of different groups had high structural and functional similarities, and the genes related to cotton fiber development were identified. It was clarified thatspecifically expressed in the secondary wall synthesis stage, and closely related to the difference in fiber strength of different upland cotton materials.
cotton; FLA; fiber development; gene family; expression analysis

10.3864/j.issn.0578-1752.2023.23.004
2023-02-16;
2023-04-20
河北省农林科学院基本科研业务费(2021070205)、河北省农林科学院科技创新专项课题(2022KJCXZX-MHS-1,2023KJCXZX-MHS-11)
唐丽媛,E-mail:liyuaner05@163.com。通信作者张建宏,E-mail:mhszjh@126.com
(责任编辑 李莉)
