基于深度学习的多模态医学图像分割综述
2023-11-29窦猛陈哲彬王辛周继陶姚宇
窦猛,陈哲彬,王辛,周继陶,姚宇*
基于深度学习的多模态医学图像分割综述
窦猛1,2,陈哲彬1,2,王辛3,周继陶3,姚宇1,2*
(1.中国科学院 成都计算机应用研究所,成都 610213; 2.中国科学院大学 计算机科学与技术学院,北京 100049; 3.四川大学华西医院 腹部肿瘤科,成都 610041)( ∗ 通信作者电子邮箱 Casitmed2022@163.com)
多模态医学图像可以为临床医生提供靶区(如肿瘤、器官或组织)的丰富信息。然而,由于多模态图像之间相互独立且仅有互补性,如何有效融合多模态图像并进行分割仍是亟待解决的问题。传统的图像融合方法难以有效解决此问题,因此基于深度学习的多模态医学图像分割算法得到了广泛的研究。从原理、技术、问题及展望等方面对基于深度学习的多模态医学图像分割任务进行了综述。首先,介绍了深度学习与多模态医学图像分割的一般理论,包括深度学习与卷积神经网络(CNN)的基本原理与发展历程,以及多模态医学图像分割任务的重要性;其次,介绍了多模态医学图像分割的关键概念,包括数据维度、预处理、数据增强、损失函数以及后处理等;接着,对基于不同融合策略的多模态分割网络进行综述,对不同方式的融合策略进行分析;最后,对医学图像分割过程中常见的几个问题进行探讨,并对今后研究作了总结与展望。
深度学习;多模态;医学图像;图像融合;图像分割
0 引言
在多模态医学成像技术不断发展的背景下,多模态医学图像分割方法也被广泛研究。多模态医学图像分割指融合多模态图像的信息以提高分割性能[1]。常见的医学图像主要有计算机断层扫描(Computed Tomography, CT)、磁共振成像(Magnetic Resonance Imaging, MRI)和正电子发射断层扫描(Positron Emission computed Tomography, PET)等。相较于单模态医学图像,多模态医学图像可以为医生提供丰富的互补信息。其中,CT图像是肌肉及骨骼疾病,如骨肿瘤、骨折等疾病的常用诊断成像,MRI图像能提供较好的软组织对比度[2]。功能性影像(例如PET)缺乏解剖特征,但能提供疾病的定量代谢和功能信息。多模态MRI(图1)还可根据采集参数的差异提供补充信息,包括T1加权成像(T1 weighted imaging, T1w)、对比增强T1加权成像(T1 weighted contrast enhanced imaging, T1ce)、T2加权成像(T2 weighted imaging, T2w)以及流体衰减反转恢复(Fluid attenuated inversion recovery, Flair)图像。以脑肿瘤为例,T2w与Flair适用于显示瘤周水肿肿瘤,T1w图像与T1ce图像适用于肿瘤核心无瘤周水肿肿瘤。因此应用多模态图像进行分割能降低信息不确定性,提高临床诊断与分割精度[3]。
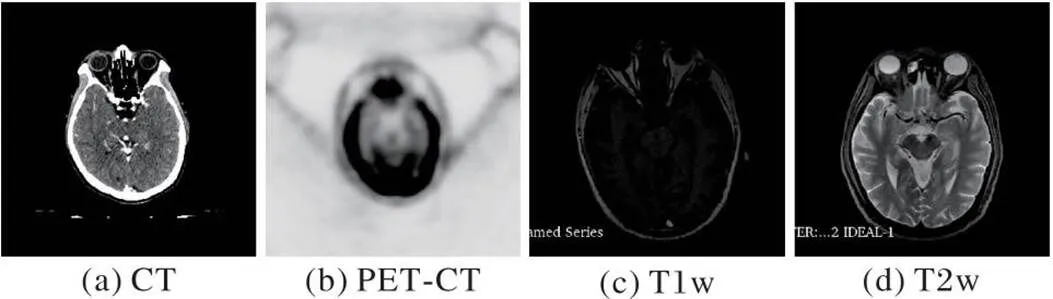
图1 不同模态的医学图像示例
如何从多模态医学图像中自动、有效地融合不同模态图像的信息完成分割任务,是多模态医学图像分割任务的关键。早在20世纪90年代,就有很多图像融合策略被国内外研究者[4]提出,其中最重要的有:基于概率论的融合[5]、基于复杂性理论的融合[6-7]、基于置信函数的融合[8-9]和基于机器学习的融合[10-13]。对于上述方法,不同模态的图像的统计特性存在差异,使浅层模型建模较为困难,性能也难以得到保证。从2012年开始,深度学习开始应用于计算机视觉领域,研究人员先后提出了AlexNet (Alex Network)[14]、VGG-Net (Visual Geometry Group Network )[15]、GoogleNet[16]、残差网络(Residual Network, ResNet)[17]、密集连接网络(Densely connected Network, DenseNet)[18]、全卷积神经网络(Fully Convolutional Neural Network, FCNN)[19]与U-Net[20]等网络模型,不仅在图像分类、分割、目标检测与跟踪等任务中获得了最佳性能,也为多模态医学图像分割问题提供了新思路。
随着深度学习的成功,国内外研究者致力于利用深度学习方法实现包括脑[21-23]、肺[24]、胰腺[25-26]、前列腺[27]和多器官[28-29]在内的医学图像分割。医学图像分割作为医学图像分析的一个重要的研究领域,对于诊断、检测、治疗都非常重要,目的在于给图像的每一个像素赋予一个标签。基于深度学习的方法已经在医学图像分割任务上获得了比传统方法更好的表现。为了得到更加精确的分割结果以便更好地诊断,融合多模态医学图像进行分割已经成了研究热点。
1 基础概念
1.1 基于深度学习的医学图像分割
深度学习模型一般指由多层非线性处理单元组成的神经网络模型,各个层以上一层的输出为输入,通过多层非线性处理单元的堆叠,可以从海量数据中抽取复杂特征信息[30]。近几年,深度学习已经在图像分类、识别、目标检测以及医学图像分析等领域得到显著发展,并获得与人类专家不相上下甚至更优越的性能。计算机视觉领域中最成功的算法为卷积神经网络(Convolutional Neural Network, CNN)模型。2012年,AlexNet[14]问世,并获得ImageNet挑战赛冠军,top-5错误率由26%下降至16.4%。在随后的几年中,多个经典的基于CNN的架构被相继提出,包括VGG-Net[15]、GoogleNet[16]、ResNet[17]和DenseNet[18],极大推动了相关研究的发展。
深度学习技术在医学图像分割领域也得到了广泛的应用。其中,FCNN将CNN中的全连接层替换为卷积层,同时通过反卷积实现上采样,从而实现像素级别的分类,但FCNN没有考虑到像素之间的对应关系,对于图像细节的分割结果不理想。U-Net在FCNN的基础上增加上采样的次数及跳跃,通过跳跃连接实现了网络中高维语义特征与低维语义特征的融合,提升了分割精度。U-Net(图2[20])是应用最广泛的深度学习分割模型,且作为其他研究的基准模型进行改进,达到了更优的表现。

图2 U-Net结构
然而,由于CNN中卷积核大小有限,它对远程信息建模的性能不是最佳的,这可能导致对大小可变的目标分割存在缺陷。另一方面,Transformer模型在自然语言处理和计算机视觉等多个领域的长距离信息建模方面表现出了卓越的能力。Hatamizadeh等[31]提出了一种新颖的分割模型Swin UNETR。具体地,该模型将多模态输入数据投影到1D嵌入序列中,并用作Swin UNETR编码器的输入;Swin UNETR编码器利用移位窗口计算自注意力来提取5种不同分辨率的特征,并通过跳跃连接在每个分辨率下连接到基于卷积的解码器。该模型在BraTS 2021分割挑战赛中跻身表现最佳的方法之列。
1.2 多模态医学图像分割数据集
由于分割目标在尺寸、形状、位置等方面具有较大的差异,因此医学图像分割在医学图像分析领域中具有非常高的挑战性。目前,已经提出的分割网络架构多种多样,但是很难比较不同算法的性能,这是因为大多数算法都是在不同的数据集上评估并以不同的指标进行评价。为公平地比较不同方法的表现,先后公布了多个公开数据集,例如脑肿瘤分割(Brain Tumor Segmentation, BraTS)数据集[21]、缺血性中风病变分割(Ischemic Stroke LEsion Segmentation, ISLES)数据集、MR脑图像分割(MR Brain image Segmentation, MRBrainS)数据集[32]、新生儿脑分割(Neonatal Brain Segmentation, NeoBrainS)数据集[33]、组合(CT-MR)腹部器官分割(Combined CT-MR Healthy Abdominal Organ Segmentation, CHAOS)数据集、婴儿脑MRI分割(6-month Infant brain MRI segmentation, Iseg)数据集[34]和多模态MR图像自动椎间盘定位分割(automatic InterVertebral Disc localization and segmentation from 3D MR images, IVDM3Seg)数据集。详细资料见表1。
2 医学图像分割的关键概念
2.1 数据维度

2.2 数据预处理
数据预处理对后续分割任务有重要影响;尤其对多模态医学图像进行分割时,由于图像具有强度不一、对比度各异、噪声较大等特点,为使图像像素值分布更统一、网络训练更平滑,需要将图像经过预处理操作后再送入分割网络。如图3所示,典型的预处理技术主要有图像配准、偏置场校正、图像重采样、强度值归一化等。针对BraTS数据集,研究人员在公开数据集之前进行了图像配准,同时利用N4ITK对MRI图像进行偏置场校正。强度归一化就是用图像像素强度的平均值减去标准偏差来独立地标准化每个患者的每个模态。
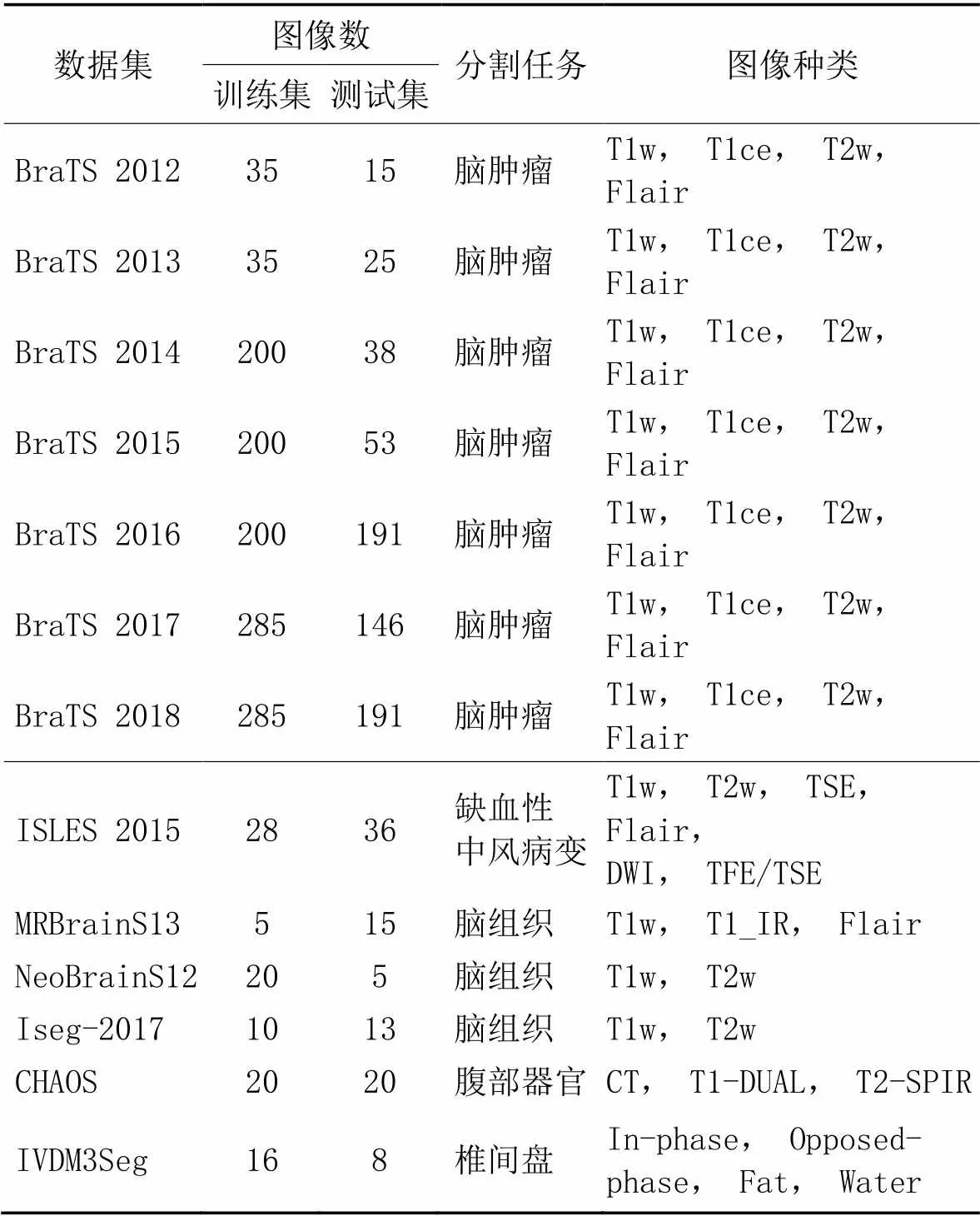
表1 多模态医学图像分割领域常用数据集
2.3 数据增强
医学图像分割任务很难得到训练用的大量有标签数据。对数据进行标注的过程通常需要本领域专家来完成,成本高且费时。在利用小样本数据对深度学习模型进行训练的过程中往往存在过拟合的问题。数据增强就是通过提高训练数据量来降低过拟合[43]。如图4,数据增强通过对训练数据集中的图像进行变换(旋转、平移、缩放、翻转、扭曲以及加入某些噪声如高斯噪声等),增加了可用的训练数据。数据增强技术在医学图像的分割任务中有着广泛的应用。
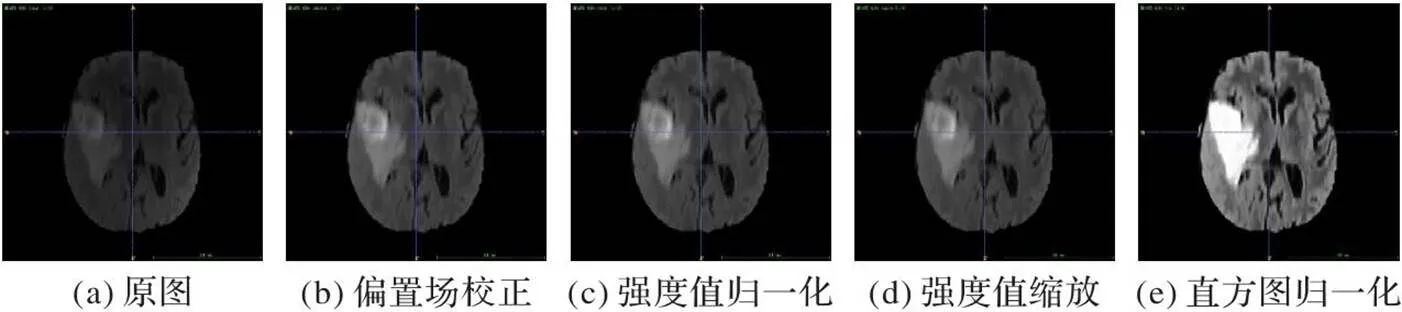
图3 预处理方式可视化

图4 数据增强方式可视化
2.4 损失函数
损失函数是用来度量模型的预测值()与真实值的差异程度的运算函数。在模型的训练阶段,每个批次的训练数据送入模型后,通过前向传播输出预测值,然后损失函数会计算出预测值和真实值之间的差异值,也就是损失值。得到损失值之后,模型通过反向传播去更新模型参数,来降低真实值与预测值之间的差异,从而达到学习的目的。在多模态医学图像分割中用到的损失函数与单模态医学图像分割中所用的损失函数大致相同,有以下几种:
1)交叉熵损失(Cross-Entropy loss, CE)函数:交叉熵损失是最常用的损失函数之一,对每个像素的类别预测进行单独评估,然后对所有像素点进行平均,通过式(1)计算。Long等[19]提出对不同类别的损失进行加权,以缓解类不平衡问题。

2)加权交叉熵损失(Weighted Cross-Entropy loss, WCE)函数:在医学图像分割任务中由于大部分像素点属于背景类,因此在计算损失时对不同类别的像素点赋予不同的权重是合理的,并可以缓解类别不平衡问题,定义如式(2):

3)骰子损失(DICE loss):骰子损失同样是医学图像分割中常用的损失函数之一。它可以衡量预测结果和真实结果之间重合度,通过式(3)计算:

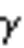


2.5 数据后处理
数据后处理通常被用来校正分割网络预测结果。3D模型分割过程中常常存在部分误分割像素,而分割结果中的最大体素块通常是最满足要求的。基于此前提,采用形态学方法去除误分割像素和保留最大体素块,并可依据检测区域结构设计若干后处理技术。文献[40]中将3D全连接条件随机场应用到后处理中,有效去除错误分割像素,细化分割结果。
3 多模态医学图像分割网络
近年来,研究人员已经提出了多种基于深度学习的多模态医学图像分割方法,如FCNN和U-Net。根据融合策略,这些方法可以分为输入级融合网络、中间级融合网络、决策级融合网络。对于每种融合网络,本文分别进行了总结,如图5所示。
3.1 输入级融合网络
输入级融合网络通常将不同的多模态图像在像素级别通过拼接操作得到多通道图像,输入多模态分割网络进行训练。现有的多模态医学图像分割网络大多为输入级融合网络[44-47],直接将多模态图像在原始输入空间进行融合。图6描述了输入层融合分割网络的通用网络架构。本文以多模态MRI图像作为输入,CNN作为分割网络,脑肿瘤分割作为分割任务进行说明。通过使用输入级融合策略,可以在神经网络的所有层中充分利用来自不同模态的丰富特征信息。这种融合通常使用五种技术,多任务分割、多视图分割、多尺度分割、生成对抗网络(Generative Adversarial Network, GAN)和Transformer网络。

图5 融合策略的通用分类
在BraTS数据集上,Wang等[39]提出了一种级联全卷积神经网络(图7[39]),将脑肿瘤划分为3个子区域,包括整体肿痛、肿瘤核心和增强肿瘤核心。该模型将4种模态MRI图像(T1、T1c、T2和Flair)直接集成到多通道输入中,再根据子区域层次把多类分割问题分解成3个连续的二分类分割问题:1)分割整体肿痛;2)利用分割结果包围盒分割肿瘤核心;3)根据肿瘤核心的分割结果包围盒对增强肿瘤核心进行分割。
此外,为了利用3D上下文信息,对于每个单独的任务,通过平均单个任务的Softmax输出融合来自3个不同正交视图(水平位、冠状位、矢状位)的分割结果。在BraTS 2017测试集上的实验结果表明,该方法在增强肿瘤核心、整体肿痛和肿瘤核心方面的平均DICE得分分别为0.783、0.874和0.775,在BraTS 2017挑战赛中获得第二名。多任务分割把多类分割中复杂的任务分解为多个较简单的分割任务,同时利用肿瘤子区域层次结构提高了分割精度。
级联策略虽然表现良好,但是会导致系统复杂性增加,且忽略模型间相关性。为了克服这些缺陷,Zhou等[47]提出了注意力机制指导下的多任务分割网络,将输入空间中的多模态MRI图像逐通道地进行融合,然后将脑肿瘤的分割分解成三个不同却又相互联系的工作,每项工作具有单独的卷积层、分类层、损失层、输入数据各不相同。为更加有效地优化网络,他们还设计了跨任务指导注意力的模块,通过沿用以往任务给出的预测结果引导,该模块能够根据具体类统计数据对通道特征响应进行自适应再标定。该方法在BraTS 2015测试集上排名第一,在BraTS 2017数据集上取得了最好的性能。
由于地域文化差异和社会发展水平的不同,外来订单影响了广彩的色彩倾向,逐渐成为热烈华丽的釉上彩。十八世纪的广彩受到欧洲古典画派的影响,迎合当时盛行的洛可可风格,表达出温馨、欢快的情调。1784年,美国的“中国皇后”号抵达广州港,开始大批量的贸易活动,至此美国也成为外销瓷的主要市场。此后的广彩瓷愈加浓烈奔放,以红、绿、金、蓝等色彩为主,描绘出一幅幅热闹的景象,特点鲜明并影响至今。
为了对脑肿瘤进行更加精确且快速的分割与重建,Zhao等[48]提出Multi-View Pointwise U-Net模型,该方法沿用编码器-解码器的结构,且该结构中的3D卷积被3个2D多视点卷积代替(图8[48]),这些卷积接收3个正交视图以学习空间特征并通过1个逐点卷积以学习通道方向特征。在BraTS 2020测试集中,对于增强肿瘤核心、整体肿痛和肿瘤核心,该方法的平均DICE得分分别为0.715、0.839和0.768。
为了进一步利用这些数据集中嵌入的3D信息,Ding等[49]提出了一种多视角动态融合框架,以提高脑肿瘤分割的性能,该框架由3个关键部分组成。首先提出一种含有多个学习网络的多视点深度神经网络结构来对脑肿瘤进行不同视角的分割;其次使用动态融合方法对多角度分割结果进行融合;再次,提出多视点融合损失(由分割损失、过渡损失、决策损失等构成),保证分割结果与医生标注在外观和空间上的一致性。在BraTS 2015和BraTS 2018测试集上,该方法以更高效率达到较好的分割效果。
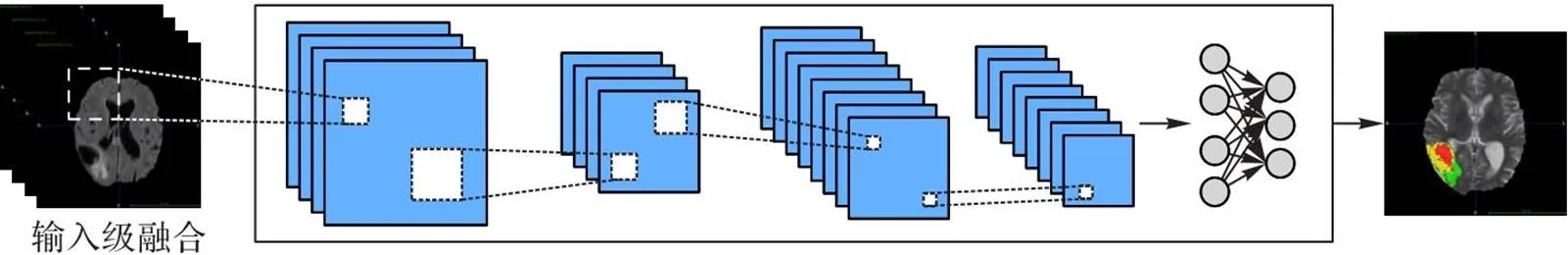
图6 输入级融合网络结构
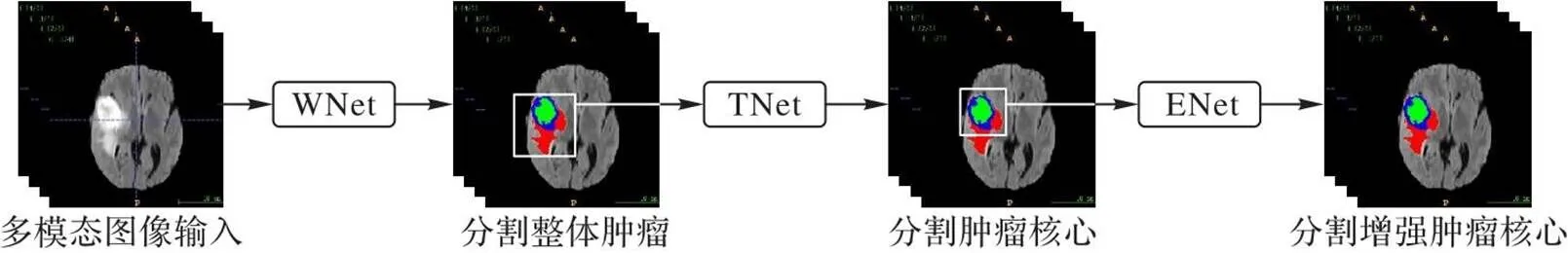
图7 多任务分割网络结构

图8 多视角卷积模块
在分割医学图像中的不同区域时,可能需要不同大小的感受野。大尺寸目标可能需要全局信息,而小目标可能需要高分辨率的局部信息。因此,Qin等[50]提出了自动聚焦卷积层以增强神经网络的多尺度处理能力。自动聚焦卷积层根据处理后的上下文自适应地改变感受野的大小,并结合了一种注意力机制来学习最佳的感受野尺度。该方法在盆腔CT中的多器官分割和MRI中的脑肿瘤分割等具有挑战性的任务中取得了可喜的表现。Lin等[51]则提出了路径聚合U-Net(Path Aggregation U-Net, PAU-Net)模型,该模型包含一个自底向上的路径聚合(Path Aggregation, PA)编码器、增强型解码器(Enhanced Decoder, ED)和有效特征金字塔(Effective Feature Pyramid, EFP)组成的路径聚合U-Net,在MRI影像的脑肿瘤分割中取得了较好的性能。
生成对抗网络[52]包含生成器和鉴别器两个子网络,其中生成器生成图片并输入鉴别器,由鉴别器判断输入图片是标签图还是生成图,二者基于对抗性过程进行训练。Yang等[53]通过融合多模态图像作为多通道输入,训练了两个独立的网络:以U-Net为基干的生成器网络和鉴别器网络,生成器网络用于预测分割结果,而鉴别器网络将区分分割掩膜是模型预测结果还是真实值。该方法(图9[53])在BraTS 2018测试集上进行了评估并取得了有竞争力的结果,说明在对抗训练中加入额外的约束可以改进原始分割结果。Huo等[54]使用Patch-GAN[55]作为额外的鉴别器来监督网络的训练过程。基于GAN的方法由于判别器的额外约束可以获得更加鲁棒的分割结果,但是训练额外的判别器需要更多的内存。
鉴于Transformer结构在自然语言处理领域所取得的成功[56],研究人员将它迁移到计算机视觉领域,并先后提出了Vision Transformer (ViT)[57]和Swin Transformer[58],在图像分类任务上获得了良好的性能。部分研究人员根据多模态医学影像的特点,将Transformer结构引入到多模态医学图像分割任务中,取得了优异的表现[59-63]。其中,Wang等[61]将CNN和Transformer结构相结合以更好地对局部信息和全局信息进行建模(图10[61])。为了获取局部的3D上下文信息,编码器首先利用3D CNN提取特征图,然后送入Transformer进行全局特征建模,解码器利用Transformer输出的特征并结合上采样来预测分割图。在BraTS 2019和2020测试集上的实验结果表明,该方法均取得了最优的结果。
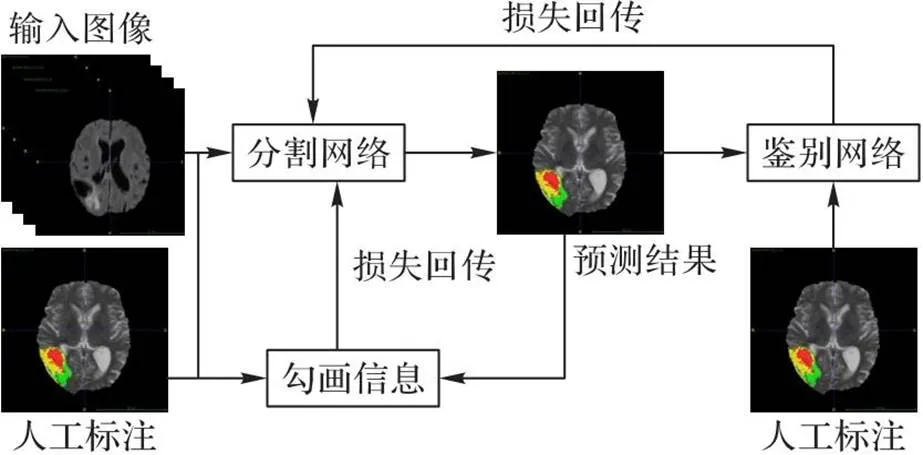
图9 生成对抗分割网络结构

图10 Trans-BTS结构
输入级融合策略可以最大限度地保留原始图像信息并学习图像的内在特征。使用CNN与不同的策略相结合,例如多任务、多视图、多尺度、GAN和Transformer等,可以充分利用多模态图像的特征表示。
3.2 中间级融合网络
中间级融合网络将不同模态的图像分别作为分割网络的输入,再将所学特征融入网络各层,最后,融合结果传输到决策层,获得最终分割结果。中间级融合网络能够有效地集成并充分利用多模态图像[64-66]。图11是中间级融合网络的网络架构。

图11 中间级融合网络结构

图12 超密集连接网络结构
尽管中间级融合网络已经取得了令人鼓舞的表现,但特征融合通常是利用简单的求和或拼接来实现,这限制了模型性能的提升。Li等[67]提出了一种有监督的图像融合方法,通过注意力模块可以选择性地融合来自不同模态的有效信息并抑制各自的噪声信号(图13[67]),并通过注意力机制从不同的模态中选择对结果贡献最大的一种模态作为主模态,监督其他辅助模态的信息选择。该方法在乳房肿块分割实验中验证了有效性,与其他先进的方法相比,获得了更好的分割结果。此外,基于注意力机制进行不同模态之间的特征融合也被应用于肝脏肿瘤分割[68]。

图13 注意力融合网络结构
研究提取不同模态图像间的跨模态信息,并将它们应用到多模态图像分割中,同样受到较多关注[69-70]。Dou等[69]提出了CT和MRI多模态分割的模型Chilopod-net,通过CT和MRI之间共享所有的卷积核来共享特征,同时,通过与模态相关的批归一化(Batch Normalization, BN)层,减小了不同模态数据之间的差别。通过对心脏结构分割和腹部器官分割这两个任务进行实验,结果显示该方案总是比单模态训练及以往多模态分割方法效果更好。
总之,中间级融合网络中如何有效地实现特征的融合是一个关键的问题。密集连接应用最广泛,该方法有利于提高全网信息流及梯度并减轻梯度消失。基于注意力机制的特征融合在近年来也得到了广泛的研究,这种方法能够有效地融合不同模式下的有用信息,并且对噪声具有抑制作用。最后通过对跨模态信息的学习,为多模态图像融合提供一种创新方法,与前述方法相比能有效降低模型的复杂度。
3.3 决策级融合网络
决策级融合网络与中间级融合网络相同,都是把不同模态的医学图像分别送入相应的分割网络进行训练。单个网络能较好地利用对应模态的特征信息,接着融合各网络的输出,得到最终分割结果。决策级融合网络的目的是自主地学习不同模态下的互补信息,这是因为不同图像采集技术使多模态图像很少有直接互补信息存在于各自原始图像空间。图14是决策级融合分割网络的一般网络架构。

图14 决策级融合网络结构
例如,为了有效地利用来自T1、T2和分数阶各向异性图像的多模态特征信息,Nie等[38]提出了一种新的多FCN结构,应用于婴儿脑组织分割的对象是:白质(White Matter WM)、灰质(Grey Matter, GM)、脑脊液(Cerebro-Spinal Fluid, CSF)等。这种方法并不将从输入空间中获取到的3个模态数据进行简单结合,而是对每一个模态的影像单独训练一个分割网络,再将从各个网络获取到的结果进行加权。结果表明,所建模型的精度明显高于以往方法。
关于决策级融合网络的融合策略,研究者们进行了诸多研究[71],如基于平均投票的融合策略、多数投票等。在平均策略方面,Kamnitsas等[72]分别对3个网络进行训练,再针对每个网络的输出取平均值,以该平均值作为融合网络输出结果。对大多数投票策略而言,体素的最终标签为各个网络的投票结果中占多数的标签。
不同模态图像在统计属性上有差别,导致单个模型难以对跨模态特征进行直接研究。因此,在决策级融合分割网络中可以训练多个分割网络以充分利用多模态特征,充分发挥多模态特征的优势。Aygün等[73]研究了脑肿瘤分割问题的不同融合方法在计算资源和模型性能等方面的差异,提出了一种新的脑肿瘤分割方法。从资源占用的角度来看,决策级融合策略对资源占用的要求较高;但从性能上看,由于各模态图像由独立的网络提取特征,决策级融合网络能够取得较好的效果,比输入级融合网络能够学习到复杂而又相互补充的特征信息。
4 面临的问题
基于深度学习的多模态医学影像分割面临的主要问题如下:
1)多模态医学图像融合的复杂性。不同模态的医学图像蕴含着具有互补性的特征,设计合适的模型充分利用不同模态的医学图像信息并进行融合是需要解决、也是研究人员正在努力攻克的首要难题。目前主流的融合方式包括:输入级融合、中间层融合以及决策级融合,这三种方式都能够捕获不同模态的医学图像特征,利用不同模态之间的互补信息。但是,这三种方式各有优缺点:输入级融合模型结构简单,但是无法充分发现模态之间的互补信息;中间层融合以及决策级融合网络模型较复杂,对计算设备的要求较高。这些问题导致现有模型距离实际临床应用仍存在较大距离。因此,后续研究可以继续探索不同模态图像的有效融合方式,尤其是针对中间层融合,将它与网络结构搜索技术结合,使模型能够自动学习最优的特征融合方式,即在哪个阶段及采用何种方式进行融合。
2)多模态医学图像分割的类别不均衡问题。在医学图像分析中,另一个难题就是数据类别不均。比如对脑肿瘤或者白质病变进行划分时正常脑区域要比病变区域大得多,利用类不平衡数据训练将造成分割网络的不稳定性,使网络更倾向于对像素分配多数类的标签。例如在BraTS 2017数据集上,训练集中正类像素与负类像素的比例是1∶99,背景素占据了绝大部分,所以损失函数选取对于分割网络来说非常关键。前文介绍了目前医学图像分割网络普遍采用的单独或联合应用的几类损失函数。在数据层面上,类别不平衡问题可通过重新采样解决,主要包括三种采样方法:负类欠采样、正类过采样[74]与人工少数类过采样法(Synthetic Minority Over-sampling TEchnique, SMOTE)[75]。这些方法虽然易于实施,但是可能删除某些重要的数据或者在训练集中加入冗余的数据。基于补丁块(patch)的采样方式也能减轻数据不平衡。比如Kamnitsas等[40]提出的平衡策略用50%的概率从病变或者健康体素中抽取训练补丁以减轻数据不平衡;Clèrigues等[76]采用了以病灶为核心的策略,在该策略中,训练补丁全部来自病灶体素核心区域。此外将随机偏移加入采样以避免病变像素始终位于patch的中心位置同样会有利于部分数据的增强。
5 讨论
本文总结了当前较先进的基于深度学习的多模态医学图像分割网络,并展示在表2中。
目前用于医学图像分割任务的公开可用的多模态医学图像数据集还很少,其中最常用的BraTS数据集还是2012年开始提出的。2022年表现最好的方法是文献[61]中提出的,模型使用输入级融合策略直接整合输入空间中的不同模态,同时为了获取局部的3D上下文信息,首先利用3D CNN提取特征图,然后,将特征图送入Transformer进行全局特征建模,解码器利用Transformer输出的特征并结合上采样来预测分割图。在BraTS 2019和2020数据集上的实验结果表明,该方法均取得了最优的结果。与之前的工作相比,文献[61]中证明了Transformer结构的引入能够学习全局语义相关性从而提升网络性能。为定量比较不同融合策略的性能,表2同时比较了使用三种不同策略的网络模型的性能。在BraTS 2018数据集上,本文列举了采用三种不同融合策略的网络模型,从分割精度来看使用输入级融合的模型取得了最优的表现,优于中间级融合以及决策级融合;决策级融合模型在增强肿瘤核心时,分割精度达到0.831,但是在分割肿瘤核心时,分割性能最差;中间级融合网络的分割性能整体低于输入级融合网络,与决策级融合网络差异不大。
对于多模态医学图像分割,融合策略对于网络模型的性能起着至关重要的作用。传统的图像融合策略通过学习图像的统计特性实现,由于不同模态的图像具有不同的统计特性,导致使用浅层模型难以对图像进行建模。为此,基于深度学习的方法进行多模态图像分割开始受到关注。基于深度学习的方法主要有三点优势:第一,基于深度学习的网络可以针对多模态图像学习复杂而抽象的特征表示,克服了手动特征设计的困难;第二,基于深度学习的网络可以通过使用不同的网络层来呈现不同模态之间的复杂关系,例如中间层融合策略;第三,传统融合策略中的图像融合策略可以通过训练深度学习模型实现,这样就可以研究一些潜在的深度学习网络架构来设计有效的图像融合策略。因此,基于深度学习的方法具有比传统方法产生更好融合结果的巨大潜力。
如何选择有效的深度学习融合策略仍然是困扰学界的难题。在2013—2020 BraTS挑战中,所有表现优秀的方法都使用了输入级融合策略,将输入网络的不同MRI图像直接融合,以保留图像的固有特征,并专注于后续的分割网络架构设计,例如多任务、多视图、多尺度、基于GAN的策略和基于Transformer的策略。对于中间级融合,典型的融合策略往往以DenseNet作为基础网络,通过密集连接的方式捕获模态内部与不同模态之间的复杂关系,这可以帮助分割网络学习更多有价值的信息并获得比输入层融合模型更好的性能。对于决策级融合策略,与输入级融合相比,每种模态的图像分别输入不同的网络以学习独立的特征表示,这需要大量的内存和计算时间。由此可见,虽然目前这些表现优秀的方法大多是基于输入级融合策略,但是中间级融合以及决策级融合仍有待进一步探索。

表2 不同融合方式的比较
尽管基于深度学习的多模态医学图像分割已经取得了极大的进展,但是仍有一些关键问题需要解决。众所周知,多模态融合网络在分割任务中通常比单模态网络表现更好,关键问题是如何融合不同模态的图像以获得更加精确的分割结果,如在哪一层融合以及采用何种方式融合等。网络结构搜索(Network Architecture Search, NAS)允许网络从预定义好的网络空间中学习出最优的子空间[77],如果将NAS技术与多模态图像融合技术相结合,使模型可以自动学习最优的图像融合方式将是一个极富前景的探索方向。
其他问题主要涉及数据问题。首先,由于标注成本较高导致大规模数据集获取比较困难,有限的训练数据很容易导致过度拟合问题。为了解决这个问题,降低网络架构的复杂性或增加训练数据已被证明可以缓解该问题。其次,使用类别不平衡数据进行训练会导致分割网络不稳定,尤其是对于小病变或小目标分割。
6 结语
近年来,基于深度学习的多模态医学图像分割方法被广泛研究,并取得了很多很好的成果。本文介绍了深度学习和多模态医学图像分割等基本概念,概括了多模态医学图像分割过程中涉及的数据预处理、数据增强、数据后处理等技术,总结了三种目前主流的多模态图像分割方法,并进一步对这些方法进行了细粒度的划分和概括,概述了不同方法的优劣;本文同时还阐述了基于深度学习的多模态医学影像分割面临的主要问题,其中的小样本性和复杂性使现有研究距离实际应用仍有一定距离;最后,本文在分析现有模型不足的基础上,总结了现有的前沿的研究结果,并展望了未来的研究方向。研究基于深度学习的多模态医学影像分割方法具有非常重要的意义,通过该方法可以实现更加精准的病变和目标分割,有助于减轻临床医生的负担,提升诊断的精度和速度,这将是未来需要关注的重要研究领域。
[1] 范立南,王晶,王银玲.多模态医学图像融合技术综述[J].沈阳大学学报,2009,21(3):96-100.(FAN L N, WANG J, WANG Y L. A summary of multimodality medical image fusion technology[J]. Journal of Shenyang University, 2009, 21(3): 96-100.)
[2] 邹诚实,岑婷.腹部CT及MRI图像融合配准在临床中的应用[J].上海医学影像,2002,11(4):250-253.(ZOU C S, CEN T. Abdomen CT and MRI image fusion technique and its clinical application [J]. Shanghai Medical Imaging, 2002,11(4): 250-253.)
[3] GUO Z, LI X, HUANG H, et al. Deep learning-based image segmentation on multimodal medical imaging [J]. IEEE Transactions on Radiation and Plasma Medical Sciences, 2019, 3(2): 162-169.
[4] KHALEGHI B, KHAMIS A, KARRAY F O, et al. Multisensor data fusion: a review of the state-of-the-art [J]. Information Fusion, 2013, 14(1): 28-44.
[5] 赵澄东,王旭辉,杨静宇.一种基于改进的概率图像融合方法[J].计算机工程与应用,2012,48(8):182-184.(ZHAO C D, WANG X H, YANG J Y. Method of image fusion based on improved probability theory [J]. Computer Engineering and Applications, 2012, 48(8): 182-184.)
[6] DAS S, KUNDU MK. A neuro-fuzzy approach for medical image fusion [J]. IEEE Transactions on Biomedical Engineering, 2013, 60(12): 3347-3353.
[7] BALASUBRAMANIAM P, ANANTHI V P. Image fusion using intuitionistic fuzzy sets [J]. Information Fusion, 2014, 20: 21-30.
[8] SMETS P. The combination of evidence in the transferable belief model [J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 1990, 12(5): 447-458.
[9] LIAN C, RUAN S, DENŒUX T, et al. Joint tumor segmentation in PET-CT images using co-clustering and fusion based on belief functions [J]. IEEE Transactions on Image Processing, 2018, 28(2): 755-766.
[10] VÁZQUEZ-REINA A, GELBART M, HUANG D, et al. Segmentation fusion for connectomics [C]// Proceedings of the 2011 International Conference on Computer Vision. Piscataway: IEEE, 2011: 177-184.
[11] 肖儿良,周莹,简献忠.迁移学习与GAN结合的医学图像融合模型[J].小型微型计算机系统,2020,41(9):1972-1978.(XIAO E L, ZHOU Y, JIAN X Z. Medical image fusion model combining transfer learning and generative adversarial network [J]. Journal of Chinese Computer Systems,2020,41(9):1972-1978.)
[12] 李鸿,邹俊颖,谭茜成,等.面向医学图像分割的多注意力融合网络[J].计算机应用,2022,42(12):3891-3899.(LI H, ZOU J Y, TAN X C, et al. Multi-attention fusion network for medical image segmentation [J]. Journal of Computer Applications, 2022, 42(12): 3891-3899.)
[13] ZHANG N, RUAN S, LEBONVALLET S, et al. Kernel feature selection to fuse multi-spectral MRI images for brain tumor segmentation [J]. Computer Vision and Image Understanding, 2011, 115(2): 256-269.
[14] KRIZHEVSKY A, SUTSKEVER I, HINTON G E. ImageNet classification with deep convolutional neural networks [J]. Communications of the ACM, 2017, 60(6): 84-90.
[15] SIMONYAN K, ZISSERMAN A. Very deep convolutional networks for large-scale image recognition [EB/OL]. [2023-04-24]. https://arxiv.org/pdf/1409.1556.pdf.
[16] SZEGEDY C, LIU W, JIA Y, et al. Going deeper with convolutions [C]// Proceedings of the 2015 IEEE Conference on Computer Vision and Pattern Recognition. Washington, DC: IEEE Computer Society, 2015: 1-9.
[17] HE K, ZHANG X, REN S, et al. Identity mappings in deep residual networks [C]// Proceedings of the 2016 European Conference on Computer Vision, LNIP 9908. Cham:Springer, 2016: 630-645.
[18] HUANG G, LIU Z, MAATEN L van der, et al. Densely connected convolutional networks [C]// Proceedings of the 2017 IEEE Conference on Computer Vision and Pattern Recognition. Washington, DC: IEEE Computer Society, 2017: 2261-2269.
[19] LONG J, SHELHAMER E, DARRELL T. Fully convolutional networks for semantic segmentation [C]// Proceedings of the 2015 IEEE Conference on Computer Vision and Pattern Recognition. Washington, DC: IEEE Computer Society, 2015: 3431-3440.
[20] RONNEBERGER O, FISCHER P, BROX T. U-Net: convolutional networks for biomedical image segmentation [C]// Proceedings of 18th International Conference on Medical Image Computing and Computer-Assisted Intervention, LNCS 9351. Cham: Springer, 2015: 234-241.
[21] MENZE B H, JAKAB A, BAUER S, et al. The multimodal brain tumor image segmentation benchmark (BRATS) [J]. IEEE Transactions on Medical Imaging, 2015, 34(10): 1993-2024.
[22] PEREIRA S, PINTO A, ALVES V, et al. Brain tumor segmentation using convolutional neural networks in MRI images [J]. IEEE Transactions on Medical Imaging, 2016, 35(5): 1240-1251.
[23] HAVAEI M, DAVY A, WARDE-FARLEY D, et al. Brain tumor segmentation with deep neural networks [J]. Medical Image Analysis, 2017, 35: 18-31.
[24] KALINOVSKY A, KOVALEV V. Lung image segmentation using deep learning methods and convolutional neural networks [C]// Proceedings of the 13th International Conference on Pattern Recognition and Information Processing. Berlin: Springer, 2016: 21-24.
[25] ROTH H R, LU L, FARAG A, et al. DeepOrgan: multi-level deep convolutional networks for automated pancreas segmentation [C]// Proceedings of 18th International Conference on Medical Image Computing and Computer-Assisted Intervention, LNIP 9349. Cham: Springer, 2015: 556-564.
[26] FU M, WU W, HONG X, et al. Hierarchical combinatorial deep learning architecture for pancreas segmentation of medical computed tomography cancer images [J]. BMC Systems Biology, 2018, 12: No.56.
[27] YU L, YANG X, CHEN H, et al. Volumetric ConvNets with mixed residual connections for automated prostate segmentation from 3D MR images [C]// Proceedings of the 31st AAAI Conference on Artificial Intelligence. Palo Alto, CA: AAAI Press, 2017: 66-72.
[28] ZHOU X, TAKAYAMA R, WANG S, et al. Deep learning of the sectional appearances of 3D CT images for anatomical structure segmentation based on an FCN voting method [J]. Medical Physics, 2017, 44(10): 5221-5233.
[29] STOYANOV D, TAYLOR Z, CARNEIRO G, et al. Deep learning in medical image analysis and multimodal learning for clinical decision support [C]// Proceedings of the 4th International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, DLMIA 2018, and 8th International Workshop, ML-CDS 2018, Held in Conjunction with MICCAI 2018, LNCS 11045. Cham: Springer, 2018: 3-11.
[30] ZHOU T, RUAN S, CANU S. A review: deep learning for medical image segmentation using multi-modality fusion [J]. Array, 2019, (3/4): No.100004.
[31] HATAMIZADEH A, NATH V, TANG Y, et al. Swin UNETR: Swin transformers for semantic segmentation of brain tumors in MRI images [C]// Proceedings of the 7th International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, BrainLes 2021, Held in Conjunction with MICCAI 2021, LNCS 12962. Cham: Springer, 2022: 272-284.
[32] MENDRIK A M, VINCKEN K L, KUIJF H J, et al. MRBrainS challenge: online evaluation framework for brain image segmentation in 3T MRI scans [J]. Computational Intelligence and Neuroscience, 2015, 2015: No.813696.
[33] IŠGUM I, BENDERS M J N L, AVANTS B, et al. Evaluation of automatic neonatal brain segmentation algorithms: the NeoBrainS12 challenge [J]. Medical Image Analysis, 2015, 20(1): 135-151.
[34] WANG L, NIE D, LI G, et al. Benchmark on automatic six-month-old infant brain segmentation algorithms: the iSeg-2017 challenge [J]. IEEE Transactions on Medical Imaging, 2019, 38(9): 2219-2230.
[35] DOLZ J, GOPINATH K, YUAN J, et al. HyperDense-Net: a hyper-densely connected CNN for multi-modal image segmentation [J]. IEEE Transactions on Medical Imaging, 2019, 38(5): 1116-1126.
[36] CUI S, MAO L, JIANG J, et al. Automatic semantic segmentation of brain gliomas from MRI images using a deep cascaded neural network [J]. Journal of Healthcare Engineering, 2018, 2018: No.4940593.
[37] DOLZ J, DESROSIERS C, BEN AYED I. IVD-Net: intervertebral disc localization and segmentation in MRI with a multi-modal UNet [C]// Proceedings of 5th International Workshop and Challenge, CSI 2018, Held in Conjunction with MICCAI 2018, LNCS 11397. Cham: Springer, 2019: 130-143.
[38] NIE D, WANG L, GAO Y, et al. Fully convolutional networks for multi-modality isointense infant brain image segmentation[C]// Proceedings of 2016 IEEE International Symposium on Biomedical Imaging. Piscataway: IEEE, 2016: 1342-1345.
[39] WANG G, LI W, OURSELIN S, et al. Automatic brain tumor segmentation using cascaded anisotropic convolutional neural networks [C]// Proceedings of the Third International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, BrainLes 2017, Held in Conjunction with MICCAI 2017, LNCS 10670. Cham: Springer, 2018: 178-190.
[40] KAMNITSAS K, LEDIG C, NEWCOMBE V F J, et al. Efficient multi-scale 3D CNN with fully connected CRF for accurate brain lesion segmentation [J]. Medical Image Analysis, 2017, 36: 61-78.
[41] ZHAO X, WU Y, SONG G, et al. A deep learning model integrating FCNNs and CRFs for brain tumor segmentation [J]. Medical Image Analysis, 2018, 43: 98-111.
[42] MLYNARSKI P, DELINGETTE H, CRIMINISI A, et al. 3D convolutional neural networks for tumor segmentation using long-range 2D context [J]. Computerized Medical Imaging and Graphics, 2019, 73: 60-72.
[43] WANG J, PEREZ L. The effectiveness of data augmentation in image classification using deep learning [EB/OL]. [2022-05-09]. http://vision.stanford.edu/teaching/cs231n/reports/2017/pdfs/300.pdf.
[44] ISENSEE F, KICKINGEREDER P, WICK W, et al. Brain tumor segmentation and radiomics survival prediction: Contribution to the BraTS 2017 challenge [C]// Proceedings of the Third International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, BrainLes 2017, Held in Conjunction with MICCAI 2017, LNCS 10670. Cham: Springer, 2018: 287-297.
[45] ISENSEE F, KICKINGEREDER P, WICK W, et al. No new-net [C]// Proceedings of the 4th International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, BrainLes 2018, Held in Conjunction with MICCAI 2018, LNCS 11384. Cham: Springer, 2019: 234-244.
[46] MYRONENKO A. 3D MRI brain tumor segmentation using autoencoder regularization [C]// Proceedings of the 4th International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, BrainLes 2018, Held in Conjunction with MICCAI 2018, LNCS 11384. Cham: Springer, 2019: 311-320.
[47] ZHOU C, DING C, WANG X, et al. One-pass multi-task networks with cross-task guided attention for brain tumor segmentation [J]. IEEE Transactions on Image Processing, 2020, 29: 4516-4529.
[48] ZHAO C, ZHAO Z, ZENG Q, et al. MVP U-Net: multi-view pointwise U-net for brain tumor segmentation [C]// Proceedings of the 6th International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, BrainLes 2020, Held in Conjunction with MICCAI 2020, LNCS 12659. Cham: Springer, 2021: 93-103.
[49] DING Y, ZHENG W, GENG J, et al. MVFusFra: a multi-view dynamic fusion framework for multimodal brain tumor segmentation [J]. IEEE Journal of Biomedical and Health Informatics, 2021, 26(4): 1570-1581.
[50] QIN Y, KAMNITSAS K, ANCHA S, et al. Autofocus layer for semantic segmentation [C]// Proceedings of 21th International Conference on Medical Image Computing and Computer-Assisted Intervention, LNCS 11072. Cham: Springer, 2018: 603-611.
[51] LIN F, WU Q, LIU J, et al. Path aggregation U-Net model for brain tumor segmentation [J]. Multimedia Tools and Applications, 2021, 80(15): 22951-22964.
[52] GOODFELLOW I, POUGET-ABADIE J, MIRZA M, et al. Generative adversarial networks [J]. Communications of the ACM, 2020, 63(11): 139-144.
[53] YANG H-Y, YANG J. Automatic brain tumor segmentation with contour aware residual network and adversarial training[C]// Proceedings of the 4th International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, BrainLes 2018, Held in Conjunction with MICCAI 2018, LNCS 11384. Cham: Springer, 2019: 267-278.
[54] HUO Y, XU Z, BAO S, et al. Splenomegaly segmentation on multi-modal MRI using deep convolutional networks [J]. IEEE Transactions on Medical Imaging, 2019, 38(5): 1185-1196.
[55] ISOLA P, ZHU J Y, ZHOU T, et al. Image-to-image translation with conditional adversarial networks [C]// Proceedings of the 2017 IEEE Conference on Computer Vision and Pattern Recognition. Washington, DC: IEEE Computer Society, 2017: 5967-5976.
[56] VASWANI A, SHAZEER N, PARMAR N, et al. Attention is all you need [C]// Proceedings of the 31st International Conference on Neural Information Processing Systems. Red Hook, NY: Curran Associates Inc., 2017: 6000-6010.
[57] DOSOVITSKIY A, BEYER L, KOLESNIKOV A, et al. An image is worth 16x16 words: Transformers for image recognition at scale [EB/OL]. [2023-04-24]. https://arxiv.org/pdf/2010.11929.pdf.
[58] LIU Z, LIN Y, CAO Y, et al. Swin Transformer: hierarchical vision Transformer using shifted windows [C]// Proceedings of the 2021 IEEE/CVF International Conference on Computer Vision. Piscataway: IEEE, 2021: 10012-10022.
[59] JIA Q, SHU H. BiTr-Unet: a CNN-Transformer combined network for MRI brain tumor segmentation [C]// Proceedings of the 7th International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, BrainLes 2021, Held in Conjunction with MICCAI 2021, LNCS 12963. Cham: Springer, 2022: 3-14.
[60] CHEN J, LU Y, YU Q, et al. TransUnet: Transformers make strong encoders for medical image segmentation [EB/OL]. [2023-04-25]. https://arxiv.org/pdf/2102.04306.pdf.
[61] WANG W, CHEN C, DING M, et al. TransBTS: multimodal brain tumor segmentation using transformer [C]// Proceedings of the 24th International Conference on Medical Image Computing and Computer Assisted Intervention, LNIP 12901. Cham: Springer, 2021: 109-119.
[62] HATAMIZADEH A, NATH V, TANG Y, et al. Swin UNETR: Swin Transformers for semantic segmentation of brain tumors in MRI images [C]// Proceedings of the 7th International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, BrainLes 2021, Held in Conjunction with MICCAI 2021, LNCS 12962. Cham: Springer, 2022: 272-284.
[63] JIANG Y, ZHANG Y, LIN X, et al. SwinBTS: a method for 3D multimodal brain tumor segmentation using Swin Transformer [J]. Brain Sciences, 2022, 12(6): No.797.
[64] CHEN L, WU Y, DSOUZA A M, et al. MRI tumor segmentation with densely connected 3D CNN [C]// Proceedings of the 2018 Medical Imaging: Image Processing, SPIE 10574. Bellingham, WA: SPIE, 2018: 357-364.
[65] CHEN Y, CHEN J, WEI D, et al. OctopusNet: a deep learning segmentation network for multi-modal medical images [C]// Proceedings of the First International Workshop on Multiscale Multimodal Medical Imaging, LNCS 11977. Cham: Springer, 2020: 17-25.
[66] YUE M, DAI Z, HE J, et al. MRI-guided automated delineation of gross tumor volume for nasopharyngeal carcinoma using deep learning[C]// Proceedings of the 35th IEEE International Symposium on Computer-Based Medical Systems. Piscataway: IEEE, 2022: 292-296.
[67] LI C, SUN H, LIU Z, et al. Learning cross-modal deep representations for multi-modal MR image segmentation [C]// Proceedings of the 22nd International Conference on Medical Image Computing and Computer Assisted Intervention, LNCS 11765. Cham: Springer, 2019: 57-65.
[68] ZHANG Y, YANG J, TIAN J, et al. Modality-aware mutual learning for multi-modal medical image segmentation [C]// Proceedings of the 24th International Conference on Medical Image Computing and Computer-Assisted Intervention, LNCS 12901. Cham: Springer, 2021: 589-599.
[69] DOU Q, LIU Q, HENG P A, et al. Unpaired multi-modal segmentation via knowledge distillation [J]. IEEE Transactions on Medical Imaging, 2020, 39(7): 2415-2425.
[70] WANG H, ZHANG J, CHEN Y, et al. Uncertainty-aware multi-modal learning via cross-modal random network prediction[C]// Proceedings of the 17th European Conference on Computer Vision, LNCS 13697. Cham: Springer, 2022: 200-217.
[71] ROKACH L. Ensemble-based classifiers [J]. Artificial Intelligence Review, 2010, 33(1/2): 1-39.
[72] KAMNITSAS K, BAI W, FERRANTE E, et al. Ensembles of multiple models and architectures for robust brain tumour segmentation [C]// Proceedings of the Third International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, BrainLes 2017, Held in Conjunction with MICCAI 2017, LNCS 10670. Berlin: Springer, 2018: 450-462.
[73] AYGÜN M, ŞAHIN Y H, Ünal G. Multi modal convolutional neural networks for brain tumor segmentation [EB/OL]. [2023-04-24]. https://arxiv.org/pdf/1809.06191.pdf.
[74] DOUZAS G, BACAO F. Effective data generation for imbalanced learning using conditional generative adversarial networks [J]. Expert Systems with Applications, 2018, 91(C): 464-471.
[75] CHAWLA N V, BOWYER K W, HALL L O, et al. SMOTE: synthetic minority over-sampling technique [J]. Journal of Artificial Intelligence Research, 2002, 16(1): 321-357.
[76] CLÈRIGUES A, VALVERDE S, BERNAL J, et al. Acute and sub-acute stroke lesion segmentation from multimodal MRI [J]. Computer Methods and Programs in Biomedicine, 2020, 194: No.105521.
[77] HE Y, YANG D, ROTH H, et al. DiNTS: differentiable neural network topology search for 3D medical image segmentation [C]// Proceedings of the 2021 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Washington, DC: IEEE Computer Society, 2021: 5841-5850.
[78] CHANG P D. Fully convolutional neural networks with hyperlocal features for brain tumor segmentation [C]// Proceedings of the 19th International Conference on Medical Image Computing and Computer-Assisted Intervention. Berlin: Springer, 2016: 4-9.
[79] ZHOU T, RUAN S, VERA P, et al. A tri-attention fusion guided multi-modal segmentation network [J]. Pattern Recognition, 2022, 124(C): No.108417.
[80] ZHANG D, HUANG G, ZHANG Q, et al. Cross-modality deep feature learning for brain tumor segmentation [J]. Pattern Recognition, 2021, 110: No.107562.
Review of multi-modal medical image segmentation based on deep learning
DOU Meng1,2, CHEN Zhebin1,2, WANG Xin3, ZHOU Jitao3, YAO Yu1,2*
(1,,610213,;2,,100049,;3,,610041,)
Multi-modal medical images can provide clinicians with rich information of target areas (such as tumors, organs or tissues). However, effective fusion and segmentation of multi-modal images is still a challenging problem due to the independence and complementarity of multi-modal images. Traditional image fusion methods have difficulty in addressing this problem, leading to widespread research on deep learning-based multi-modal medical image segmentation algorithms. The multi-modal medical image segmentation task based on deep learning was reviewed in terms of principles, techniques, problems, and prospects. Firstly, the general theory of deep learning and multi-modal medical image segmentation was introduced, including the basic principles and development processes of deep learning and Convolutional Neural Network (CNN), as well as the importance of the multi-modal medical image segmentation task. Secondly, the key concepts of multi-modal medical image segmentation was described, including data dimension, preprocessing, data enhancement, loss function, and post-processing, etc. Thirdly, different multi-modal segmentation networks based on different fusion strategies were summarized and analyzed. Finally, several common problems in medical image segmentation were discussed, the summary and prospects for future research were given.
deep learning; multi-modal; medical image; image fusion; image segmentation
1001-9081(2023)11-3385-11
10.11772/j.issn.1001-9081.2022101636
2022⁃11⁃04;
2023⁃04⁃26;
国家自然科学基金资助项目(82073338); 四川省科技计划项目重点研发项目(2022YFS0217)。
窦猛(1993—),男,山东滨州人,博士研究生,主要研究方向:医学图像分析、深度学习; 陈哲彬(1993—),男,贵州遵义人,博士研究生,主要研究方向:医学图像分析、深度学习; 王辛(1977—),女,四川成都人,教授,博士,主要研究方向:直肠肿瘤诊断与治疗; 周继陶(1985—),女,四川成都人,主治医师,博士,主要研究方向:直肠肿瘤诊断与治疗; 姚宇(1980—),男,四川宜宾人,教授,博士,主要研究方向:医学图像分析、机器学习。
TP183
A
2023⁃05⁃04。
This work is partially supported by National Natural Science Foundation of China (82073338), Key Research and Development Project of Sichuan Science and Technology Plan (2022YFS0217).
DOU Meng, born in 1993, Ph. D. candidate. His research interests include medical image analysis, deep learning.
CHEN Zhebin, born in 1993, Ph. D. candidate. His research interests include medical image analysis, deep learning.
WANG Xin, born in 1977, Ph. D., professor. Her research interests include diagnosis and treatment of rectal tumors.
ZHOU Jitao, born in 1985, Ph. D., attending physician. Her research interests include diagnosis and treatment of rectal tumors.
YAO Yu,born in 1980, Ph. D., professor. His research interests include medical image analysis, machine learning.
