小麦穗长主效QTL—qSl-2D的遗传和育种选择效应解析
2023-11-18董继梓陈林渠郭浩儒张梦宇刘志霄韩磊田赵飒爽徐宁浩郭庆杰黄振洁杨傲宇赵春华吴永振孙晗秦冉崔法
董继梓,陈林渠,郭浩儒,张梦宇,刘志霄,韩磊,田赵飒爽,徐宁浩,郭庆杰,黄振洁,杨傲宇,赵春华,吴永振,孙晗,秦冉,崔法
小麦穗长主效QTL的遗传和育种选择效应解析
董继梓,陈林渠,郭浩儒,张梦宇,刘志霄,韩磊,田赵飒爽,徐宁浩,郭庆杰,黄振洁,杨傲宇,赵春华,吴永振,孙晗,秦冉,崔法
鲁东大学农学院/山东省高等学校作物高产抗逆分子模块重点实验室,山东烟台 264025
【目的】通过对小麦穗长稳定主效QTL进行遗传及育种选择效应分析,明确其对产量性状的遗传效应,评价其未来育种应用潜力,为后续基因挖掘和小麦分子育种提供依据。【方法】利用科农9204×京411构建的重组自交系(recombinant inbred lines derived from the cross of Kenong 9204 and Jing 411,KJ-RIL)群体定位到一个多环境稳定表达的穗长主效QTL,命名为;利用双亲靶区间序列差异InDel位点开发出2个与该QTL紧密连锁的分子标记;结合分子标记及55K芯片基因型数据,分别进行基于KJ-RIL、MY-F2、NILs及自然作图群体的产量相关性状遗传效应分析;基于自然作图群体基因分型,分析单倍型在各麦区及不同年代的育种选择效应。【结果】QTL定位结果表明,可在7/10组环境数据中被检测到,可解释4.02%—10.10%的表型变异。其中,5/10组环境数据的LOD峰值均位于608.75 Mb处。遗传效应分析结果表明,增效等位基因型在4个群体遗传背景下均能显著增加穗长。此外,其在大部分群体背景下对穗粒数、株高有正向效应,而对千粒重、穗粒重和单株产量有负向效应。对KJ-RIL群体株高进一步分析发现,增效等位基因型对株高效应未达到显著水平的原因在于其对除穗下节间长以外的各节间长都有降秆效应;单倍型分析结果表明,长穗单倍型Hap-AA-GG在不同麦区中选择利用率差异较大,在北部冬麦区中选择利用率最高,占比24%;而短穗单倍型Hap-CC-CC在大部分麦区中占比30%以上。此外,随着年代的递进,长穗单倍型选择利用率逐渐降低,而短穗单倍型一直保持较高的选择利用率。【结论】定位到一个稳定主效的穗长QTL——,其增效等位基因型可在不同遗传背景下显著增加穗长,同时对其他产量相关性状有一定的遗传效应。靶区段开发的紧密连锁分子标记可用于小麦穗长及相关产量性状的遗传改良。
小麦(L.);穗长;主效QTL;遗传效应解析;分子标记
0 引言
【研究意义】小麦是一种在世界各地广泛种植的谷类作物,是世界上重要的粮食作物之一,在所有的人类食物中提供约20%的蛋白质和卡路里[1]。目前,全球极端、恶劣气候频发,城市扩大不断挤压耕地,人口数量激增,小麦产量每年需要以1.7%的速度增长(https://iwyp.org)才能满足全球口粮需求。因此,小麦单产的提高成了保障粮食增产、人民基本物质生活的前提。穗粒数、千粒重和亩穗数是小麦产量构成三要素[2]。穗长是小麦穗部的一个重要农艺性状,影响小穗着生的空间,与小麦穗粒数密切相关[3]。许多育种家将穗长性状作为育种过程中考查的一个重要指标。对穗长QTL精细定位及遗传效应分析,可为进一步解析小麦穗长遗传调控机制奠定基础,同时也为现代小麦分子育种提供基因标记资源。【前人研究进展】目前,已报道的诸多与小麦穗部性状相关的QTL分布于小麦21条染色体[4-11]。Ma等[12]基于RIL群体,检测到一个穗长相关的主效QTL,定位于7D染色体—,解释了29.7%—36.3%的表型变异。该QTL在F2群体中也表现出较大的效应,解释了31.4%的表型变异。Wu等[13]研究发现,与穗长相关的主效或稳定QTL,分布在2D、3A、4A、4B、5A、6A、6B、7A、7B和7D染色体上。王佳佳[14]利用MS-BC3F2:3、MT群体在4B染色体—区间检测到1个QTL簇,涉及穗粒数、不育小穗数、可育小穗数、穗长和株高。Zhang等[15]利用M8008×YN15(MY群体)和M8008×石家庄54(MS群体)2个RIL群体进行QTL分析,在—区间检测到1个控制穗长、粒宽、籽粒长宽比和千粒重的QTL簇。其中,和优异等位基因来自M8008,和优异等位基因来自YN15。Zhai等[16]利用191个家系组成的F9重组自交系群体经多环境联合分析,发现穗长QTL——与矮秆基因定位区间一致,可能为相同的基因,在不改变小穗数的情况下,可降低穗长长度。程瑞如[17]发现是1个控制穗长的基因,位于2DS染色体的0.9 cM区间。李聪等[18]利用3年8个环境表型数据共检测出44个控制穗长和株高的QTL。其中,稳定的穗长QTL有5个,分布于2D、4B和5B染色体上。柴岭岭[19]以普通小麦豫麦8679和京411及其衍生的剩余杂合体和近等基因系为材料,精细定位了2D染色体短臂上的一个同时控制株高和穗长的QTL,即,进一步明确了单位点水平上的遗传效应,并采用图位克隆策略鉴定出区间内的候选基因,初步确定了功能变异位点。水志杰等[20]以普通小麦品种西农389×人工合成小麦材料KU98衍生的F7:8RIL群体为试验材料,基于小麦55K SNP芯片对该群体进行基因分型,对小麦穗长进行了QTL定位,在1A、2D、3A、5A和7B染色体上共检测到10个与穗长性状相关的QTL。姚俭昕[21]以小偃81和西农1376构建的包含190个株系的F10:11RIL群体为材料,检测到3个控制穗长的QTL,分别为、和。吕栋云[22]基于50K和90K SNP芯片,以分别含有198和102个株系的西农1376×小偃81和周麦8425B×小偃81 2个RIL群体为材料,共检测到36个控制穗长的位点。Ji等[23]利用13F10×CM42构建的RIL家系在5A染色体上将穗长QTL定位到518.43—525.12 Mb。Li等[9]利用CM42×CKM1构建的CK1群体检测到2个与穗长相关的主效稳定QTL,分别解释12.27%—29.49%和7.13%—9.37%的表型变异,2个位点的优异等位基因由CM42贡献。陈黄鑫等[24]以硬粒小麦矮兰麦和野生二粒小麦LM001构建的F8RIL群体为材料检测到17个与穗长相关的QTL,分布在2A、2B、3A、4A、4B、5A和6B染色体上,可解释6.52%17.10%的表型变异。胡文静等[25]以扬麦13和CIMMYT引进种质人工合成小麦衍生系C615为亲本构建重组自交系群体,经QTL分析,检测到1个每穗结实总小穗数、2个穗长、2个结实小穗着生密度和3个株高QTL,并发现多个QTL共定位的现象,如每穗结实总小穗数位点与株高位点、穗长位点、结实小穗着生密度位点与株高位点及穗长位点与结实小穗着生密度位点。【本研究切入点】小麦穗长为复杂的数量性状,受多基因控制,由于其遗传力相对较高[26],可利用数量遗传学方法对控制穗长的主效基因进行挖掘。但小麦基因组庞大复杂,因此,关于穗长相关基因的定位和克隆报道较少,且其对产量性状的遗传效应及育种选择效应研究还不够深入。前期利用KJ-RIL群体,结合高密度遗传连锁图谱基因型值及多环境表型数据,挖掘了一批小麦产量性状QTL位点[8, 27-28]。【拟解决的关键问题】基于前期研究,本研究将遗传图谱中标记序列比对至科农9204基因组,获得基于科农9204基因组的物理图谱。基于物理图谱和表型数据对小麦穗长重新精准定位,多环境中均能在小麦2D染色体检测到一个控制穗长的主效QTL——;本研究进一步解析对产量性状的遗传效应,分析其育种选择效应特征,并开发与紧密连锁的可用于小麦分子育种的分子标记。
1 材料与方法
1.1 试验材料
供试材料包括以科农9204×京411构建的F8重组自交系群体,命名为KJ-RIL群体。该群体包含188个家系,由于基因型取舍,KJ129家系未用于本研究;以绵37×烟农999构建的F2群体,命名为MY-F2群体,该群体包含196个家系;靶区段剩余杂合体经自交获得近等基因系(near-isogenic line,NILs)群体。此外,鲁东大学小麦分子育种团队收集并保存了190份小麦育成品种(系)组成的自然作图群体[29-31],其中,北部麦区29份材料,黄淮冬麦区93份材料,青藏春冬麦区23份材料,西南冬麦区30份材料,长江中下游冬麦区6份材料,西北春麦区2份材料,海外地区7份材料。
1.2 田间设计及农艺性状考种
KJ-RIL群体种植环境涉及3个试验基地:石家庄、北京和新乡。每个地点设置高氮(high nitrogen,HN)和低氮(low nitrogen,LN)2个氮素水平,在每个高氮地块中,播种前,施磷酸二铵300 kg·hm-2、尿素150 kg·hm-2,在拔节阶段,施用尿素150 kg·hm-2。在LN样地中,整个生育期均未施用氮肥。各个环境根据种植时间顺序命名为E1—E8。其中,E1、E3、E5、E7为低氮环境,分别为2011—2012年石家庄、2012—2013年石家庄、2012—2013年北京、2013—2014年新乡;E2、E4、E6、E8为高氮环境,分别为2011—2012年石家庄、2012—2013年石家庄、2012—2013年北京、2013—2014年新乡[32]。190份小麦育成品种(系)组成的自然作图群体在5个环境下进行性状表型鉴定,分别为2017—2018年鲁东大学栖霞试验基地、2018—2019年潍坊农科院试验基地、2018—2019年烟台瀑拉谷试验基地环境一和环境二、2019—2020年鲁东大学试验基地。对上述2个群体所鉴定的性状包括穗长、小穗数、小穗密度、株高、穗粒数、单株穗数、穗粒重、千粒重和单株产量;此外,对KJ-RIL群体,还测定了不同环境下(E9—E12)的节间长度,包括穗下节间长、倒二节间长、倒三节间长、倒四节间长和倒五节间长,其中,E9、E10环境为2013—2014年石家庄,E11、E12环境为2014—2015年石家庄[33]。选用的MY-F2群体于2020—2021年种植在烟台瀑拉谷试验基地,所鉴定的性状包括穗长、株高、穗粒数、每穗小穗数、千粒重和小穗密度。选用的NILs群体于2021—2022年种植在烟台市农业科学院试验基地,所鉴定的性状包括穗长、株高、穗粒数、每穗小穗数、单株穗数、千粒重和小穗密度。
以上试验材料的种植均遵循随机完全区组设计,2次重复。行长2 m,行距0.25 m,田间管理均参照当地管理规格进行。相关材料的产量相关性状调查方法参考Fan等[8]报道。
1.3 基因型的获取、QTL定位及分子标记开发
利用CTAB法提取小麦叶片DNA。采用660K SNP芯片对科农9204、京411和187个KJ-RIL家系基因分型,采用小麦55K Affymetrix芯片对190份育成品种(系)基因型分型。
基于KJ-RIL群体小麦660K芯片基因型数据,前期获得小麦高密度遗传连锁图谱[27]。利用本地BLAST(https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/)对SNP侧翼序列进行比对分析,获得基于科农9204基因组每个SNP的物理位置信息[34]。通过去冗余分析,获得包含7 141个标记的物理图谱,用于穗长QTL定位。按照IciMapping v4.2中BIP格式将187个KJ-RIL家系的基因型数据及各环境表型数据进行整理。QTL检测以1.0 Mb为步长,进行1 000次置换检验,值包含阈值为0.001,I型误差为0.05,最终确定LOD阈值。
利用科农9204和京411全基因组重测序数据,获得亲本间序列插入缺失(insertion-deletion,InDel)位点信息;选择目标QTL区段内双亲间序列差异≥5 bp的InDel位点,利用多组学网站WheatOmics1.0(http://202.194.139.32/)中的PrimerServer工具进行多态性引物的设计。利用开发的分子标记对KJ-RIL群体及双亲DNA样本进行PCR反应,产物用非变性聚丙烯酰胺凝胶电泳检测。PCR反应程序为95 ℃ 5 min;95 ℃ 30 s,57 ℃ 30 s,72 ℃ 40 s,34个循环;72 ℃ 5 min,12 ℃保存。
1.4 数据处理与分析
利用分子标记对群体基因型进行鉴定,将KJ-RIL群体中与科农9204基因型一致的命名为Hap-9204,与京411基因型一致的命名为Hap-J411;NILs群体中与科农9204基因型一致的命名为NIL-KN9204,与京411基因型一致的命名为NIL-J411;将MY-F2群体中与M37基因型一致的命名为Hap-M37,与YN999基因型一致的命名为Hap-YN999。利用LOD峰值附近的2个SNP标记(A/C)和(C/G)对自然群体进行基因型分类,将与京411基因型相同的Hap-AA-GG定义为长穗单倍型,与科农9204基因型相同的Hap-CC-CC定义为短穗单倍型,Hap-AA-CC或Hap-CC-GG定义为重组单倍型,基因位点存在缺失的定义为缺失单倍型,基因位点存在杂合的定义为杂合单倍型。
将KJ-RIL群体8个环境下的穗长及其他表型数据按照高氮、低氮分类汇总,运用QGA Station[35]分别获得高氮、低氮水平下的最佳线性无偏估计值(best linear unbiased estimator,BLUE),记为HN-BLUE和LN-BLUE。联合上述基因型值及各环境的表型数据进行QTL定位分析和基于KJ-RIL群体遗传效应分析;另对190个育成品种(系)组成的自然群体5个环境下的穗长及其他表型数据进行BLUE分析,分别获得对应性状的BLUE值,用于的遗传效应和育种选择效应分析研究。利用Excel软件对数据进行统计分析,采用Student’s检验进行显著性分析。
2 结果
2.1 穗长主效QTL——qSl-2D精准定位及紧密连锁PCR分子标记开发
为了对穗长QTL进行精准定位,利用基于科农9204基因组的物理图谱对KJ-RIL群体10组穗长数据进行QTL分析,发现在7组数据中均能检测到小麦2D染色体上控制穗长的QTL,包括3个低氮环境(E1、E3和E7)、2个高氮环境(E4和E8)及高、低氮BLUE值数据。定位区间为KN2D:576.25—610.25 Mb,其中,6/10组环境数据将QTL定位于—间2 Mb的区间内,对应科农9204基因组KN2D:608.25—610.25 Mb,5/10组数据(E1、E3、E4、E8、HN-BLUE)的LOD峰值均位于608.75 Mb(表1和图1)。其LOD值为5.63—10.13,可解释4.02%— 10.10%的表型变异,加性效应为-0.25—-0.19,表明其增效等位基因型来自京411。以上结果表明,QTL是一个多环境下稳定表达的穗长主效QTL。
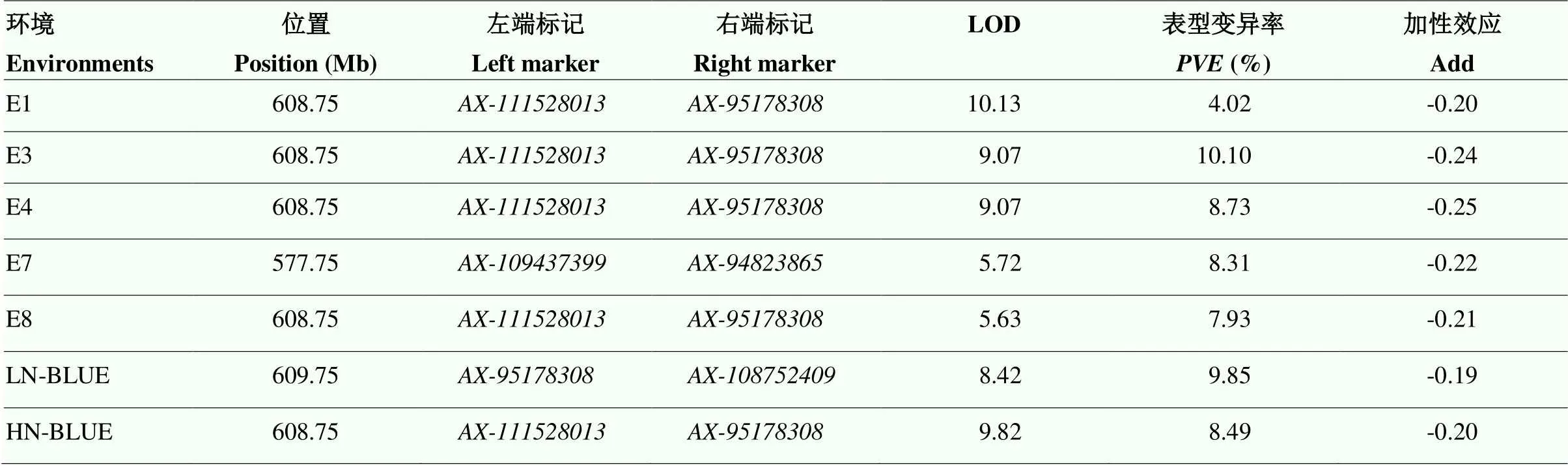
表1 多环境下穗长QTL——qSl-2D初级定位分析
位置代表LOD峰值在科农9204基因组上的物理位置。LN:低氮环境;HN:高氮环境
Position represents the physical location of the LOD peak on Kenong 9204 genome. LN: low nitrogenenvironment; HN: high nitrogen environment

横坐标为660K芯片SNP标记对应科农9204基因组物理位置。下面红色染色体区段表示定位区间
利用科农9204和京411基因组重测序数据筛选出靶区间内的InDel位点591 382 416和608 064 987 bp,基于网站WheatOmics1.0的PrimerServer功能设计出双亲间多态性分子标记(F:5′-GGGGTCGA CGTTCTTGATCT-3′和R:5′-CCGGCACCTTCAACCT TCAA-3′)和(F:5′-TGCCTCGCTGAAACTGAC TT-3′和R:5′-GTCTGACCCCTGTGTTCCTG-3′)。对KJ-RIL亲本及群体家系进行PCR扩增。结果表明,2个标记均能扩增出清晰条带,且分离效果较好(图2)。其中,在科农9204和京411中分别扩增出240和209 bp的目的片段,在科农9204和京411中分别扩增出323和347 bp的目的片段。将2个分子标记基因型数据和表型数据关联分析,发现来自京411的增效等位基因型在8/10组数据中均可显著增加穗长(图3),包括3个高氮环境(E2、E4和E8)、3个低氮环境(E1、E3和E7)及高、低氮BLUE值数据。以上结果表明,2个紧密连锁的分子标记可以与穗长显著关联,因此,可用于穗长分子标记辅助选择育种及相关性状遗传效应分析。

M:Marker;1—46:KJ-RIL群体部分家系扩增结果;K:科农9204,J:京411
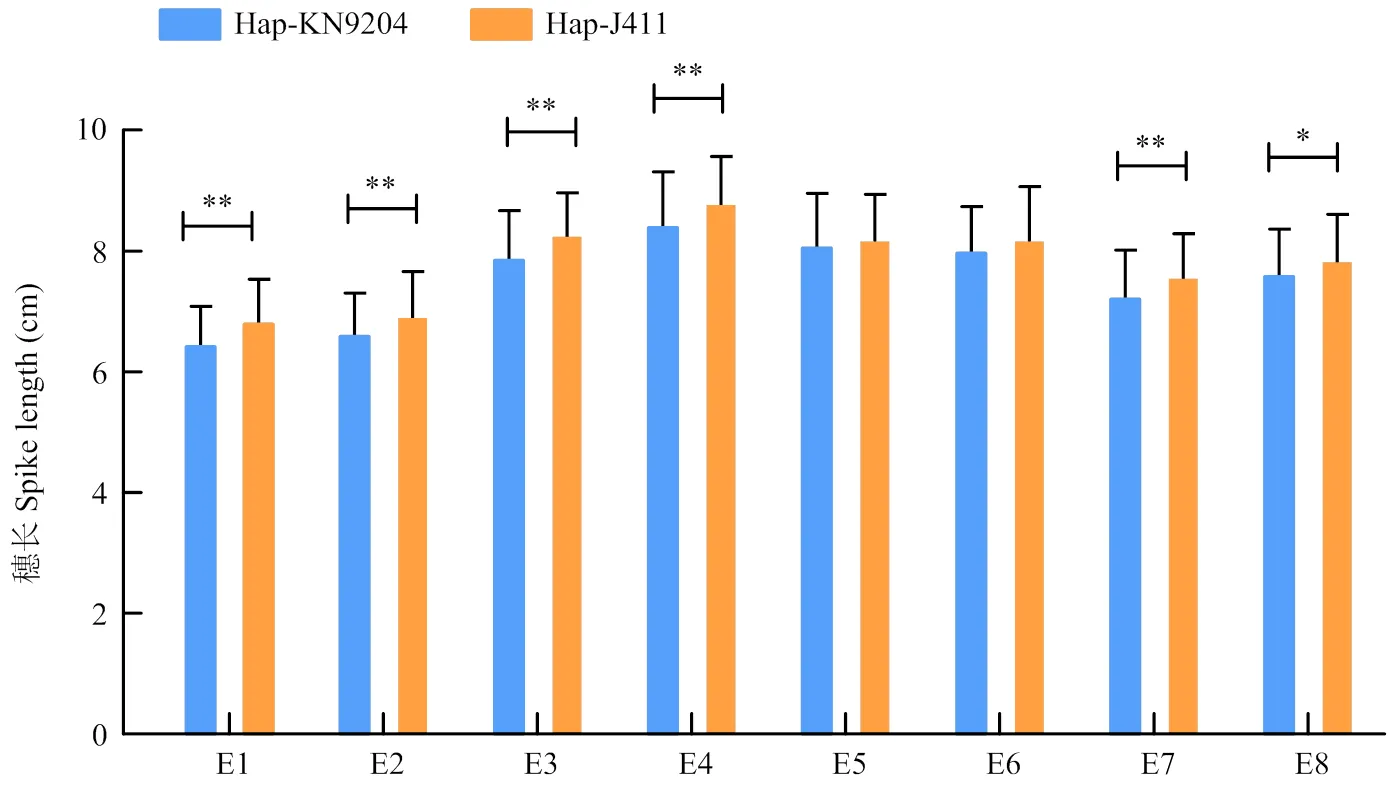
Hap-KN9204:与科农9204相同基因型,Hap-J411:与京411相同的基因型;*、**分别表示在P<0.05和P<0.01水平差异显著。下同
2.2 qSl-2D对小麦产量相关性状遗传效应解析
2.2.1 基于KJ-RIL群体对小麦产量相关性状遗传效应分析 利用靶区段2个分子标记在KJ-RIL群体的基因型数据与穗长等其他产量性状BLUE值进行关联分析,结果发现,高、低氮条件下,来自京411的增效等位基因型均能显著增加穗长(图4)。其对穗粒数有正向效应,对穗粒重、千粒重、株高及单株产量有负效应,但仅有高、低氮环境下千粒重及高氮环境下穗粒重达到显著水平。因为株高由穗长及各节间长度共同决定,所以继续分析了该位点对各节间长的遗传效应。结果表明,增效等位基因型与穗下节间长呈正相关,但与倒二节间长、倒三节间长、倒四节间长和倒五节间长呈负相关(表2)。

图4 基于KJ-RIL群体qSl-2D对产量相关性状的遗传效应分析
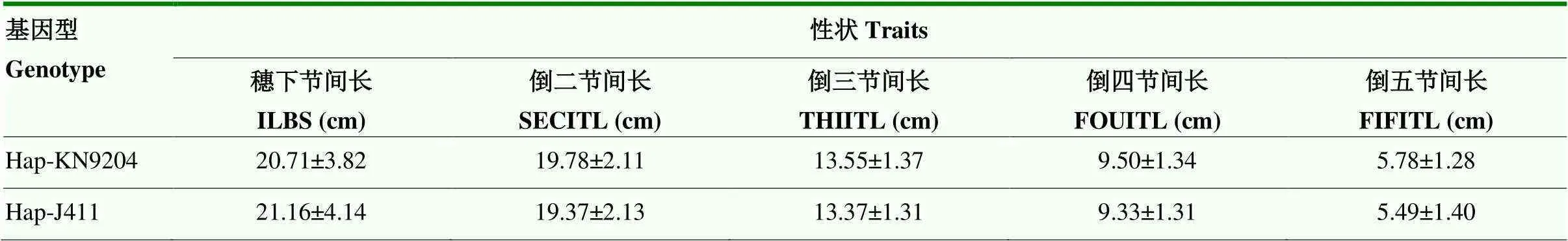
表2 基于KJ-RIL群体qSl-2D对各节间长度遗传效应分析
ILBS:穗下节间长;SECITL:倒二节间长;THIITL:倒三节间长;FOUITL:倒四节间长;FIFITL:倒五节间长
ILBS: Internode length below the spike; SECITL: Second internode length from up; THIITL: Third internode length from up; FOUITL: Fourth internode length from up; FIFITL: Fifth internode length from up
2.2.2 基于NILs群体对小麦产量相关性状遗传效应分析 在靶区段剩余杂合体自交构建的NILs分离群体中,利用和分子标记分别鉴定到短穗基因型NIL-KN9204和长穗基因型NIL-J411。比较发现,来自京411的增效等位基因型可以显著增加穗长(表3)。此外,其对株高、穗粒数、每穗小穗数和单株穗数有增加的效应,对千粒重和小穗密度有降低的效应,但均未达到显著水平。

表3 基于NILs群体qSl-2D对产量相关性状的遗传效应分析
NIL-KN9204:与科农9204相同的基因型;NIL-J411:与京411相同的基因型;SL:穗长;PH:株高;KNPS:穗粒数;SNPS:每穗小穗数;SN:单株穗数;TKW:千粒重;SD:小穗密度。*、**分别表示在<0.05和<0.01水平差异显著。下同
NIL-KN9204: genotype identical to Kenong 9204, NIL-J411: genotype identical to Jing 411; SL: Spike length; PH: Plant height; KNPS: Kernel number per spike; SNPS: Spikelet number per spike; SN: Spikes number; TKW: Thousand kernel weight; SD: Spikelet density. *, **: Significant differences at<0.05 and<0.01 levels, respectively. The same as below
2.2.3 基于MY-F2群体对小麦产量相关性状遗传效应分析 为了进一步解析的遗传效应,利用和对MY-F2群体进行基因型鉴定。通过与表型数据关联分析发现,来自绵37的增效等位基因型可以显著增加穗长、株高和小穗密度(表4)。此外,其对穗粒数、每穗小穗数和千粒重有增加的效应,但未达到显著水平。

表4 基于MY-F2群体qSl-2D对产量相关性状的遗传效应分析
Hap-M37:与绵37相同的基因型;Hap-YN999:与烟农999相同的基因型
Hap-M37: genotype identical to Mian 37; Hap-YN999: genotype identical to Yannong 999
2.2.4 基于自然作图群体对小麦产量相关性状遗传效应分析 为进一步验证的遗传效应,选择190份不同年代育成品种(系)组成的自然作图群体进行分析。在靶区间内选择LOD峰值附近的2个SNP标记(A/C)和(C/G),根据190份育成品种(系)基因型值进行分类汇总,对进行单倍型分析。结果表明,与短穗单倍型Hap-CC-CC相比,长穗单倍型Hap-AA-GG穗长显著增加。此外,其对穗粒数、单株穗数和株高有正向效应,而对小穗密度、穗粒重、千粒重和单株产量有负向效应,但仅小穗密度达到显著水平(图5)。
2.3 qSl-2D在小麦育种中的选择效应分析
为了解在小麦育种中的选择效应,分别从地域分布和时间跨度上对自然作图群体进行单倍型分析。首先,根据品种来源和地域分布信息将190份自然群体划分为7个大区,分别是北部冬麦区、黄淮冬麦区、青藏春冬麦区、西南冬麦区、长江中下游冬麦区、西北春麦区和海外地区。由于长江中下游冬麦区、西北春麦区、海外地区的材料较少,不具备代表性,本研究未进行计算。结果表明,北部冬麦区对长穗单倍型的选择利用率最高,长穗单倍型Hap-AA-GG占比为24%,其次为青藏春冬麦区(22%)、黄淮冬麦区(15%)、西南冬麦区(10%)(图6);除北部冬麦区(14%)外,其他3个麦区中靶区段短穗单倍型Hap-CC-CC占比均在30%以上,说明其在大部分地区受到了强烈选择。

Hap-CC-CC:短穗单倍型;Hap-AA-GG:长穗单倍型;图中数字表示平均值,图中横线表示均值线
其次,根据品种审定时间对190份自然群体材料进行分类,分析单倍型在各年代内的育种选择效应及育种应用情况。在20世纪90年代,长穗单倍型Hap-AA-GG选择利用率最高,占比为22%,短穗单倍型Hap-CC-CC占比22%;在21世纪00年代,长穗单倍型占比16%,短穗单倍型占比45%;而在21世纪10年代,长穗单倍型占比降到13%,短穗单倍型占比38%。结果表明,随着年代的递进,长穗单倍型Hap-AA-GG选择利用率逐渐减少(图7),而短穗单倍型Hap-CC-CC一直保持较高的选择利用率。
3 讨论
3.1 qSl-2D是一个稳定主效QTL
国内外学者在不同环境下鉴定了大量的小麦穗长QTL,并对此进行了解析[12-25]。基于构建的高密度遗传连锁图谱[27],课题组前期利用去除冗余后包含4 959个标记的遗传连锁图谱,在KJ-RIL群体中检测到了穗长QTL——[8],对应中国春染色体2D:592.66—623.65 Mb置信区间;为了获得精准的QTL位置,本研究基于660K SNP芯片构建的高密度遗传图谱转化而成的基于科农9204基因组的物理图谱,对进行了重新精准定位。此位点在7/10组环境数据中均能被检测到,置信区间为KN2D:576.25— 610.25 Mb。在5/7组环境数据(71.4%)中将其定位于KN2D:608.25—610.25 Mb的2 Mb区间内,对应中国春2D:607.55—609.55 Mb,与前期定位结果相比区间进一步缩小。随后,对小麦2D染色体已发表穗长QTL进行了汇总比对(表5)[10, 13, 15, 17-21, 36-39],发现其与前人的定位区间存在部分重叠,可能为相同位点。如Zhang等[15]利用烟农15突变体群体在593.74 Mb附近检测到一个穗长QTL,李聪等[18]在602.14—606.92 Mb检测到穗长位点。Liu等[39]在将穗长定位于585.69—601.05 Mb,但均未见该位点进一步遗传解析的相关报道。本研究与前人定位结果存在重叠,说明该位点在不同遗传背景下均能被检测到,说明其是一个稳定主效的QTL,也暗示其在调控穗长方面发挥了重要作用。的初级定位结果可为其精细定位及基因候选提供重要参考信息。
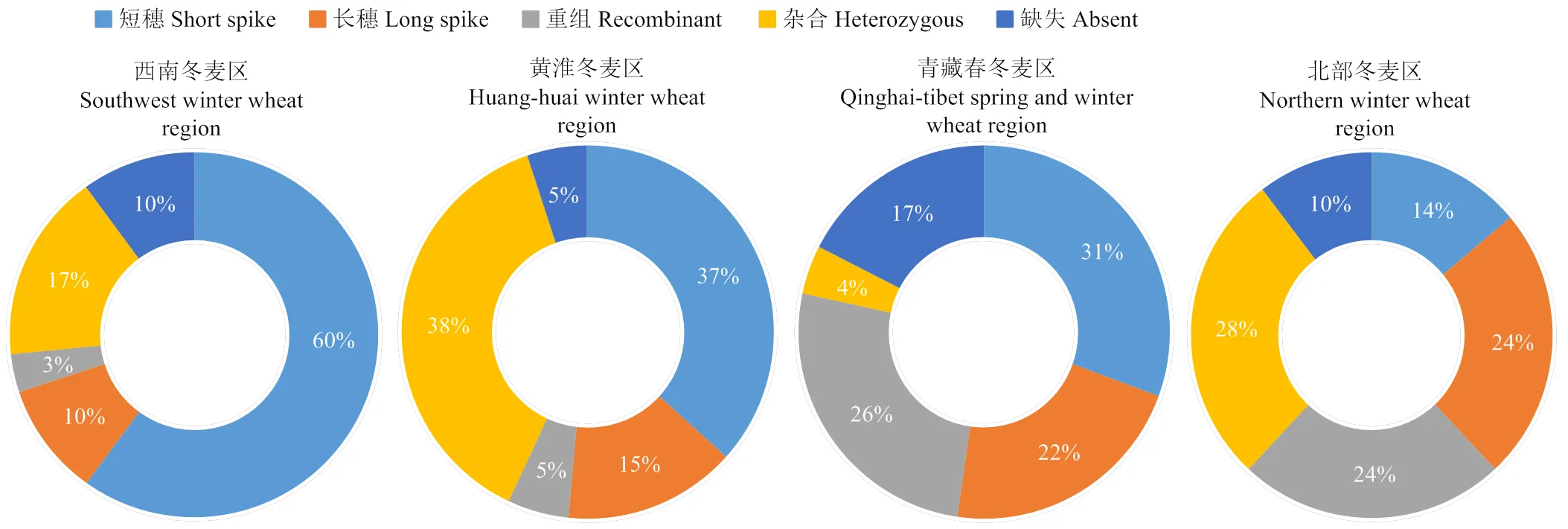
图6 qSl-2D单倍型在不同麦区选择效应分析
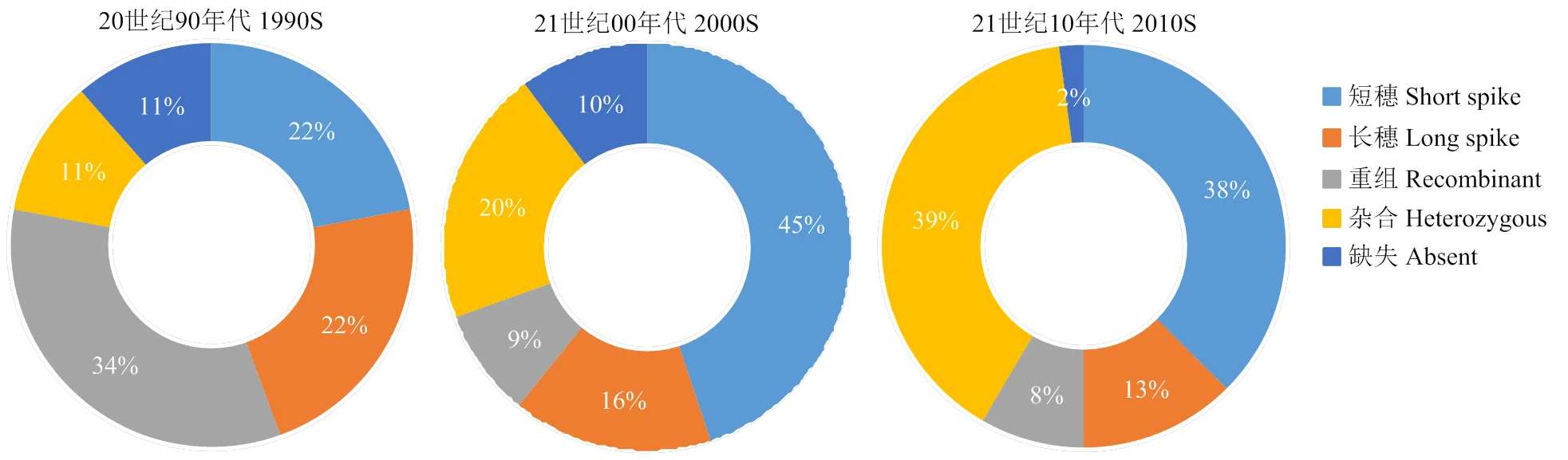
图7 qSl-2D单倍型在不同年代审定品种中的选择效应分析
3.2 QTL——qSl-2D的遗传效应
在KJ-RIL群体中,来自亲本京411的增效等位基因型在高、低氮条件下均能显著增加穗长,虽对株高有降低趋势但未达到显著水平。针对这一问题,对KJ-RIL群体株高的各节间长进行分析,发现增效等位基因型在除穗下节间长以外的其他节间都有一定的降杆作用。为进一步验证遗传效应选择NILs群体、绵37×烟农999构建的MY-F2群体和190份育成品种(系)组成的自然作图群体进行分析。与KJ-RIL群体结果一致,增效等位基因型可显著增加穗长。不同的是,MY-F2群体中增效等位基因型可使株高显著增加,而在NILs群体和自然群体中该效应未达到显著水平;此外,在KJ-RIL群体、NILs群体及自然群体中,即使增效等位基因型可以增加每穗小穗数,但由于对穗长有显著增效作用,而对小穗密度表现出负效应,且在后者中达到显著水平。MY-F2群体中则表现出小穗密度显著增加的趋势;在千粒重方面,KJ-RIL群体、NILs群体和自然群体中,增效等位基因型对千粒重呈负效应,且前者达到显著水平,但在MY-F2群体中表现出结果相反的趋势;在不同背景群体中,增效等位基因型均可增加穗粒数,这也得益于穗长的增加有利于小穗生长空间的扩大及颖花数的增加。但由于增效等位基因型对千粒重的负向效应导致单株产量下降,但未达到显著水平。综上,在不同群体背景下遗传效应基本一致,存在的轻微变化可能是由于受环境和群体背景的影响导致的。因此,推测是一个受环境、基因型和遗传背景多条件影响的位点,以上结果为其未来分子应用提供参考。
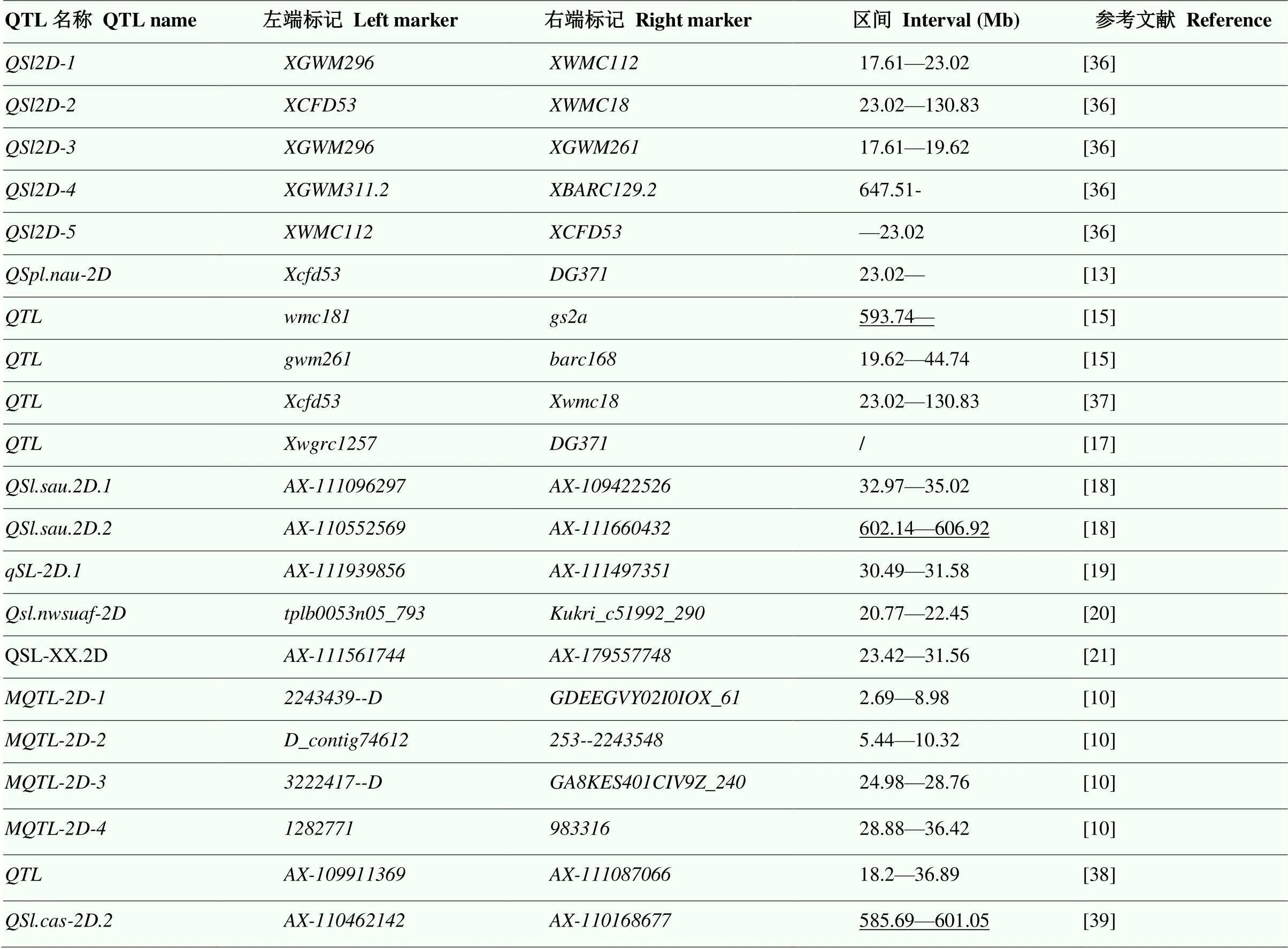
表5 小麦2D染色体上已报道穗长QTL定位信息
物理区间参考中国春基因组(IWGSC RefSeq v1.0);下划线表示与前人定位区间存在重叠
The physical interval referring to Chinese spring genome (IWGSC RefSeq v1.0); The intervals underlined indicate thatoverlaps with previous positioning intervals
3.3 QTL——qSl-2D的育种选择效应
在190份自然群体育种选择效应分析中,长穗单倍型Hap-AA-GG在北部冬麦区、西南冬麦区、黄淮冬麦区及青藏春冬麦区利用率均较低(10%—24%)(图6)。短穗单倍型Hap-CC-CC在除北部冬麦区(14%)外的其他3个麦区中占比均在30%以上,说明其受到了强烈选择;此外,190份自然群体在不同年代中的单倍型分析结果表明,随着时间的递进,育成品种数量不断增加,但长穗单倍型Hap-AA-GG的利用率逐渐下降,而短穗单倍型Hap-CC-CC却在不同年代中均占有较大比例(22%— 45%),推测可能由于长穗单倍型对单株产量和千粒重有负向效应,导致其在现代育成品种(系)中不被育种家重点选择。短穗单倍型Hap-CC-CC对株高有负效应,这与育种家选择半矮秆优良小麦品种的改良目标相契合,因此,在不同地域及年代受到强烈选择。株高的降低往往可以增强小麦的抗倒伏能力,提高收获指数,保障小麦的稳产。20世纪中叶以来,()和()等矮秆基因的育种利用带来了小麦的“绿色革命”,为粮食产量的大幅度增加带来契机,解决了大范围的全球粮食危机[40-41]。本研究穗长等位基因相关遗传研究及分子标记的开发可为基于分子育种的小麦遗传改良提供依据和标记资源。
4 结论
穗长QTL——是一个稳定主效的QTL,其增效等位基因型可在不同遗传背景下显著增加穗长,同时对其他产量相关性状有一定的遗传效应。通过开发的靶区段紧密连锁分子标记可用于小麦穗长及相关产量性状的遗传改良。
[1] 范涛, 李治, 蒋庆, 陈姝霖, 欧霞, 陈永艳, 任天恒. 小麦单位面积穗数和粒长主效QTL紧密连锁KASP标记的开发及其效应评价. 中国农业科学, 2021, 54(14): 2941-2951.
FAN T, LI Z, JIANG Q, CHEN S L, OU X, CHEN Y Y, REN T H.Development and effect evaluation of KASP markers closely linked to major QTLs of spike number per unit area and grain length in wheat. Scientia Agricultura Sinica, 2021, 54(14): 2941-2951. (in Chinese)
[2] 刘凯, 邓志英, 李青芳, 张莹, 孙彩铃, 田纪春, 陈建省. 利用高密度SNP遗传图谱定位小麦穗部性状基因. 作物学报, 2016, 42(6): 820-831.
LIU K, DENG Z Y, LI Q F, ZHANG Y, SUN C L, TIAN J C, CHEN J S. Mapping QTLs for wheat panicle traits with high density SNP genetic map. Acta Agronomica Sinica, 2016, 42(6): 820-831. (in Chinese)
[3] 杨光. 四倍体和六倍体小麦穗部形态性状的比较及其演化规律研究[D]. 杨凌: 西北农林科技大学, 2021.
YANG G. Comparison of morphological characters of ear between tetraploid and hexaploid wheat and study on their evolution law[D]. Yangling: Northwest A&F University, 2021. (in Chinese)
[4] Huang X Q, Kempf H, Ganal M W, RÖDER M S. Advanced backcross qtl analysis in progenies derived from a cross between a German elite winter wheat variety and a synthetic wheat (L.). Theoretical and Applied Genetics, 2004, 109(5): 933-943.
[5] Deng S M, Wu X R, Wu Y Y, Zhou R H, Wang H G, Jia J Z, LIU S B. Characterization and precise mapping of a qtl increasing spike number with pleiotropic effects in wheat. Theoretical and Applied Genetics, 2011, 122(2): 281-289.
[6] CUI F, DING A M, LI J, ZHAO C H, WANG L, WANG X Q, QI X L, LI X F, LI G Y, GAO J R, WANG H G. QTL detection of seven spike-related traits and their genetic correlations in wheat using two related RIL populations. Euphytica, 2012, 186(1): 177-192.
[7] CUI F, ZHAO C H, DING A M, LI J, WANG L, LI X F, BAO Y G, LI J M, WANG H G. Construction of an integrative linkage map and QTL mapping of grain yield-related traits using three related wheat RIL populations. Theoretical and Applied Genetics, 2014, 127(3): 659-675.
[8] FAN X L, CUI F, JI J, ZHANG W, ZHAO X Q, LIU J J, MENG D Y, TONG Y P, WANG T, LI J M. Dissection of pleiotropic QTL regions controlling wheat spike characteristics under different nitrogen treatments using traditional and conditional QTL mapping. Frontiers in Plant Science, 2019, 10: 187.
[9] LI T, DENG G B, SU Y, YANG Z, TANG Y Y, WANG J H, QIU X, PU X, LI J, LIU Z H, ZHANG H L, LIANG J J, YANG W Y, YU M Q, WEI Y M, LONG H. Identification and validation of two major QTLs for spike compactness and length in bread wheat (L) showing pleiotropic effects on yield-related traits. Theoretical and Applied Genetics, 2021, 134(11): 3625-3641.
[10] YANG Y, AMO A, WEI D, CHAI Y M, ZHENG J, QIAO P F, CUI C G, LU S, CHEN L, HU Y G. Large-scale integration of meta-QTL and genome-wide association study discovers the genomic regions and candidate genes for yield and yield-related traits in bread wheat. Theoretical and Applied Genetics, 2021, 134(9): 3083-3109.
[11] XU H W, ZHANG R Q, WANG M M, LI L H, YAN L, WANG Z, ZHU J, CHEN X Y, ZHAO A J, SU Z Q, XING J W, SUN Q X, NI Z F. Identification and characterization of qtl for spike morphological traits, plant height and heading date derived from the D genome of natural and resynthetic allohexaploid wheat. Theoretical and Applied Genetics, 2022, 135(2): 389-403.
[12] Ma Z Q, Zhao D M, Zhang C Q, Zhang Z Z, XUE S L, LIN F, KONG Z X, TIAN D G, Luo Q Y. Molecular genetic analysis of five spike-related traits in wheat using RIL and immortalized F2populations. Molecular Genetics and Genomics, 2007, 277(1): 31-42.
[13] WU X Y, CHENG R R, XUE S L, KONG Z X, WAN H S, LI G Q, HUANG Y L, JIA H Y, JIA J Z, ZHANG L X,MA Z Q. Precise mapping of a quantitative trait locus interval for spike length and grain weight in bread wheat (L). Molecular Breeding, 2014, 33(1): 129-138.
[14] 王佳佳. 小麦基部小穗不育突变体穗部性状的QTL定位[D]. 泰安: 山东农业大学, 2014.
WANG J J. QTL mapping of panicle traits of spikelet sterile mutant at the base of wheat [D]. Taian: Shandong Agricultural University, 2014. (in Chinese)
[15] Zhang G Z, Wang Y Y, GUO Y, ZHAO Y, KONG F M, LI S S. Characterization and mapping of QTLs on chromosome 2D for grain size and yield traits using a mutant line induced by EMS in wheat. The Crop Journal, 2015, 3(2): 135-144.
[16] ZHAI H J, FENG Z Y, LI J, LIU X Y, XIAO S H, NI Z F, SUN Q X. QTL analysis of spike morphological traits and plant height in winter wheat (L) using a high-density SNP and SSR-based linkage map. Frontiers in Plant Science, 2016, 7: 1617.
[17] 程瑞如. 小麦粒形的分子遗传分析及穗长基因的精细定位[D]. 南京: 南京农业大学, 2018.
CHENG R R. Molecular genetic analysis of wheat kernel dimensions and fine mapping of spike length QTL[D]. Nanjing: Nanjing Agricultural University, 2018. (in Chinese)
[18] 李聪, 马建, 刘航, 丁浦洋, 杨聪聪, 张涵, 秦娜娜, 兰秀锦. 基于小麦55K SNP芯片检测小麦穗长和株高性状QTL. 麦类作物学报, 2019, 39(11): 1284-1292.
LI C, MA J, LIU H, DING P Y, YANG C C, ZHANG H, QIN N N, LAN X J. Detection of QTLs for spike length and plant height in wheat based on 55K SNP array. Journal of Triticeae Crops, 2019, 39(11): 1284-1292. (in Chinese)
[19] 柴岭岭. 普通小麦2D染色体株高和穗长QTL的精细定位与图位克隆[D]. 北京: 中国农业大学, 2019.
CHAI L L. Fine mapping and map-based cloning of QTL controlling plant height and spike length on chromosome 2D in common wheat (L) [D]. Beijing: China Agricultural University, 2019. (in Chinese)
[20] 水志杰, 安沛沛, 刘天相, 吴洪启, 刘乐, 史雪, 王中华. 利用人工合成小麦RIL群体进行小麦穗长和穗宽性状的QTL分析. 麦类作物学报, 2020, 40(6): 656-664.
SHUI Z J, AN P P, LIU T X, WU H Q, LIU L, SHI X, WANG Z H. QTL analysis of spike length and width using RIL population of synthetic wheat. Journal of Triticeae Crops 2020, 40(6): 656-664. (in Chinese)
[21] 姚俭昕. 基于50K芯片的小麦穗部及籽粒相关性状的QTL定位[D]. 杨凌: 西北农林科技大学, 2020.
YAO J X. QTL mapping of wheat ear and kernel related traits based on 50K Chips[D]. Yangling: Northwest A&F University, 2020. (in Chinese)
[22] 吕栋云. 基于两个RIL群体的小麦产量相关性状的QTL定位[D]. 杨凌: 西北农林科技大学, 2021.
LÜ D Y. QTL mapping for yield-related traits in wheat based on two RIL populations[D]. Yangling: Northwest A&F University, 2021. (in Chinese)
[23] JI G S, XU Z B, FAN X L, ZHOU Q, YU Q, LIU X F, LIAO S M, FENG B, WANG T. Identification of a major and stable QTL on chromosome 5A confers spike length in wheat (L). Molecular Breeding, 2021, 41(9): 56-68.
[24] 陈黄鑫, 李聪, 吴坤燕, 王岳, 牟杨, 唐华苹, 唐力为, 兰秀锦, 马建. 四倍体小麦株高和穗长性状的QTL定位及其遗传效应分析. 麦类作物学报, 2022, 42(7): 799-807.
CHEN H X, LI C, WU K Y, WANG Y, MU Y, TANG H P, TANG L W, LAN X J, MA J. Detection of QTLs for plant height and spike length in tetraploid wheat and analysis of their genetic effect. Journal of Triticeae Crops, 2022, 42(7): 799-807. (in Chinese)
[25] 胡文静, 李东升, 裔新, 张春梅, 张勇. 小麦穗部性状和株高的QTL定位及育种标记开发和验证. 作物学报, 2022, 48(6): 1346-1356.
HU W J, LI D S, YI X, ZHANG C M, ZHANG Y. Molecular mapping and validation of quantitative trait loci for spike-related traits and plant height in wheat. Acta Agronomica Sinica, 2022, 48(6): 1346-1356. (in Chinese)
[26] 杨在君, 彭丽娟. 小麦三雌蕊突变体主要农艺性状的遗传力分析. 西华师范大学学报(自然科学版), 2013, 34(1): 1-4.
YANG Z J, PENG L J. Heritability analysis of main agronomic traits in common wheat line three pistils. Journal of China West Normal University (Natural Science), 2013, 34(1): 1-4. (in Chinese)
[27] Cui F, Zhang N, Fan X L, Zhang W, Zhao C H, Yang L J, Pan R Q, Chen M, Han J, Zhao X Q, Ji J, Tong Y P, Zhang H X, Jia J Z, Zhao G Y, Li J M. Utilization of a Wheat660K SNP array-derived high-density genetic map for high-resolution mapping of a major QTL for kernel number. Scientific Reports, 2017, 7: 3788.
[28] Cui F, Fan X L, Chen M, Zhang N, Zhao C H, Zhang W, Han J, Ji J, Zhao X Q, Yang L J, Zhao Z W, Tong Y P, Wang T, Li J M. QTL detection for wheat kernel size and quality and the responses of these traits to low nitrogen stress. Theoretical and Applied Genetics, 2016, 129(3): 469-484.
[29] 刘朦朦, 张萌娜, 张倩倩, 刘锡建, 郭宇航, 孙靳惠, 武亚瑞, 王素容, 吴永振, 孙晗, 崔法, 赵春华. 小麦旗叶宽主效QTL-遗传效应解析. 麦类作物学报, 2019, 39(12): 1399-1405.
Liu M M, Zhang M N, Zhang Q Q, Liu X J, Guo Y H, Sun J H, Wu Y R, Wang S R, Wu Y Z, Sun H, Cui F, Zhao C H. Genetic analysis of a major stable QTL-for wheat flag leaf width. Journal of Triticeae Crops, 2019, 39(12): 1399-1405. (in Chinese)
[30] 崔俊鹏, 赵慧, 张倩倩, 宫娜, 刘朦朦, 张萌娜, 侯玉竹, 刘成, 李林志, 周芳婷, 吴永振, 孙晗, 赵春华, 崔法. 小麦穗粒数主效QTL-遗传效应解析. 分子植物育种, 2019, 17(11): 3632-3640.
Cui J P, Zhao H, Zhang Q Q, Gong N, Liu M M, Zhang M N, Hou Y Z, Liu C, Li L Z, Zhou F T, Wu Y Z, Sun H, Zhao C H, Cui F. Genetic effects analysis of major QTL-for kernel per spike in common wheat. Molecular Plant Breeding, 2019, 17(11): 3632-3640. (in Chinese)
[31] 张倩倩, 闫学梅, 刘锡建, 张萌娜, 刘朦朦, 周芳婷, 吴永振, 孙晗, 赵春华, 崔法. 小麦穗粒数主效 QTL-遗传效应解析. 分子植物育种, 2020, 18(15): 5003-5009.
Zhang Q Q, Yan X M, Liu X J, Zhang M N, Liu M M, Zhou F T, Wu Y Z, Sun H, Zhao C H, Cui F. Genetic analysis of: a major stable QTL for kernel number per spike in common wheat. Molecular Plant Breeding, 2020, 18(15): 5003-5009. (in Chinese)
[32] Cui F, Fan X L, Zhao C H, ZHANG W, Chen M, Ji J, Zhang W, Li J M. A novel genetic map of wheat: utility for mapping QTL for yield under different nitrogen treatments. BMC Genetics, 2014, 15: 57.
[33] Zhang N, Fan X L, Cui F, Zhao C H, Zhang W, Zhao X Q, Yang L J, Pan R Q, Chen M, Han J, Ji J, Liu D C, Zhao Z W, Tong Y P, Zhang A M, Wang T, Li J M. Characterization of the temporal and spatial expression of wheat (L.) plant height at the QTL level and their influence on yield-related traits. Theoretical and Applied Genetics, 2017, 130(6): 1235-1252.
[34] Shi X L, Cui F, Han X Y, He Y L, Zhao L, Zhang N, Zhang H, Zhu H D, Liu Z X, Ma B, Zheng S S, Zhang W, Liu J J, Fan X L, Si Y Q, Tian S Q, Niu J Q, Wu H L, Liu X Y, Chen Z, Meng D Y, ZHANG H, Wang X Y, Song L Q, Sun L J, Han J, Zhao H, Ji J, Wang Z G, He X Y, Li R L, Chi X B, Liang C Z, Niu B, Xiao J, Li J M, Ling H. Comparative genomic and transcriptomic analyses uncover the molecular basis of high nitrogen-use efficiency in the wheat cultivar Kenong 9204. Molecular Plant, 2022, 15(9): 1440-1456.
[35] 朱志翔. 遗传分析软件QGAStation2.0和GMDR-GPU的开发[D]. 杭州: 浙江大学, 2012.
ZHU Z X. Development of the genetic analysis software QGAStation 2.0 and GMDR-GPU[D]. Hangzhou: Zhejiang University, 2012. (in Chinese)
[36] 崔勇. 不同施氮期对小麦主要农艺性状影响的QTL分析[D]. 泰安: 山东农业大学, 2013.
CUI Y. QTL mapping for main agronomic traits in Wheat under various nitrogen supplying dates[D]. Taian: Shandong Agricultural University, 2013. (in Chinese)
[37] 刘书含, 侯立江, 华冠勋, 宋瑜龙, 牛娜, 马守才, 宋亚珍, 王军卫, 张改生. 大穗材料高麦1号/密小穗F2群体穗长性状的QTL初步定位. 麦类作物学报, 2016, 36(4): 409-414.
LIU S H, HOU L J, HUA G X, SONG Y L, NIU N, MA S C, SONG Y Z, WANG J W, ZHANG G S. Quantitative trait loci mapping of spike length using F2,population of Gaomai 1/Mixiaosui in Wheat (L.). Journal of Triticeae Crops, 2016, 36 (4): 409-414. (in Chinese)
[38] Xu X, Li X J, Zhang D H, Zhao J S, Jiang X L, Sun H L, Ru Z G. Identification and validation of QTLs for kernel number per spike and spike length in two founder genotypes of wheat. BMC Plant Biology, 2022, 22(1): 146.
[39] Liu H, Shi Z P, Ma F F, Xu Y F, Han G H, Zhang J P, Liu D C, An D G. Identification and validation of plant height, spike length and spike compactness loci in common wheat (L.). BMC Plant Biology, 2022, 22(1): 568.
[40] HEDDEN P. The genes of the Green Revolution. Trends in Genetics, 2003, 19(1): 5-9.
[41] PENG J, RICHARDS D E, HARTLEY N M, MURPHY G P, DEVOS K M, FLINTHAM J E, BEALES J, FISH, L J, WORLAND A J, PELICA F, SUDHAKAR D, CHRISTOU P, SNAPE J W, Gale M D, HARBERD N P. “Green revolution” genes encode mutant gibberellin response modulators. Nature, 1999, 400: 256-261.
Analysis of genetic andbreedingselection effects of amajor QTL-forwheat spike length
DONG JiZi, CHEN LinQu, GUO HaoRu, ZHANG MengYu, LIU ZhiXiao, Han Lei, TIAN ZhaoSaShuang, XU NingHao, GUO QingJie, HUANG ZhenJie, YANG AoYu, ZHAO ChunHua, WU YongZhen, SUN Han, QIN Ran, CUI Fa
School of Agriculture, Ludong University/Key Laboratory of Molecular Module-Based Breeding of High Yield and Abiotic Resistant Plants in Universities of Shandong, Yantai 264025, Shandong
【Objective】By analyzing the genetic and breeding selection effects of the stable major QTL for spike length in wheat, its genetic effects on yield-related traits were clarified, and thefuture breeding application potential was evaluated. The results could provide a basis for subsequent gene mining and molecular breeding of wheat. 【Method】A major QTL for spike length, named, was detected in multiple environments using a recombinant inbred lines population derived from the cross of Kenong9204 and Jing411, denoted as KJ-RIL; Two molecular markers closely linked towere developed by using the InDel sites in target interval. The genetic effects of yield-related traits based on KJ-RIL, MY-F2, NILs and natural mapping populations, were analyzed by combining genotype data of molecular markers or wheat 55K array, respectively. By genotyping the natural mapping population, the breeding selection effect ofhaplotype was parsed across different wheat regions and different ages. 【Result】QTL mapping results showed thatcould be detected in 7/10 sets of environmental data, and could explain 4.02%-10.10% of the phenotypic variation. The peak LOD of 5/10 sets of environmental data was positioned at 608.75 Mb. The results of genetic effect analysis showed that the enhancing allele ofcould significantly increase spike length in the four populations with different genetic backgrounds. In addition, it has positive effects on kernel number per spike and plant height, but has negative effects on thousand kernel weight, kernel weight per spike and yield per plant in most population backgrounds. Further analysis of plant height in KJ-RIL population showed thattheenhancing allele had rod lowering effect on all internode lengths except the internode length below spike, which resulted inthe insignificant increase in plant height. The results ofhaplotype analysis showed that the utilization rates of the long-spike haplotype Hap-AA-GG varied greatly in different wheat regions, with the highest utilization rate in the northern winter wheat region, accounting for 24%; while the short-spike haplotype Hap-CC-CC accounted for more than 30% in most wheat regions. Moreover, the utilization rate oflong-spike haplotype showed a gradual decrease over time, while that of short-spike haplotype consistently maintained a higher selection trend. 【Conclusion】A stable major QTL-for spike length was identified, the enhancing allele ofcould significantly increase spike length under different genetic backgrounds, and had certain genetic effects on yield-related traits. The closely linked molecular markers developed in the target region can be used for the genetic improvement of wheat spike length and yield-related traits in wheat.
wheat (L.); spike length; major QTL; genetic effects analysis; molecular marker

10.3864/j.issn.0578-1752.2023.20.001
2023-04-10;
2023-05-23
国家自然科学基金(32101726,32072051,32272119)、山东省重点研发计划(重大创新工程)(2022LZG002-2)
董继梓,E-mail:1324758770@qq.com。陈林渠,E-mail:1531484513@qq.com。董继梓和陈林渠为同等贡献作者。通信作者秦冉,E-mail:ranqin89@163.com。通信作者崔法,E-mail:sdaucf@126.com
(责任编辑 李莉)
