利用长链非编码RNAs与mRNAs联合分析半番鸭及番鸭胚胎期性腺发育差异
2023-11-14李丽章琳俐辛清武缪中纬朱志明邱君志郝晓娜黄勤楼郑嫩珠
李丽,章琳俐,辛清武,缪中纬,朱志明,邱君志,郝晓娜,黄勤楼,郑嫩珠*
(1.福建省农业科学院畜牧兽医研究所,福建 福州 350013;2.福建省畜禽遗传育种重点实验室,福建 福州 350013;3.福建农林大学生命科学学院,福建 福州 350002;4.福建农林大学动物科学学院(蜂学学院),福建 福州 350002)
半番鸭是番鸭(Cairina moschata)与家鸭(Anas platyrhynchosvar.domestica)属间远缘杂交的后代,兼有双亲的优点并具有很强的杂种优势,如生长速度快、肉质细嫩和瘦肉率高等[1]。但在实际生产中,半番鸭仍存在一个显著问题,即没有实际种用价值,生产中往往采用父母本杂交繁殖,自然交配率低,公鸭利用率差,主要依赖人工授精技术,其过程烦琐且培育成本较高,极大地限制了半番鸭的生产。从性成熟期和胚胎期系统对比分析半番鸭与繁殖力正常的父母本性腺的差异基因和转录本,更易于发现半番鸭不育性状的遗传基础。长链非编码RNAs(long non-coding RNAs, lncRNAs)是一类长度大于200 nt 并且一般不编码蛋白质的RNA 分子,长期被当作无用的转录物而被忽视[2]。远缘杂交不育的遗传基础较复杂。目前,学者们先后从解剖形态学、染色体学说、生理生化(包括激素含量)和差异基因等方面对半番鸭的不育机制进行了分析。许玉德等[3]认为半番鸭性腺中激素分泌不足导致其不育。檀俊秩等[4]发现家鸭和番鸭的染色体核型不一致,从染色体组学角度解析了半番鸭不育的原因。随着功能基因组时代的到来,学者们运用高通量测序技术研究畜禽性腺发育的分子机制,大量基因和转录本得以挖掘。PENG 等[5]发现lncRNAs通过调控FOLX2、Wnt6等靶基因参与卵母细胞减数分裂等过程,进而影响鸡卵泡发育。AYERS 等[6]发现钙蛋白酶5 基因(calpain 5 gene,CAPN5)、G 蛋白偶联受体56 基因(G protein coupled receptor 56 gene,GPR56)和成纤维细胞生长因子受体3 基因(fibroblast growth factor receptor 3 gene,FGFR3)等与鸡性别分化相关。REN 等[7]筛选到6 个与鸭卵巢在不同阶段转化显著相关的lncRNAs。随着高通量测序技术的快速发展,尽管众多研究都强调了差异转录本、微RNAs(microRNAs, miRNAs)及基因在畜禽性腺发育方面的重要作用,但关于半番鸭不育性状的生物学意义还鲜为人知,尤其是lncRNAs 在半番鸭不育方面的作用还鲜有报道。近年来,我们在半番鸭不育性状方面进行了一系列的研究,完成了半番鸭和家鸭染色体差异分析,以及半番鸭和亲本在性成熟期精巢与卵巢组织形态学差异及基因表达差异分析。结果发现,与父母本北京鸭或番鸭相比,半番鸭在性成熟期生殖系统发育不全,多表现为精巢或输卵管缺陷,或缺乏性激素分泌关键基因和在减数分裂中发挥重要作用的着丝粒蛋白基因,从而直接导致半番鸭繁殖力低下,且其成年后的表型取决于胚胎期组织器官的分化发育[8-9]。为进一步探究半番鸭不育性状分化发育差异的根源,本研究对半番鸭和番鸭雄性性腺组织进行全转录组高通量测序,探究半番鸭及其亲本在胚胎期性腺发育差异的关键lncRNAs和mRNAs,阐释半番鸭不育的分子机制,深入了解半番鸭及番鸭胚胎期性腺发育差异,为禽类性腺发育机制和生殖发生研究提供基础资料,为解决半番鸭繁育难题提供科学的参考依据。
1 材料与方法
1.1 试验材料
选取半番鸭(福建千里洋种禽有限公司)和番鸭(漳州昌龙农牧有限公司)的受精蛋各50 枚进行预试验,在相同条件下孵化,番鸭孵化期为35 d,半番鸭孵化期为30 d 左右。在预试验中,分别取3~22 d(7、8、9、10、11、12、13 d 为重要时间节点)的鸭胚,于无菌超净台中提取其胚胎性腺,对各鸭胚的外观形态和性腺进行观察和拍照。其中,7 d时可见半番鸭性腺,而由于番鸭孵化期较长,7~9 d时其性腺不可见,10 d 时可在显微镜下隐约分辨其性腺,12.5 d 时可完整采集2 个鸭品种的性腺。在前期观察胚胎发育预试验基础上,选择入孵后第12.5 d 的胚蛋,每个品种各取10枚。在无菌超净台中对胚蛋进行消毒后,将鸭胚置于显微镜下,提取胚体并置于装有磷酸盐缓冲液(phosphate buffered saline,PBS)(武汉赛维尔生物科技有限公司)的培养皿中,借助SMZ745T体视显微镜(日本Nikon公司)、显微剪和显微镊,对各组性腺组织的外观形态进行观察,结果发现,雄性性腺为两侧发育,雌性性腺因右侧卵巢退化而左右大小不同;剥离性腺组织并迅速置于液氮中,之后转移至-80 ℃冰箱中,待用;剪下腿部并置于-20 ℃冰箱中,待用。利用聚合酶链反应(polymerase chain reaction, PCR)再次鉴定性别后,每个品种各取3 个雄性性腺组织进行lncRNAs和mRNAs转录组测序。试验于福建省农业科学院畜牧兽医研究所动物房内进行,并参照国家实验动物处理行为准则,由福建省农业科学院畜牧兽医研究所动物实验伦理委员会批准(批准号:FAASIAHV-AEC-2020-0310)。
1.2 雌雄鉴定
参考美国国家生物技术信息中心基因和基因组信息数据库(NCBI GenBank)中鸟类CHD1基因序列,提取供试鸭胚腿部DNA用于性别鉴定。引物序列如下:CHD1-F,5´-AGTGCATTGCAGAAGCA ATATT-3´;CHD1-R,5´-GCCTCCTGTTTATTATAG AATTCAT-3´。PCR 扩增体系(50 μL):灭菌去离子水1 μL,CHD1-F(10 μmol/L)1 μL,CHD1-R(10 μmol/L)1 μL,基因组模板2 μL,Platinum®PCR SuperMix High Fidelity 45 μL。PCR 扩增条件:94 ℃预变性2 min;94 ℃变性30 s,56 ℃退火30 s,68 ℃延伸30 s,35个循环。
1.3 总RNA 提取及cDNA 文库构建和测序
以TRIzol试剂(美国Thermo Fisher Scientific公司)提取供试鸭胚性腺组织总RNA,用DNA酶Ⅰ去除残余的基因组DNA,并对RNA 样品进行严格质控:1)采用琼脂糖凝胶电泳分析样品RNA完整性及是否存在DNA 污染;2)利用Nanodrop 微量分光光度计(美国Thermo Fisher Scientific 公司)进行初步定量,检测RNA浓度及纯度;3)利用2100生物分析仪(美国Agilent公司)精确检测RNA的完整度。
采用去除核糖体RNA 的方法构建链特异性文库。先用Qubit 荧光定量仪(美国Thermo Fisher Scientific 公司)对文库进行初步定量,并稀释至1 ng/μL;然后用2100生物分析仪对文库的插入片段长度进行检测,发现片段长度为250~300 bp,符合预期;接着,使用实时荧光定量聚合酶链反应(real-time fluorescence quantitative polymerase chain reaction,qRT-PCR)方法对文库的有效浓度进行准确定量,结果表明,文库的有效浓度>2 nmol/L,保证了样本文库的质量;最后,根据得到的文库有效浓度及数据产出需求,采用Illumina 平台进行PE150 测序。建库和测序由北京诺禾致源科技股份有限公司完成。
1.4 数据过滤及转录水平分析
为了保证数据分析的质量及可靠性,利用FastQC 软件过滤掉原始数据中少量带有测序接头或测序质量较低的片段。利用TopHat2 软件将测序得到的高质量读长(clean reads)比对到参考基因组[http://www.ensembl.org/Anas_platyrhynchos/Info/Index,BGI_duck_1.0(GCA_000355885.1)]上。基于比对到基因组上的结果,使用Stringtie 软件将片段拼接成转录本并进行定量。使用Cuffmerge软件对拼接得到的各样本转录本进行合并,并和已知数据库进行比较,过滤掉数据库中已知的转录本,然后对筛选到的新转录本进行编码潜能预测,最终得到Novel_lncRNAs和Novel_mRNAs,并采用edgeR软件进行表达差异显著性分析。将矫正后的p值(padj)<0.05作为差异显著性标准(padj是当假阳性率较高时对p值进行多重检验校正后得到的值)。
1.5 差异基因功能富集分析
采用GOseq 软件进行基因本体(gene ontology,GO)富集分析,分为分子功能(molecular function)、生物过程(biological process)和细胞组分(cellular component)3 部分;采用KOBAS 2.0 软件的京都基因和基因组数据库(Kyoto Encyclopedia of Genes and Genomes, KEGG)进行通路富集分析,从而确定候选靶基因参与的最主要的生化代谢途径和信号转导途径。
1.6 lncRNAs 靶基因预测与功能分析
通过lncRNAs 与蛋白编码基因的位置关系(co-location)和表达相关性(co-expression)预测lncRNAs的靶基因:根据lncRNAs与mRNAs的位置关系进行cis靶基因预测,筛选范围为100 kb 以内;根据lncRNAs 与mRNAs 的表达相关性进行靶基因预测,筛选条件是相关系数大于0.95(当样品数<5时不进行该项分析)。然后对差异lncRNAs 的靶基因进行GO/KEGG 功能富集分析,预测lncRNAs 的主要功能。
1.7 测序结果可靠性验证
为了验证测序结果的可靠性,对差异表达的mRNAs 和lncRNAs 进行验证。用Primer Premier 6.0和Beacon Designer 7.8 软件进行qRT-PCR 引物设计,然后由生工生物工程(上海)股份有限公司负责合成,引物序列如表1 所示。在基因表达差异统计分析中,每个样品重复3次,各基因的相对表达水平以值进行统计分析。

表1 qRT-PCR引物序列信息Table 1 qRT-PCR primer sequence information
1.8 重组载体17β-HSD3-pcDNA3.1 3×Flag的构建
1.8.1 17β-羟基类固醇脱氢酶3基因(17β-hydroxysteroid dehydrogenase 3 gene,17β-HSD3)的PCR扩增
以cDNA 为模板,设计引物17β-HSD3-F 和17β-HSD3-R 并进行PCR 扩增。引物序列如下:17β-HSD3-F,5´-TTGGTACCGAGCTCGGATCCG CCACCATGCATGCTAGCTTGCTGCCTT-3´(下划线处为BamHⅠ酶切位点);17β-HSD3-R,5´-CTGT GCTGGATATCTGCAGAATTCGGGCTTCCTCCAC CTTTTCT-3´(下划线处为EcoRⅠ酶切位点)。PCR扩增体系(50 μL):去离子水1 μL,17β-HSD3-F(10 μmol/L)1 μL,17β-HSD3-R(10 μmol/L)1 μL,cDNA 模板2 μL,Platinum®PCR SuperMix High Fidelity 45 μL。PCR 扩增条件:94 ℃预变性2 min;94 ℃变性30 s,60 ℃退火30 s,68 ℃延伸1 min,35个循环。
1.8.2 载体和目的片段重组(In-Fusion 法)
PCR 扩增产物采用QIAquick PCR & Gel Cleanup 试剂盒进行切胶回收(具体操作参照试剂盒说明书),然后采用GeneArt®Seamless Cloning &Assembly Enzyme Mix 进行重组质粒的无缝克隆。17β-HSD3蛋白和pcDNA3.1融合反应体系:灭菌去离子水2 μL,17β-HSD3 蛋白纯化产物2 μL,pcDNA3.1(BamHⅠ/EcoRⅠ 50 ng,美国Thermo Fisher Scientific公司)1 μL,GeneArt®2×Enzyme Mix 5 μL。室温放置20 min 后,在冰上放置2~3 min,然后转化大肠埃希菌DH5α感受态细胞。
1.9 靶向关系验证
1.9.1 17β-HSD3-pcDNA3.1 3×Flag 免疫共沉淀
鸭胚成纤维细胞采用α-MEM 培养液[含10%胎牛血清(fetal bovine serum, FBS)和青霉素-链霉素双抗]进行复苏,在37 ℃、5% CO2条件下培养至汇合度为90%左右;加入0.25%胰酶-乙二胺四乙酸进行消化,以5×105mL-1细胞密度接种于6 cm细胞培养皿中进行传代。按LipofectamineTM3000说明书进行转染,待细胞裂解后,进行优质预偶联蛋白磁珠A/G和抗体复合物制备,RNA结合蛋白免疫沉淀(RNA binding protein immunoprecipitation,RIP)试验按EZ-Magna RIP 试剂盒(美国Millipore公司)说明书进行。
1.9.2 反转录试验
TCONS_00246198 cDNA 模板合成采用SuperScriptTMⅢ First Strand Synthesis SuperMix 系统(美国Thermo Fisher Scientific 公司)。第1 链cDNA 合成反应体系:分离纯化的RNA 4 μL,2×RT Reaction Mix 5 μL,RT Enzyme Mix 1 μL。反应条件:25 ℃孵育10 min,50 ℃孵育30 min,85 ℃终止反应5 min。将反转录产物保存在-20 ℃冰箱中,备用。
1.9.3 qRT-PCR 扩增
采用PowerUpTMSYBRTMGreen Master Mix 系统(美国Thermo Fisher Scientific 公司)进行qRT-PCR扩增。引物序列:TCONS_00246198-F,5´-CAAAT CTAGGCAACACCACA-3´;TCONS_00246198-R,5´-CAGCAACAATCATAGGAGGC-3´(扩增长度94 bp,熔解温度60 ℃)。反应体系(20 μL):去离子水8 μL,Power SYBR®Green Master Mix 10 μL,TCONS_00246198-F(10 μmol/L)0.5 μL,TCONS_00246198-R(10 μmol/L)0.5 μL,稀释1 倍后的cDNA 1 μL。反应条件:95 ℃预变性1 min;95 ℃变性15 s,63 ℃退火25 s,40 个循环。收集荧光,分析55~95 ℃的熔点曲线。
2 结果与分析
2.1 鸭胚雌雄鉴定
利用CHD1和PCR 进行雌雄鉴定,结果如图1所示,呈现明显双条带的为雌性(ZW型),呈现单一条带的为雄性(ZZ型)。在本试验中,选取单一且较亮条带的半番鸭和番鸭样本各3 个进行测序,其中半番鸭标记为BF1、BF2、BF3,番鸭标记为F1、F2、F3。
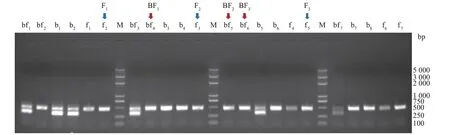
图1 半番鸭和番鸭雌雄鉴定Fig.1 Female and male identification of the mulard duck and the muscovy duck
2.2 测序数据的质量及比对分析
本研究通过Illumina PE150 对半番鸭和番鸭的性腺组织进行测序,获得13.25~15.94 Gb的数据量(原始碱基数),Q20(碱基错误识别概率小于1%)均≥97.97%,Q30(碱基错误识别概率小于0.1%)均≥94.25%(表2)。测序数据经过滤后,平均69.57%的高质量读长比对到了鸭参考基因组上,说明测序数据的质量良好,可用于后续分析。

表2 测序数据的质量及比对分析Table 2 Quality and alignment analysis of sequencing data
2.3 mRNAs 及lncRNAs 差异表达分析
以padj<0.05 且|log2差异倍数[log2(fold change),log2(FC)]|>1作为显著差异基因的筛选条件,在此条件下,从半番鸭和番鸭中共筛选出1 109个差异基因。相对于番鸭,半番鸭中上调表达基因共857个,下调表达基因共252个。其中,与繁殖相关的基因有醛酮还原酶家族1 成员D1 基因(aldo-keto reductase family 1 member D1 gene,AKR1D1)、17β-HSD3、前列腺素F2 受体负调节因子基因(prostaglandin F2 receptor negative regulator gene,PTGFRN)和胆固醇侧链裂解酶基因(cholesterol side-chain cleavage enzyme gene,P450scc)等,提示这些基因可能影响半番鸭性腺组织的分化发育。
为了分析lncRNAs是否参与胚胎期鸭的性腺发育,本研究筛选了半番鸭和番鸭雄性性腺组织中差异表达的lncRNAs。结果(图2)显示,半番鸭和番鸭性腺组织中共有733个显著差异表达的lncRNAs,相对于番鸭,半番鸭中显著上调的lncRNAs共660个,显著下调的lncRNAs共73个。
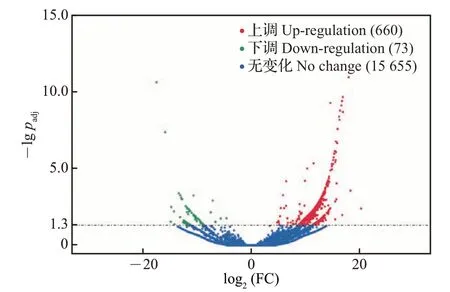
图2 半番鸭中相对于番鸭的差异表达的lncRNAsFig.2 lncRNAs differentially expressed in the mulard duck compared with the muscovy duck
表3和表4分别表示差异倍数前10位的上调和下调的差异基因和lncRNAs,反映了正常番鸭和不育半番鸭性腺组织的差异mRNAs 和lncRNAs 的表达谱。

表4 差异倍数前10位的差异表达的lncRNAsTable 4 Differentially expressed lncRNAs at top 10 of fold changes
2.4 lncRNAs 靶基因预测
lncRNAs 生物学功能丰富,广泛参与生物体各类重要生理过程,可在转录水平和转录后水平上对靶基因的表达进行调控,并找出不同处理产生的差异功能通路。lncRNAs主要通过位置关系或表达相关性调控靶基因的表达。针对位置关系调控机制共发现1 587 个靶基因,针对表达相关性调控机制共发现4 507个靶基因。
选取靶基因并与差异表达的mRNAs 进行分析。通过靶基因预测和差异基因维恩图(图3)可知,共有136 个下调的lncRNAs 和893 个上调的lncRNAs 参与调控差异基因表达,并可能存在潜在的调控关系。
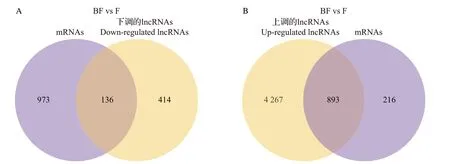
图3 差异lncRNAs靶向差异mRNAs的维恩图Fig.3 Venn diagram of differential lncRNAs targeting differential mRNAs
在靶基因预测结果中,已知与繁殖相关的lncRNAs TCONS_00246198靶向17β-HSD3,TCONS_00229529 靶向四次穿膜蛋白2 基因(tetraspanin-2 gene,TSPAN2),XR_217588.4 靶向TSPAN5,TCONS_00087816靶向5α-类固醇还原酶2型基因(5α-steroid reductase type 2 gene,SRD5A2),TCONS_00223199 靶向类固醇激素合成急性调节蛋白基因(steroidogenic acute regulatory protein gene,STAR),XR_003497885.1、XR_001193797.2、XR_003497886.1、TCONS_00139307靶向骨形态发生蛋白受体1A 基因(bone morphogenetic protein receptor 1A gene,BMPR1A),XR_003494073.1、TCONS_00048332、TCONS_00048329、TCONS_00240705 靶向辅肌动蛋白α2 基因(actinin alpha 2 gene,ACTN2),XR_003494639.1、XR_003494505.1 靶向AKR1D1。推测以上lncRNAs 或差异基因参与了鸭胚胎期性腺发育。
2.5 差异基因和差异lncRNAs靶基因功能富集分析
GO功能注释结果(图4)显示,有43个生物过程显著富集,主要参与细胞蛋白定位、DNA代谢过程、细胞大分子定位等。在差异lncRNAs靶基因表达相关性的GO分析中,蛋白质结合、S-腺苷甲硫氨酸依赖性甲基转移酶活性、自噬、O-甲基转移酶活性、蛋白质糖基化、蛋白质修饰过程等显著富集。在差异lncRNAs 靶基因位置关系的GO 分析中,蛋白质ADP-核糖基化、蛋白质糖基化、糖基化、糖蛋白生物合成过程、糖蛋白代谢过程、转录因子TFIID复合物等显著富集。
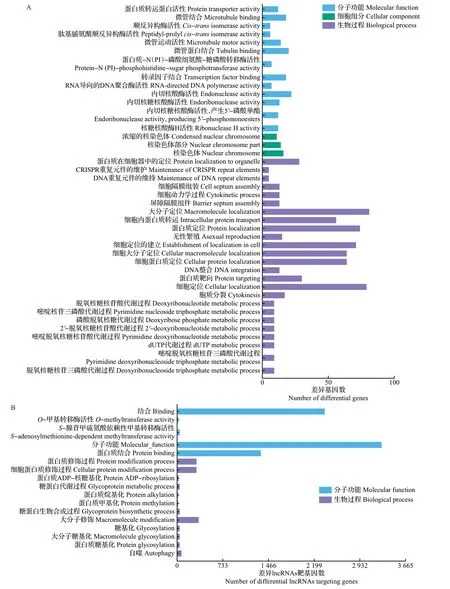
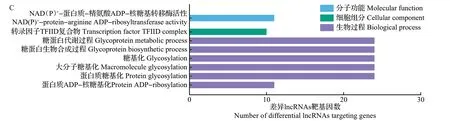
图4 差异基因和差异lncRNAs靶基因的GO富集过程Fig.4 GO enrichment process of differential genes and differential lncRNAs targeting genes
为明确半番鸭的不育性状是否存在某些特殊的过程,对差异基因和差异lncRNAs 靶基因进行KEGG 通路注释,结果共鉴定出13 条显著通路(图5),主要包括内质网中蛋白质加工、类固醇激素生物合成、谷胱甘肽代谢、自噬调节、细胞外基质(extracellular matrix, ECM)-受体相互作用和三磷酸腺苷结合盒转运体等。在差异lncRNAs 靶基因表达相关性的KEGG分析中,显著富集的有真核生物中核糖体生物发生和ECM-受体相互作用。在差异lncRNAs 靶基因位置关系的KEGG 分析中,鞘糖脂生物合成——乳糖和新乳糖系列显著富集。ECM-受体相互作用在差异基因和差异lncRNAs靶基因中均显著富集。


图5 差异基因和差异lncRNAs靶基因的KEGG富集过程Fig.5 KEGG enrichment process of differential genes and differential lncRNAs targeting genes
由差异基因和差异lncRNAs 靶基因功能富集注释结果可知,类固醇激素生物合成通路可能与性腺发育有关,该通路上包含AKR1D1、17β-HSD3和睾丸细胞色素P450家族1 亚家族B1 等显著差异基因。另外,ECM-受体相互作用也可能在鸭性腺发育中起到了重要作用,该通路上包含差异基因突触囊泡糖蛋白2B基因。
2.6 测序数据验证
为了验证测序结果,分别挑选4 个差异基因和4 个lncRNAs 进行qRT-PCR 验证,其中,AKR1D1、17β-HSD3和PTGFRN可能与性腺分化发育相关,而LOC101794158、TCONS_00189165、XR_003494977.1、TCONS_00087264 和XR_003498542.1 为随机挑选的不同差异倍数的差异基因或lncRNAs,以便全面验证测序结果。如图6 所示,在qRT-PCR 检测中,差异基因或lncRNAs的表达趋势均与lncRNAs测序结果中基因的表达趋势一致,即上调的AKR1D1、TCONS_00189165 和XR_003494977.1 在qRT-PCR中也为上调,下调的17β-HSD3、TCONS_00087264、XR_003498542.1 等在qRT-PCR 中均为下调,表明测序结果较为准确,可用于后续分析。
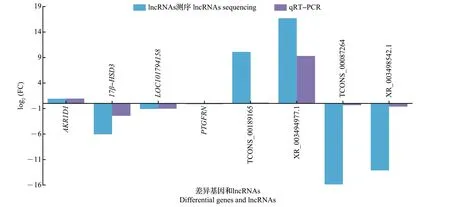
图6 qRT-PCR验证半番鸭和番鸭胚胎期性腺组织中差异表达基因和lncRNAsFig.6 Differential genes and lncRNAs in embryonic gonadal tissues of the mulard duck and the muscovy duck verified by qRT-PCR
2.7 17β-HSD3-pcDNA3.1 3×Flag 免疫共沉淀检测
为研究lncRNAs TCONS_00246198与17β-HSD3的靶向关系,通过转染17β-HSD3-pcDNA3.1 3×Flag重组质粒,采用Flag 抗体进行RIP 检测。结果(图7)表明,与IgG 相比,TCONS_00246198 富集水平达71.51 倍,说明TCONS_00246198与17β-HSD3蛋白存在直接或间接的相互作用,即两者可能存在靶向关系。
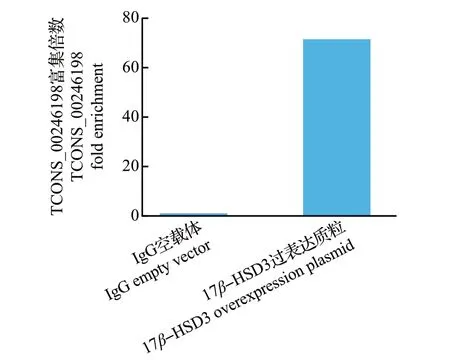
图7 17β-HSD3 蛋白对lncRNAs TCONS_00246198 的富集分析Fig.7 Enrichment analysis of lncRNAs TCONS_00246198 by 17β-HSD3 protein
3 讨论
近年来研究发现,lncRNAs 可与mRNAs 或miRNAs 共同参与诸多性腺分化发育生物学过程,如lncPGCR 靶向Btrc基因在原始生殖细胞(primordial germ cells, PGCs)发育中具有生长促进作用[10],lnc_13814 通过apla-miR-145-4 促进鸭颗粒细胞的凋亡[11],lncPGCAT-1 通过MAPK 信号转导调节原始生殖细胞的形成[12],lncRNAs可调控鸡胚胎干细胞向精原干细胞的分化过程[13]。LI等[14]通过测序探究了鸡胚胎干细胞向精原干细胞分化过程中与lncRNAs 相关的调控机制。近些年,笔者对半番鸭与亲本性成熟期精巢和卵巢组织形态学差异和基因差异进行了系统研究,为了进一步探究半番鸭不育性状分化发育差异的根源,本研究以不育半番鸭和繁殖性状正常番鸭的胚蛋为研究对象,对胚胎期性腺组织进行高通量测序,筛选出可能与胚胎期性腺发育相关的差异基因和差异lncRNAs,通过功能注释推测其参与繁殖过程的代谢通路。
在雄性动物中,睾酮是主要的雄激素[15],由睾丸间质细胞分泌,可影响精巢组织生长发育并维持精子发生[16],其合成相关酶主要来自羟基类固醇脱氢酶家族(如17β-HSD3)和细胞色素P450 家族(如P450scc)[17]。本研究中,TCONS_00246198 的靶基因为17β-HSD3,推测两者为半番鸭性腺发育的lncRNAs 和关键基因。17β-HSD3 蛋白可将由胆固醇转化而成的雄烯二酮最终氧化为睾酮[18],对睾丸激素合成和雄性繁育性状至关重要[19]。在人类医学研究中,46,XY性别分化障碍患者缺乏17β-HSD3且体内雄激素水平较低[20]。RHOUMA 等[21]和ENGELI等[22]研究发现,17β-HSD3错义突变导致睾酮含量过低,并使46,XY 性别分化障碍。以上研究提示,17β-HSD3可能与性腺发育相关。因此,可将TCONS_00246198 和靶基因17β-HSD3作为下一步研究半番鸭性腺发育的关键因子。此外,本研究发现,P450scc可能参与类固醇激素合成通路并影响半番鸭性腺发育。NURLIANI 等[23]研究发现,胆固醇侧链裂解酶(cholesterol side-chain cleavage enzyme,P450scc/Cyp11a)在睾丸间质细胞中合成雄激素;MENG 等[24]的研究进一步证实了P450scc 影响睾酮合成并可能与性腺发育相关;LIANG 等[25]通过免疫荧光分析发现,Cyp11a 位于间质细胞中,催化鱼类中所有类固醇激素的合成,并参与比目鱼的性腺分化和发育。
本研究中,lncRNAs TCONS_00229529 靶向TSPAN2,XR_217588.4 靶向TSPAN5,即可能参与鸭性腺发育,而KASHEF 等[26]研究发现,四次穿膜蛋白家族参与爪蟾胚胎协调发育过程,但未发现其参与性腺发育过程。因此,TSPAN2和TSPAN5在性腺发育方面的功能还需要进一步探究。
RENAULT 等[27]认为,BMPR1A和BMPR1B在卵巢功能中起关键作用,其新序列发生变异可导致原发性卵巢功能不全。EDSON等[28]研究发现BMPR1A和BMPR1B可调节卵巢生育力。本研究中,BMPR1A是lncRNAs XR_003497885.1、XR_001193797.2、XR_003497886.1、TCONS_00139307 的靶基因,并且在半番鸭睾丸组织中高表达,在番鸭睾丸组织中未表达,与以上研究结果不尽相同。因此,lncRNAs XR_003497885.1、XR_001193797.2、XR_003497886.1、TCONS_00139307靶向BMPR1A是促进还是抑制半番鸭性腺发育,还有待进一步研究。
PTGFRN是促性腺激素抑制剂。AHMED等[29]研究发现,PTGFRN为miRNAs miR-7 的直接靶标,通过调节前列腺素和骨形态生成蛋白信号通路来负调节垂体激素转录和分泌,进而调节动物性成熟和生殖功能。本研究中,PTGFRN在半番鸭睾丸组织中未表达,在番鸭睾丸组织中高表达,还需要通过定量试验来进一步验证PTGFRN在半番鸭和番鸭睾丸组织中的表达情况。
已有学者研究了特异性磷酸酶细胞分裂周期蛋白14B(cell division cycle protein 14B, CDC14B)在动物性腺及胚胎发育中的作用。如ZHANG 等[30]检测到CDC14B、FANCC和STARD4等基因可能是影响鸡睾丸生长发育重要的潜在候选基因;SCHINDLER等[31]研究表明,CDC14B能抑制小鼠卵母细胞减数分裂。以上研究提示,CDC14B可能影响性细胞减数分裂,而本研究发现,TCONS_00238912的靶基因为CDC14B,推测其通过靶向CDC14B来影响鸭染色体减数分裂过程,进而影响鸭性腺发育,后续可进一步研究该基因的功能和调控机制。
SRD5A2的失活突变会导致广泛的雄性化缺陷[32]。研究表明,人46,XY性别分化障碍与SRD5A2突变有关[33-34],但病因仍无法确定[35]。本研究中,lncRNAs TCONS_00087816的靶基因为SRD5A2,其对半番鸭性腺的影响有待深入研究,并可作为后续研究的关键基因。
ZHANG 等[36]的研究结果表明,由类固醇生成的急性调节蛋白STAR 和STAR-a 在公猪雄性发育中起着必不可少的作用。在禽类中,ESTIENNE等[37]研究表明,低表达的STAR 可通过抑制趋化因子样受体1 基因(chemokine-like receptor 1 gene,CMKLR1)来抑制公鸡睾丸激素的产生。HAYMAN等[38]则认为,STAR、CYP11B和SOX9a等为深水鱼类睾丸分化的最早且最可靠的标记。本研究中,STAR为lncRNAs TCONS_00223199的靶基因,表明其可作为分析半番鸭雄性性腺分化的候选因子。
综上所述,本研究筛选到可能影响胚胎期性腺发育的差异基因和差异lncRNAs,如TCONS_00246198 靶向17β-HSD3,TCONS_00229529 靶向TSPAN2,TCONS_00087816 靶向SRD5A2。其中,17β-HSD3是类固醇激素合成途径中的关键差异基因,已有相关报道将17β-HSD3视为性腺发育的靶基因[21-22],表明该关键基因可能影响半番鸭胚胎期性腺发育过程。另外,关键lncRNAs TCONS_00246198 对生殖细胞发生的作用也还需要进一步验证。未来将继续研究上述关键基因和关键lncRNAs对细胞增殖或凋亡的作用,从分子水平科学解释属间杂交后代半番鸭不育的分子遗传机制,为半番鸭不育研究提供新的途径。
4 结论
本研究以福建省特色优势杂交品种半番鸭为试验对象,依据父本番鸭正常性腺分化发育规律,获得了一批可能影响鸭胚胎期性腺发育的关键mRNAs 和lncRNAs,同时,确定了关键lncRNAs TCONS_00246198 和17β-HSD3的靶向关系。本研究结果有助于了解半番鸭不育分子机制、鸭胚胎期性腺发育差异及禽类性腺发育机制。
