解磷真菌产紫青霉菌SW-10 全基因组测序及功能分析
2023-10-20刘峰张培雨姜雯刘树堂孙雪芳赵子铉刘湘孙青
刘峰,张培雨,姜雯,刘树堂,孙雪芳,赵子铉,刘湘,孙青
(1. 青岛农业大学农学院,山东青岛 266109;2. 青岛农业大学资源与环境学院,山东青岛 266109)
磷是组成植物体内核酸和磷脂等物质的重要矿质元素,缺磷往往会导致植株矮小、瘦弱、抗逆性差和产量降低等[1]。 我国耕地土壤全磷含量一般在0.2~1.1 g/kg,土壤磷素存量大,但其中约95%不能被植物直接吸收利用,而土壤中磷素供给不足常常是制约作物生长发育的重要原因之一[2]。 我国约有74%的土壤缺磷,生产上通常采用外施大量磷肥补充作物对磷的需求,但大部分磷肥因土壤金属离子的吸附作用被固定成难溶磷在土壤中大量积累,只有1%~5%的土壤总磷是以可被植物吸收利用的可溶性形式存在,因此能够被植株吸收利用的量很低,磷肥利用率只有10%~25%。 磷肥施用量高而利用率低的问题不仅造成了肥料的浪费,对土壤质量以及水质也带来了威胁[3]。 另据报道,磷肥的生产原料磷矿石预计将在未来50~100 年间消耗殆尽[4-5]。 因此,通过绿色、高效、经济的解磷措施挖掘土壤潜在磷库对解决作物缺磷、实现作物对磷的高效利用、减轻土壤面源污染及维持生态系统的稳定具有重要意义。
解磷微生物因其高效的解磷能力被认为是降解土壤难溶磷的有效途径之一。 研究表明,土壤中存在大量的解磷微生物,解磷细菌包括芽孢杆菌属(Bacillus)、假单胞菌属(Pseudomonas)和不动杆菌属(Acinetobactersp.)等[6],解磷真菌包括青霉菌属(Penicillium)和曲霉菌属(Aspergillus)等[7]。 其中,解磷真菌的解磷能力一般是解磷细菌的几倍甚至更高,而且遗传性状更加稳定,对植酸钙、磷酸钙等难溶磷源均有较强解磷能力,具备解磷效率高、适应性强、多重解磷机制共同解磷的特点[8],同时,解磷真菌还有促进植株生长和提高抗逆性等作用,被广泛开发成生物菌肥用于土壤潜在磷库的挖掘与利用[9-10]。 如李豆豆等[11]从番茄根际土中分离出草酸青霉(Penicillium oxalicum),发现其对磷酸钙、磷酸铝等难溶性无机磷具有较好解磷效果;李小军等[12]从烟草根际土壤中筛选出一株烟曲霉(Aspergillus fumigatiaffinis),能够有效提高土壤中有效磷含量,对烟草具有较好的促生效果。 目前,关于解磷真菌的研究主要集中于解磷菌的筛选、解磷机理及其对作物生长的影响,对其解磷的分子机理研究较少。
本课题组前期从玉米根际土中筛选到一株对难溶性无机磷和有机磷均具有较好解磷效果的解磷真菌SW-10,经形态学及分子鉴定确定为产紫青霉菌(Penicillium purpurogenum)。 目前关于产紫青霉菌的报道多集中于其解磷条件的优化,如Scervino 等[13]研究了产紫青霉菌解无机磷的最适pH 值为6.5,碳源为葡萄糖,氮源为(NH4)2SO4,能通过产生有机酸溶解难溶性无机磷;Della Mónica 等[14]报道了产紫青霉菌产生胞外磷酸酶的最适pH 值为6.0~6.5,磷源为卵磷脂。 但对于产紫青霉菌解磷的分子机制尚无报道。 本研究基于Illumina NovaSeq 平台对该SW-10 菌株进行全基因组序列测序分析,并对解磷相关基因及代谢通路等进行初步分析,以期为阐明产紫青霉菌解磷机制、挖掘与利用功能基因提供遗传信息基础。
1 材料与方法
1.1 供试菌株
产紫青霉菌(Penicillium purpurogenum)SW-10 由青岛农业大学玉米生理生态实验室分离,已于中国普通微生物菌种保藏中心登记保藏(CGMCC No.20737)。
1.2 基因组DNA 提取
将菌株SW-10 的分生孢子接种至灭菌PDA液体培养基,置于200 r/min、28 ℃的培养箱培养48 h 后过滤收集菌丝,无菌水冲洗3 遍后用液氮速冻研磨,采用上海生工生物公司的Ezup 柱式真菌DNA 提取试剂盒提取基因组DNA。
1.3 测序方法
选取质量检测合格的DNA 用Covaris 打断,使其片段化,并通过磁珠纯化分选目标片段;对DNA 片段做末端修复和3′末端加A,连接测序接头,并用磁珠纯化连接产物。 利用PCR 选择性地富集两端连有接头的DNA 片段,同时扩增DNA文库;随即利用Picogreen 定量文库及Agilent Bioanalyzer 2100 对PCR 富集片段进行质控,随后通过Illumina NovaSeq 平台测序。
1.4 基因组拼接
原始数据经过接头污染去除等[15]处理后过滤生成高质量Reads 序列。 采用Falcon、CANU 软件对测序数据进行从头拼装,并使用Pilon (v1.18)[16]软件校正结果,随后利用BUSCO(v3.0.2)评估基因组拼装的完整性。 采用RepeatModler(v1.0.4)和RepeatMasker(v4.0.5)[17]软件分别进行从头注释和同源注释,以完成重复序列的分析与屏蔽、构成与种类统计。
1.5 蛋白编码基因预测
利用 Augustus (v3.03)[18]、 Glimmer HMM(v3.0.1)[19]和Gene Mark-ES(v4.35)[20]三个软件对菌株SW-10 的基因组序列进行从头预测;利用Exonerate 软件(v2.2.0)和近缘物种的蛋白质序列进行同源预测。 从头预测和同源预测的结果采用EVidenceModeler 软件[21]整合,生成菌株SW-10 的预测结果。
1.6 基因注释
蛋白编码基因的序列比对采用Diamond(v0.9.10.111)软件来完成,将预测得到的蛋白编码基因序列与NR、eggNOG、KEGG、Swiss-Prot、GO、P450、TCBD 数据库包含的蛋白质序列进行比对,序列比对的临界值选取为10-6。
GO 注释采用InterPro(v66.0)软件来完成,将InterPro 注释得到的结果对应到InterPro2GO 序列ID 当中并提取得到GO 号。 GO Slim 注释结果采用Map2Slim 完成。 KO 及Pathway 注释主要采用KEGG 的KAAS(v2.1)自动化注释系统[22]完成,基因集选择“For Eukaryotes”,基因KO 判别规则选取bi-directional best hit(BBH),注释完成后将KO 映射到相应KEGG Pathway 通路。 KOG 注释利用eggNOG-mapper 软件完成,比对数据库为eggNOG(v4.5)。
2 结果与分析
2.1 基因组测序与组装
本研究利用Illumina NovaSeq 测序平台对菌株SW-10 进行全基因组测序,结果如表1 所示。 菌株SW-10 基因组测序数据量总计4 145 395 200 bp,原始Reads 总数为27 635 968 个,过滤后获得27 257 154 个高质量Reads,GC 含量为47.10%,K-mer 预估其基因组大小为29.10 Mb。 SW-10基因组测序数据拼装完成后,共获得699 个scaffolds,最小序列长度为504 bp,最大序列长度为2 390 315 bp,N50 是551 686 bp。 基因预测结果(表2)显示,SW-10 基因组共有9 195 个蛋白编码基因,总基因序列长度15 597 887 bp,每个基因平均长度1 696 bp,CDS 平均长度1 554 bp,平均每个基因含3 个外显子,外显子平均长度505 bp,内含子平均长度68 bp。

表1 产紫青霉菌SW-10 基因组测序与组装

表2 产紫青霉菌SW-10 基因组基因预测结果
2.2 基因功能注释
通过NCBI 的NR 数据库共注释到基因9 123个,占预测基因总数的99.22%;利用KOG 数据库和KEGG 数据库分别注释到基因8 817 个和3 878个,分别占预测基因总数的95.89%和42.18%;通过SwissProt 数据库共注释到基因7 026 个,占预测基因总数的76.41%;通过GO 数据库共注释到基因6 608 个,占预测基因总数的71.87%;利用P450 数据库和TCDB 数据库分别注释到基因9 003 个和1 513 个,分别占预测基因总数的97.91%和16.45%(图1)。

图1 产紫青霉菌SW-10 基因功能注释数据库分布情况
2.2.1 GO 功能注释 对菌株SW-10 的基因组利用InterPro 注释共得到6 608 个基因,按基因功能分为3 大亚类,即生物学过程47 个分支、分子功能27 个分支和细胞组分15 个分支(图2)。 生物学过程亚类中涉及基因较多的是生物过程和细胞氮化合物代谢过程,分别为6 044 个(91.46%)和1 712 个(25.91%);细胞组分亚类中涉及基因最多的是细胞过程,达到2 592 个(39.21%);分子功能亚类涉及基因较多的是分子功能和离子结合过程,分别为5 535 个(83.76%)和2 321个(35.12%)。 其中,具有柠檬酸合成酶活性基因5 个,苹果酸脱氢酶活性基因4 个,碱性磷酸酶活性基因2 个,酸性磷酸酶活性基因15 个,植酸酶活性基因1 个,均参与分子功能过程。

图2 产紫青霉菌SW-10 基因组GO 功能分类结果
2.2.2 KEGG 功能注释 对菌株SW-10 进行KEGG Pathway 注释后发现能对应至代谢通路的基因共3 878 个,富集在52 条代谢通路中(图3)。涉及基因数量较多的代谢通路主要有以下7 条:遗传信息处理代谢通路,共2 420 个基因(62.40%);信号和细胞过程通路,共646 个基因(16.66%);代谢通路,共542 个基因(13.98%);信号转导通路,共549 个基因(14.16%);碳水化合物代谢通路,共474 个基因(12.22%);氨基酸代谢通路,共430 个基因(11.09%);运输和分解代谢通路,共377 个基因(9.72%)。 其中,与菌株SW-10 解磷相关基因包括柠檬酸合成酶基因5个、苹果酸脱氢酶基因5 个、酸性磷酸酶基因7个、碱性磷酸酶基因2 个和磷酸盐转运蛋白5 个,主要参与碳水化合物代谢、信号和生物学过程等代谢通路。

图3 产紫青霉菌SW-10 基因组KEGG 数据库主要代谢通路分析
2.2.3 KOG 功能注释 通过eggNOG 数据库对菌株SW-10 进行基因注释及KOG 预测分类,共注释到8 817 个基因,分为25 个大类(图4)。 除功能未知一类外,涉及到基因数量较多的分别是:碳水化合物运输和代谢类(G),共691 个基因,占比为7.84%;翻译后修饰、蛋白质周转及分子伴侣类(O),共493 个基因,占比为5.59%;次级代谢物的生物合成、运输和分解代谢类(Q),共490 个基因,占比为5.56%;转录类(K),共389 个基因,占比为4.41%。 另外,无机离子运输与代谢类(P)和细胞壁/膜/包膜生物合成类(M)分别涉及161 个和97 个基因,占比分别为1.83%和1.10%。其中,与菌株SW-10 有机酸分泌相关的基因包括柠檬酸合成酶基因5 个和苹果酸脱氢酶基因4个,参与能量生产和转化;与有机磷矿化水解相关的基因包括3-植酸酶基因1 个、酸性磷酸酶基因10 个和碱性磷酸酶基因2 个,参与脂质代谢与运输、碳水化合物运输和代谢等过程;磷酸盐转运蛋白基因6 个,参与无机离子运输与代谢过程。

图4 产紫青霉菌SW-10 基因组KOG 功能分类结果
2.3 碳水化合物活性酶基因分析
采用hmmscan 软件预测菌株SW-10 基因组序列中存在的CAZy 酶类基因,发现其中有209个糖苷水解酶(glycoside hydrolases,GH)基因、86个糖基转移酶(glycosyl transferases,GT)基因、83个碳水化合物酯酶(carbohydrate esterases,CE)基因、80 个辅助活性酶(auxiliary activities,AA)基因、12 个碳水化合物结合模块(carbohydratebinding modules,CBM)基因和2 个多糖裂解酶(polysaccharide lyases,PL)基因(图5)。
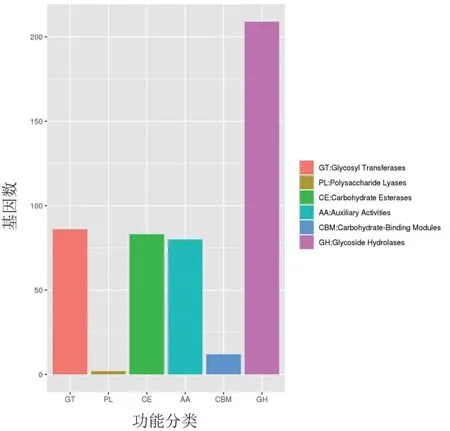
图5 产紫青霉菌SW-10 基因组CAZyme 基因数目
2.4 解磷及磷酸盐转运相关基因分析
解磷微生物可以通过产生和分泌有机酸(包括柠檬酸、苹果酸等)、磷酸酶(包括碱性磷酸酶和酸性磷酸酶)及植酸酶等将土壤中难溶性磷转化为可溶性磷,从而有利于植物对磷的吸收。经过蛋白编码基因的NR 序列比对、KEGG、GO 及KOG 功能分析,本研究发现了2 个柠檬酸合成酶基因、2 个苹果酸脱氢酶基因、2 个磷酸盐转运蛋白基因、2 个碱性磷酸酶基因、7 个酸性磷酸酶基因和1 个植酸酶基因,推测其参与了解磷过程(表3)。

表3 产紫青霉菌SW-10 解磷及磷酸盐转运相关基因
3 讨论
活化土壤中的难溶性磷、增强土壤有效磷的供给能力对农业可持续发展具有重要意义。 土壤解磷微生物能够通过酸解、酶解作用等将土壤中无效磷转化为有效磷供植物吸收利用,从而促进植物生长发育。 解磷微生物改善土壤磷素营养是一项有利于资源节约、环境友好的重要农业措施[23]。 本课题组前期从玉米根际土壤中分离出一株产紫青霉菌SW-10,研究发现该菌株对难溶性无机磷和有机磷均具有较好的解磷效果,并且能够促进玉米生长,接种产紫青霉菌后地上部鲜重和干重、根部鲜重和干重以及株高均显著增加,较对照分别增加36. 33%、37. 93%、31. 20%、31.25%、13.03%,地上部和根部的磷含量分别增加了32.86%和9.77%[24]。 为深入了解该菌株解磷的生物学特性,挖掘具有促进解磷的功能基因,本研究利用Illumina NovaSeq 测序平台获得菌株SW-10 的全基因组序列,确定了该菌株的基因组大小为29.10 Mb,与已报道的烟曲霉、构巢曲霉等基因组大小相近[25];同时将菌株SW-10 基因组测序数据在NR、GO、KEGG、eggNOG 等数据库比对分析,在分子生物学层面揭示其解磷过程中可能涉及到的基因及代谢通路。
解磷真菌可通过产生苹果酸、柠檬酸等有机酸促进难溶性无机磷的磷酸根离子释放[26]。Cunningham 等[27]发现比莱青霉在溶解磷酸钙时产生大量草酸和柠檬酸等有机酸。 唐超西等[28]自黑曲霉H1 获得解磷基因psgA和psgB,均能促进甲酸、乙酸和苹果酸的合成,同时分别可以诱导合成α-酮戊二酸和柠檬酸。 Lü 等[29]研究发现将一株草酸青霉的苹果酸脱氢酶mMDH基因导入大肠杆菌后能够有效提高大肠杆菌的解磷能力。 本研究发现产紫青霉菌SW-10 基因组具有2 个柠檬酸合成酶基因和2 个苹果酸脱氢酶基因,表明其对难溶性无机磷具有降解潜力。
解磷真菌也可通过分泌磷酸酶、植酸酶等加快有机磷矿化过程。 Yadav 等[30]研究发现解磷真菌在溶解有机磷时其分泌的植酸酶和磷酸酶活性分别可达19.9EU 和0.29EU;吴静[31]发现黑曲霉N-2 含有植酸酶基因phyA,能够促进真菌快速分泌植酸酶并提高植酸酶活性,进而加快植酸钙溶解。 本研究发现菌株SW-10 含有2 个碱性磷酸酶基因、7 个酸性磷酸酶基因和1 个植酸酶基因,说明该菌株能够通过分泌植酸酶和磷酸酶等多种途径矿化土壤中难溶性有机磷,增加土壤磷的有效性,进而促进作物对养分的吸收。
研究表明,碳水化合物活性酶(CAZymes)与抑菌作用相关[32]。 本研究通过hmmscan 软件预测到菌株SW-10 中与碳水化合物活性酶相关基因共计460 个,包括糖苷水解酶(GH)、糖基转移酶(GT)、碳水化合物酯酶(CE)、辅助活性酶(AA)、多糖裂解酶(PL)五类碳水化合物活性酶基因,这与Mardones 等[33]报道产紫青霉Penicillium purpurogenum具有大量的木质纤维素降解酶相一致。 另外,Echeverría 等[34]从Penicillium purpurogenum基因组中鉴定出糖苷水解酶基因(XynD),表明菌株SW-10 可能具有较好的细胞壁水解酶活性,可用于木质纤维素的降解过程。
4 结论
本研究采用Illumina NovaSeq 测序平台对解磷真菌产紫青霉菌SW-10 进行全基因组测序,得到菌株的基因组大小为29.10 Mb。 通过在NR、GO、KEGG、KOG 及Swiss-Prot 数据库比对,发现其含有2 个柠檬酸合成酶基因、2 个苹果酸脱氢酶基因、2 个磷酸盐转运蛋白基因、2 个碱性磷酸酶基因、7 个酸性磷酸酶基因和1 个植酸酶基因,从基因角度证实了菌株SW-10 具有较强的解磷能力;同时,预测菌株SW-10 基因组含有碳水化合物活性酶相关基因460 个,推测其具有较好的细胞壁水解酶活性。 本研究可为揭示产紫青霉菌的解磷分子机制、推动其在生物解磷方面的进一步开发利用及改良其遗传信息提供理论基础。
