基因组光学图谱技术在产前诊断中的应用
2023-10-11许伊云张沁欣季修庆周冉罗春玉王艳孟露露胡平许争峰南京医科大学附属妇产医院产前诊断中心南京210004
许伊云,张沁欣,季修庆,周冉,罗春玉,王艳,孟露露,胡平,许争峰(南京医科大学附属妇产医院产前诊断中心,南京 210004)
基因组光学图谱技术(optical genome mapping,OGM)被称为新一代细胞遗传学技术,是一种通过对超高分子量(ultra-high molecular weight,uHWM)DNA 进行荧光特异性标记以及线性化高分辨率成像,从而构建全基因组物理图谱的新兴技术。目前认为,OGM 以其大于230 kb 的长读长,能够在单次检测中识别各种主要类型的结构变异(structural variation,SV),最高分辨率可达500 bp[1],近年来已逐步被应用于生殖遗传领域[2-3]。然而,OGM 在产前诊断中的相关研究较少,仅有少数学者对其在产前诊断中的应用进行了回顾性分析及评述,认为其在未累及着丝粒的染色体变异中的检测效能与常规方法完全一致[4-5]。本研究以染色体微阵列分析(chromosomal microarray analysis,CMA)联合核型分析为对比,对OGM 在产前诊断中的可行性及有效性进行评估,以期进一步完善OGM 在产前诊断中的应用方案,深入剖析其在各类染色体变异中的检测效能及优势。
1 材料和方法
1.1 研究对象 收集2022 年6 月至8 月于南京市妇幼保健院进行产前遗传咨询的病例60 例,年龄(29.3±4.1)岁,中位穿刺孕周23+3周(18~31+3周)。纳入标准:(1)胎儿结构畸形;(2)超声软指标异常,包括颈项透明层(nuchal translucency,NT)增厚(≥3.0 mm)、鼻骨发育异常和轻度侧脑室增宽(10~15 mm)[6]。排除标准:(1)多胎妊娠;(2)孤立性室间隔缺损、孤立性右位主动脉弓、孤立性永存左上腔静脉、孤立性足内翻;(3)穿刺失败、样本量不足、培养或DNA 提取失败等原因导致无法进行OGM、CMA 检测。介入性产前诊断指征主要包括NT 增厚15 例,多发软指标异常13 例,心血管畸形9 例,结构畸形合并软指标异常7 例,其他超声异常16 例。取得患者知情同意后获取羊水样本30 mL,由3 组实验人员平行进行OGM、CMA 检测及染色体核型分析,并对检测结果进行盲法分析及致病性评估。本研究经南京市妇幼保健院医学伦理委员会批准(批准文号:2021KY-118),患者及家属知情同意。
1.2 主要仪器及试剂 羊水细胞培养基(德国Cytogen GmbH 公司),Quibit 双链DNA 定量试剂盒、Quibit 荧光定量仪(美国Thermo Fisher Scientific 公司),超长片段DNA 抽提试剂盒、DLS DNA 标记试剂盒、Saphyr DNA 单分子成像平台(美国Bionano Genomics 公司),DNA 提取试剂盒(德国QIAGEN公司),基因芯片试剂盒及基因芯片平台、杂交炉、洗涤工作站、扫描仪(美国Affymetrix 公司),FISH试剂盒(北京金菩嘉公司)。
1.3 OGM 检测及分析取约5 mL 羊水接种于T25 培养瓶中,经培养及分瓶传代至铺满2 个培养瓶后进行细胞冻存,其中约1/2 细胞用于uHMW DNA 提取,剩余细胞冻存备份。按照超长片段DNA 抽提试剂盒的标准化操作流程从>50 万的羊水细胞中提取uHMW DNA,并利用Quibit 双链DNA 定量试剂盒、荧光定量仪以及脉冲场凝胶电泳完成基因组DNA 的定量及长度质控,要求大部分DNA 分子长度>500 kb,浓度36~150 ng/μL。DLS技术(Direct Label and Stain)序列特异性标记使用DLS DNA 标记试剂盒完成,其主要流程包括基因组CTTAAG 6 个碱基序列标记,游离荧光基团的清除,均质化及DNA 骨架染色过夜,标记后产物定量。浓度4~12 ng/μL、标准差<0.25 的样本可直接上机。标记后的样本置于4 ℃避光保存。标记后DNA 载入Saphyr 芯片,在Saphyr DNA 单分子成像平台中实现线性化及成像。下机数据符合Bionano官方推荐的质控标准(https:/ /bionanogenomics.com/wp-content/uploads/2018/04/30223-Saphyr-
Molecule-Quality-Report-Guidelines.pdf):N50 读长>230 kb,比对率≥70%,每100 kb 14~17 个标记,有效数据深度>80×,阳性标记偏差率3%~10%,阴性标记偏差率6%~15%。在芯片扫描完成后,利用Bionano Access 软件(v1.7)进行基因组的从头组装,并通过与参考基因组(hg19)比对,实现各种类型SV 的检出。数据分析前设置过滤条件如下:(1)CNV 算法:CNV 置信度>95%,CNV 大小≥500 kb,关闭CNV masking 过滤器;(2)SV 算法:SV在对照人群中发生频率≤1%,关闭SV masking 过滤器。过滤后检索国际公认数据库,包括ClinGen数据库、OMIM 数据库、Ensemble 数据库、DECIPHER 数据库、DGV 数据库、PubMed 数据库,对变异进行注释及分析,并根据美国医学遗传学与基因组学学会(American College of Medical Genetics and Genomics,ACMG)指南[7]完成变异致病性的判读。
1.4 CMA 检测及分析 按照DNA 提取试剂盒说明书从10 mL 羊水中提取基因组DNA。根据Affymetrix 基因芯片平台的标准操作流程进行酶切、接头连接、PCR 扩增、磁珠纯化、纯化产物测定、DNA片段化、片段末端标记、杂交、洗涤染色、扫描。利用ChAS 软件(v3.2)进行数据分析。检索UCSC 基因浏览器、ClinGen 数据库、DECIPHER 数据库、gnomAD 数据库、DGV 数据库、PubMed 数据库等国际公认数据库,根据ACMG 指南[7-8]将拷贝数变异(copy number variation,CNV)分为致病、可能致病、临床意义未明(variants of uncertain significance,VOUS),可能良性和良性,并主要报告>100 kb 的致病、可能致病的CNV、VOUS 以及涉及明确印记基因致病的6、7、11、14、15、20 号染色体上>10 Mb的纯合区域。
1.5 染色体核型分析 取15 mL 羊水接种于2 个T25 培养瓶中双线培养。按照G 显带染色体核型分析的常规流程[9]进行试验,并参考国际细胞遗传命名系统(International System for Human Cytogenetic Nomenclature,ISCN)2020 对染色体核型进行分析[10]。
1.6 荧光原位杂交(fluorescence in situ hybridization,FISH)将备份的羊水细胞培养至分裂中期,加入秋水仙素破坏纺锤体,再经低渗、预固定和固定后,根据OGM 提示的易位片段选择商业化FISH探针与细胞杂交。选择特异性荧光染料复染后进行观察和分析。
1.7 统计学分析 统计学分析由SPSS 23.0 软件完成。对病例的一般资料及OGM 检测数据进行描述性统计分析。计量资料根据是否符合正态分布用或中位数(四分位数)[M(P25,P75)]表示;计数资料用频数或率表示。采用卡方检验进行两组间率的比较。以P<0.05 为差异有统计学意义。
2 结果
2.1 OGM 基因组组装信息及变异检出情况 60例样本DNA 分子平均长度为(291.65±19.05)kb,平均DNA 分子N50 长度为(309.41±30.66)kb。经过滤后,单个样本中CNVs 的中位检出量为1(0~6)个,SVs 的平均检出量为(76.9±11.0)个,累及基因的SVs 的平均检出量为(39.2±6.4)个。
2.2 OGM 与CMA 联合核型分析的检测效能 60例病例中各类染色体变异的检出情况见表1。OGM 和CMA 联合核型分析的检出率差异无统计学意义(P>0.05)。以hg19 为参考基因组进行组装,OGM 漏检1 例15q11.2 区段约507 kb 的致病性微缺失,其在染色体非整倍体、CNV、三倍体检测中的敏感性为93.8%,特异性为100.0%。将该漏检病例的OGM 数据再次以hg38 为参考基因组进行组装,该例CNV 能够被OGM 检出。

表1 OGM 与CMA 联合核型分析在染色体非整倍体、拷贝数变异及三倍体中的检出率比较[n(%)]
2.3 OGM 明确重复插入的位置及方向 在60 例病例中,共检出6 例病例存在染色体微重复。OGM提示该6 例病例均为原位串联重复。6 例病例的CMA、OGM 结果及OGM 提示的断点区间见表2。以病例059 为代表病例,OGM 图谱见图1。
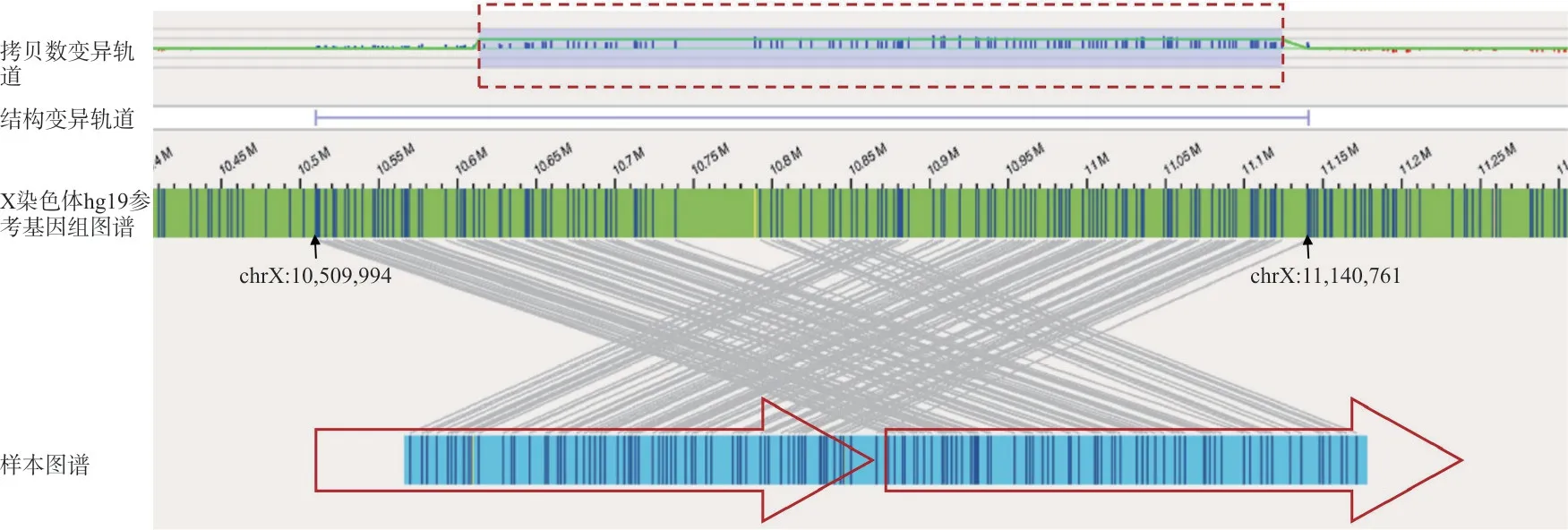
图1 染色体微重复代表病例(病例059)的OGM 检测结果

表2 染色体微重复病例CMA 及OGM 结果及断点区间
2.4 OGM 额外检出隐匿性易位 在60 例病例中,OGM 较CMA 联合核型分析额外检出了2 例(3.3%)隐匿性易位,包括1 例隐匿性非平衡易位和1 例隐匿性平衡易位(表3),且均经FISH 验证证实。其中病例039 孕妇既往曾有2 次稽留流产史。经亲代溯源,该平衡易位被证实为父源遗传。病例039 的OGM 及FISH 验证结果见图2。

图2 隐匿性平衡易位病例(病例039)OGM 及FISH 验证结果

表3 OGM 额外检出隐匿性易位的断点信息
2.5 OGM 检测周期及检测成功率 用于OGM 检测的中位初始羊水投入量为5.5(4~8.5)mL,羊水细胞培养的中位天数为12(11~19)d。OGM 检测及分析周期约为5 d。OGM 检测成功率为100%。
3 讨论
OGM 是一种能够一站式识别各种主要类别SVs 的综合性检测技术。根据本研究数据,以hg19为参考基因组,OGM 漏检1 例15q11.2 区段的复发性CNV,该CNV 两侧断点位于片段重复区。DNA分子在该区段组装困难,无法生成跨越两侧断点的OGM 图谱,因此导致假阴性结果的出现。但总体而言,本研究中OGM 与CMA 联合核型分析的检出率差异无统计学意义。尤其在以hg38 为参考基因组组装后,OGM 的敏感性和特异性均达到100%。本研究再次证实了OGM 在产前诊断中的有效性及可靠性。
OGM 能够明确重复插入的位置及方向。染色体微重复的致病性可能与三倍剂量敏感、基因内复制导致阅读框破坏、基因间复制断裂点破坏基因或导致基因融合相关。研究发现,83%的重复为原位串联重复,另有17%为易位插入、复杂结构重排等其他类型的重复,提示了在对染色体微重复的致病性评估中结合断点分析的重要性[10]。相较于CMA,OGM 的一大突出优势为SV 算法的直观性,其能够可视化描绘重排模式。本研究中,OGM 提示6 例染色体微重复均为原位串联重复。尽管本研究未发现重复导致基因破坏或基因融合的病例,但已有相关研究报道提示了OGM 在全面评估重复插入片段致病性中的优势所在[11]。
OGM 能够检测核型分析无法识别的隐匿性染色体重排。平衡性结构重排能够通过破坏基因或影响远程调控相互作用而导致表型。近年来,已有多篇研究[2-3]将OGM 用于不明原因多次不良孕产史以及不孕不育患者的诊断,揭示了OGM 在精细化断裂点区间的优势所在,认为其可作为临床检测隐匿性平衡性染色体重排的一线方法。本研究通过OGM 诊断了1 例隐匿性平衡易位,并描绘了断裂点区间,提示5 号染色体断点位于WWC1基因内,但该基因与胎儿异常表型无关。此外,经过溯源,发现该平衡易位为父源遗传,从而为该孕妇两次稽留流产找到了原因。此对夫妻再生育可考虑植入前遗传学检测,从而避免不良妊娠结局的发生。由此可见,OGM 在SV 检测中的另一大优势为高分辨率检测隐匿性染色体重排、精细化断裂点区间协助SV 致病性判读。
目前OGM 对产前样本的要求仍然较高,无法使用未经培养的羊水样本直接提取DNA。本研究中羊水细胞中位培养天数为12 d,检测成功率为100%,提示了OGM 应用于产前诊断的可行性。目前OGM 检测的限速步骤主要在于羊水细胞培养以及上机后数据产出及分析过程。本研究中初始羊水投入量较少,临床应用时可通过增加初始羊水投入量以及羊水细胞接种面积缩短细胞培养时间。此外,Saphyr 系统若能实现通量的提升,则可进一步缩短诊断周期,以利于OGM 的产前应用。
目前OGM 产前应用限制主要在于以下几个方面。首先,羊水细胞培养存在失败风险,诊断周期有待进一步缩短;其次,对于检出的部分SV 无法直接进行致病性评估,需结合测序数据进行深入分析;再者,其检测效能,尤其是对于断点位于片段重复区的SV 检测的准确性仍需更多前瞻性数据积累及评估。然而,随着技术的完善、算法的优化以及Saphyr 系统的进一步升级,OGM 有望成为产前SV检测的一线方案。
