基于m7G相关lncRNA识别胃癌的预后及免疫特征
2023-09-25王振张乐刘虹汝张钰哲
王振,张乐,刘虹汝,张钰哲,2,3
(1.大理大学基础医学院生物化学与分子生物学教研室,云南 大理 671000;2.云南抗病原药用植物筛选重点实验室,云南 大理 671000;3.云南省昆虫生物医药重点实验室,云南 大理 671000)
胃癌是全球最常见的癌症之一[1],幽门螺杆菌感染是其最主要病因[2]。虽然胃癌的预后及生存期已明显改善,且发病率在逐步降低,但在50岁以下人群中却呈增长趋势[3]。N7-甲基鸟苷 (N7-methylguanosine,m7G) 修饰是最常见的RNA修饰之一[4-7],在肿瘤的发生发展过程中发挥关键作用[8-13]。甲基转移酶1 (methyltransferase 1,METTL1) 是目前研究最多的m7G调控因子,可通过在微RNA (microRNA,miRNA)、转运RNA (transfer RNA,tRNA) 和信使RNA(messenger RNA,mRNA) 中安装m7G修饰行使其功能[14-17]。长链非编码RNA (long non-coding RNA,lnc-RNA) 是一类长度超过200个核苷酸的非编码RNA(non-coding RNA,ncRNA)[18],其差异表达与肿瘤的发生、转移和耐药密切相关[19-21]。目前对胃癌中m7G相关lncRNA的功能未知,故本研究拟构建m7G相关lncRNA预后模型,以评估、改善胃癌患者的预后;并通过公共数据库进一步分析m7G相关lncRNA与胃癌的免疫功能、肿瘤免疫逃逸 (tumor immune escape,TIE)、药物敏感性预测和肿瘤突变负荷 (tumor mutation burden,TMB)。
1 材料与方法
1.1 数据收集
通过癌症基因组图谱 (The Cancer Genome Atlas,TCGA) 数据库 (https://portal.gdc.cancer.gov/),获取胃癌组织和正常胃组织的STAR-Counts数据和相关临床数据,包括343例肿瘤及30例正常组织样品。其中339例临床病理数据完整,用于后续分析。见图1。

图1 基于m7G 相关lncRNA识别胃癌的预后及免疫特征的工作流程图Fig.1 Workflow chart for identifying the prognosis and immune features of gastric cancer based on m7G-related lncRNAs
1.2 m7G及相关lncRNA筛选
在Genecards (https://www.genecards.org/)、NCBI(https://www.ncbi.nlm.nih.gov/)、Gene Set Enrichment Analysis (GSEA,http://www.gseamsigdb.org/gsea/index.jsp) 数据库中分别输入“m7G”“N7-methylguanosine”“7-methylguanosine”,删除重复值后共获得143个m7G相关基因。用GENCODE注释文件从TCGA数据库 (http://cancergenome.nih.gov/abouttcga) 中获取16 876个lncRNA的表达文件。应用皮尔森相关分析识别m7G相关lncRNA,基于“limma”R 包 (|coefficients|>0.5,P< 0.001)。最终筛选出805个m7G相关lncRNA用于后续分析。见图1。
1.3 lncRNA预测模型构建与评估
将患者从完整数据集 (n= 339) 以1︰1比例随机分为训练集 (n= 170) 和测试集 (n= 169),用训练集数据构建预后模型,再用测试集数据进行验证。首先,用单变量Cox回归 (P< 0.05) 确定潜在的预后lncRNA;然后,通过最小绝对收缩和运算符选择(least absolute shrinkage and selection operator,LASSO)回归算法,每个周期随机刺激1 000次,减少过拟合基因;最后,通过多变量Cox回归建立胃癌的预后模型。计算风险评分=表达量 (lncRNA)×系数(lncRNA)。同时,根据打分的中位数将患者分为m7G低风险组和高风险组。用χ2检验验证临床特征与风险组的相关性。用单变量Cox和多变量Cox回归分析确定风险评分是否为胃癌患者总生存期 (overall survival,OS) 的独立危险因素。用受试者操作特征 (receiver operating characteristic,ROC) 曲线和一致性指数 (concordance index,C-index) 进一步评估模型的预测准确性。见图1。
1.4 预测性列线图构建和校准
使用“rms”R语言包,根据胃癌患者的m7G风险评分、年龄、性别、肿瘤分级、T分期、远处转移、淋巴结播散情况和肿瘤分期结果构建了列线图,用于预测患者1年、3年和5年的OS。并绘制了校准曲线,用于验证列线图模型的预测能力。见图1。
1.5 主成分分析 (principal component analysis,PCA)、基因本体 (Gene Ontology,GO) 和京都基因与基因组数据库 (Kyoto Encyclopedia of Genes and Genomes,KEGG) 富集分析
通过PCA分析m7G相关的lncRNA表达模式为样本进行分类。根据m7G风险特征将所有患者分为高风险组和低风险组,以log2FC>1和P< 0.05为阈值筛选2组间差异表达基因。对差异表达基因进行GO和KEGG富集分析 (P< 0.05,FDR<0.05)。见图1。
1.6 免疫功能、TIE和药物敏感性分析
用单样本基因集富集分析 (single-sample gene set enrichment analysis,ssGSEA) 算法分析免疫功能,以评估m7G高、低风险组中免疫功能的差异。通过肿瘤免疫功能障碍和排除 (tumor immune dysfunction and exclusion,TIDE) 算法 (http://tide.dfci.harvard.edu/) 分析m7G高、低风险组间TIE潜能和免疫治疗效果。使用R语言“pRRophetic”包,通过癌症药物敏感性基因组学 (Genomics of Drug Sensitivity in Cancer,GDSC)的半抑制浓度 (half-maximal inhibitory concentration,IC50) 评估m7G高风险组和低风险组患者的治疗效果,见图1。
1.7 TMB分析
从TCGA数据库中获取胃癌相关样本的肿瘤突变数据。根据m7G突变风险评分,将患者的突变注释格式 (mutation annotation format,MAF) 分为2组。根据2组患者的体细胞突变数据,分别计算TMB评分,比较TMB高、低风险组间生存情况的差异。见图1。
2 结果
2.1 m7G相关lncRNA筛选
基于143个m7G相关基因及胃癌样本中lncRNA的表达水平,通过皮尔森相关分析,共获取805个m7G相关lncRNA。临床统计分析验证结果显示,训练集和测试集数据之间无统计学差异 (P> 0.05),并通过训练集数据构建预后模型。
2.2 m7G相关lncRNA预后模型的构建
通过单因素Cox比例风险分析,筛选出22个m7G相关的lncRNA与胃癌患者的生存率显著相关 (P<0.05),包括2个低风险lncRNA[风险比 (hazard ratio,HR) <1]和20个高风险lncRNA (HR>1),见图2A。通过LASSO回归分析,找到11个与预后显著相关的lncRNA。通过多因素Cox 回归建立由6个lncRNA(AC090425.3,AC004817.3,AC023590.1,C3orf36,AC012055.1,LINC01854) 组成的预测性m7G相关风险模型。风险评分= [AC090425.3表达量× (-0.434 18) ]+(AC004817.3表达量×0.618 419) +[AC023590.1表达量×(-1.510 279 758)]+ (C3orf36表达量×0.636 472 436) +(AC012055.1表达量×1.367 919 436) + (LINC01854表达量×0.927 740 399)。见图2A~2C。
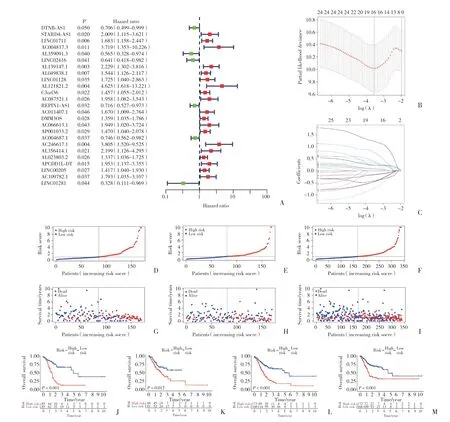
图2 m7G 相关 lncRNA预后模型的构建和训练集、测试集、完整数据集中样本的存活率Fig.2 Construction of m7G-related lncRNA prognostic model and survival rate of samples in training set,test set,and complete data set
2.3 m7G相关lncRNA预后模型的验证与评估
根据计算风险评分的中位值,将所有患者分为低风险组或高风险组。在完整数据集、训练集和验证集中,高风险组患者的总生存期均显著低于低风险组 (图2D~2I)。Kaplan-Meier生存曲线显示,高风险组胃癌患者的总生存时间明显较低风险组患者缩短 (图2J~2L),m7G低风险组胃癌患者的无进展生存期 (progression-free survival,PFS) 也显著高于高风险组 (图2M)。1年、3年和5年ROC曲线下面积 (area under curve,AUC) 分别为0.672、0.658和0.673 (图3A)。风险模型中,1年ROC风险得分较其他临床病理特征预测能力更强 (图3B),1年期C-index为0.672(图3C)。单因素Cox回归分析表明,年龄 (HR:1.027,P< 0.004)、T分期 (HR:1.527,P< 0.001)、风险评分(HR:1.048,P< 0.021) 与OS显著相关 (图3D)。多变量Cox回归分析显示,年龄 (HR:1.036,P< 0.001)、T分期 (HR:1.690,P< 0.001) 和风险评分 (HR:1.063,P< 0.005) 是OS的独立危险因素 (图3E)。
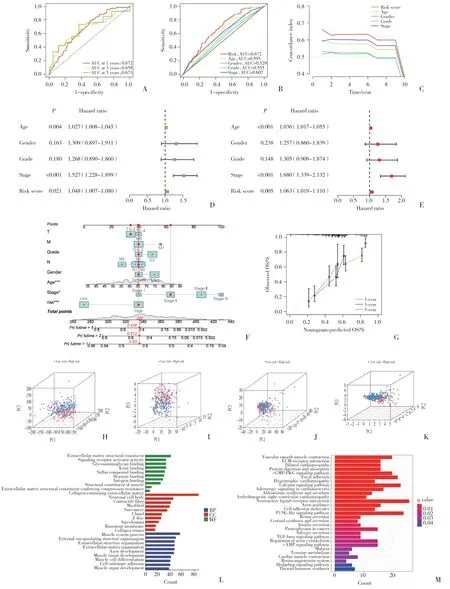
图3 预后风险评估模型验证、列线图、模型校准曲线、PCA 和功能分析Fig.3 Verification of prognosis risk assessment model,nomogram,calibration curves of the model,the PCA and functional analyses
2.4 列线图构建
根据风险评分和临床病理因素,开发了用于预测胃癌患者1年、3年和5年OS的列线图 (图3F)。1年、3年和5年的校准曲线显示出较好的预测准确性,表明该模型具有良好的预测能力 (图3G)。
2.5 PCA和生物途径分析
PCA的三维散点图分别显示了m7G相关lncRNA不同模式的分布情况。PCA分布结果表明,根据风险模型中m7G相关lncRNA的分组具有明显的聚集特征 (图3H~3K)。
根据上述风险特征将所有患者分为m7G高、低风险组,并以log2FC>1和P< 0.05为标准筛选了634个差异表达m7G相关基因。功能富集分析提供了对这些基因的生物学理解。GO分析表明,这些基因分别与肌肉系统过程、肌肉收缩、细胞外基质组织、细胞外结构、外封装结构组织和轴突发育显著相关 (图3L)。KEGG富集分析显示,这些基因与血管平滑肌收缩、ECM-受体相互作用和扩张型心肌病显著相关 (图3M)。
2.6 免疫功能、TIE及免疫治疗药物敏感性
通过ssGSEA方法对肿瘤样本进行打分,发现m7G高、低风险组间的免疫功能也存在差异,高风险组Ⅱ型干扰素 (interferon,IFN) 反应、趋化因子受体 (chemokine receptor,CCR)、抗原递呈细胞 (antigen-presenting cell,APC) 共刺激显著上调 (图4A)。高风险组患者的TIE得分高于低风险组 (图4B)。通过“pRRophetic”R包共找到88种化疗药物的IC50值在高、低风险组间有显著差异。其中,71种药物在低风险组敏感性更高,17种药物则在高风险组中更敏感 (图4C~4J)。
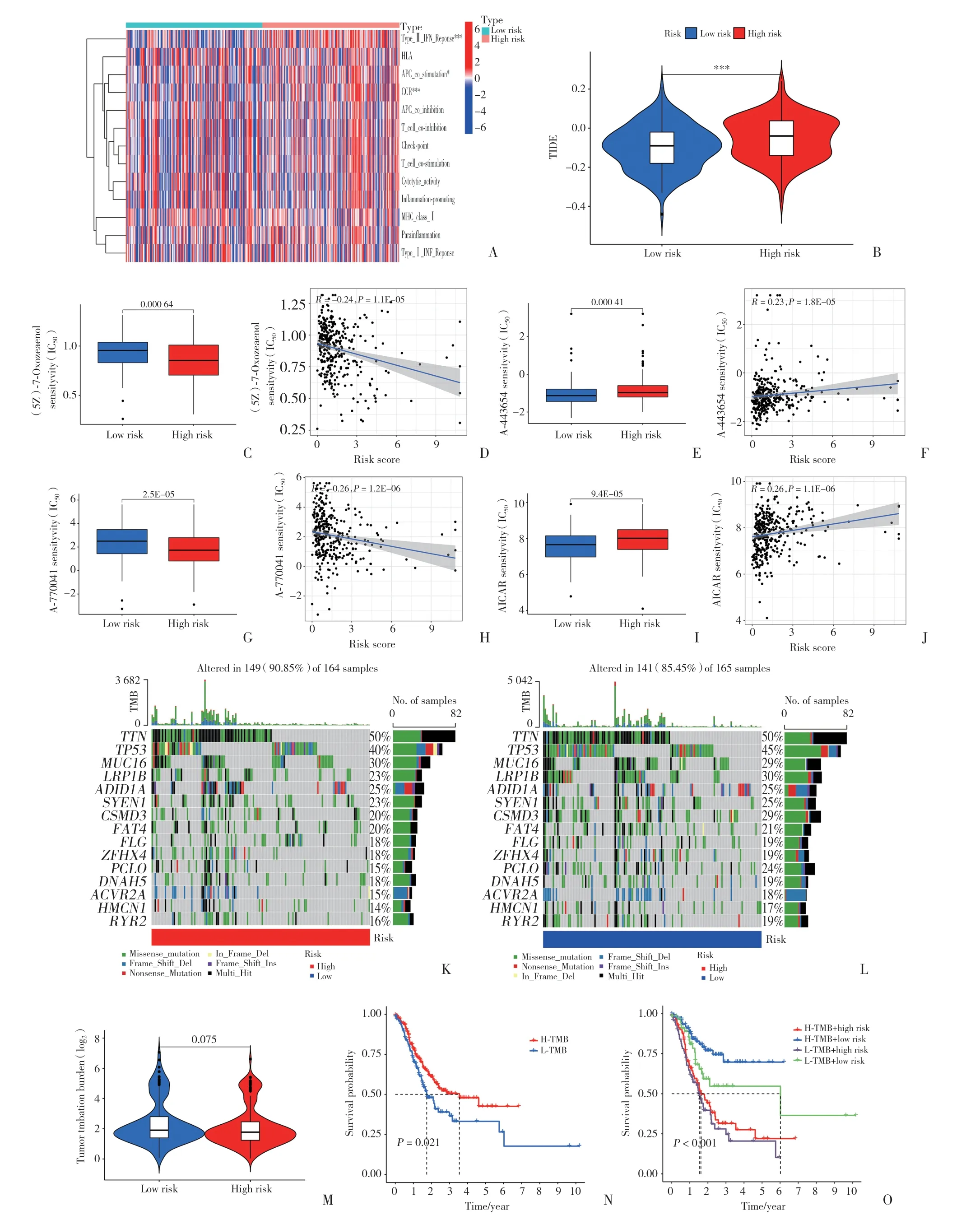
图4 m7G高、低风险组的免疫特征和TMB分析Fig.4 Immune characteristics and TMB analysis of the m7G high-risk and low-risk groups
2.7 TMB
m7G高、低风险组中体细胞突变差异分析显示,突变率最高的10个基因是TTN、TP53、MUC16、LRP1B、ARID1A、SYNE1、CSMD3、FAT4、FLG和ZFHX4(图4K、4L)。与低风险组相比,高风险组患者TMB更低 (图4M、4N)。此外,与其他m7G风险和TMB组相比,m7G高风险组和低TMB组的患者预后最差 (图4O)。
3 讨论
胃癌的发生是环境因素与遗传因素共同作用的结果。在基因及表观遗传学层面研究胃癌发生发展的机制十分重要。m7G修饰参与多种病理生理活动,尤其是肿瘤的发生和进展。目前尚未见胃癌中m7G修饰作用的相关报道。lncRNA已被证实参与肿瘤相关的细胞途径,对肿瘤的诊断及预后具有良好的预测能力[22]。因此,本研究尝试建立一种以m7G相关lncRNA为基础的预测模型,并探讨胃癌中m7G风险水平与生物途径、免疫功能、TIE、免疫治疗药物敏感性和TMD之间的关系。
本研究中,使用多因素Cox风险回归基于6个m7G相关的lncRNA构建了风险模型,通过风险评分中位值将患者分为高、低风险组,结果发现低风险组通常预后更好。通过ROC曲线和C-index曲线证实该模型准确可靠。通过m7G风险情况和临床信息构建了线列图,用于预测患者的生存情况,校准图证明该模型具有良好的预测准确性,可用于预测胃癌患者生存期。
本研究还对m7G高、低风险组的差异基因进行了GO和KEGG富集分析。GO分析结果表明,肌肉系统过程、肌肉收缩、外部封装结构组织等生物学过程在胃癌的预后方面起重要作用。KEGG富集结果显示,m7G高、低风险组的差异基因主要在血管平滑肌收缩、ECM-受体相互作用、扩张型心肌病等通路中富集。并发现肌肉相关过程 (如肌肉系统过程、肌肉收缩、肌肉组织发育、肌细胞分化、肌肉的结构成分及心肌收缩等) 在GO和KEGG富集中均有重要作用。肌肉减少症是一种以骨骼肌质量和力量的进行性和全身性丧失为特征的复杂综合征[23]。衰老通常是原发性肌肉减少症的直接原因,而继发性少肌症则由废用、营养不良、器官衰竭、侵入性干预或恶性肿瘤引起[24]。已有文献[25]报道了胃癌与肌肉减少症之间的联系。此外,有证据表明肌肉减少症可作为胃癌术后并发症和OS的独立预后风险因素[26]。本研究发现,m7G高、低风险组间的差异基因在肌肉相关生物学过程和通路之间富集,因此推测m7G修饰可能在胃癌患者发展为肌肉减少症中发挥作用,并且可能成为m7G高风险组患者预后较差的原因之一。
胃癌具有非常特别的肿瘤微环境 (tumor microenvironment,TME),可促进肿瘤进展和转移[27]。通过对风险模型进行免疫功能分析发现,IFN反应、CCR、APC共刺激在高风险组中的表达均高于低风险组。其中,CCR3、CCR4、CCR5、CCR7已被证明与胃癌的不良预后有关[28],这也验证了本研究结果的准确性和可靠性。
研究[29]发现,TME可作为免疫检查点阻断 (immune checkpoint blockade,ICB) 治疗效果的生物标志物。TMB增加意味着参与转录与翻译的基因突变的可能性也随之增加,因此,新抗体产生的可能性也随之增加,并诱导T细胞毒性和抗肿瘤反应,从而提高ICB治疗的敏感性[30-31]。本研究发现,m7G低风险组的整体TMB高于高风险组,高TMB也预示着更好的预后,但t检验显示2组TMB无统计学差异 (P=0.075)。通过高TMB和低TMB组之间的Kaplan-Meier生存曲线可以看出,高TMB组的OS显著优于低TMB组,这表明TMB对胃癌患者的生存情况具有重要影响,但m7G风险评分与TMB没有显著的相关性。
TIDE 算法被认为是预测癌症中ICB反应的方法之一[32]。TIDE评分越高,ICB反应越差,患者的免疫治疗效果也越差。在本研究中,m7G高风险组TIDE评分显著高于低风险组,表明低风险组具有更好的ICB反应,预示着免疫治疗效果好于高风险组,且预后更好,这也与TMB分析结果一致。此外,本研究用“pRRophetic”R包共筛选出88种在m7G高、低风险组中治疗效果具有显著差异的药物,其中71种药物在低风险组中敏感性更高,只有17种药物在高风险组中敏感性更高,这也进一步证实了本研究对TMB和TIDE的猜想,可为临床胃癌治疗提供参考。
综上所述,本研究通过6个m7G相关lncRNA构建了1个预后模型,用于预测胃癌患者的预后,并得到了校准曲线的验证;通过GO和KEGG富集分析预测m7G修饰在胃癌患者发展为肌肉减少症中可能发挥潜在作用;通过TMB、TIDE和药物敏感性分析证实m7G低风险组具有更高的ICB反应及更好的药物敏感性。本研究存在一定的局限性,本研究为回顾性研究,数据处理和样本选择存在不可避免的偏差,未来仍需通过其他数据库或体内外实验对本研究结果进行验证。
