长双歧杆菌W13 的全基因组序列分析
2023-09-19许婷婷司静闫梦娜逯彬刘翔王艳萍耿伟涛
许婷婷,司静,闫梦娜,逯彬,刘翔,王艳萍,耿伟涛
(天津科技大学 食品科学与工程学院,天津 300457)
哺乳动物的胃肠道内定植着复杂的微生物群落,这些微生物在调控宿主营养吸收、肠道功能及免疫应答等方面具有重要作用[1]。其中,益生菌作为胃肠道中的有益细菌,有助于改善肠道微生态平衡、促进人体健康[2-3]。据统计,目前人体中所含益生菌种类超过400 种,其中主要有4 类:乳酸菌、双歧杆菌、酵母菌和芽孢杆菌[4]。
双歧杆菌是较早发现的一种重要的益生菌。目前鉴定的绝大多数双歧杆菌来源于人类和其他哺乳动物的胃肠道,其在胃肠道中的数量取决于个体的年龄和饮食方式[5]。Arboleya 等[6]发现腹泻、绞痛、过敏、坏死性小肠结肠炎、肥胖等疾病患者的胃肠道中双歧杆菌丰度显著降低,这表明双歧杆菌对肠道菌群稳态具有重要贡献[7]。
随着高通量测序技术的发展,对双歧杆菌的研究进入了基因组学时代[8]。了解菌株的基因组基本信息,可以更进一步认识基因与蛋白质、代谢功能、个体行为之间的关系。比较基因组是在高通量测序的基础上发展而来,通过比较不同物种、菌株或同一物种的不同菌株的基因组的差异和相似性,能够了解基因功能之间的关系[9]。
本课题组在前期试验中从婴儿粪便中筛选分离得到了一株具有抗氧化特性的长双歧杆菌。长双歧杆菌作为人体肠道内存在的分布最广、丰度最高的一种双歧杆菌,且具有一定的益生特性,近年来引起了广泛的关注。本文通过生物信息学技术研究长双歧杆菌W13 的基因组特征,挖掘菌株的功能特性基因,以期为长双歧杆菌长亚种资源的开发与应用提供参考。
1 材料与方法
1.1 材料
长双歧杆菌W13:天津科技大学发酵食品与益生菌资源开发实验室前期自婴儿粪便中分离得到。菌株W13 的基因组序列已提交至美国国立生物技术信息中心(national center for biotechnology information,NCBI)的GenBank 数据库,登录号为CP096771.1。
1.2 方法
1.2.1 菌株W13 的基因组特性
自NCBI 下载菌株W13 的全基因组序列,利用CGVIEW(https://proksee.ca/projects/new)预测基因组的基因结构,并进行CRISPR 序列的预测。
1.2.2 生物信息学分析
使用本地Blast 工具将预测得到的编码基因蛋白序列与直系同源蛋白分组比对(evolutionary genealogy of genes:non-supervised orthologous groups,eggNOG)数据库、基因组百科全书(kyoto encyclopedia of genes and genomes,KEGG)进行比对,获得编码基因的功能信息;通过碳水化合物活性酶(carbohydrate-active enzymes,CAZy)数据库的比对分析基因组中的碳水化合物活性酶。通过使用子系统技术快速注释(rapid annotation using subsystem technology,RAST)数据库、抗性基因数据库(comprehensive antibiotic resistance database,CARD)完成菌株W13 基因组中耐药基因的预测。使用IslandViewer4 (https://www.pathogenomics.sfu.ca/islandviewer/upload/)对菌株W13 中存在的基因组岛进行预测[10]。利用PHASTER(https://phaster.ca/)在线网站分析鉴定和注释微生物基因组内的噬菌体序列[11-13]。
对于系统发育树的构建,先使用软件BPGA 预测核心基因,再使用软件MEGA 11.0.13 构建系统发育进化树。根据核心基因建树结果,选取与菌株W13 亲缘关系较近的7 株菌,利用EZBioCloud 网站(https://www.ezbiocloud.net/tools/ani) 比较菌株W13 与7 株菌之间的平均核苷酸一致性(average nucleotide identity,ANI)[14],并绘制热图。长双歧杆菌长亚种的泛基因组和核心基因组预测使用PanGP 软件完成[15],通过BRIG 软件比较分析基因组。
2 结果与分析
2.1 菌株W13 基因组特性
2.1.1 菌株W13 基因组基本特征
对菌株W13 的基因组序列进行分析,其基本特征见表1。
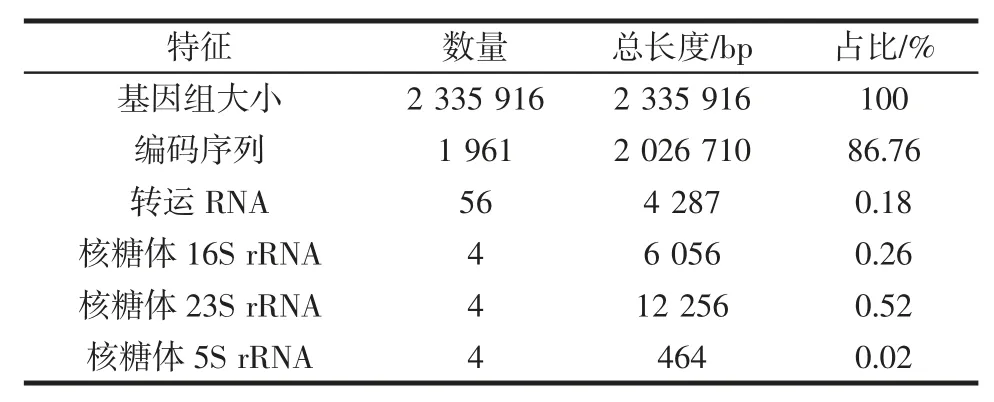
表1 W13 基因组基本特征Table 1 Basic characteristics of Bifidobacterium longum W13 genome
由表1 可知,长双歧杆菌W13 的基因组序列全长为2 335 916 bp。此外,长双歧杆菌W13 的GC 含量(GC 含量指DNA 中鸟嘌呤和胞嘧啶所占比例的比率)为60.95%。对菌株W13 的基因组基本特征做可视化处理,绘制菌株W13 的染色体基因组圈图,见图1。
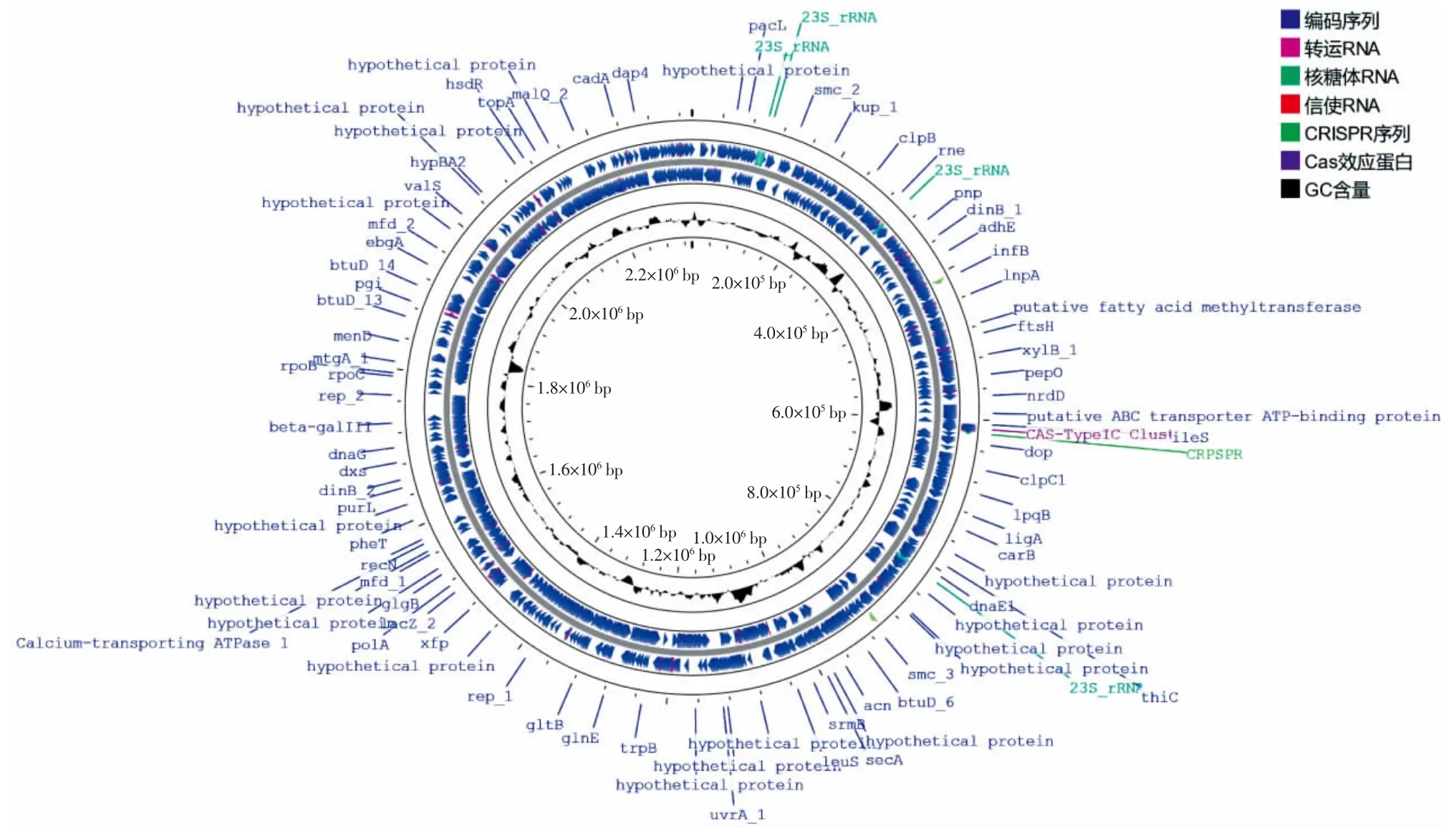
图1 长双歧杆菌W13 的基因组圈图Fig.1 Genomic cycle of Bifidobacterium longum W13
图1 展示了预测的GC 含量、编码序列区(coding sequence,CDS)功能、转运RNA(transfer RNA,tRNA)、核糖体RNA(ribosomal RNA,rRNA)、信使RNA(transfermessenger RNA,tmRNA)、成簇规律间隔的短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)。该菌株的基因组由一个不包含质粒的完整环状染色体构成,依据CGVIEW 预测菌株W13 基因组中的编码基因,共包含1 961 个编码基因,占整个基因组的86.76%。CRISPR 序列的具体排布方式见图2。
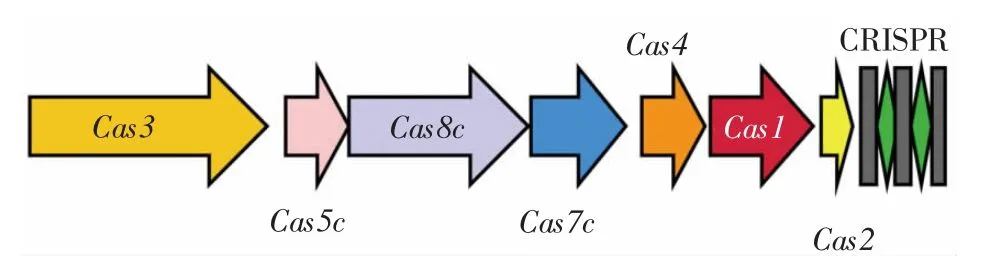
图2 菌株W13 中的CRISPR 基因簇Fig.2 CRISPR gene cluster in strain W13
由图2 可知,在菌株W13 中,依据其Cas 效应蛋白可将菌株含有的内源CRISPR 系统分类为Ⅰ-C 型。CRISPR 序列广泛分布在原核生物中,是大多数细菌及古细菌中一种不断进化适应的免疫防御机制,其分布具有菌株特异性,可用于进行菌株的分型。
2.1.2 菌株W13 的基因组岛预测
基因组岛是微生物基因组中可水平转移的基因簇,基因组岛的转移是微生物基因交换的一种方式,可以提高微生物的多样性和对环境的适应性。
使用IslandViewer4 基因组岛在线预测网站得到的菌株W13 基因组岛预测结果见图3。
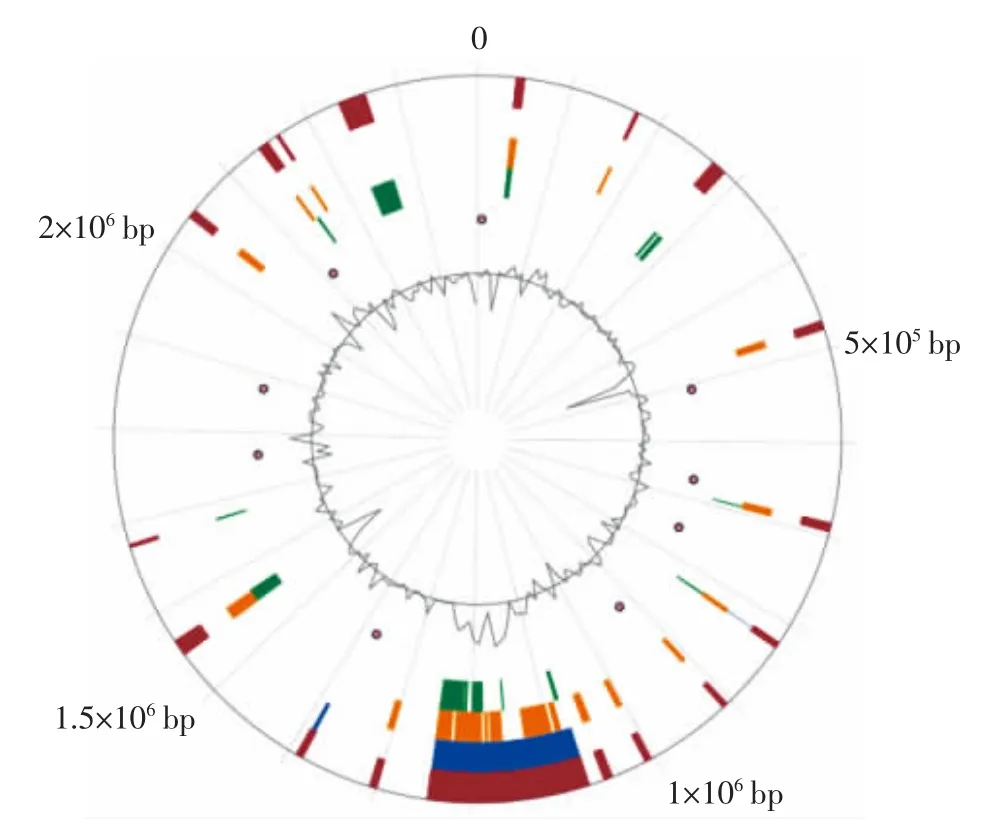
图3 菌株W13 的基因组岛预测结果Fig.3 Predicted genomic islands in strain W13
从图3 可以看出,菌株W13 的染色体上共含有33 个基因组岛。在这33 个基因组岛中,岛4 是最长的基因组岛,长度为25 714 bp;岛13 是最短的基因组岛,长度为4 321 bp。
其中,岛23 属于碳水化合物活性酶GH2 家族;岛16 属于碳水化合物活性酶GH25 家族;岛2、岛26 均属于碳水化合物活性酶GH43 家族;岛4、5、6 均属于碳水化合物活性酶GT2 家族。碳水化合物活性酶相关基因占总基因组岛的21%,这表明菌株W13 具有较高的糖合成能力。
此外,岛7 与CRISPR 序列有关,包含Cas 效应蛋白,这与2.1.1 的分析对应。
2.2 菌株W13 的基因功能注释
2.2.1 eggNOG 数据库注释
eggNOG 数据库收集了全面的物种和大量的蛋白序列数据,并且能进行同源基因分类以及功能注释,是目前最先进、最完善的数据库。
通过分析,通过eggNOG 数据库注释,预测菌株W13 的蛋白质功能见图4。

图4 eggNOG 数据库注释分类统计图Fig.4 eggNOG database annotation classification statistics
从图4 可以看出,在菌株W13 的基因组中,除占比最多的假定蛋白外,关于碳水化合物的运输和代谢(G)、氨基酸的运输和代谢(E)的相关基因最多,分别占比10.30%、9.95%,表明菌株可能具有较高的糖代谢和氨基酸代谢能力;其次为K(转录)、L(复制、重组和修复)、J(翻译、核糖体结构和生物发生),分别占比9.36%、9.07%、8.37%。
2.2.2 KEGG 数据库注释
KEGG 数据库是收集了生物的基因组、通路和化合物信息的综合性的数据库,该数据库主要将基因组信息分为三大类,分别为环境信息处理(environmental information processing)、新陈代谢(metabolism)、遗传信息处理(genetic information processing),通过KEGG 数据库注释,菌株W13 中关于环境信息处理、新陈代谢、遗传信息处理中分别涉及基因数目为151、630、90 个。将这三大类中涉及较多的基因统计,得到的注释信息见图5。
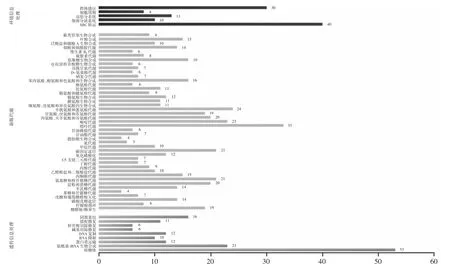
图5 KEGG 数据库注释分类统计图Fig.5 KEGG database annotation classification statistics
从图5 可以看出,环境信息处理类占比最多的为腺苷三磷酸结合盒转运蛋白(ATP-binding cassette transporter proteins)。该家族蛋白是已知最大的蛋白质家族之一,广泛存在于细菌、古菌和真核生物中。特别地,群体感应(quorum sensing,QS)相关基因占比较多,QS 是细菌根据细胞密度变化进行基因表达调控的一种生理行为,可通过微生物的信息交流调控肠道屏障功能与营养素代谢,进而维持机体对营养素的吸收和肠道健康,促进肠道稳态。新陈代谢类数量最多的为嘌呤代谢途径相关基因,很多细菌在人体嘌呤代谢中起着重要作用,人体内三分之二的尿酸是由肾脏排出的,其余的主要由肠道排出,尿酸酶由尿酸转化为尿囊素和尿素,广泛存在于乳酸菌中。因此菌株W13 可能具有延缓肾脏疾病进程的潜力。遗传信息处理类数量最多的为核糖体相关基因,主要负责基因的翻译。
2.2.3 RAST 数据库注释
RAST 数据库可用于注释细菌和古菌基因组,该服务器通过识别蛋白质编码基因、rRNA 和tRNA 基因,来预测不同基因的功能,使用这些信息重建代谢网络[16]。
在菌株W13 的基因组中,RAST 数据库注释到了28%的基因,预测结果见图6。
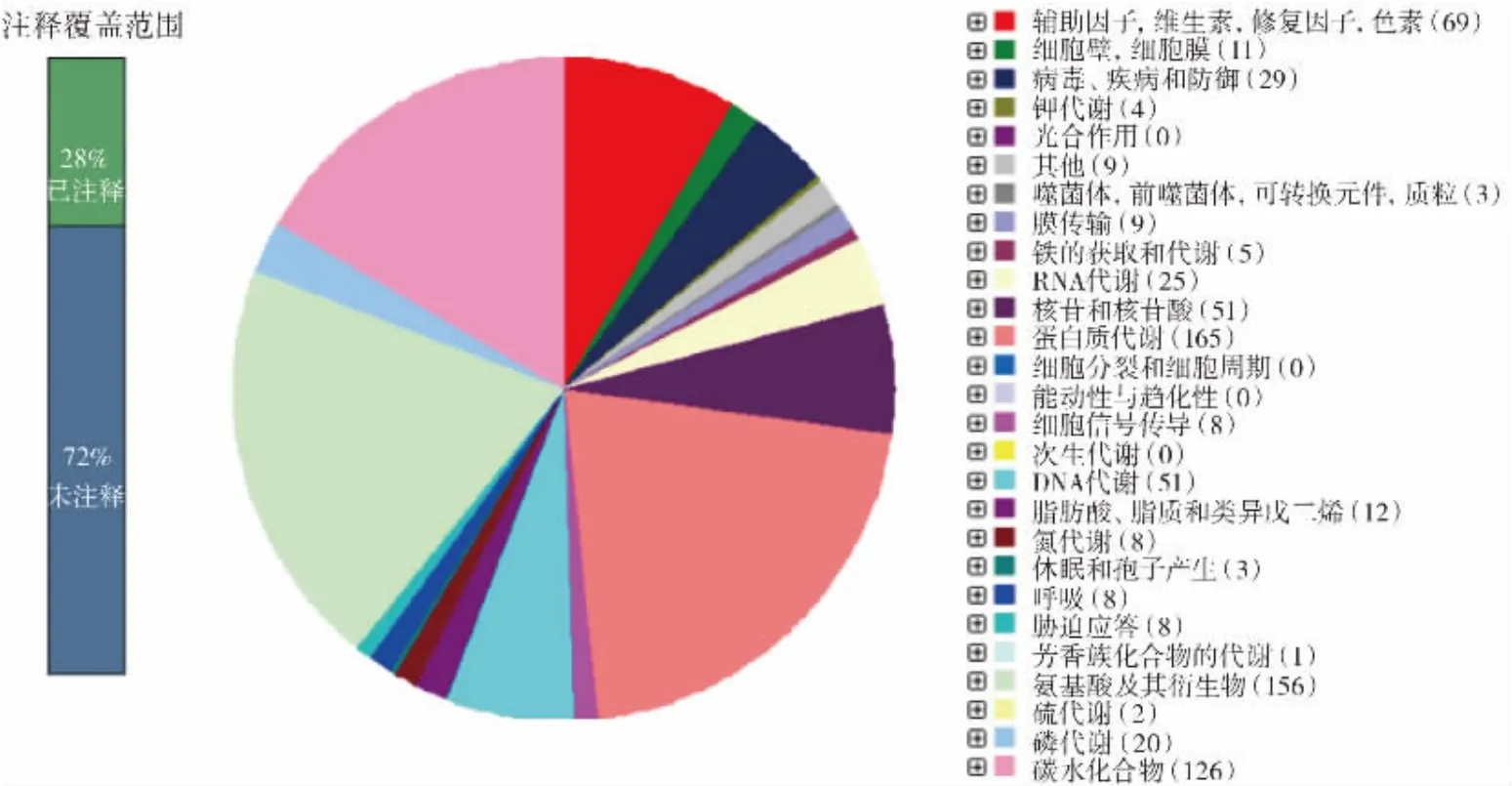
图6 RAST 数据库注释Fig.6 RAST database annotation
从图6 可以看出,在菌株W13 的基因组功能预测结果中,占比最高的三大类分别为蛋白质代谢(protein metabolism)、氨基酸及其衍生物(amino acids and derivatives)、碳水化合物(carbohydrates),分别占比21%、19.92%、16.09%。特别地,在胁迫应答调控基因类下注释到了谷胱甘肽氧化还原循环相关酶,谷胱甘肽是一种三肽,在微生物的大多数活细胞中以高浓度存在,谷胱甘肽在众多细胞功能中起着关键作用,包括清除自由基、参与氧化还原反应、参与脱氧核糖核苷酸的形成等,这表明菌株W13 可能具有抗氧化潜力,印证了本课题组前期对菌株W13 抗氧化能力的研究[17]。同时,还预测到了胆盐水解酶相关基因,这表明菌株W13 可能具有耐胆盐的能力。
2.2.4 CAZy 数据库注释
CAZy 数据库是收录碳水化合物活性酶的数据库。该数据库将蛋白质按功能类别分为六大类:糖苷水解酶(glycoside hydrolases,GH)、糖基转移酶(glycosyl transferases,GT)、多糖裂解酶(polysaccharide lyases,PL)、碳水化合物酯酶(carbohydrate esterases,CE)、辅助氧化还原酶(auxiliary activities,AA)和非催化的结合碳水化合物的功能域(carbohydrate-binding modules,CBM)。菌株W13 的注释结果见图7。
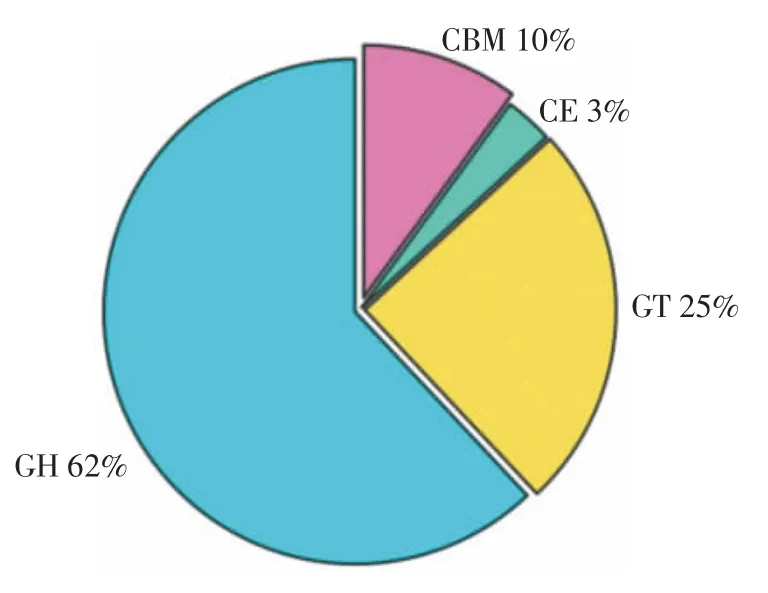
图7 碳水化合物酶分布比例图Fig.7 Distribution ratio of carbohydrate enzymes
在菌株W13 中共注释到80 个GH 家族,其中占比较多的是GH2、GH3、GH13 和GH43,数量分别为5、6、13 和8。GH2 分布在哺乳动物组织、植物和微生物中,能够产生具有转糖基活力的β-半乳糖苷酶,这是以母乳或奶粉为主要营养来源的婴幼儿体内最重要的消化酶,GH2 家族的β-半乳糖苷酶包括LacZ 型和异二聚体LacLM 型酶,双歧杆菌菌株多含有LacZ 型酶[18]。在食品工业中,β-半乳糖苷酶因其水解乳糖和合成低聚半乳糖的能力而被利用;GH3 家族包含β-葡萄糖苷酶,该酶在微生物中尤为普遍,可使乳糖水解为葡萄糖和半乳糖。GH3 和GH43 家族含具有β-木糖苷酶活性的糖苷水解酶,该酶是重要的木糖分解酶,用于许多生物技术过程,可应用于海洋产品加工;GH13 为淀粉水解酶家族,是双歧杆菌属糖生物组中最丰富的GH 家族之一[19-21]。
此外,在菌株W13 中共注释到32 个GT 家族,其中占比较多的糖基转移酶家族是GT2 和GT4,数量分别为17、9。多种合成型糖基转移酶属于GT2 家族,例如纤维素、几丁质、透明质酸或葡聚糖合酶。GT4 具有蔗糖合成酶、蔗糖磷酸合成酶等活性[22],综上说明W13有较高的糖合成能力,具备合成胞外多糖的潜力。
2.2.5 抗生素抗性基因预测
将菌株W13 基因的蛋白序列上传到CARD,使用工具软件RGI 进行比对并预测潜在的抗性基因。预测结果见表2。
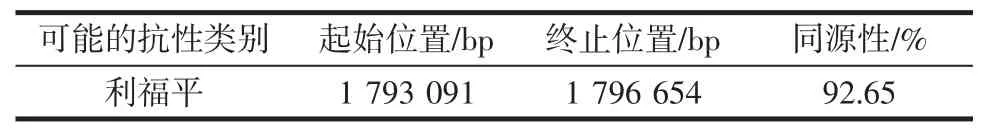
表2 CARD 预测抗性基因Table 2 Resistance genes predicted by CARD
根据表2 的比对发现,菌株W13 中含有利福平(rifampicin,RFP)抗性基因,与青春双歧杆菌rpoB 突变体的序列相似性达到了92.65%。
2.2.6 前噬菌体预测
一部分微生物的横向转移基因是通过噬菌体的侵染而获得的。在PHASTER 中共预测出2 条前噬菌体片段,见表3。

表3 前噬菌体预测基本情况Table 3 Basic information of prophage prediction
由表3 可知,菌株W13 中所包含的2 条前噬菌体长度均小于30 kbp(1 kbp=1 000 bp),且不完整,因此认为这2 条噬菌体不具有活性。将预测到的前噬菌体片段对比基因组岛分析,发现基因组岛中包含部分与前噬菌体相关的基因片段,但不具有噬菌体活性。因此认为菌株W13 中并无可进行水平转移的致病岛。
2.3 比较基因组学分析
2.3.1 基于核心基因的系统发育进化树分析
利用BPGA 软件将菌株W13 的蛋白序列与8 株长双歧杆菌猪亚种、8 株长双歧杆菌婴儿亚种及8 株长双歧杆菌长亚种的蛋白序列分析比对,结果见图8。
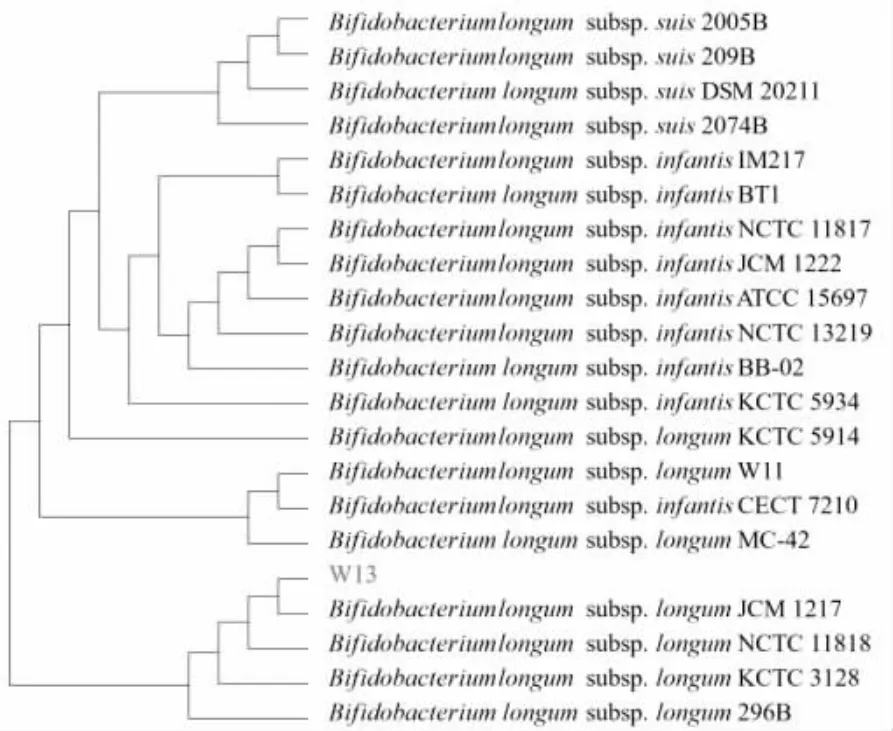
图8 基于核心基因建立的菌株W13 系统发育树Fig.8 Phylogenetic tree of strain W13 based on core gene
图8 的比对结果显示,菌株W13 的核心基因数为990,必需基因数为857。利用核心基因建立系统发育树,由图8 可知,菌株W13 与模式菌株Bifidobacterium longum subsp. longum JCM 1217 进化遗传距离最近,推测菌株W13 为长双歧杆菌长亚种。Bifidobacterium longum subsp.infants CECE 7210 与长亚种的进化遗传距离较近,这一结果与Blanco 等[24]的研究结果一致,是由于CECE 7210 被错误地分类到了婴儿亚种中。
2.3.2 平均核苷酸一致性分析
基于核心基因组,选定了7 株与菌株W13 亲缘关系较近的菌株,在NCBI 数据库中下载的这7 株长双歧杆菌的基因组数据见表4。
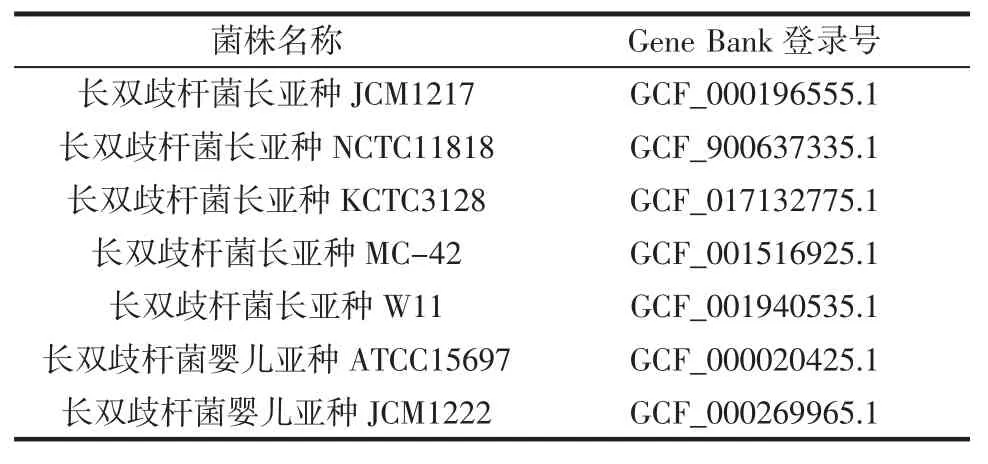
表4 ANI 分析选用的7 株菌基因组信息Table 4 Genome information of 7 strains selected for ANI analysis
将菌株W13 与表4 中的7 株菌进行了ANI 值的计算,对比结果见图9。
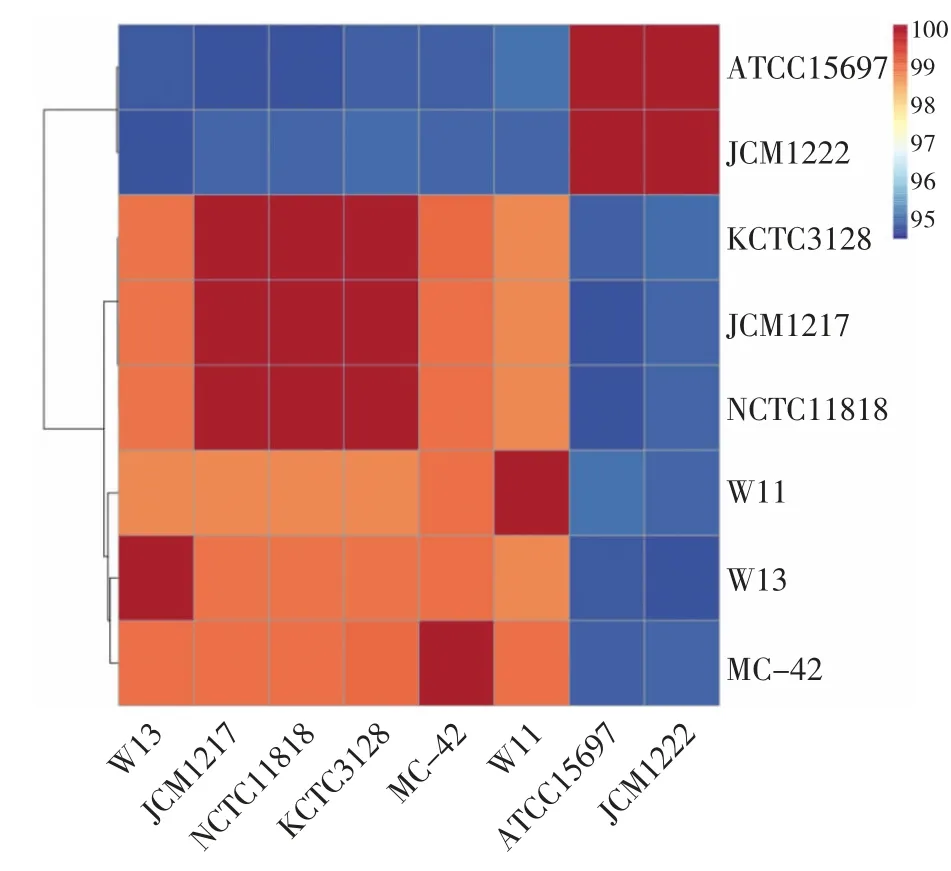
图9 菌株W13 与亲缘关系较近的8 株菌的ANI 值Fig.9 ANI value of strain W13 and 8 strains with close relationship
由图9 可知,菌株W13 与5 株长双歧杆菌长亚种的ANI 值高于98%,而与2 株长双歧杆菌婴儿亚种的ANI 值均在95%以下,与系统发育树的结果具有一致性。这说明菌株W13 属于长双歧杆菌长亚种。
2.3.3 泛基因组、核心基因组分析
使用PanGP 软件预测了长双歧杆菌长亚种的泛基因组和核心基因组随基因组数目的变化,结果见图10。
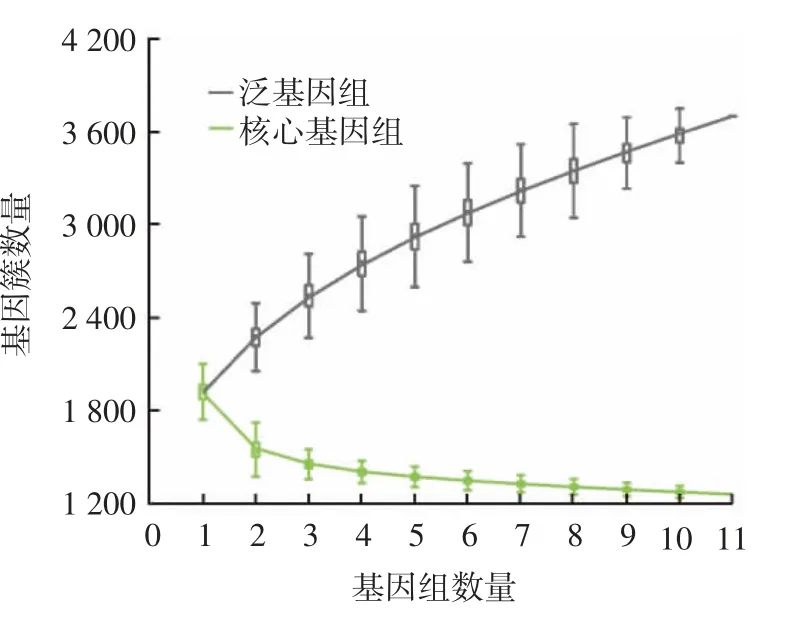
图10 Bifidobacterium longum subsp. longum 的泛基因组特征曲线Fig.10 Pan-genome characteristic curves of Bifidobacterium longum subsp. longum
由图10 可以看出,随着菌株数量的增加,泛基因组的拟合曲线呈上升趋势,这说明长双歧杆菌长亚种的泛基因组是开放性的,这可能是由于长双歧杆菌长亚种在自然界中分布广泛,与外界各种遗传物质发生交换,说明该物种有较高的遗传多样性。相反,其核心基因的数量随菌株数量增加逐渐趋于稳定,说明该物种的稳定性较高。
2.3.4 长双歧杆菌长亚种的基因组比较
利用BRIG 软件对菌株W13 与8 株长双歧杆菌长亚种的全基因组进行了比较,这8 株菌的基因组信息见表5。
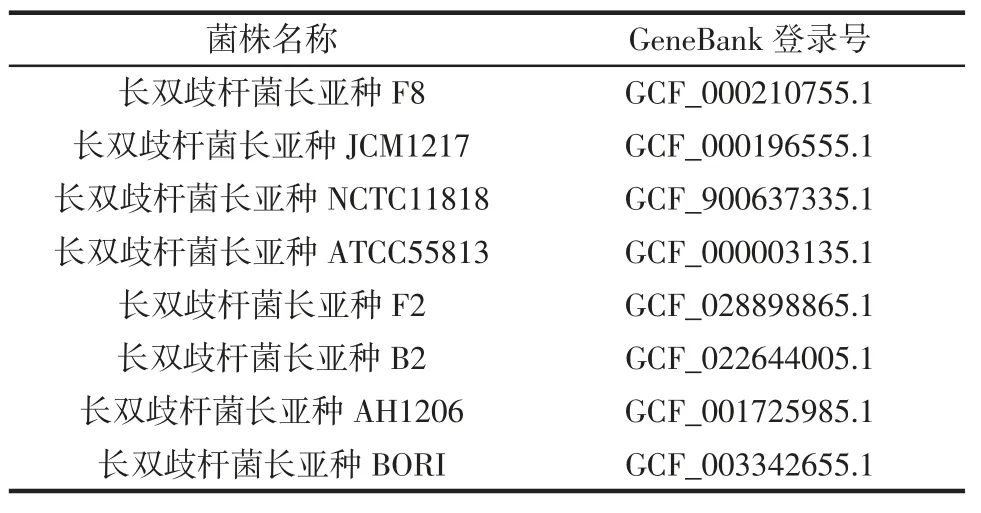
表5 BRIG 软件比较基因组使用的菌株信息Table 5 Strain genome information compared by BRIG software
以菌株W13 作为参考基因组,菌株W13 与从NCBI 数据库下载的8 株长双歧杆菌长亚种的基因组进行比较,结果见图11。
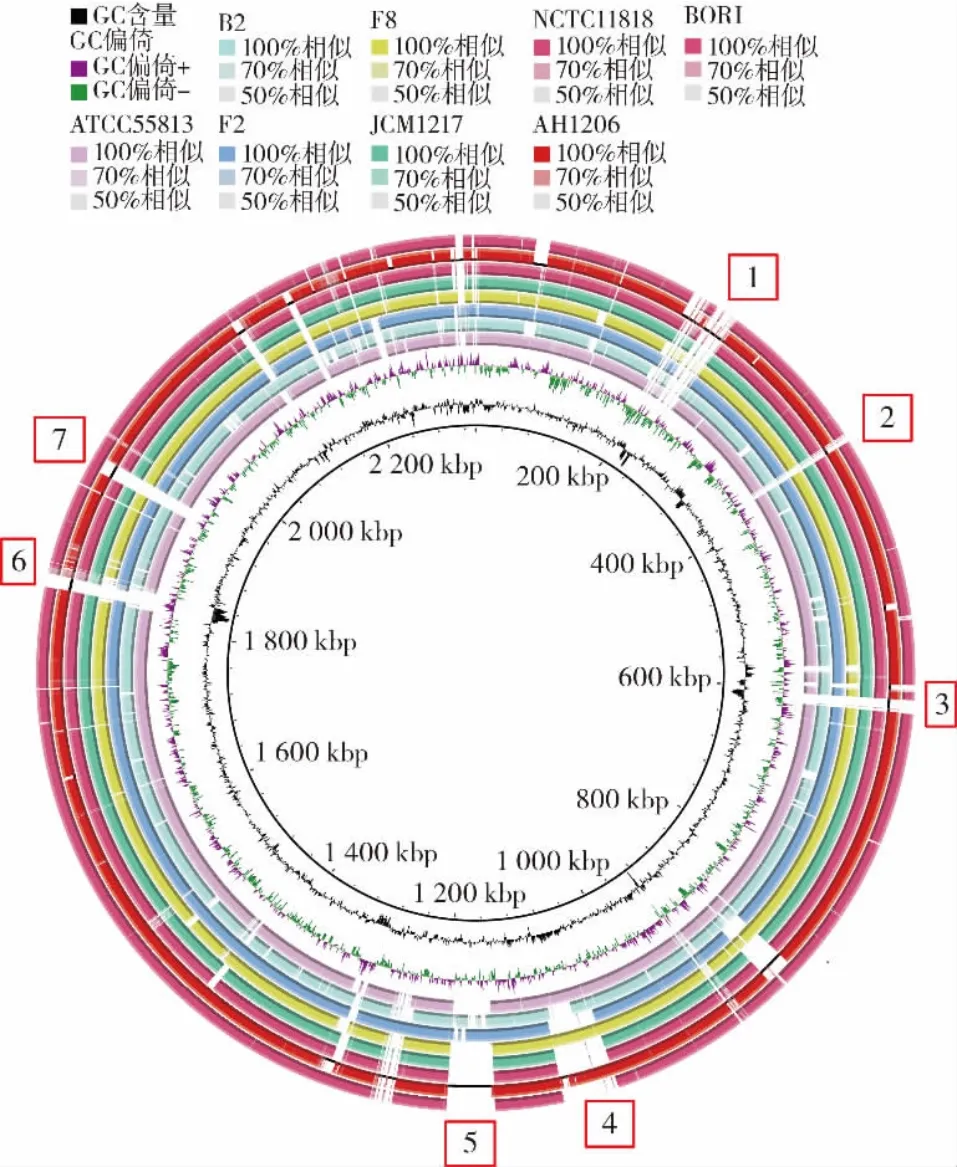
图11 比较基因组图Fig.11 Comparative genome map
由图11 可以看出,菌株W13 的染色体与其它菌株相比有其特定的功能基因。图中空白区域代表在参考基因组中存在而在其它基因组中不存在的基因,即区域1~7 是菌株W13 所特有的。经过分析,区域2、4、7 为假定蛋白,区域1、5 与复制、重组和修复区域;细胞壁/膜/包膜生物发生有关;区域3 为CRSPR 序列区域;区域6 与辅酶转运与代谢相关,表明W13 可能具有更强的抗氧化、抗衰老、抗炎等能力。
3 结论
本研究从基因组特性出发,对长双歧杆菌W13 进行了系统化的分析。通过对菌株W13 进行全基因组测序,在KEGG、eggNOG、RAST 数据库中系统分析了其功能基因与代谢通路,发现该菌株可能具有抗氧化、耐胆盐的能力,W13 含有较多的群体感应基因,可促进肠道稳态。除此之外,W13 含有内源Ⅰ-C 型CRISPR 系统,可用于进行菌株的分型,未来可应用于进行内源CRISPR 系统基因编辑。通过分析碳水化合物活性酶基因,预测该菌株具有合成胞外多糖的潜力,能够高产胞外多糖。通过基因组学分析,认为长双歧杆菌长亚种在自然界中分布广泛,有较高的遗传多样性和稳定性。综上,认为长双歧杆菌W13 是一株益生功能强、具有商业化价值的菌株,为长双歧杆菌的开发与应用提供了参考。
