饥饿胁迫对拟穴青蟹大眼幼体的比较转录组分析
2023-09-14胡伟胜马凌波张凤英刘志强马春艳
胡伟胜,王 伟,马凌波,张凤英,刘志强,赵 明,马春艳
(1.中国水产科学研究院东海水产研究所遗传育种与生物技术实验室,上海 200090;2.上海海洋大学水产与生命学院,水产科学国家级实验教学示范中心,上海 201306)
自然界中,食物匮乏是甲壳类时常面临的环境胁迫之一。甲壳类面对短期或长期的营养限制时经常通过调整代谢、行为和消耗内部储存的氨基酸、脂质和碳水化合物以维持基本的生理功能并实现有限的生长和繁殖[1-7]。饥饿胁迫下,颗粒新厚蟹(Neohelicegranulata)肌肉组织的氨基酸代谢和肝胰腺的甘油异生发生明显改变[1-2]。近期的研究表明,饥饿胁迫激活了中华绒螯蟹(Eriocheirsinensis)肝胰腺的脂质代谢和糖原代谢[3]。饥饿压力下,艾氏滨蟹(Carcinus aestuarii)免疫功能显著改变[4],凡纳滨对虾(Litopenaeusvannamei)免疫酶的活性显著升高[5]。饥饿压力下,甲壳类行为也会发生适应性改变。例如,在饥饿压力下,三疣梭子蟹(Portunustrituberculatus)和日本蟳(Charybdis japonica)的捕食率呈先升后降的趋势[6],凡纳滨对虾的游泳速度明显放缓[7]。因甲壳动物的肠道微生物群落与宿主的消化和免疫功能紧密相关[8-9],在饥饿状态下的凡纳滨对虾肠道微生物的多样性和群落结构与健康虾虽无统计学差异,但是饥饿虾的肠道中机会病原体丰度更高,导致虾生病几率大大增加[5]。
拟穴青蟹(Scyllaparamamosain)俗称青蟹,隶属于节肢动物门(Arthropoda),甲壳纲(Crustacea),十足目(Decapoda),梭子蟹科(Portunidae),青蟹属。属广温性和广盐性海洋经济甲壳动物,是我国东南沿海地区重要的海水养殖蟹类。迄今为止,关于梭子蟹科的饥饿胁迫研究主要侧重于表观指标的呈现。例如,饥饿后复投喂降低了锯缘青蟹[10]和三疣梭子蟹[11]Ⅰ期溞状幼体的生长指标和存活率。对于饥饿胁迫分子机理的研究目前相对较少见,特别是幼体阶段的个体。本文对正常投喂和饥饿状态下的拟穴青蟹大眼幼体作比较转录组分析,以探究饥饿胁迫对拟穴青蟹大眼幼体代谢过程的影响,以期为拟穴青蟹苗种的健康培育提供理论参考,并丰富拟穴青蟹的分子生物学基础数据。
1 材料与方法
1.1 样本采集
实验用蟹采自中国水产科学研究院东海水产研究所浙江宁海研究中心,正式实验前将即将蜕壳的Ⅴ期溞状幼体从育苗池转移至塑料圆柱形桶(底面半径25 cm,高80 cm)中,待其顺利蜕壳后捞取大眼幼体开展后续实验。本实验选用的大眼幼体来源于课题组培育的同一亲本“东方1号”。实验期间用1 L的玻璃烧杯养殖大眼幼体,保持温度27.5~30.0℃、盐度24~26、pH 8.0~8.4,饵料为孵化的丰年虫无节幼体。每天上午8∶00采用滴管滴入的方式投喂饵料。共设2个处理组,分别为对照组(正常投喂3 d组,F3S0)和饥饿组(饥饿3 d组,S3F0),每组3个重复,每个重复8只大眼幼体。处理3 d后采用滴管吸入的方式采集大眼幼体,并将其置于RNA保存液中4℃过夜,-80℃保存。
1.2 RNA提取
8只大眼幼体作为一个样品,用TRIzol法提取样品RNA,利用Nanodrop 2000超微量分光光度计(Thermo,美国)检测RNA浓度和纯度,用琼脂糖凝胶电泳检测RNA完整性,用Agilent 2100生物分析仪(Agilent,美国)测定RNA完整值(RNA integrity number,RIN)。OD260/280介于1.9~2.1。
1.3 文库构建
利用带有Oligo(dT)的磁珠与mRNA上的poly-A结合纯化mRNA,采用离子打断的方式,将mRNA分成200~300 bp的片段。以mRNA作为模板,反转录合成cDNA第一链,再以第一链cDNA为模板合成双链cDNA。双链cDNA经末端修复、加接头、连接、纯化、PCR扩增和cDNA回收构建cDNA文库。使用Agilent 2100质检文库,利用Pico green检测文库浓度,用qRT-PCR定量检测有效文库。文库基于Illumina HiSeq测序平台测序(Illumina HiSeq 3000)。
1.4 转录组De novo组装
Illumina测序得到原始序列(raw reads)。对原始序列进行数据过滤,将原始序列中的接头序列和低质量序列去除,获得高质量序列(clean reads)。使用Trinity软件对高质量序列进行拼接得到转录本和unigene。为获得全面的基因功能信息,将拼接得到的unigene与NR、GO、KEGG、Pfam、eggNOG和Swissprot等数据库进行比对注释。
1.5 差异基因表达
采用DESeq软件分析基因表达差异,将筛选条件设定为:|log2FoldChange|>1,显著性P<0.05。使用topGO对差异基因进行GO富集分析。利用KOBAS数据库(KOBAS网址:http://kobas.cbi.pku.edu.cn/)对差异基因进行KEGG富集分析和KO分析。当矫正后的P值<0.05时,认为该功能显著富集。
1.6 实时荧光定量PCR(qRT-PCR)验证
为了验证测序结果的准确度,实验随机挑选了Met、MIH、E75、α-amylase和Trypsin基因进行qRT-PCR验证。利用NCBI Primer-Blast软件设计荧光定量引物,内参选择18S rRNA(表1)。引物送至上海杰李生物技术有限公司合成。使用ABI QuantStudioTM6 Flex System 仪和TaKaRa SYBR Premix ExTaqⅡ试剂分析F3S0和S3F0差异基因的表达水平。反应体系采用TaKaRa SYBR Premix ExTaqⅡ说明书中20μL体系标准,PCR反应程序为:95℃30 s;95℃5 s,60℃34 s,40个循环;95℃15 s,60℃1 min,95℃15 s。Met、MIH、E75、α-amylase和Trypsin基因的相对表达分析采用2-△△Ct法。数据处理使用SPSS26.0 软件进行单因素方差分析(one-way ANOVA)和Duncan检验,显著性水平设置为P<0.05。
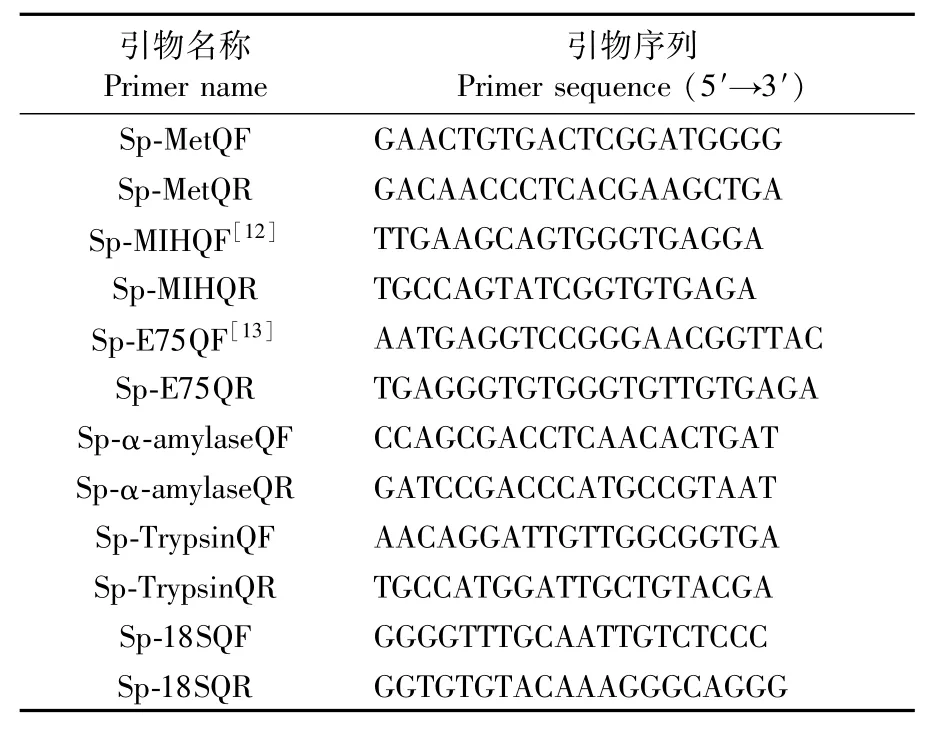
表1 qRT-PCR引物序列Tab.1 Primers for quantitative RT-PCR
2 结果与分析
2.1 转录组测序和De novo组装
对正常投喂和饥饿3 d的拟穴青蟹大眼幼体进行测序,分别获得162 469 532和139 222 024条原始序列。原始数据过滤(去除接头序列和低质量序列)后分别获得 150 103 944 和128 924 890条高质量序列(表2)。使用Trinity软件对高质量序列做De novo组装,获得158 201个转录本。将转录本去冗余后获得79 581个unigene,最大长度20 651 bp,平均长度1 065.44 bp(表3)。后续分析全部以unigene序列作为参考序列。
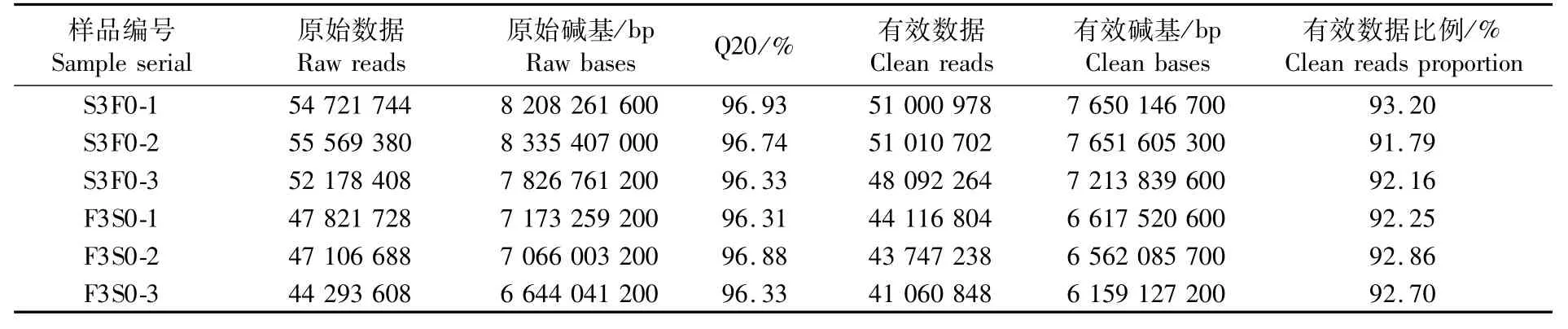
表2 转录组测序数据统计Tab.2 Statistic summary of sequencing data
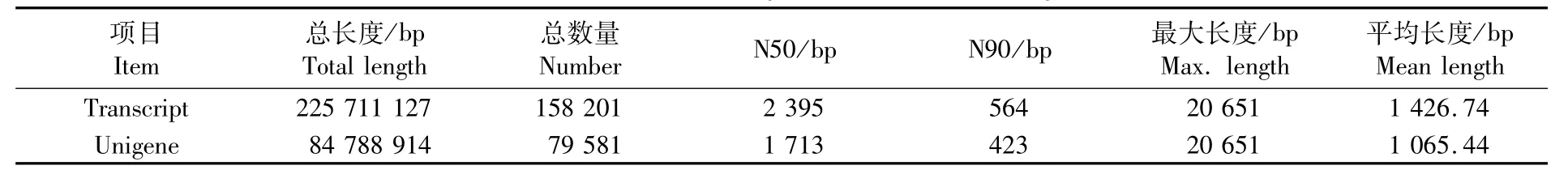
表3 De novo组装结果统计Tab.3 Statistic summary of De novo assembly data
2.2 功能注释
Nr、GO、KEGG、Pfam、EggNOG和Swissprot数据库的注释信息统计结果见表4。注释到GO数据库的unigene共7 294个(表4)。GO功能注释分为三大类:生物过程(biological process)、细胞组分(cellular component)和分子功能(molecular function)。生物过程中,代谢过程(GO:0008152 metabolic process)、细胞过程(GO:0009987 cellular process)和单一生物过程(GO:0044699 single-organism process)是主要的子类;细胞组分中,细胞(GO:0005623 cell)、细胞部分(GO:0044464 cell part)、细胞器(GO:004322 6 organelle)和膜(GO:0016020 membrane)是主要的子类;分子功能中,催化活性(GO:0003824 catalytic activity)和结合(GO:0005488 binding)是主要的子类(图1)。

图1 GO分类功能注释Fig.1 Functional classification annotation of GO database
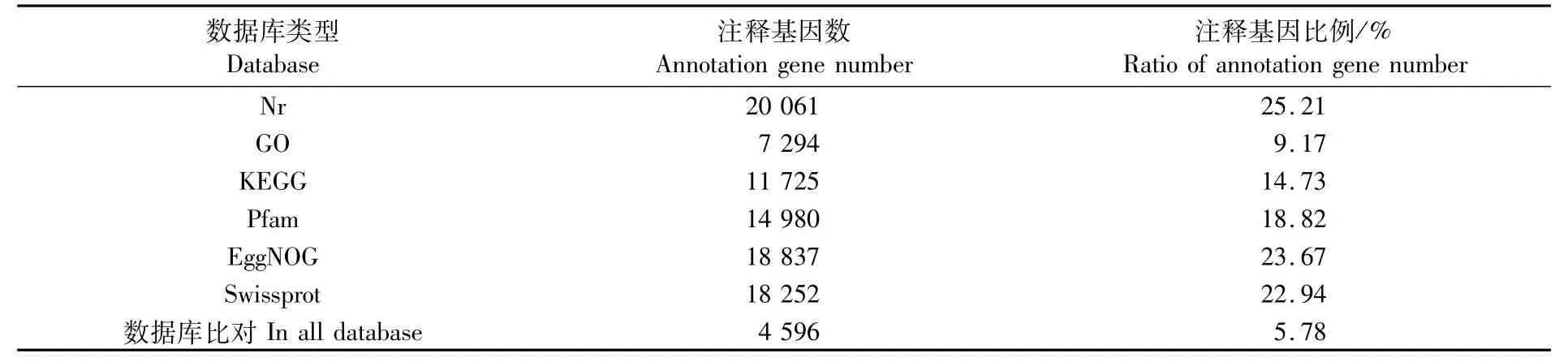
表4 基因注释数量统计Tab.4 Statistic summ ary of annotation gene number
注释到KEGG数据库的unigene共11 725个(表4),level1分为5类,即代谢(metabolism)、遗传信息处理(genetic information processing)、环境信息处理(environmental information processing)、细胞过程(cellular process)和有机系统(organismal system),其注释的unigene数量分别为4 055、2 272、1 823、2 112和3 602个(图2)。

图2 KEGG分类注释Fig.2 Functional classification annotation of KEGG database
2.3 表达差异分析和富集分析
转录组测序共获得5 621个差异表达基因,饥饿组与对照组相比,上调2 641个,下调2 980个。GO富集分析显示,共254条GO Term得到显著性富集(矫正后的P值小于0.05)。本文仅列出富集最显著的前20个GO Term(图3),其中前5条GO Term 分别是催化活性(catalytic activity)、碳水化合物代谢过程(carbohydrate metabolic process)、氧化还原过程(oxidationreduction process)、 氧 化 还 原 酶 活 性(oxidoreductase activity)和DNA复制起始(DNA replication initiation)。
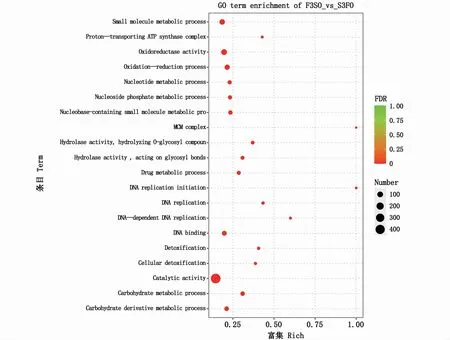
图3 差异表达基因富集最显著的前20条GO TermFig.3 Top 20 significantly enriched GO term s based on differentially expressed genes
KEGG 富集分析显示,共 60 条 KEGG pathway得到显著性富集(矫正后的P值小于0.05)。本文仅列出富集最显著的前20条KEGG pathway(图4),其中前5条KEGG pathway分别是DNA复制(DNA replication)、缬氨酸、亮氨酸和异亮氨酸降解(valine,leucine and isoleucine degradation)、细胞周期(cell cycle)、淀粉和蔗糖代谢(starch and sucrosemetabolism)、碱基切除修复(base excision repair)。值得注意的是,DNA复制通路富集的21个差异表达基因全部下调(图5)。此外,KEGG显著富集的通路中氨基酸代谢、脂质代谢和碳水化合物代谢的占比相对较大(表5)。

图4 差异表达基因富集最显著的前20条KEGG通路Fig.4 Top 20 significantly enriched KEGG pathways based on differentially expressed genes
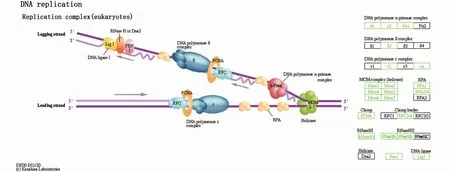
图5 差异表达基因在DNA复制通路中的分布Fig.5 Distribution of differentially expressed genes in DNA replication pathway

表5 与氨基酸代谢、脂质代谢和碳水化合物代谢相关的显著富集的KEGG通路Tab.5 Significantly enriched KEGG pathways related to am ino acid metabolism,lipid metabolism and carbohyd ratemetabolism
2.4 饥饿胁迫下消化系统、嗅觉和昼夜节律相关基因的筛选
基于KEGG通路结合以往的报道,筛选了部分与消化系统、嗅觉和昼夜节律相关的显著差异表达基因(表6)。
2.5 差异表达基因qRT-PCR验证
选取上调基因Met、MIH、E75和下调基因αamylase和Trypsin进行qRT-PCR验证(图6)。结果显示,验证结果与测序结果是一致的,表明转录组的测序结果和生物信息学分析是可靠的。
3 讨论
拟穴青蟹作为一种重要的海水养殖蟹,大眼幼体期的营养状态对其后期生长发育是十分重要的[17]。但目前关于饥饿是如何影响拟穴青蟹生长发育的分子研究未见报道,因而通过转录组测序挖掘相关的代谢通路是十分必要的。本研究测序获得79 581个unigene,平均长度1 065.44 bp,以Nr数据库注释的unigene数最多(20 061个),但也仅占总数的25.21%,可见大部分unigene未得到有效注释。
以往的研究表明,饥饿状态下锯缘青蟹、三疣梭子蟹和远海梭子蟹(Portunuspelagicus)Ⅰ期溞状幼体的生化组成发生了改变,主要体现为蛋白质、脂质的减少[14-16]。本研究中,多数氨基酸代谢和脂质代谢相关通路同样得到显著性富集。不同的是,锯缘青蟹[14]和三疣梭子蟹[15]Ⅰ期溞状幼体在饥饿状态下碳水化合物的含量基本保持不变,而本研究中有8条碳水化合物代谢相关通路得到显著性富集。此外,锯缘青蟹Ⅰ期溞状幼体在饥饿状态下蛋白酶、淀粉酶和酯酶的活性明显下降[18]。本研究中拟穴青蟹大眼幼体的胰蛋白酶、淀粉酶和三酰甘油酯酶基因的表达水平是显著下降的。可见,由于饥饿原因,青蟹大眼幼体下调了自身的消化酶分泌水平。
DNA复制是细胞最基本的生命活动之一,是生物体生存和繁殖的基础。KEGG富集分析显示,DNA复制通路富集是最显著的,且富集的21个差异表达基因全部下调。此外,碱基切除修复、错配修复、非同源末端连接和细胞周期途径同样显著富集。以上这些结果表明,饥饿干扰了拟穴青蟹大眼幼体的DNA复制和修复,进而影响了细胞周期。此外,细胞凋亡代谢通路是显著富集的。以往的研究有过相似的结果,例如饥饿胁迫诱导了中华绒螯蟹肝胰腺的细胞凋亡[3]。由此可知,饥饿一定程度干扰了拟穴青蟹大眼幼体细胞的分裂和凋亡。
睡眠和觅食是高度保守、相互关联的行为,对机体生存至关重要。已有研究发现饥饿抑制了果蝇(Drosophilamelanogaster)的睡眠,而饥饿后再次摄食果蝇的睡眠时间增加[19]。但对于饥饿调控睡眠的机理目前依然知之甚少。过去的研究揭示,果蝇大脑中的丝氨酸代谢调控饥饿诱导的睡眠抑制[20]。而最近的研究显示,果蝇胰岛素样肽Dilp2是饥饿引起果蝇睡眠深度增加的关键神经肽,但其不参与睡眠时间的调控[21]。根据先前的报道,果蝇Dilp或者其受体DInR的突变都将抑制睡眠,而饥饿降低了Dilp2的表达水平[22]。本文测序分析结果显示,饥饿状态下拟穴青蟹昼夜节律基因per和tim表达水平显著变化,这表明饥饿干扰了拟穴青蟹的睡眠。与此同时,本文丝氨酸代谢通路显著富集,部分胰岛素样肽受体相关基因差异表达,与果蝇中的发现类似。但对于饥饿是否直接通过上述途径干扰拟穴青蟹睡眠则需要更多的实验加以验证。此外,以往研究中还发现饥饿调节果蝇的嗅觉行为[23],而本文的转录组测序同样发现部分嗅觉相关基因在饥饿状态下是显著差异表达的,但饥饿与拟穴青蟹嗅觉行为的关系仍需进一步的验证。
qRT-PCR结果表明,测序结果与验证结果是一致的,因而转录组基于测序结果进行的生信分析是可信的。本研究通过转录组分析,获得了拟穴青蟹大眼幼体的转录组序列和基因注释结果,筛选了与饥饿胁迫相关的一系列代谢通路,例如氨基酸代谢、脂质代谢、碳水化合物代谢和细胞生长和死亡等,分析了饥饿状态下部分与消化系统、嗅觉和昼夜节律相关的基因表达水平的差异。研究表明,饥饿胁迫干扰了拟穴青蟹大眼幼体的细胞分裂和凋亡,影响了体内的氨基酸代谢、脂质代谢和碳水化合物代谢。本研究的相关发现有助于推进拟穴青蟹饥饿胁迫的分子机制研究。
