抗菌肽Hepcidin诱导草鱼肾细胞miRNA表达分析
2023-09-09岳小真常娇娇常新月李槿年
岳小真 常娇娇 常新月 李槿年
(安徽农业大学动物科技学院, 安徽省兽医病理生物学与疫病防控重点实验室, 合肥 230036)
Hepcidin(Hepatic bactericidal proteins)又称铁调素, 是一类富含半胱氨酸, 兼有广谱抗菌、免疫调节和铁代谢调节作用的抗菌肽[1]。自Shike等[2]从杂交条纹鲈(Morone chrysops)中分离到Hepcidin以来, 国内外学者对鱼源抗菌肽Hepcidin的抗菌活性进行了广泛研究。研究结果表明该抗菌肽对嗜水气单胞菌(Aeromonas hydrophila)、副溶血弧菌(Vibrio parahaemolyticus)、鲍曼不动杆菌(Acinetobacter baumannii)和海豚链球菌(Streptococcusiniae)等多种水产生物病原菌均具有较强的体外抑菌活性[3,4]。同时发现, 注射或饲投该抗菌肽可通过调节宿主免疫功能而发挥体内抗迟缓爱德华氏菌(Edwardsiella tarda)或柱状黄杆菌(Flavobacterium columnare)作用[5,6]。因此, 鱼源Hepcidin被认为是最有前景的新型抗菌感染药物之一。但是, 目前关于鱼源Hepcidin的抗菌作用机制和免疫调节机制尚不完全清楚, 这限制了其开发应用。
微小核糖核酸(miRNA)是由内源性基因编码的长度在18—25 nt的单链非编码小分子 RNA[7]。在脊椎动物中, miRNA能够通过与靶基因mRNA的3′端非翻译区互补结合, 引起靶标mRNA降解或翻译抑制, 从而在转录后水平调控基因表达, 并参与细胞增殖、分化、凋亡、免疫等诸多生物学过程[8]。已有研究报道, 鱼类miRNA可以通过靶向免疫信号通路中的多个分子调控先天免疫应答, 从而在宿主受病原菌感染及疫苗诱导的免疫应答中发挥着重要作用。如在斜带石斑鱼(Epinephelus coioides)中,miR-146a通过靶向负调控TRAF6表达促进石斑鱼神经坏死病毒(RGNNV)的复制[9]。Huo等[10]研究表明, 鲫(Carassius auratus)鳃中miR-17、miR-26a、miR-144和miR-146a等关键miRNAs在嗜水气单胞菌感染后的局部免疫反应中发挥了关键作用。曹守林[11]研究发现加州鲈(Carassius auratus)免疫接种鰤鱼诺卡氏菌双载体核酸疫苗后, 脾脏中显著下调的ola-miR-139靶向TRAF6激活NF-κB通路, 从而增强疫苗诱导的免疫应答水平。然而, 迄今为止鱼源抗菌肽Hepcidin过表达能否诱导细胞miRNA表达谱变化, 以及细胞中关键miRNA在Hepcidin免疫调节机制中的作用尚未见报道。
我们在前期研究中已证实草鱼Hepcidin(CiHep)具有良好的体外抗拟态弧菌活性[12], 且体内注射的CiHep基因重组真核表达质粒可通过调节机体免疫功能而保护65%草鱼抗拟态弧菌感染(结果未发表),但其免疫调节机制尚不明确。为此, 本研究借助高通量测序和生物信息学软件分析CiHep过表达前后CIK细胞中miRNA差异表达谱, 挖掘富集在免疫信号通路中的关键差异表达miRNA及其靶基因, 以期为从miRNA角度深入研究抗菌肽CiHep的免疫调节机制打下基础。
1 材料与方法
1.1 实验材料
草鱼肾细胞系由本实验室保存; 大肠埃希氏菌DH5α菌株(Escherichia coliDH5α)感受态细胞购于北京擎科新业生物技术有限公司; 真核表达载体pmCherry-N1由上海兽医研究所刘光清研究团队惠赠。
1.2 CiHep基因的重组表达质粒构建
根据草鱼Hepcidin(CiHep)基因成熟肽序列(NO.JQ246442.1)设计1对带有酶切位点的特异性引物(表1), 用于构建重组表达质粒pmCherry-N1-Cihep。以CIK细胞cDNA为模板, PCR扩增CiHep基因。PCR反应体系(50 μL)为: 2×TaqPCR MasterMix 25 μL,上、下游引物各1 μL, DNA模板2 μL, ddH2O 21 μL。PCR反应条件为: 95℃预变性5min; 95℃变性30s,65℃退火30s, 72℃延伸30s, 30个循环; 72℃ 3min。将纯化的PCR产物与空载体pmCherry-N1依次进行XhoI与EcoR I双酶切、酶连及转化E. coliDH5α感受态细胞。取转化液均匀涂布于含100 μg/mL Kana的LB培养基, 37℃培养16h。随机挑取3个菌落进行扩大培养后, 抽提重组质粒进行PCR与双酶切鉴定。
1.3 CiHep基因最佳过表达时间的筛选
为了以转染后CiHep基因相对表达量及其蛋白表达量达到最大值的CIK细胞作为测序样品, 需要筛选CiHep基因的最佳过表达时间点。首先将生长状态良好且密度为2×106个/孔的CIK细胞接种至6孔细胞培养板, 用MEM完全培养液于28℃、5% CO2条件下培养至80%汇合度, 弃去孔内培养液, 更换为不含血清与双抗的细胞培养液, 使用Lipofectamine 3000脂质体转染试剂将重组表达质粒pmCherry-N1-Cihep转染至细胞中, 转染后每一检测时间点(48h、72h和96h)做6个复孔(用于检测基因转录与蛋白表达各3孔)。转染体系为: 2.5 μg质粒、3.75 μL转染试剂和250 μL不含血清与双抗的细胞培养液。
分别于转染后不同时间点(48h、72h和96h)收集细胞样品, 用TRIzol试剂提取细胞总RNA, 按照FastKing RT Kit(With gDNase)反转录试剂盒说明书合成cDNA作为实时荧光定量PCR(Quantitative Real-time PCR, qPCR)检测用模板。根据CiHep基因序列设计用于荧光定量PCR(RT-qPCR)检测的特异性引物, 并选用β-actin作为内参基因(表1)。qPCR反应体系: 2×SuperReal PreMix Plus 10 μL, cDNA模板100 ng, 上下游引物各0.5 μL, RNase free ddH2O补足至20 μL。反应条件: 95℃ 预变性15min、95℃10s, 60℃ 30s, 共计40个循环, 每个样品做3个技术重复。在反应结束后, 使用2-∆∆Ct法[14]计算各检测时间点(转染后48h、72h和96h)目的基因相对转录水平, 并进行单因素方差分析。
进一步, 分别于转染后不同时间点(48h、72h和96h), 弃去细胞孔内培养液, 磷酸盐缓冲溶液(PBS)洗涤3次, 用4%多聚甲醛室温固定细胞15min,PBS洗涤3次, 再用DAPI染核10min, PBS洗涤3次,在荧光倒置显微镜下观察荧光融合蛋白mCherry-CiHep的表达情况。
1.4 测序样品采集与细胞总RNA提取
按1.3方法将重组表达质粒pmCherry-N1-Cihep转染CIK细胞, 共转染3个孔(转染组), 同时做3孔PBS对照(PBS对照组)。转染后72h, 收集各孔细胞,用TRIzol®Reagent提取细胞总RNA。然后, 利用NanoDrop ND-2000 Spectrophotometer (Thermo Scientific)测定总RNA浓度与纯度, 使用Agilent Bioanalyzer2100( Agilent Technologies)评估RNA完整性(RIN)。若A260/A280>1.8且RIN >7, 表明RNA质量合格。
1.5 sRNA文库构建与miRNA测序
将质检合格的二组各3个RNA样品等量混合为2个测序样品(编号TRH与TRQ)送至北京百迈客生物科技有限公司进行miRNA测序。即PAGE电泳切胶得到大小为18—30 nt目的片段, 通过单链DNA接头连接, 加入消化酶去除3′接头, 以带UMI的RT引物进行反转录延伸, 合成cDNA; 高敏聚合酶扩增富集cDNA, 再进行文库定量和pooling环化, 构建sRNA文库后, 利用Illumina HiSeq 2500 测序平台进行高通量测序, 测序读长为测序读长为single-end(SE) 50 nt。
1.6 miRNA的鉴定与预测
测序得到的原始数据为图像文件经过base calling, 得到以fastq格式存储的raw reads。利用FastQC软件[15]去除raw reads中带有接头与低质量序列, 统计序列的总数、种类及长度, 得到纯净序列clean reads用于生物信息学分析。
运用Bowtie软件[16]将长度为18—30 nt的Clean Reads比对至草鱼参考基因组(http://www.ncgr.ac.cn/grasscarp/), 通过与mRNA的外显子和内含子进行比对, 筛选来自mRNA降解片段的sRNA, 并按照已知成熟的miRNA (Known miRNA)>rRNA>tRNA>snRNA>snoRNA>repeat>gene>新miRNA (Novel miRNA)顺序对每个sRNA进行注释, 获得过表达CiHep前后CIK细胞的miRNA表达谱。再将有效reads与miRBase 22.0数据库中miRNA前体与成熟肽序列进行比对, 完全匹配的序列认定为known miRNA。对于未注释的序列, 则依据miRNA前体的发夹结构、二级结构和最低自由能等信息, 运用miRDeep2软件[17]预测novel miRNA。
1.7 差异表达miRNA筛选及其靶基因功能分析
计算测序样品中miRNA的表达量, 使用TPM(每百万次读取的转录本)进行归一化处理。以|log2(Fold change)|≥0.585, 且FDR(错误发现率)≤0.05为筛选标准, 利用edgeR软件[18]筛选抗菌肽过表达前后CIK细胞中显著差异表达的miRNA (DEmiRNA)。
为挖掘miRNA的下游靶基因, 联合利用TargetScan和miRanda软件[19]对DEmiRNA进行靶基因预测, 取两者的交集作为潜在靶基因。进而用基因本体论数据库(Gene Ontology, GO)和京都基因和基因组百科全书数据库(Kyoto Encyclopedia of Genes and Genomes, KEGG)对潜在靶基因进行功能注释和通路富集分析。为了进一步分析DEmiRNA可能在调控网络中发挥的作用, 运用在线分析软件Cytoscape(http://www.cytoscape.org/)[20]绘制与免疫过程相关的miRNA-mRNA-免疫调控网络互作图。
1.8 差异表达miRNA的qPCR验证
为了验证筛选的差异表达miRNA的准确性, 随机取12个已知DEmiRNA(7个下调、5个上调)进行PolyA加尾法qPCR检测。即先委托北京擎科生物科技有限公司设计与合成miRNA上游引物, 下游引物则为miRNA第一链cDNA合成(加尾法)试剂盒中提供的通用引物。进而用该试剂盒将测序生物公司返回的RNA样品反转录为cDNA, 并以此为模板,按照1.3方法中体系与反应条件进行qPCR检测,U6作为内参基因。反应结束后采用2-∆∆Ct法计算miRNA相对表达量。
1.9 数据分析
所有的qPCR检测结果均用均数±标准差(mean±SD)表示。利用SPSS 26.0软件中的单因素方差(One-Way ANOVA)法分析miRNA和mRNA表达量在对照组与过表达组中的差异水平(P<0.05, 差异显著;P<0.01, 差异极显著;P<0.001, 差异极极显著), 并采用Graphpad Prism 8作图。
2 结果
2.1 重组表达质粒的PCR与双酶切鉴定
采用常规酶切酶连法构建重组表达质粒pmCherry-N1-Cihep, 抽提质粒进行PCR和双酶切鉴定。结果如图1所示, 以质粒DNA为模板扩增到目的基因CiHep(图1a泳道, 99 bp); 双酶切反应后均获得两条DNA条带, 其大小分别与空载体和目的基因大小基本一致(图1b泳道3和4), 说明重组表达质粒pmCherry-N1-Cihep构建成功。
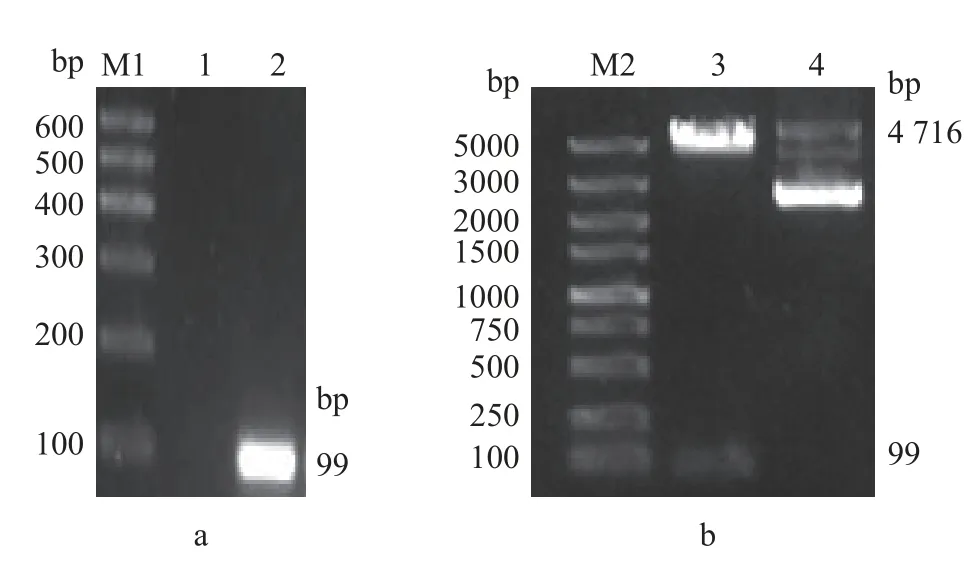
图1 重组表达质粒 pmCherry-N1-Cihep的PCR(a)与双酶切鉴定(b)Fig. 1 PCR and enzyme digestion Identification of recombinant expression plasmid pmCherry-N1-Cihep by PCR (a) and double enzyme digestion (b)
2.2 CiHep基因的最佳过表达时间
通过检测瞬时转染后不同时间点CiHep基因转录情况(mRNA表达水平)及其荧光融合蛋白表达情况, 综合分析其最佳过表达时间。结果如图2所示,与未转染的细胞对照组相比, 转染组CiHep基因mRNA表达水平在各检测时间点均显著上调(P<0.01),且转染后72h的mRNA表达水平极显著高于48h(P<0.01)和极极显著高于96h(P<0.001)。荧光倒置显微镜观察到各检测时间点红色荧光融合蛋白mCherry-CiHep在胞质与胞核中均有表达, 且以转染后72h细胞发出的红色荧光强度最高(图3)。结果说明CiHep基因在CIK细胞中最佳过表达时间为转染后72h, 应选择该时间点收集细胞样品用于miRNA测序。
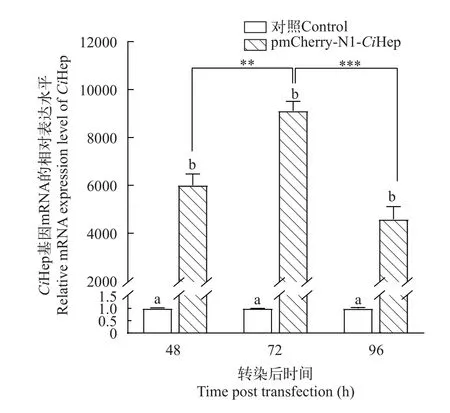
图2 转染后不同时间CiHep基因在CIK细胞中的相对转录水平Fig. 2 Relative transcription levels of CiHep gene in CIK cells at different times after transfection
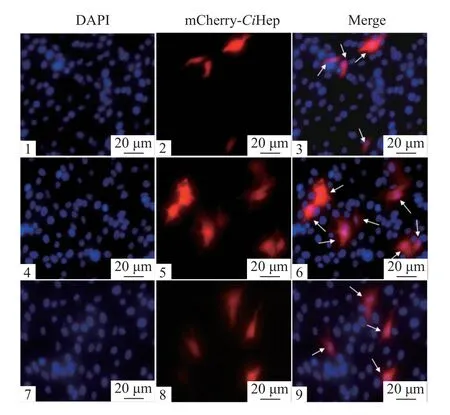
图3 转染后不同时间荧光融合蛋白mCherry-CiHep在CIK细胞中的表达Fig. 3 Expression of fluorescent fusion protein mCherry-CiHep in CIK cells at different times after transfection
2.3 测序数据统计结果
结果如表2所示, 抗菌肽CiHep过表达前后的2个sRNA文库中分别有10414716条和15695371条miRNA原始序列。去除低质量序列、接头序列和短序列后, 分别筛选出10030808和14869867条Clean reads, 分别占总Read的96.31%和94.74%。Q30碱基百分比分别为96.72%和97.09%, 错误率均为0.1%。对照组(TRQ)和过表达组(TRH)能够定位到草鱼基因组上的Unique序列分别有9921271条和14635340条, 匹配率分别为92.83%和91.96%。2个文库中miRNA长度分布在18—24 nt, 其中序列长度为22 nt所占比例最大, 与典型的miRNA长度分布相符(图4)。以上结果表明, 测序数据质量良好, 可用于后续分析。
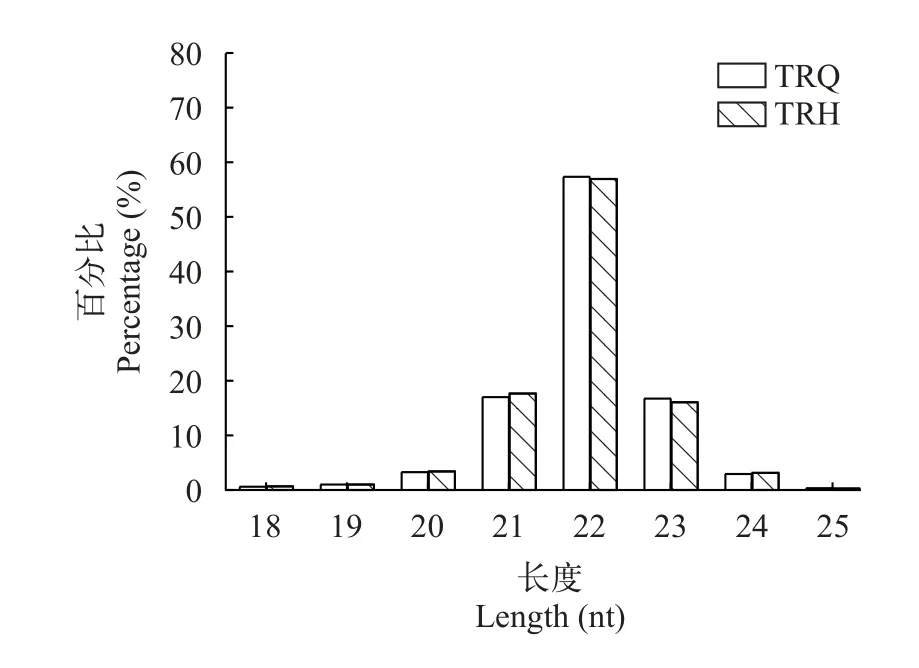
图4 2个文库中miRNA长度分布Fig. 4 Length distribution of miRNA in two libraries
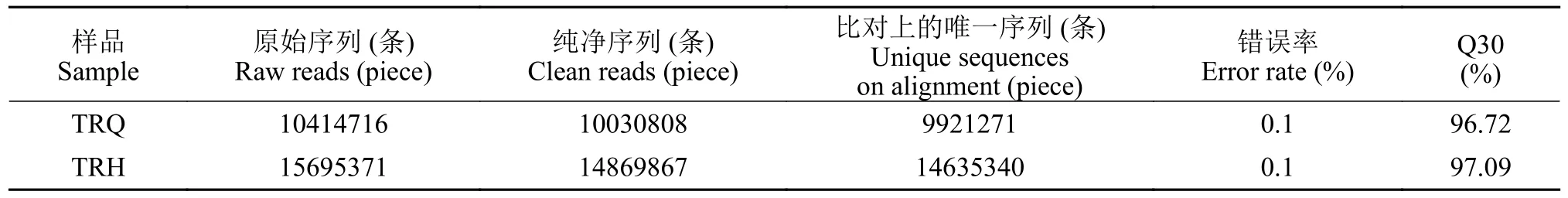
表2 测序数据统计Tab. 2 Sequencing data statistics
2.4 sRNA分类注释及miRNA的鉴定与预测
sRNA分类注释结果显示, 2个文库(TRQ和TRH)中miRNA所占比例最高, 分别为 98.91%和98.41%,其余依次是tRNA (0.49%和0.89%)、rRNA (0.36%和0.51%)和Repeat (0.22%和0.16%; 图5)。将有效reads与miRBase数据库比对, 从对照组(TRQ)与过表达组(TRH)分别鉴定到1850和2013种已知成熟的miRNA, 其中有10个已知miRNAs仅在对照组中出现, 173个已知miRNAs只在过表达组中检测到。对于比对上草鱼基因组但未注释上任何RNA类型的Clean reads则通过miRDeep2软件分析, 在对照组与过表达组分别预测到1266和1347种新miRNA, 其中有1260个新miRNA为二组共有。
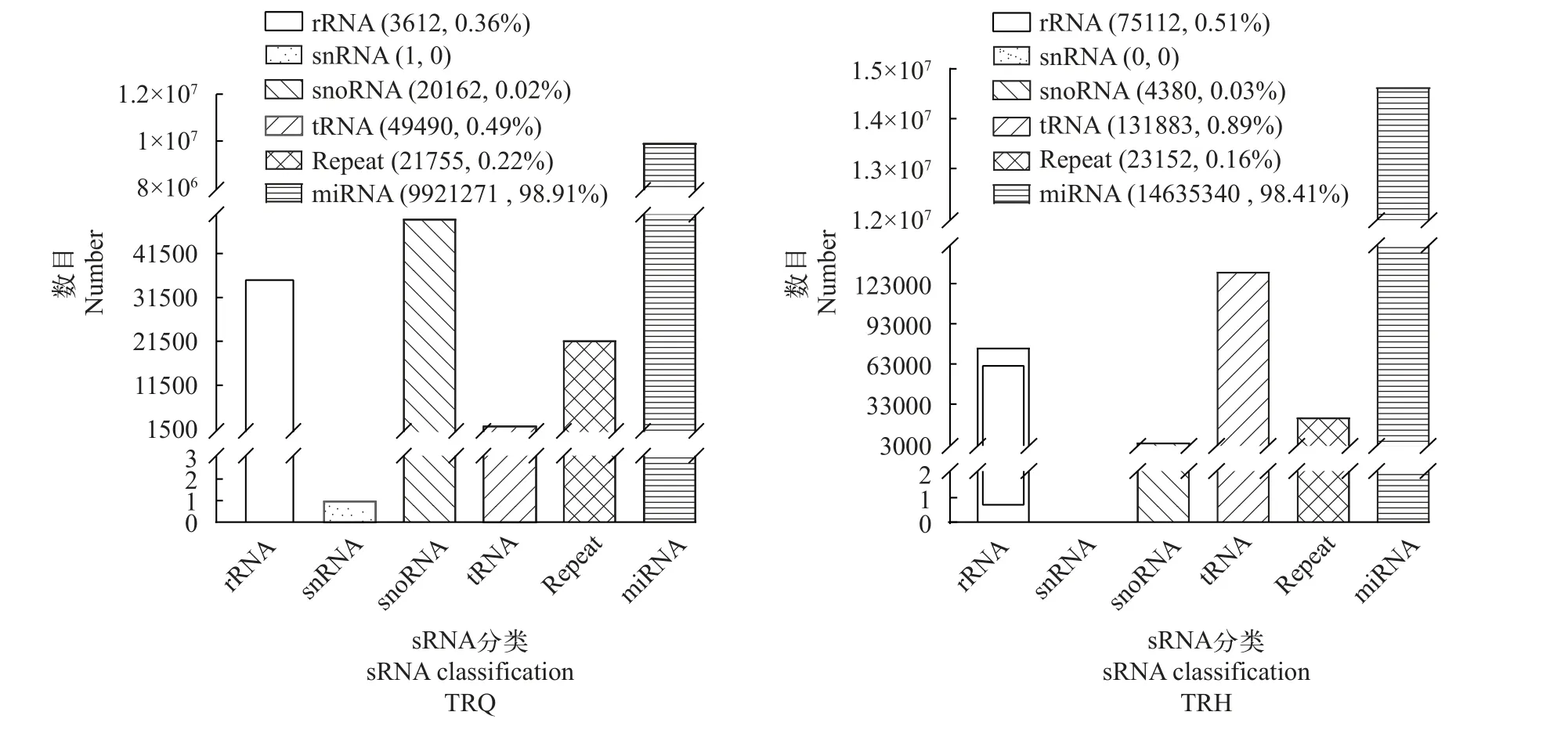
图5 过表达前后2个文库中sRNA的分类注释Fig. 5 Classified annotations of sRNA in the two libraries before and after overexpression
2.5 CiHep过表达后CIK细胞中差异表达的miRNA
以|log2(Fold change)|≥0.585且FDR≤0.05 为筛选标准, 对CiHep过表达前后CIK细胞中差异表达miRNA进行筛选。结果如图6所示, 与对照组相比, 过表达组细胞中共出现460个DEmiRNA, 其中392个显著上调、68个显著下调, 已知DEmiRNA 222个、新预测的DEmiRNA 238个, 在已知miRNA中pma-miR-199b-5p为下调倍数最大(3.76倍)的DEmiRNA。
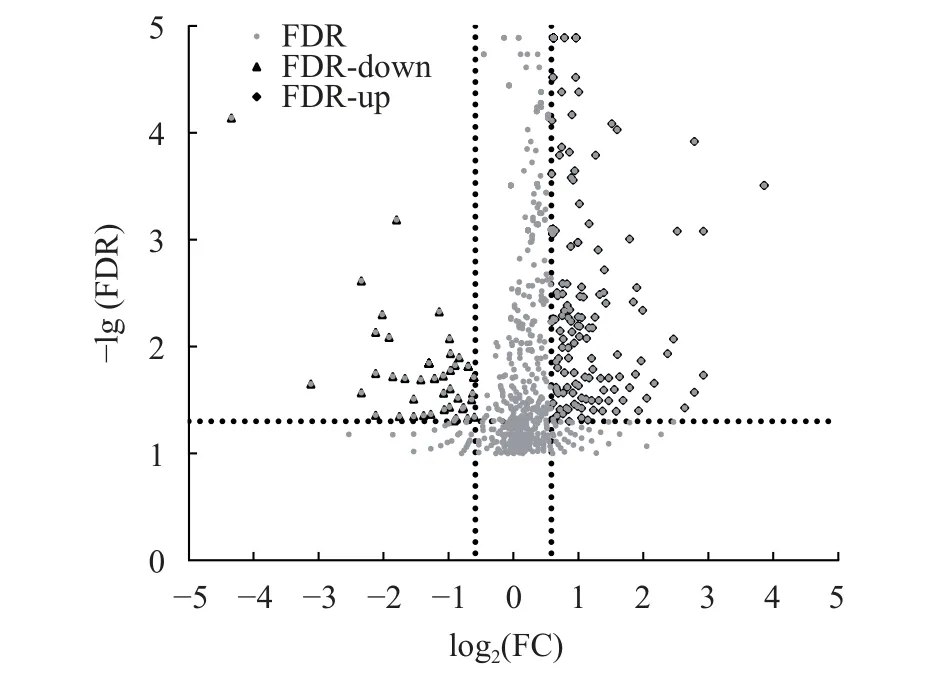
图6 CIK细胞中差异表达miRNA火山图Fig. 6 Volcano plot of differentially expressed miRNA in CIK cells
2.6 差异表达miRNA的靶基因及其功能分析
利用TargetScan和miRanda 两款软件对460个DEmiRNA进行靶基因预测, 共得到5150个潜在靶基因, 其中有436个潜在靶基因得到GO注释, 可富集到细胞、细胞部分和膜等16个细胞组件, 参与细胞进程、生物学调节和单细胞生物进程等22个生物学过程, 发挥结合、催化和信号转导活性等13种分子功能(图7)。KEGG通路富集分析结果显示, 有157个潜在靶基因富集于54条KEGG通路, 其中属于细胞进程、环境信息处理、遗传信息处理、人类疾病、新陈代谢及生物系统6类通路, 主要发现C型凝集素受体(C-type lectinreceptor, CLR)、PI3KAkt通路、NOD样受体(NLRs)、内吞作用(Endocytosis)、细胞骨架调节(Regulation of actin cytoskeleton)、神经活性配体受体相互作用(Neurolactive ligand recepto interaction)、细胞黏附分子(Cell adhesion moleclules)、细胞凋亡(Apoptosis)和丝裂原活化蛋白激酶(Mitogen-activated protein kinase,MAPK)等免疫相关信号通路(图8)。互作分析结果如图9所示, 29条DEmiRNA(2条已知、27条新DEmiRNA)、23个潜在的靶基因以及13个免疫相关信号通路构成了miRNA-mRNA-免疫互作网络。互作网络中1个miRNA可以同时靶向多个宿主细胞基因, 1个宿主细胞基因也能受到多个miRNA的调控,2条已知DEmiRNA富集在PI3K-Akt通路和NOD样受体(NOD-like receptors, NLRs)通路。
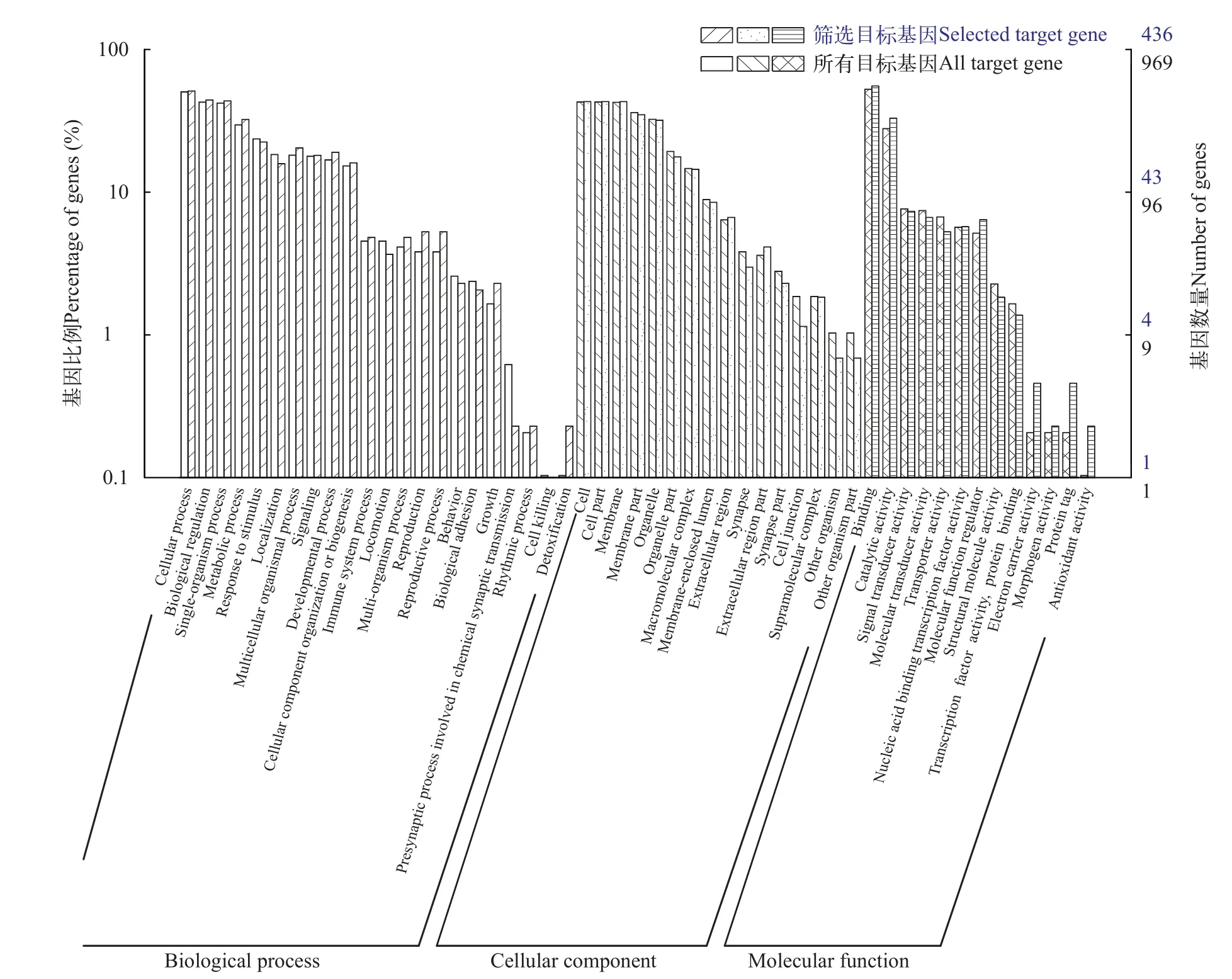
图7 差异表达miRNA靶基因GO分析Fig. 7 GO analysis of target genes corresponding to differentially expressed miRNA
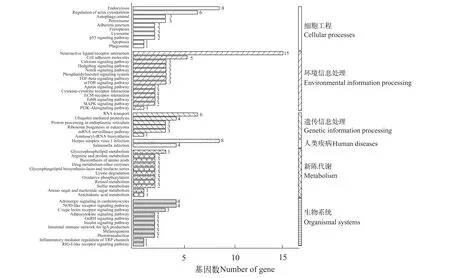
图8 差异表达miRNA靶基因KEGG通路富集分析Fig. 8 Enrichment analysis of KEGG pathway of target genes corresponding to differentially expressed miRNA
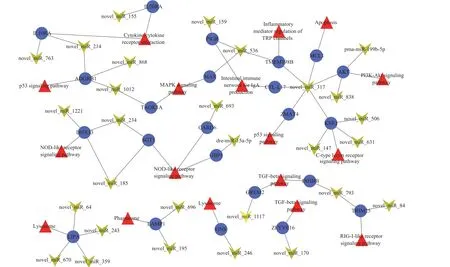
图9 miRNA-mRNA-免疫信号通路互作网络图Fig. 9 miRNA-mRNA- immune signaling pathway interaction network map
2.7 qPCR验证结果
随机选择了12个已知DEmiRNA进行qPCR验证。结果如图10所示, 与对照组相比, 过表达组中cfa-miR-99a显著下调(P<0.05); dre-miR-34c-5p、pma-miR-199b-5p、abu-miR-24a、mze-miR-187、ccr-miR-24和abu-miR-192极显著下调(P<0.01); dremiR-301a与ccr-miR-140-5p显著上调(P<0.05); dremiR-19c-3p、dre-miR-221-5p和ccr-miR-124b极显著上调(P<0.01)。各检测miRNA的表达模式均与高通量测序结果完全一致, 表明本次测序结果有较高的准确性和可信度。
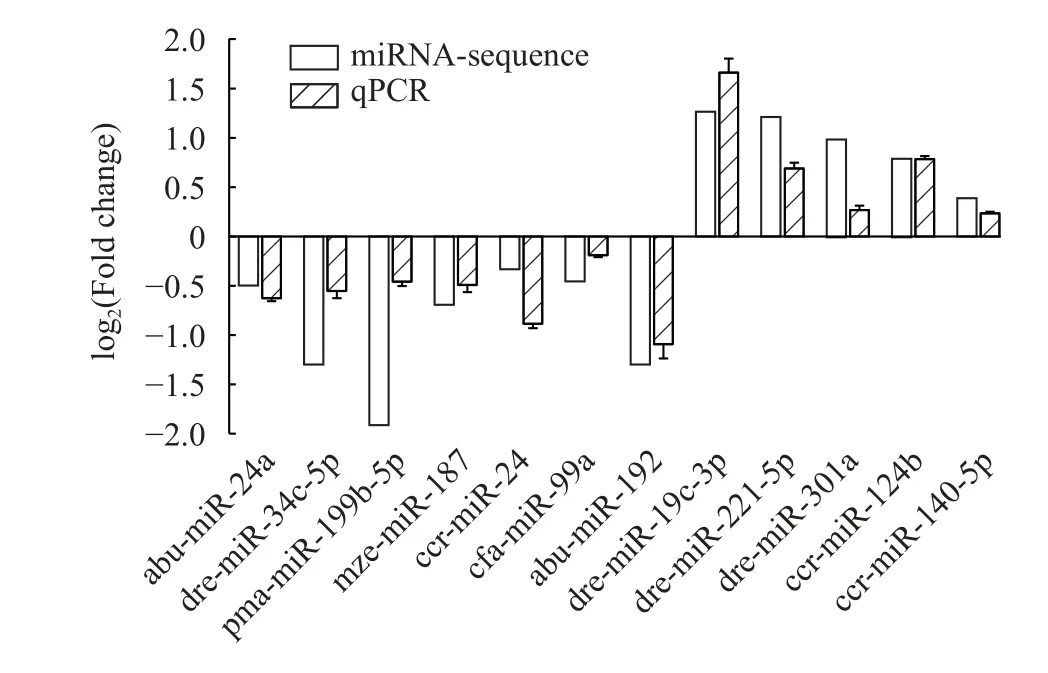
图10 差异表达miRNA的qPCR验证Fig. 10 Verification of differentially expressed miRNA by qPCR
3 讨论
抗菌肽既是免疫效应因子, 也是免疫调节因子。已有研究表明, 体内抗菌肽的浓度通常很低(远小于其最低抑菌浓度), 并不表现出明显的直接抑菌或杀菌作用, 而是通过调节机体的促炎和抗炎反应, 激活吞噬细胞, 增强 T、B 淋巴细胞活性, 诱导趋化因子与细胞因子产生等来发挥间接抗菌作用的[21]。对昆虫抗菌肽免疫调节机制研究发现, 当机体受到外来病原体感染时, 昆虫抗菌肽可以启动天然免疫信号通路机制调节机体免疫功能[22]。miRNA是鱼类复杂免疫应答调控网络中的关键组成部分。近年来, 随着miRNA高通量测序技术和生物信息学分析技术的快速发展, 对参与鱼类免疫应答的miRNA类型和特征也有了一些认识[23]。但是, 鱼源抗菌肽Hepcidin过表达能否诱导细胞miRNA表达谱变化, 以及其中关键miRNA在Hepcidin免疫调节机制中发挥的作用尚不清楚。为此, 本研究首先筛选CiHep最佳过表达时间, 当CiHep基因相对转录水平和蛋白表达量达到最大值时收集细胞样品, 进而借助miRNA测序技术和生物信息学软件首次对CiHep过表达前后的CIK细胞miRNA差异表达谱进行分析。结果显示,CiHep过表达可引起CIK细胞miRNA表达谱发生明显变化, 共获得460个DEmiRNA, 其中392个显著上调、68个显著下调, 已知DEmiRNA 222个、预测的新DEmiRNA 238个以及免疫相关DEmiRNA 48个。进一步随机取12个已知DEmiRNA进行qPCR验证, 发现其表达变化趋势与测序结果一致。这些结果不仅可丰富草鱼miRNA的种类与数量, 而且鉴定获得的免疫相关DEmiRNA可为从miRNA角度深入研究CiHep的免疫调节机制提供素材。
为了深入挖掘CiHep过表达后细胞中潜在的miRNA-mRNA-免疫调控网络, 联合利用TargetScan和miRanda软件预测DEmiRNA对应的靶基因,并对潜在靶基因进行GO功能注释和KEGG通路富集分析。GO功能注释分析结果显示, 潜在靶基因富集在生物过程条目中的生物学调节和分子功能条目中的细胞信号转导活性均与免疫调控过程相关。KEGG通路富集分析结果表明,有157个潜在靶基因富集于6类54条KEGG通路, 其中29条DEmiRNA及其潜在的23个靶基因及13个免疫相关信号通路构成miRNA-mRNA-免疫调控网络。韩朝辉等[24]报道miR-199b-5p是一种肿瘤抑制因子, 可靶向透明质酸蛋白聚糖连结蛋白1增强顺铂对人宫颈癌细胞的杀伤力。曾妮[25]研究发现胞核内miR-199b-5p能够靶向细胞周期依赖性激酶9, 上调心肌纤维化相关基因的表达, 促进心肌纤维化过程。本研究中已知miRNA中下调倍数最大的pma-miR-199b-5p与2个差异表达的新miRNA(novel_miR_317和novel_miR_838)具有共同预测靶基因蛋白激酶B(又称为AKT), 并在miRNA-mRNA-免疫调控网络中富集于PI3K-Akt信号通路。这些研究结果表明相同的miRNA可能通过直接或间接靶向多个靶基因调控多个不同信号通路。在哺乳动物中已证实AKT是一种丝/苏氨酸蛋白激酶, 也是PI3K-Akt通路中关键信号分子, AKT通过磷酸肌醇3-激酶(Phosphoinositide 3-kinase, PI3K)依赖方式活化后,磷酸化下游靶蛋白, 最终激活PI3K-Akt信号通路[26]。PI3K/Akt信号通路作为细胞内重要的信号转导通路, 能够调控细胞代谢、凋亡、存活和增殖等, 尤其是促进B细胞和T细胞的发育、激活和分化[27]。然而, pma-miR-199b-5p是否真正靶向草鱼AKT基因, 草鱼AKT蛋白是否能够激活PI3K-Akt信号通路是后续需要深入研究的问题。
NOD样受体(NOD-like receptors, NLRs)是一类胞质模式识别受体, NLRs能识别进入细胞内的微生物相关分子模式(Microbe-associated molecular patterns, MAMPs)并激活非特异性免疫应答, 在抗微生物感染和控制炎症反应中发挥重要作用[28]。在哺乳动物中鉴定出至少22个NLR家族成员, 并且miR-223通过靶向NLRP3基因抑制炎性小体中IL-1β 的产生[29]。近年来, 参与调控NLRs信号通路的一些鱼类miRNA也陆续被发现, 如miR-144与miR-217可以靶向硬骨鱼NOD1基因, 并在 LPS 刺激下抑制促炎因子表达, 从而避免过度的炎症反应[30]。斑点叉尾鮰(Ietalurus punetaus)的NLRC5是ipu-miR-24b、ipu-miR-101a、ipu-miR-3618、ipu-miR-7547、ipu-miR-7556、ipu-miR-7559、ipu-miR-7564、ipumiR-7567和ipumiR-7574的靶基因[31]。本研究发现过表达CiHep后, 显著上调的dre-miR-15a-5p的潜在靶基因为GBP1(鸟苷酸结合蛋白1)基因、novel_miR_693与novel_miR_1221可能分别靶向CARD6(CARD域蛋白6)和DHX33(ATP依赖的RNA解旋酶)、novel_miR_185与novel_miR_234可能同时靶向DHX33和SGT1(SKP1G2等位基因抑制因子), 并且它们在miRNA-mRNA-免疫调控网络中富集于NLRs信号通路, 提示其参与调控NLRs信号通路。
目前miRBase 22.0数据库中已经收录了271个物种的miRNA前体38589个, 编码48885条成熟miRNA[32]。但是, 许多miRNA的功能仍不清楚, 已知miRNA靶基因的数量也非常有限, 因而进一步发掘新miRNA及其靶基因显得尤为重要。根据本次测序结果, 我们累计预测出过表达抗菌肽后可产生238个差异表达的新miRNA, 其中27个新DEmiRNA富集在免疫相关信号通路, 尤其是novel_miR_317与novel_miR_536可同时富集到TRP通道炎症介质调节通路与肠IgA产生免疫调节通路。这些预测的novel miRNA是否真正存在, 还需要进一步克隆鉴定, 也是进行后续相关功能研究的基础。
综上, 本研究在明确CiHep最佳过表达时间的基础上, 解析了过表达前后CIK细胞miRNA差异表达谱特征, 构建了潜在的miRNA-mRNA-免疫调控网络, 挖掘出pma-miR-199b-5p、dre-miR-15a-5p 、novel_miR_317和novel_miR_536在免疫调控中发挥潜在重要功能。研究结果从miRNA角度为深入探究CiHep的免疫调节机制奠定了良好基础。
