肉牛剩余采食量相关下丘脑差异lncRNA筛选及特征分析
2023-09-02苏总华周小南王晓薇杨朝云丁燕玲张岩峰李承隆明文轩史远刚康晓龙
苏总华,周小南,王晓薇,杨朝云,丁燕玲,张岩峰,李承隆,曾 灵,明文轩,史远刚,康晓龙
(宁夏大学 动物科技学院,宁夏 银川 750021)
在家畜饲养过程中,饲料利用率是评价生产成本最重要的指标之一。在肉牛养殖过程中,饲料成本占总成本支出的70%以上[1]。因此,提高动物饲料利用率能够直接降低饲料成本,减少甲烷排放[2]。使用剩余采食量(Residual feed intake,RFI)不仅能够精准测定家畜的饲料利用效率[3],而且能够排除动物生长性状以及生长速率的影响[4];RFI具有中等遗传力(h2=0.29~0.46)[5-8],并在众多畜禽饲料效率研究中得到开展,通过获得RFI相关的SNP位点[9]、表达调控相关基因[10]、QTL[11]、GWAS[12]等数据,为从遗传角度进行RFI解析提供依据。
长链非编码RNA(Long non-coding RNA)在各种生物过程中发挥着关键调节作用,与其相关染色质修饰、基因组印记、转录调控等众多生物学领域得到广泛关注[13-14]。越来越多的证据表明,lncRNA参与调控牛的多种生产性能,例如奶牛泌乳性能[15]、乳腺炎[16]、骨骼肌发育[17]、热应激[18]、酸中毒[19]和精子发生等[20]。饲料效率属于复杂性状,涉及饲料消化代谢、脂肪代谢、运动等多个层面。下丘脑作为重要的调节中枢,能够整合机体代谢、神经内分泌信号协调动物的摄食行为及能量平衡。在下丘脑相关研究中,已经确定弓状核在调节动物食欲方面具有关键作用,其通过2个神经元群调节采食量[21]。如促食欲神经肽NPY(Neuropeptide Y)和AGRP(Agouti-related peptide),能够促进动物采食[22];相反的,厌食神经肽如α-MSH(α-Melanocyte-stimulating hormone),通过产生饱腹感抑制动物进食[22]。除了神经肽,其他调节因子,如激素、受体、酶类、转录因子等多种分子能将外周组织与中枢神经系统形成相互关联的神经内分泌和代谢途径网络[23],从而对下丘脑参与调控机体食物摄入发挥关键作用。目前,已有研究分别从某一组织器官的分子功能予以揭示lncRNA相关调控分子的功能作用:骨骼肌[24]、肝脏[25-26]、肾上腺[27]、瘤胃[28],但采食中枢相关的lncRNA是否参与RFI表型变化尚不明确。本研究选取不同RFI表型安格斯肉牛,采集下丘脑组织,筛选肉牛下丘脑中与RFI相关差异表达lncRNA,预测其靶基因、相关生物学通路及其在肉牛能量平衡、饲料效率的潜在作用,研究结果将为提高动物饲料利用率及开展肉牛RFI研究提供理论支持。
1 材料和方法
1.1 研究动物与数据收集
本试验个体选自宁夏某养殖场22月龄、初生体质量一致、健康状态良好的安格斯公牛。试验群体(30头)按照NRC(2000)动物营养标准进行饲养,个体限栏饲喂,每天喂食2次,每次采食1.5 h,自由饮水。每周测量收集采食量(Feed intake,FI)数据,测定当天早晚各1次,利用试验期内(81 d)每个个体采食量观测值和预期采食量之差表示RFI。使用FI对中期代谢体质量 (Midpoint metabolic body weight,MMBW0.75)和平均日增质量 (Average daily gain,ADG)进行多元线性回归估算个体RFI,RFI统计分析模型如下[3,29]:
RFI=FI-(b0+b1ADG+b2MMBW0.75)
式中b1和b2分别为MMBW0.75和ADG的偏回归相关系数,b0为表型残差。依据计算结果,RFI大于0的个体定义为高剩余采食量组,小于0的个体定义为低剩余采食量组。
1.2 下丘脑样本采集
根据所收集数据,选取剩余采食量极端差异个体各5头,分为高剩余采食量(H组)和低剩余采食量(L组)2组,依次编号为H1~H5和L1~L5。人道方法屠宰试验牛后立即打开脑腔,采集下丘脑PBS清洗后放入无菌、无酶的冻存管,冻存于液氮中保存。
1.3 RNA提取与检测
根据制造商提供的说明书,使用TRIzol试剂对采集样本提取总RNA。对提取的总RNA分别使用1%琼脂糖凝胶电泳、Nanodrop、Agilent 2100验证RNA降解程度与是否被污染。保证样本浓度≥500 ng/μL,从而满足后续测序试验条件。
1.4 cDNA文库构建和转录组测序
样本提取合格总RNA后,使用去核糖体RNA的方法构建链特异性cDNA文库。按照Illumina TruseqTM样品试剂盒说明书进行文库构建。文库先使用Qubitu 2.0初步定量,稀释至1 ng/μL,之后使用Agilent 2100对文库的插入片段大小进行检测,随后使用Q-PCR对文库的有效浓度进行准确定量(文库有效浓度>2 nmol/L),最后利用Illumina平台测序,产生150 bp的双端原始测序数据(Raw data)。
1.5 质控、比对和转录本组装
为确保比对过程中提供高质量的数据,使用FastQC软件对Raw data进行质量评估,再利用软件Trim Galore并使用默认参数进行质控,去除接头序列(adapter)和低质量片段序列(reads)及Poly-N等[30]。将Clean data通过HISAT2[31]软件比对到牛参考基因组(ARS-UCD1.2),得到sam格式比对文件。利用samtools将sam文件转化为bam格式并排序,之后利用StringTie进行转录本拼接与定量[32],采用FPKM(Fragments Per Kilobase of exon model per Million mapped fragments)表示转录本表达量。
1.6 lncRNA的筛选
在筛选之前,先使用cuffmerge软件,将各样本拼接得到的转录本文件进行合并,去掉链方向不确定的转录本,得到完整的转录样信息。根据lncRNA的结构和编码能力特征,对合并的转录本集合设置严格的5步筛选条件[33]:①转录本exon个数筛选(筛选exon≥2的转录本);②转录本长度筛选(筛选转录本长度≥200 bp的转录本);③通过Cuffcompare软件对转录本已知注释筛选(筛选去除与数据库注释exon区域有重叠的转录本);④通过Cuffquant软件对转录本表达量筛选(筛选FPKM≥0.5的转录本);⑤使用CNCI[34]、CPC2[35]、Pfam-scan[36]3种软件判断转录本是否具有编码潜能,取3种软件的分析结果中没有编码潜能的转录本的交集用于后续分析。
1.7 主成分分析
本研究对测序后转录本使用R包factoextra进行主成分分析(Principal component analysis,PCA),用方差来衡量数据的差异性和相似性。根据PCA结果,剔除掉异常样本,平衡在试验和测序过程产生的误差,同时保证在转录组测序中至少3个生物学重复。
1.8 差异lncRNA筛选及潜在靶基因的识别
使用R包edgeR,在lncRNA转录本表达矩阵中筛选H组和L组差异表达lncRNA,设置参数为|log2(Fold Change)|>1和padj<0.05,使用层次聚类的方法对差异lncRNA表达值进行聚类。接着对筛选测得的差异lncRNA潜在靶基因进行预测,设置co-location阈值为lncRNA上下游100 kb筛选顺式靶基因。再根据样本间lncRNA与mRNA表达量相关性(pearson,r= 0.95)筛选反式靶基因。
1.9 功能富集分析及蛋白互作网络分析
使用R包clusterProfiler和enrichplot对筛选得到的顺式靶基因和反式靶基因进行GO(Gene Ontology)和KEGG(Kyoto Encyclopedia of Genes and Genomes)富集分析[37],设置筛选参数P<0.05。GO富集可以进一步明确lncRNA靶基因的主要生物学功能。KEGG通路富集可用于了解差异lncRNA通过靶基因参与调节的信号通路。根据GO和KEGG结果使用string(https://cn.string-db.org/)数据库将相关基因进行蛋白互作分析,分析靶基因蛋白在生物系统中的相互作用关系。接着将分析结果导入Cytoscape软件,使用插件MCODE设置参数(Degree Cutoff=2,Node Score Cutoff=0.2,K-core=2,max depth=100)筛选得到核心基因簇。
1.10 差异表达lncRNA的qRT-PCR验证
为了验证测序数据的准确性及可靠性,随机选取差异表达上调lncRNA:LNC_001283、LNC_014918、LNC_007183、LNC_002434;差异表达下调lncRNA:LNC_015216、LNC_015680、LNC_017103、LNC_001739进行qRT-PCR验证,选择GAPDH作为内参基因,引物信息见表1。反应体系:TB Green Taq Ⅱ 10 μL,Rox Dye Ⅱ 0.4 μL,200 ng/μL cDNA 2 μL,10 nmol/L上下游引物各0.8 μL,加ddH2O至总体积20 μL。qRT-PCR反应条件:95 ℃ 30 s;95 ℃ 10 s,55 ℃ 30 s,循环39次;65~95 ℃ 5 s,每个基因3个重复。
1.11 统计分析
相关数据使用非参数检验或t检验分析处理组与对照组间的统计学显著性,所有分析均使用R软件进行。
2 结果与分析
2.1 安格斯牛剩余采食量表型值
在饲养的30头公牛中,去除一头公牛意外死亡,剩余29头公牛测量数据如表2所示,利用采食量构建多元线性回归计算模型,计算公式如下:
RFI=FI-(6.343 5+0.070 7×MMBW0.75+0.745 4×ADG)
式中RFI(剩余采食量)、FI(采食量)、MMBW0.75(中期代谢体质量)、ADG(平均日增质量)。
根据上述公式,测序样本剩余采食量表型值如表3。
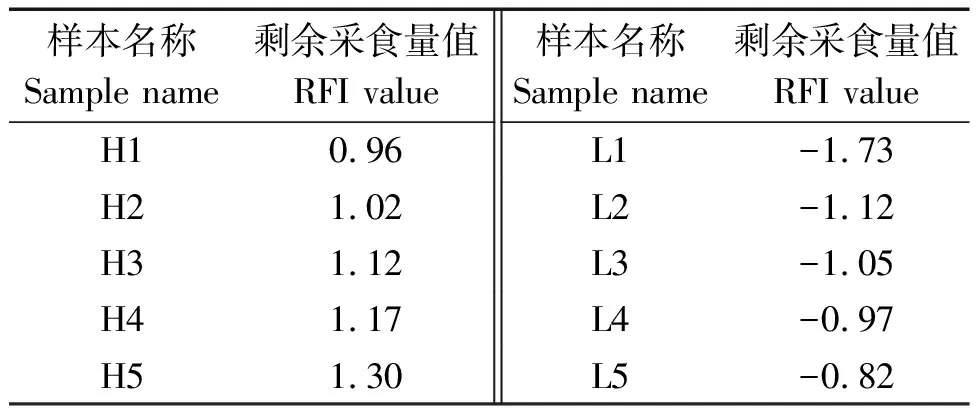
表3 测序样本RFI值
2.2 lncRNA测序数据质量及比对情况
原始数据下机后经数据质控得到Clean reads(表4)。Clean data中Q20≥95.00%,Q30≥89.00%,GC含量占比均衡、稳定,说明测序数据质量较好。之后将Clean data比对到牛参考基因组后,其中能比对到参考基因组的reads占比在93.00%以上,有唯一比对位置的reads占比在79.00%以上,且在H组和L组中比对率平均值较为接近,比对结果良好(表5)。
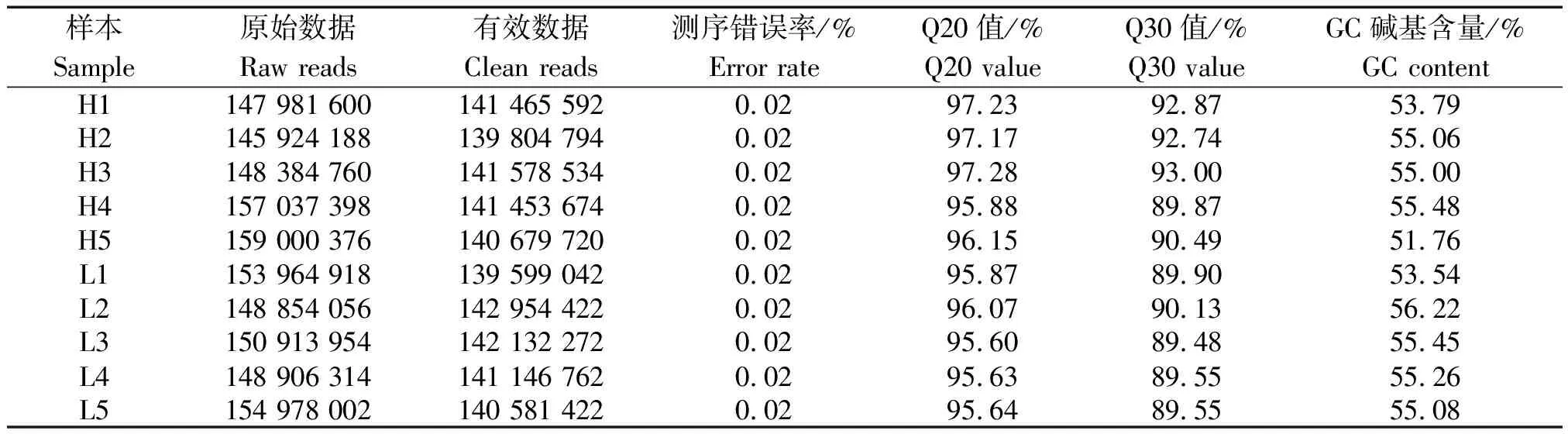
表4 样本测序数据质量控制

表5 测序reads基因组比对统计
2.3 lncRNA筛选
基于转录组拼接结果,对lncRNA分步筛选(图1-A),并将各软件识别出的非编码转录本进行统计(图1-B),最终得到20 996个lncRNA转录本集进行后续分析。
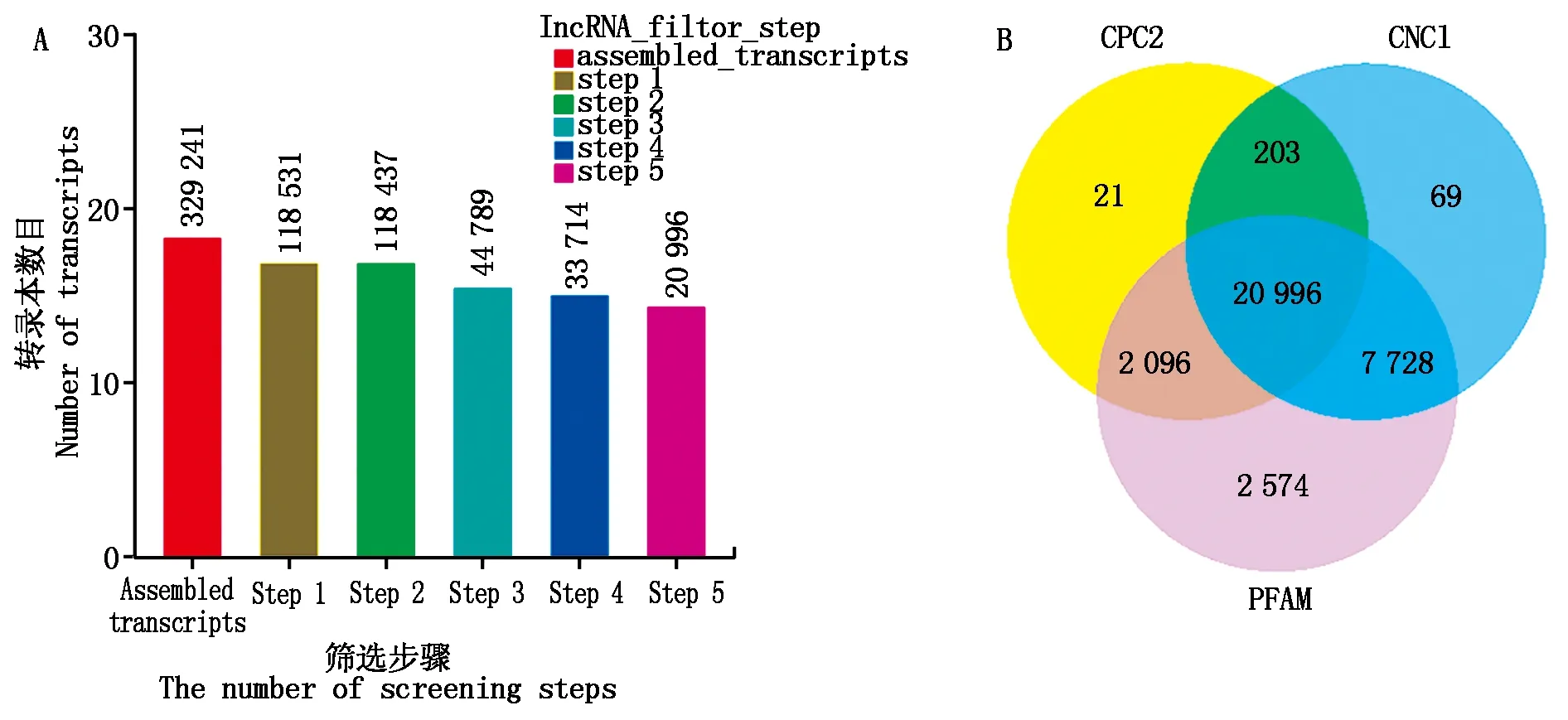
A.lncRNA筛选步骤统计图;B.lncRNA筛选结果韦恩图,表示3种软件分析到的lncRNA数目。
2.4 牛RFI相关的下丘脑lncRNA表达分布及与mRNA结构特征分析
对lncRNA进行定量分析后,共得到20 996个lncRNA,后续分析lncRNA均基于此数据集,其中antisense_lncRNA(反义lncRNA)占比9.5%,主要产生于编码链的反义链;intronic_lncRNA(内含子lncRNA)占比47.5%,主要产生于编码基因的内含子区域;lincRNA(基因间lncRNA)占比43.0%,主要产生于2个编码基因的中间区域(图2-A)。

A.lncRNA类型分布;B.lncRNA与mRNA结构特征分析(lncRNA与mRNA的转录本长度、lncRNA与mRNA外显子数目、lncRNA与mRNA ORF长度)。
将筛选得到的lncRNA及比对组装的mRNA对转录本长度、外显子个数、开放阅读框(Open reading frames,ORF)长度进行比较分析,具体分析情况以核密度图展示(图2-B)。统计分析显示,lncRNA转录本的平均长度为2 423 bp,比mRNA转录本的平均长度2 040 bp长383 bp,但根据图2-B发现lncRNA峰值转录本长度小于mRNA。lncRNA平均外显子数目为5.5个,mRNA平均外显子为10.0个,但lncRNA与mRNA外显子数目均集中于2~5个。统计分析ORF发现lncRNA平均ORF长度为255 bp,mRNA平均ORF长度为534 bp。
2.5 样本相关性和差异性
使用PCA衡量样本总体的相关性及差异性,剔除掉异常样本。结果发现,在HRFI组中H1和H5明显偏离总体水平,在LRFI组中L5偏离总体水平(图3)。为消除组内误差和增强结果可靠性,从HRFI中剔除掉H1和H5 2个样本,从LRFI组中剔除掉L3和L5 2个样本,剩余样本进行后续分析。最后对剩余表型H2、H3、H4、L1、L2、L4对应重新分组命名为HRFI组:HRFI1、HRFI2、HRFI3;LRFI组:LRFI1、LRFI2、LRFI3。

图3 测序样本PCA分析
2.6 差异lncRNA筛选及聚类分析
根据转录本表达量筛选RFI肉牛下丘脑差异lncRNA,在HRFI和LRFI组中共有105个显著差异的lncRNA转录本,其中46个lncRNA在LRFI组下调,59个lncRNA上调(图4-A),上下调中高表达lncRNA包括LNC_001285、LNC_007644、LNC_020193、LNC_013521等(表6)。

A.差异表达lncRNA火山图;B.差异表达lncRNA聚类分析,每一列代表一个样本,每一行代表一个差异lncRNA。

表6 差异上调或下调的lncRNA(Top5)
聚类分析用于判断HRFI和LRFI组中lncRNA的表达模式,根据差异lncRNA的FPKM值为表达水平做层次聚类分析,将表达模式相同或者相近的lncRNA聚集成类(图4-B)。聚类分析越接近的lncRNA,其表达规律越接近,表达模式相近的lncRNA可能有相同或者相关的功能。对差异lncRNA进行聚类分析表明,同一组样本之间的差异很小,同一基因的表达水平在相同组内具有类似表达模式。
2.7 差异lncRNA与mRNA靶向关系
根据lncRNA与mRNA的位置关系及表达量预测差异lncRNA靶向关系。结果显示,差异表达lncRNA共预测得到2 640个靶向mRNA,其中共表达预测到2 163个靶向mRNA,共定位预测到477个靶向mRNA。分析发现:显著差异的lncRNA中LNC_007569及LNC_004079可能调节促肾上腺皮质激素释放激素结合蛋白(Corticotropin releasing hormone-binding protein,CRH-BP),该蛋白是促肾上腺皮质激素释放激素(Corticotropin releasing hormone,CRH)的重要调节剂,能够抑制垂体分泌促肾上腺皮质激素(Adrenocorticotropic hormone,ACTH)[38]。同时表达变化最明显的上调和下调的前5个lncRNA中,其中共有7个lncRNA显著富集到靶向mRNA(图5)。这些潜在调控关系为研究lncRNA调控基因表达进而影响RFI提供新的素材。
2.8 差异表达lncRNA靶基因富集分析及蛋白互作分析
本研究选取差异lncRNA预测共表达及共定位靶基因进行GO富集分析,将所有基因进行富集分析,共挑选显著富集的前5条GO富集条目并展示相关靶基因(图6-A),结果发现前5条GO条目中,有4条GO条目为细胞组分(CC):线粒体被膜(Mitochondrial envelope)、线粒体内膜(Mitochondrial inner membrane)、线粒体膜(Mitochondrial membrane)、含蛋白线粒体复合物(Mitochondrial protein-containing complex),一条生物过程(BP)的GO条目:TOR信号的正向调节(Positive regulation of TOR signaling)。这些GO条目表明,差异表达lncRNA靶基因mRNA主要与线粒体功能及TOR信号通路有关。
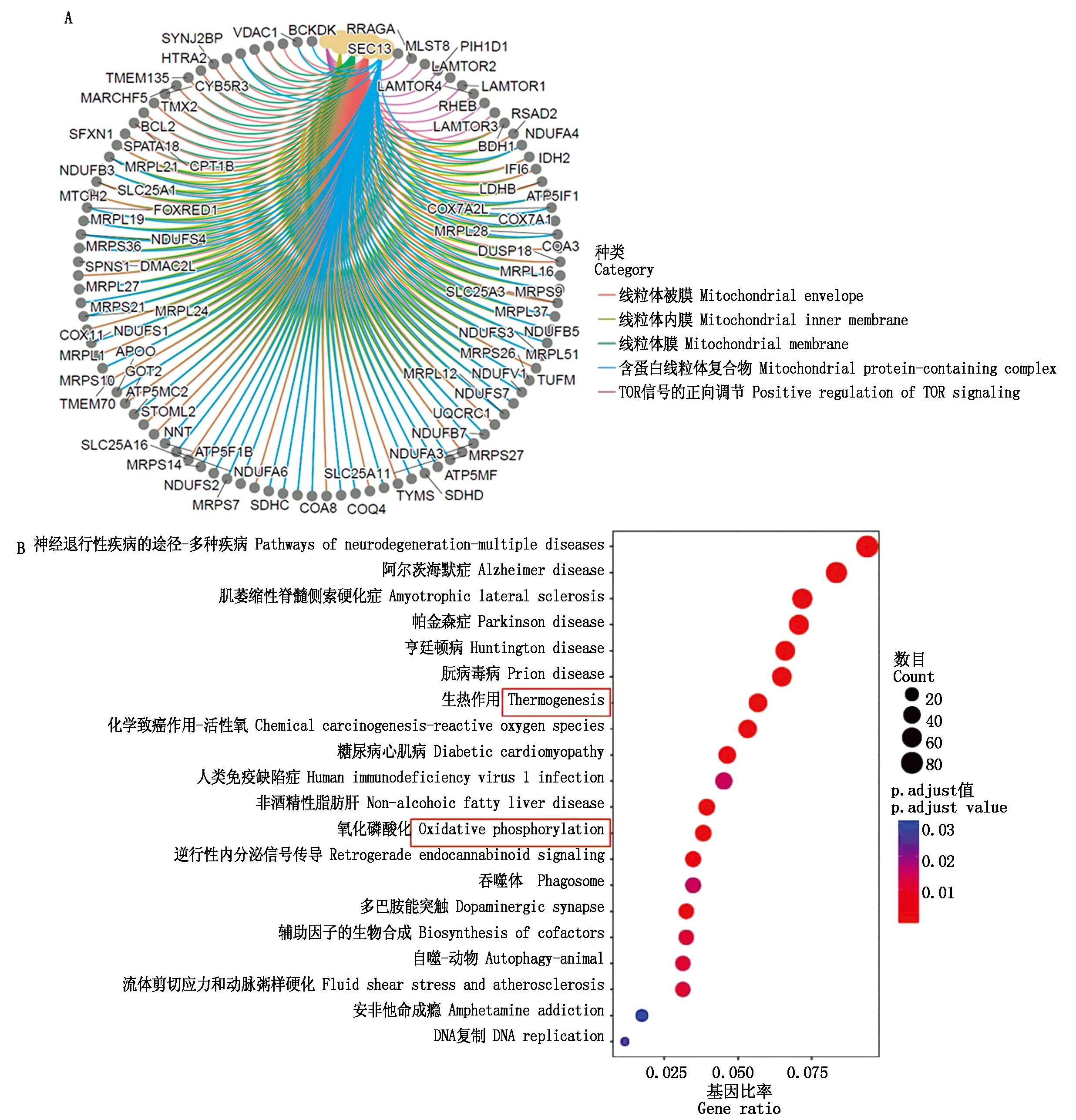
A.靶向mRNA的GO富集分析;B.靶向mRNA的KEGG富集分析。
将差异表达lncRNA靶基因进行KEGG通路富集分析,挑选富集最显著的20个通路进行分析(图6-B)。图中横坐标表示基因比率,纵坐标表示富集到的信号通路,气泡大小表示富集到此信号通路的差异lncRNA靶基因的数量。在这些信号通路中发现生热作用(Thermogenesis),氧化磷酸化(Oxidative phosphorylation)与动物的能量利用联系密切,且分析发现,上述通路中相关基因(ATP酶、呼吸链复合物)主要与差异表达lncRNA(LNC_006637、LNC_007569、LNC_012079、LNC_005449)相关。蛋白互作分析共得到46个核心基因(Hub gene),有595条相互作用关系(图7),主要为调控线粒体活性及电子呼吸链功能的线粒体核糖蛋白及泛醌氧化还原酶亚基等。例如MRPL12基因,该基因不仅是线粒体核糖体的关键成分,而且能够增强线粒体DNA编码RNA的能力[39]。NDUFA4、SDHD、UQCRH被提出在LRFI牛中表达量更高[40]。
2.9 差异表达lncRNA的qRT-PCR验证
随机选择的8个差异表达的lncRNA进行qRT-PCR验证(图8)。结果表明,这些lncRNA验证结果与测序结果表达趋势一致,说明测序数据较为可靠,为差异lncRNA的后续功能验证奠定基础。
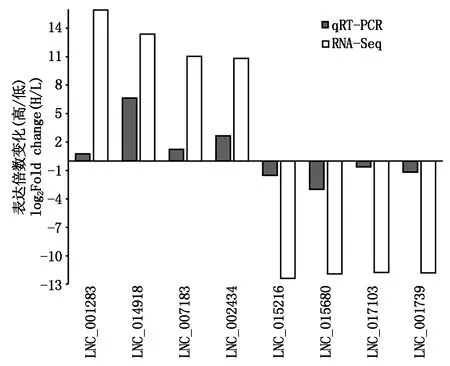
图8 差异表达lncRNA的qRT-PCR验证
3 结论与讨论
肉牛生产成本一直是养殖业最关注的问题,而动物饲料效率是影响动物生长发育速率的关键因素,提高饲料效率是集约化肉牛生产最有效的方法之一。除了研发优质饲料,深入挖掘影响肉牛饲料效率的遗传因素仍是育种工作者重要的科研方向之一。长链非编码RNA通常缺乏蛋白编码能力,通过转录和转录后调节蛋白质编码基因的表达来发挥生物学调控功能[41],目前,lncRNA如何影响靶基因转录与翻译水平,并影响肉牛剩余采食量的分子机制相对缺乏。因此,对其功能的研究仍然需要结合其他表达信息才能解释。迄今为止,许多研究表明,非编码RNA在下丘脑-垂体-肾上腺素轴[27]、脂质代谢[26]、神经元信号转导[42]中起着重要的调节作用。如在猪脂肪组织中进行转录组分析确定了在高饲料效率和低饲料效率中17个差异表达的基因间长链非编码RNA与RFI相关[43];在鸡的肝组织中,应用RNA-Seq技术也发现了影响饲料效率的差异lncRNA[26]。这些研究表明,目前主要采用测序方法对不同组织差异lncRNA进行表征,从而为解释家畜采食量差异的遗传因素提供依据,但lncRNA的调节机制复杂,对其功能的探究需要结合其他调控分子才能阐明其调控作用原理。
在本研究中,在高、低RFI肉牛中发现了20 996个lncRNA,将得到的lncRNA与编码基因比较发现,lncRNA具有更短的转录本、更少的外显子数目、更短的ORF长度,这说明lncRNA编码蛋白能力弱,符合lncRNA的表达特征[44]。在lncRNA差异表达分析中,共鉴定出差异表达lncRNA 105个,其中46个lncRNA在LRFI组下调,59个lncRNA上调。多数lncRNA不具备蛋白编码能力,主要通过结合一种或多种蛋白来实现相应的调控机能[45]。lncRNA能够与顺式或反式靶基因互作调节生物表型变异,为了明确RFI和差异表达lncRNA之间的关系,本研究选择差异表达lncRNA,预测其靶基因,其中预测到共表达靶向靶基因2 163个,共定位靶基因477个。对靶基因进行GO功能注释和KEGG通路富集分析发现,GO条目主要富集到线粒体相关细胞组分,在线粒体被膜、线粒体内膜、线粒体膜等被显著富集。在KEGG富集信号通路中,相关通路主要富集于与能量代谢有关,包括生热作用及氧化磷酸化信号通路,这与GO条目主要富集在线粒体功能上相对应。线粒体功能及产热效率方面的研究已经被证明其与家畜的生产性能及饲料效率表型差异有关[46]。富集在这些通路上的靶基因主要是ATP酶基因(ATP5F1B、ATP5IF1、ATP5MC2、ATP5MF、ATP5PO等)和呼吸链复合物(COXA3、COX7A1、NDUFB7、NDUFS3等),而这些靶基因主要由差异表达lncRNALNC_006637、LNC_007569、LNC_012079、LNC_005449调节。下丘脑是协调信号传输的重要中枢系统,能够调节机体能量稳态及形成厌食和饱食信号调控采食量。根据前人关于RFI研究,本试验进一步在下丘脑差异lncRNA靶向关系分析发现显著差异的lncRNA中LNC_007569及LNC_004079可能调节促肾上腺皮质激素释放激素结合蛋白,该蛋白是促肾上腺皮质激素释放激素的重要调节剂,能够抑制垂体分泌促肾上腺皮质激素[38]。研究表明,下丘脑-垂体-肾上腺素(Hypothalamic-pituitary-adrenal,HPA)轴是影响RFI变异的重要因素之一[47],其通过调控动物的应激反应对动物的采食量及热量消耗产生影响;HPA轴激活会导致肾上腺皮质分泌皮质醇,参与分解代谢,产生葡萄糖,激活免疫系统并降低食欲[48]。线粒体产生大约90%的细胞能量,大量存在于代谢活跃的细胞,如肝、肾、肌肉和脑细胞。线粒体功能及产热效率方面的变化已经被证明与许多畜牧业动物的生产速率和饲料效率的表型差异有关[46,49],其主要通过氧化磷酸化过程产生能量。氧化磷酸化在新陈代谢中起着核心作用,它将呼吸作用与ATP的产生联系起来,但是这种偶联不是完全紧密的,偶尔质子可以回到线粒体基质中产生热量而不是ATP,这将降低氧化磷酸化的效率[50]。先前研究表明,LRFI动物的线粒体呼吸的速率增加[51],电子传递链的偶联得到提升[47],呼吸链复合物(Ⅰ~Ⅴ)的活性更高[52],每千克代谢体质量 (BMW,Metabolic body weight)的产热量更低[2]。另外,电子传递链也被公认为ROS(Reactive oxygen species)的产生部位,升高的ROS通过增加各种细胞成分氧化损伤的敏感性,对细胞抗氧化防御系统构成严重威胁。在家禽的其他研究中发现高饲料效率的动物中,其氧化应激现象也相应较低[46,52-53]。在富集到的靶基因中,细胞色素c氧化酶(COX Ⅱ;复合物Ⅳ)在调节细胞能量产生过程中起着关键作用,催化电子从还原的细胞色素c转移到分子氧中,并参与质子的跨膜转运[54]。Kelly等[10]提出在LRFI动物中COX Ⅱ的转录丰度更高;Kong等[55]发现在高饲料效率肉牛中线粒体基因表达量更高及线粒体基因拷贝数更低,这也说明了LRFI肉牛线粒体活性可能更强、转录率更高。此外,本研究GO富集到TOR信号的正向调节(Positive regulation of TOR signaling),Cota等[56]提出mTOR(Mammalian TOR)蛋白能够通过感知能量状态的变化来调节细胞的生长周期,也证明了mTOR信号在响应营养供应、调节能量平衡的大脑机制中发挥重要作用,这也可以作为后续研究依据进行分析。
挖掘影响动物采食效率的生物机制对集约化畜牧业发展意义重大,饲料效率的提高不仅可以提高生产效益,还可以减少肉牛生产过程中产生的环境污染。RFI作为饲料效率研究的主要经济性状,对家畜RFI的遗传调控研究将为深入解析家畜RFI变异提供坚实基础,也为节粮型家畜的培育提供依据。
本研究表征了不同RFI肉牛下丘脑组织中lncRNA转录组表达谱,差异lncRNA及其主要参与生热作用和氧化磷酸化信号通路对线粒体功能调节产生重要作用,这对于理解下丘脑神经中枢对家畜采食行为的调控提供了参考依据,而这一调节过程是否通过HPA轴发挥作用,仍然需要细胞及分子试验才能明确。
