基于泛癌数据的公共驱动通路识别算法
2023-08-26潘可覃亚伦
潘可 覃亚伦

关键词:驱动通路;泛癌;基因集;调和平均数;粒子群优化;元启发式
中图分类号:TP301 文献标识码:A
文章编号:1009-3044(2023)20-0023-05
0引言
癌症是一种复杂的疾病,其发病机制涉及分子水平上的多种因素。因此,从分子层面深入认识癌症的发生发展过程,对于提高癌症的诊断、治疗和药物设计等方面的水平具有重要的意义。随着深度测序技术的飞速发展,科研人员开展了癌症基因组图谱计划[1]、国际肿瘤基因组协作组[2]等大型癌症基因组计划,获得了海量的癌症数据。如何利用这些数据来有效地识别出促进癌症发展的驱动基因,已成为生物信息学中至关重要的挑战之一。早期的研究主要致力于识别单个驱动基因[3-4],然而由于癌症基因组具有突变异质性[3-5],这类方法的识别效果并不理想。进一步的研究发现,癌症发生的标靶信号与信号通路的调控是由多个驱动基因共同作用的结果,驱动通路中任何一个驱动基因发生突变均会导致通路失调,从而引发癌症[6]。因此,在通路水平上分析突变,识别靶向同一通路的基因集合,对于揭示异质性模式和理解癌症形成更具有生物学意义[7]。驱动通路识别问题可从单癌和泛癌两个层面来研究[8-11],论文主要关注泛癌驱动通路识别问题。
识别不同癌症之间的公共驱动通路是揭开癌症奥秘的重要问题之一,也是研制靶向多种癌症的药物的关键所在。近年来,这一问题已取得一定的研究进展。2017年,Zhang等人[11]提出了ComMDP和SpeMDP两种数学规划模型,分别用于从头识别不同癌症的共同及特有驱动基因集。2021年,Wu[12]等人基于人类蛋白质相互作用网络和泛癌体细胞突变数据,提出了一种新的驱动模块识别方法ECSWalk,以解决突变基因异质性问题,提高驱动模块检测的准确性。同年,Wu[13]等人对R种癌症的公共驱动模块识别问题进行研究,提出了一种癌症公共驱动模块识别模型,并设计求解该模型的识别方法IDM-SPS。上述方法中,大多数使用了基因相互作用网络、蛋白质相互作用网络或已知通路信息等先验知识[12-13],由于目前先验知识仍然很不完整[8],使用先验知识来识别可能会阻碍发现新的突变基因组合。ComMDP算法利用驱动通路的高覆盖和高互斥特性,首先尝试从头识别方法来探测多种癌症间的公共驱动通路,获得了若干具有生物学意义的驱动通路[11]。它将用于单一癌症的最大权重子矩阵问题[9]扩展到多种癌症问题求解,即试图找到一个使每种癌症对应的权重和最大化的基因集。然而,由于不同癌症的样本数量差异很大,绝对权重值可能反映了完全相反的突变状况,同时权重累加和最大不能确保各权重都最大。针对这个问题,论文提出了一种基于相对权重值和调和平均数的新模型CDP(common drive path?way),并设计求解该模型的粒子群优化算法P-CDP。实验结果表明,识别准确度主要受到模型的影响,基于模型CDP的识别方法能够获得较基于ComMDP方法更高的识别准确度;提出的粒子群优化算法在求解大规模问题时仍具有较好的性能;与ComMDP方法相比,论文提出的识别方法能够识别出被ComMDP方法遗漏且具有生物学意义的驱动通路。
1 符号定义及问题模型
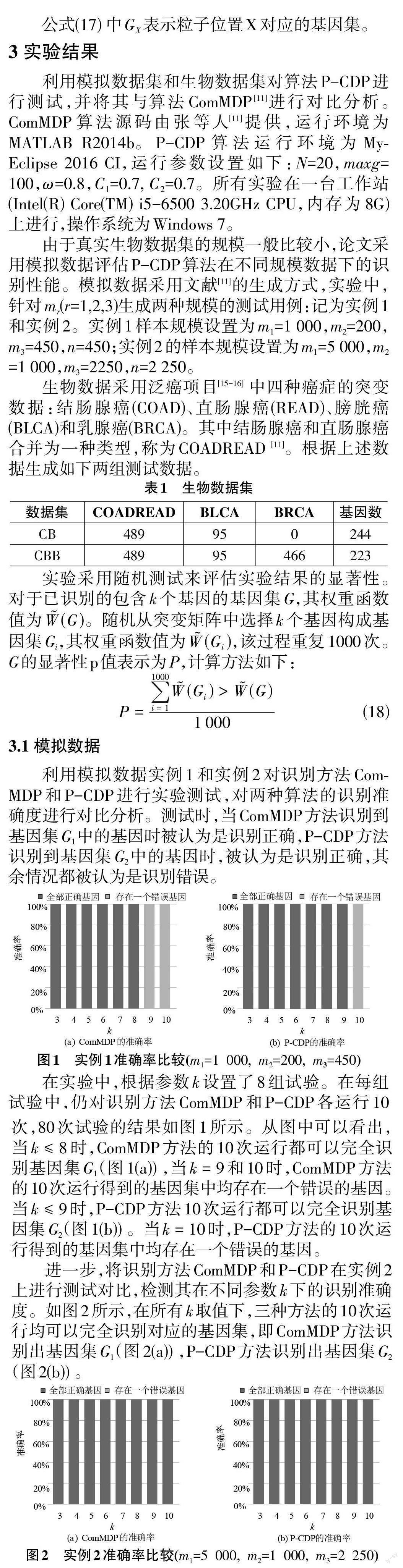
3.2 CB 数据集
在表2中,基于COADREAD和BLCA样本,针对参数k 设置了8组实验(3 ≤ k ≤ 10) 。表中字母C和P 分别表示算法ComMDP和P-CDP,后续表均以此方式表示。
如表2所示,P-CDP方法识别的基因集W?函数值较ComMDP方法识别的基因集W?函数值要大。P-CDP 方法共识别12个基因TP53, HCN1, APC, IFNA6, RB1, POLB, CTNNB1, HRAS, TGFBR2, CHD1, MAP3K1,C10orf71( 加粗基因表示仅被P-CDP方法识别),其中4个基因未被ComMDP方法所识别。ComMDP算法识别的基因涉及6 个信号通路:viral carcinogenesis 通路[17] (HRAS, RB1, CCND1), PI3K-Akt signaling pathway 通路[18] (IFNA6, HRAS, EPHA2, CCND1), thyroid hormonesignaling pathway通路[19] (HRAS, CTNNB1, CCND1), sig?naling pathways regulating pluripotency of stem cells 通路 [20] (APC, HRAS, CTNNB1), FoxO signaling pathway通路[21] (HRAS, CCND1, TGFBR2) 和hippo signaling path?way通路[22] (TGFBR2, APC, CTNNB1, CCND1)。P-CDP 方法能够识别出富集在前3 个通路的新基因:viralcarcinogenesis [17] (TP53, HRAS, RB1, POLB),PI3K-Aktsignaling pathway 通路[18] (TP53, IFNA6, HRAS), thyroidhormone signaling pathway通路[19] (TP53, HRAS, CTNNB1),且其識别的基因还富集在一个重要的致癌通路MAPK signaling pathway[23] (TP53, HRAS, TGFBR2,MAP3K1)。由于P 值均小于0.0001,因此在不同k 取值下识别的基因集均具有统计显著性。
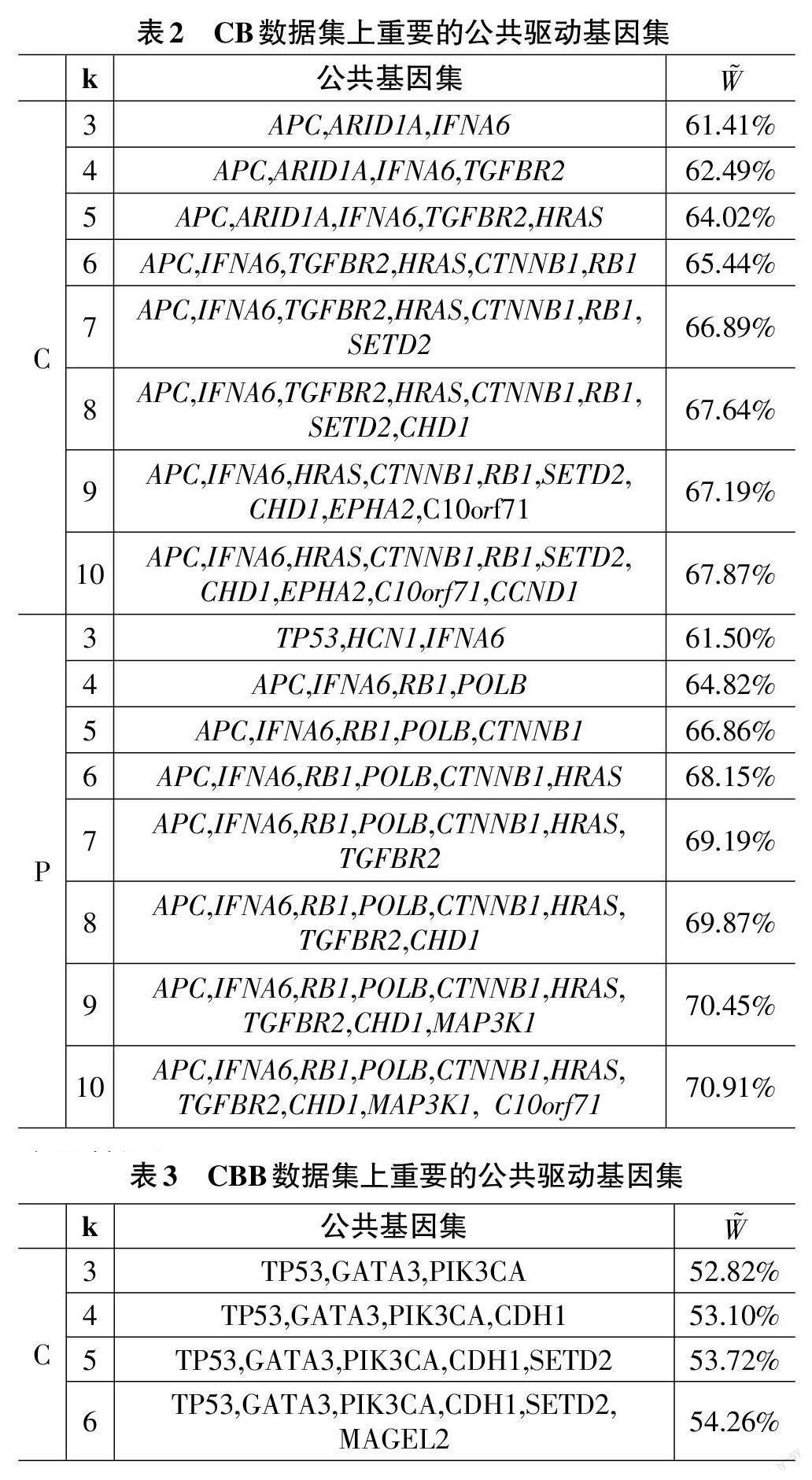
3.3 CBB数据集
将BRCA 样本整合至CB 数据集构建CBB 数据集,针对k 的不同取值识别公共驱动基因集。识别结果如表3所示,与ComMDP方法获得的基因集相比,P-CDP方法获得的基因集仍然均具有较大的W?函数值。P-CDP 方法总共识别12 个基因TP53, GATA3, CDH1, MTAP, MAP3K1, HCN1, SDK1, MAGEL2, PIK3R1, DBIL5P, BRCA2, MLL2,其中5 个基因未被ComMDP方法所识别。ComMDP方法识的基因涉及5 个重要的信号通路:apoptosis通路 [24] (TP53, PIK3CA),PI3K-Akt signaling pathway 通路[18] (TP53, PIK3CA,TNXB), neurotrophin signaling pathway 通路[25] (TP53,PIK3CA, MAP3K1), bacterial invasion of epithelial cells 通路[26] (PIK3CA, CDH1) 和 MAPK signaling pathway通路[23] (TP53, MAP3K1, NF1)。P-CDP方法能够识别富集在前4 个信号通路上的新基因:apoptosis 通路 [24](TP53, PIK3R1), PI3K-Akt signaling pathway 通路[18](TP53, PIK3R1), neurotrophin signaling pathway 通路[25](TP53, PIK3R1, MAP3K1), bacterial invasion of epithe?lial cells 通路[26] (PIK3R1, CDH1)。由于P 值均小于0.0001,因此在不同k 取值下识别的基因集均具有统计显著性。
基于上述实验,P-CDP方法共识别出11个Com?MDP 方法未识别的基因:POLB, MTAP, SDK1, PIK3R1, DBIL5P, MLL2, CHKB, GPS2, NFE2L2, CABLES1 和ATM。其中4 个基因(POLB,PIK3R1, CHKB, ATM) 参与了8个重要通路,5个基因(MTAP,MLL2,GPS2, NFE2L2, CABLES1) 已被证实与多种癌症的发病相关。例如,甲基硫代腺苷磷酸化酶MTAP是一种经常被p16编码的基因,在不同类型的肿瘤中,MTAP 缺失与肿瘤侵袭性增加和预后不良有关;赖氨酸甲基转移酶KMT2D(也称为MLL2) 是最常见的突变基因之一,其突变可能会通过扰动控制B细胞激活通路的抑癌基因的表达来促进恶性增生;GPS2 參与增殖、凋亡、DNA修复等许多生理和病理过程,在脂肪肉瘤中起着抑癌作用;NFE2L2 也称为NRF2,是常见的抑癌基因之一,NRF2的激活会加快癌细胞的增殖和生长;CABLES1 是新型垂体肿瘤易感基因,参与肾上腺垂体负反馈细胞周期调节。
4 结论
识别跨多种类型癌症的公共驱动通路是一项极其重要的任务。论文对这一问题进行了深入研究,并取得了两项重要成果:首先,提出了一种改进的数学模型,能够从头开始识别多种癌症类型之间的公共驱动基因集。该模型采用相对权重值代替绝对权重值,避免了大样本量癌症对权重值的决定作用,并引入调和平均数,使各矩阵平均权重值较高且差距较小。其次,通过引入一种短粒子编码方式,提出了一种求解该模型的粒子群优化算法 P-CDP。
利用模拟数据和真实的生物数据对提出的粒子群优化算法进行了实验测试。结果表明,该算法在求解大规模问题时仍具有较好的性能。论文对比分析了ComMDP和P-CDP两种方法的识别性能,实验结果表明,P-CDP方法能够识别出未被ComMDP方法识别的重要基因。首先,对于ComMDP方法识别的基因所富集的生物通路,算法P-CDP可以识别出富集于这些通路中的新基因;其次,算法P-CDP识别的基因还富集于其他重要的癌症相关生物通路;最后,算法PCDP能够识别出在癌症形成和发展中起至关重要作用的基因。
综上所述,实验结果表明,P-CDP能够成功地识别出ComMDP所忽略的一些具有重要生物学意义的驱动通路。因此,它有可能成为一种有效的补充工具,帮助我们更好地识别癌症通路。
