基于CRISPR/Cas系统的核酸生物传感器*
2023-08-14林昱坤宋春燕杨丹婷
陈 思 林昱坤 宋春燕 只 帅 李 毅 杨丹婷*
(1)宁波大学医学部公共卫生学院,浙江省病理生理学技术研究重点实验室,宁波 315211;2)ARC Centre of Excellence in Nanoscale Biophotonics, University of New South Wales, Sydney 2052, Australia)
CRISPR/Cas 系 统 (clustered regularly interspaced short palindromic repeats/CRISPRassociated proteins)是由成簇规律间隔短回文重复序列(CRISPR)和CRISPR 关联蛋白(Cas)组成的原核适应性免统系统,存在于大多数古菌和许多细菌中,于1987 年由Ishino 等[1]在大肠杆菌中发现,2002 年被Jansen 等[2]正式命名。CRISPR/Cas系统作为基因编辑工具被广泛应用于生物、医学、农业等多个领域[3-4]。随着研究者对CRISPR/Cas系统特异性识别靶标功能的深入研究,CRISPR/Cas系统在核酸检测领域开启了新的应用大门。目前已发表的基于CRISPR/Cas系统核酸检测的综述[5-10],主要围绕其与基于荧光、电化学和比色法结合的传感器进行介绍,少有结合表面增强拉曼光谱技术的进展综述,同时缺乏针对核酸检测系统中提高检测灵敏度策略的分类、归纳和对比。基于此,本文从提高CRISPR/Cas 系统核酸检测灵敏度的3 种策略(利用增加检测靶标量的核酸扩增技术;利用高灵敏信号转导的电化学和表面增强拉曼光谱方法;利用传感器的特殊结构设计)入手,对基于这3种方式的CRISPR/Cas 系统核酸检测生物传感器的最新研究进展进行了综述、总结和评论,以期为针对核酸检测的新一代CRISPR/Cas 生物传感器检测新方法的开发提供参考。
1 CRISPR/Cas系统的分类
CRISPR/Cas系统可分为1类系统和2类系统两大类群(表1)。1类系统包括I(Cas3)、III(Cas10)和IV(Csf1)型,由多个效应蛋白发挥作用;2类系统包括II(Cas9)、V(Cas12)和VI(Cas13)型,仅需依靠单个蛋白质发挥作用,操作简单高效,可被广泛用于核酸检测的生物传感器研究[11-12]。这里对广泛使用的3种用于核酸检测的蛋白质Cas9、Cas12和Cas13进行介绍。
Cas9 蛋白是目前被研究最为广泛的效应蛋白(图1a),能够对双链DNA(double-strand DNA,dsDNA) 进行定点编辑。其crRNA (CRISPR RNA) 和反式作用 crRNA (trans-activating CRISPR RNA,tracrRNA)中的重复序列杂交构成单向导RNA (single guide RNA,sgRNA),与Cas9蛋白形成sgRNA-Cas9复合体。在靶标核酸出现后,sgRNA-Cas9 复合体可定位到靶序列的前间隔序列邻近基序(protospacer adjacent motif,PAM)位点,使靶标序列dsDNA 被部分打开,crRNA 与互补链杂交,Cas9 蛋白中的HNH(His-Asn-His)结构域切割杂交DNA 链,RuvC 结构域切割游离链,造成目标DNA 的断裂。此外,Cas9蛋白的另一种形式, 核酸酶缺陷型Cas9(deactivated Cas9,dCas9)在酶切活性失活后也具备特异性的序列结合能力[19]。两种类型Cas9 蛋白均可用于核酸检测的生物传感器开发。
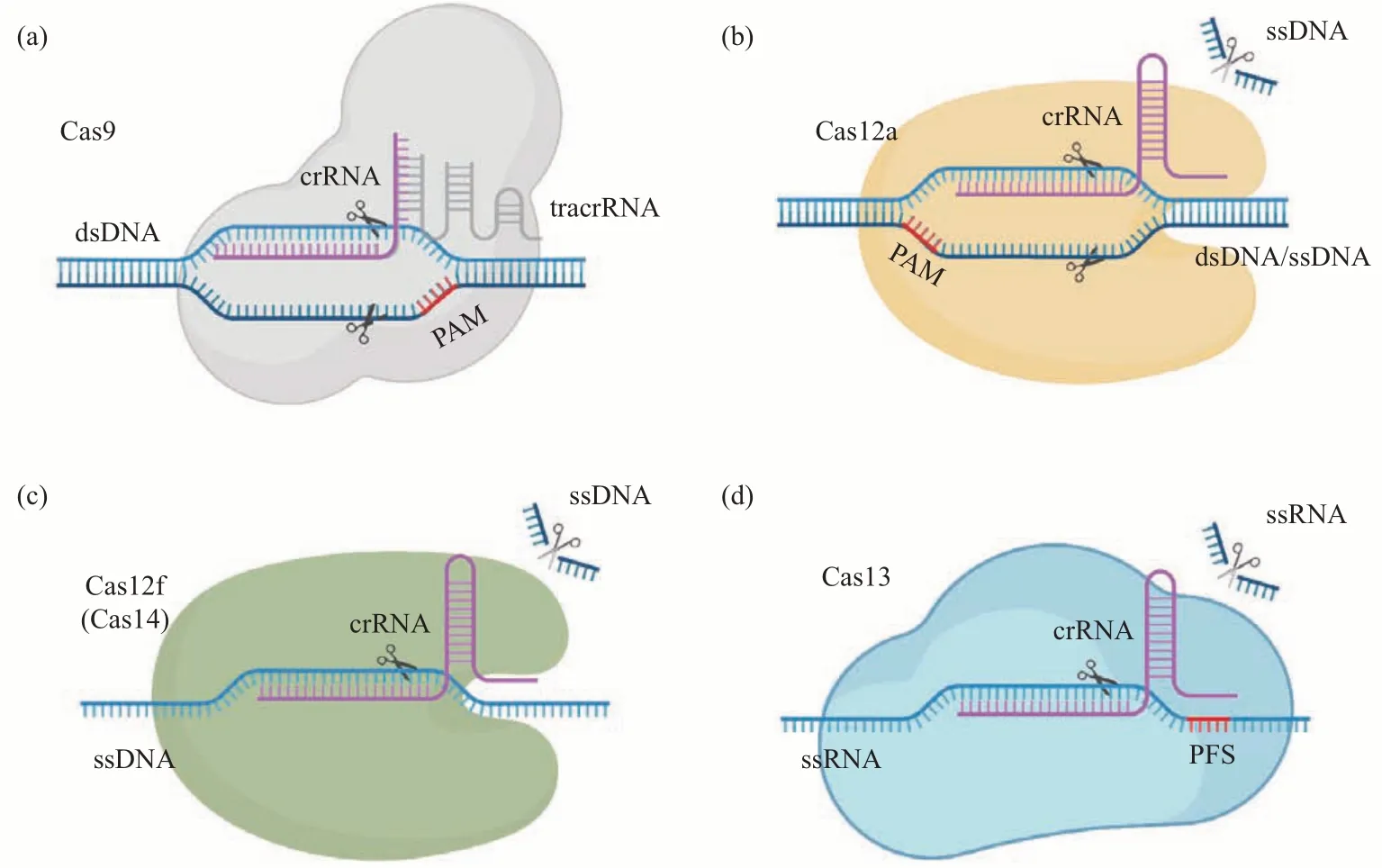
Fig. 1 Fundamental components of CRISPR/Cas9,CRISPR/Cas12a,CRISPR/Cas12f and CRISPR/Cas13图1 CRISPR/Cas9、CRISPR/Cas12a、CRISPR/Cas12f和CRISPR/Cas13结构示意图
Cas12蛋白包括Cas12a、Cas12b和Cas12f等10个亚型[13]。Cas12 由crRNA 和Cas12 核酸酶组成,与Cas9 不同,它只包含一个RuvC 结构域。在crRNA 的引导下,Cas12a 蛋白可以利用RuvC结构域识别PAM序列,特异性切割dsDNA或单链DNA(single-stranded DNA,ssDNA),同时激活非特异性的ssDNA 反式切割功能[5](图1b)。Cas12f(Cas14)蛋白的大小仅为目前已知的2 类Cas 蛋白的一半[20],和Cas12a 类似,Cas12f 识别靶标也可触发非特异性ssDNA 反式切割活性,但其不需要PAM 激活就能够实现靶向ssDNA 的切割[12](图1c)。Cas12f 与ssDNA 靶标结合的特异性比Cas12a 更高,因而更适用于开发需达到单碱基分辨率的检测方法。例如,基于Cas12f 开发的一种单核苷酸多态性基因分型系统(Cas14-DETECTR),可实现蓝眼和褐眼两种基因的鉴别[21]。
Cas13 蛋白是一种独特的蛋白质,与Cas9 和Cas12 靶向DNA 不同,Cas13 仅靶向切割单链RNA (single strand RNA,ssRNA)[22](图1d)。另外,Cas13识别目标RNA依赖的不是PAM序列,而是前间隔序列侧翼位点(protospacer flanking site,PFS)。Cas13 有两个通过构象变化结合在一起的HEPN (higher eukaryotes and prokaryotes nucleotide-binding)结构域[23],当与Cas13 crRNA间隔区互补的ssRNA 序列存在时[24],Cas13 的HEPN 结构域则会启动Cas13 的顺式和反式切割活性,实现靶标序列ssRNA 以及附近任意非特异性ssRNA 的切割[25]。Cas13a 蛋白的应用最广,其混杂切割RNA的能力[26],已被开发为体外高度灵敏的核酸检测工具。
CRISPR/Cas 系统所具备的特异性序列识别能力,以及非特异性切割的信号扩增特性,使其可根据不同核酸检测需求进行设计和选择,在开发下一代新型生物传感器方面具有显著的潜力。
2 基于CRISPR/Cas系统的核酸检测生物传感器
第一个基于CRISPR/Cas 系统的生物传感方法是Gootenberg 课题组2017 年基于Cas13a 蛋白设计的SHERLOCK (specific high sensitivity enzymatic reporter UnLOCKing) 方法[27],该方法利用了Cas13a 蛋白的靶向和非靶向的ssRNA 切割活性,非靶向的ssRNA 反式切割产生信号放大效应[28],可用于检测特定的寨卡病毒和登革病毒株,区分病原菌,对人类DNA 进行基因分型以及鉴定肿瘤DNA 突变[27]。随着DNA 靶向效应蛋白Cas12a 反式切割活性的发现,CRISPR/Cas系统对DNA的检测得到了发展[29]。这一节将根据基于CRISPR/Cas系统的核酸检测生物传感器提高检测灵敏度的3种不同策略(与增加检测靶标量的核酸扩增技术联用;与电化学、表面增强拉曼光谱等高灵敏信号转导方式联用;与特殊结构设计的传感器联用),对CRISPR/Cas 核酸检测生物传感器的应用进行分类讨论。
2.1 与核酸扩增策略联用实现高灵敏度核酸检测
常用的核酸扩增策略诸如聚合酶链反应(polymerase chain reaction,PCR)、滚动循环扩增(rolling circular amplification,RCA)、环介导等温扩 增 (loop-mediated isothermal amplification,LAMP)、指数扩增反应(exponential amplification reaction, EXPAR) 和重组酶聚合酶扩增(recombinase polymerase amplification,RPA) 等(表2)常与CRISPR/Cas系统结合[30],以实现信号增强,提高检测灵敏度。

Table 2 Nucleic acid amplification techniques-CRISPR methods表2 核酸扩增-CRISPR方法
PCR是以母链DNA为模板,在DNA聚合酶的催化下,以特定引物为延伸点,通过变性、退火、延伸等步骤复制出与母链DNA互补的子链DNA的过程[31]。作为最常用的核酸扩增方法,PCR 已被用于多种病毒和细菌的检测。Tsou等[46]证明,将CRISPR/Cas12a 系统与PCR 结合,可在不到3 h 的时间内实现H1975 癌细胞基因组DNA 中两个EGFR 杂合突变(L858R 和T790M)的检测,用时是液滴数字PCR(droplet digital PCR)的一半,可检出的突变等位基因频率最小值为0.005%,ddPCR 检测限的1/10(图2a)。Liang 等[47]通过设计PCR 引物,在靶标突变位点附近引入CRISPR/Cas12a的PAM序列,可以在没有PAM序列的情况下检测到严重急性呼吸综合征冠状病毒2(severe acute respiratory syndrome coronavirus2, SARSCoV-2)的标志性尖峰蛋白突变。Wu等[48]开发了一种结合PCR 和Cas12a 的横向流动试纸条生物传感器(lateral flow biosensor,LFB),可实现裸眼观测的7 种类型非洲猪瘟病毒(African swine fever virus,ASFV)的同步检测。他们首先用PCR对含有ASFV的样品进行预扩增,然后通过crRNA的靶标识别触发Cas12a对ssDNA报告基团的反式切割,导致报告基团不能与LFB 检测线上的互补DNA 杂交,这种效应可通过LFB 用肉眼观察到,方法简单。Peng 等[49]利用CRISPR/Cas12a 系统的可编程性、序列特异性识别和高碱基分辨优势构造了3个双输入的基本AND、OR、INHIBIT 逻辑门,利用CRISPR/Cas12a 的序列可寻址能力使该AND 逻辑门能够准确地追溯和区分输入基因,在利用PCR进行靶扩增后可实现对金黄色葡萄球菌的检测,动态范围为103~107CFU/ml。Zhang等[50]利用微型热循环仪,结合PCR 与CRISPR/Cas12a 系统,开发出一种用于副溶血弧菌检测的方法,该方法可达到102 拷贝/μl 的检测灵敏度。为了减少PCR 扩增和CRISPR/Cas 检测两步反应过程中可能造成的试剂污染,Wang等[19]提出了一种使用毛细管一锅法快速实现PCR 和Cas12a 酶切结合的方法,检测可在10 min内对最低1.28拷贝的目标序列产生明亮的荧光,并可通过比色反应实现裸眼检测。尽管PCR法能够大大提高CRISPR/Cas 系统的灵敏度,但利用PCR法进行扩增需要专门的设备,昂贵的试剂、训练有素的人员以及较长的周转时间[31-32,51],不利于CRISPR/Cas12a系统在现场检测的应用。为了克服以上困难,无需特殊温度调节的等温扩增方法诸如RCA、LAMP、RPA 等,更适用于即时检测(point-of-care-testing,POCT)技术的开发。

Fig. 2 CRISPR/Cas combined with nucleic acid amplification technology for super sensitive nucleic acid detection图2 CRISPR/Cas系统与核酸扩增技术联用用于核酸的超灵敏检测
RCA 是以环状ssDNA 为模板通过一个短的DNA 引物(与部分环状模板互补),在DNA 聚合酶催化下将脱氧核糖核苷三磷酸(deoxyribonucleoside triphosphates,dNTPs)转变成包含成百上千个重复模板互补片段ssDNA 的过程[44]。Wang 等[52]构建的RCA 辅助CRISPR/Cas9 平台能够特异性识别RCA 扩增长链ssDNA,激活CRISPR/Cas9对探针信号分子的切割活性,通过记录荧光变化实现恒温下细胞外小泡(extracellular vesicle, EV) 来源的微小RNA (microRNA,miRNA)高特异性检测(图2b)。Xu等[37]研究出一种将双适配体与RCA 辅助CRISPR/Cas12a 技术相结合检测耐甲氧西林金黄色葡萄球菌的方法,两种适配体分别用于捕获细菌和进行RCA 扩增。研究表明,此方法不仅可以特异性识别细菌表面上的蛋白质靶标,还可以将蛋白质识别转化为核酸信号,获得双倍的核酸信号扩增。Zhang 等[53]利用RCA 和PCR 扩增目的基因,并通过CRISPR/Cas13a 辅助荧光读出系统进行检测,可检测到1 拷贝/μl 的乙肝病毒共价闭合环状DNA(hepatitis B virus covalently closed circular DNA,HBVcccDNA)。然而,与定量PCR 和液滴数字PCR方法相比,该方法对于cccDNA的定量是非线性的,需要进一步优化。
LAMP 使用4~6 个引物互补于6~8 个不同的目标序列,可实现高度的靶向特异性[42]。LAMP 与CRISPR/Cas 的联合应用涉及多种Cas 蛋白。比如Song 等[54]开发了一种结合LAMP、CRISPR/Cas9和比色反应的方法,Cas9 的应用可以消除LAMP产物的假阳性信号,以实现对SARS-CoV-2及其变异基因的1 h 内检测,并且在136 个临床样本检测的盲法实验中,检测敏感度和特异性都能达到100%,该实验使用比色法,使结果可直接用裸眼观察到,方便直观。Wu等[55]以含有CaMV35S启动子的转基因大豆粉为检测对象,将CRISPR/Cas体系直接与LAMP 扩增产物在37℃下混合5 min,在紫外光下肉眼可清楚地识别检测结果。同年该课题组[56]还提出了一种基于CRISPR/Cas12a 的便携式生物传感器(Cas12a-PB),该装置具有3个通道和3个检测室,将其与LAMP结合可实现对转基因大豆粉中CaMV35S 启动子和凝集素基因的荧光检测与裸眼的双重检测,方便携带(图2c)。为了实现对RNA 的检测,Wang 等[57]提出了一种结合反转录(reverse transcription,RT)-LAMP 和Cas12a 的SARS-CoV-2 一锅法可视化检测方法,可在45 min内完成对SARS-CoV-2的检测,并达到接近单分子水平的检测灵敏度。Ali 等[44]开发的一种名为iSCAN的高灵敏SARS-CoV-2核酸检测平台,同样可以在45 min 获取可靠的SARS-CoV-2 阳性信号。Mahas 等[58]识别出一种新的Cas13 变体,并将其命名为微型Cas13(miniature Cas13,mCas13),通过mCas13 与RT-LAMP 的结合,使用STOPCovid策略中的引物设计方法,可在1 h 内检测到低至4拷贝/μl的SARS-CoV-2。
RPA 是一种依赖于重组酶、单链结合蛋白和DNA聚合酶实现的等温核酸扩增技术[33]。RPA和Cas12a 也可用来开发一锅法检测,与上述基于RT-LAMP 的一锅法检测相比,该方法用时更短,仅需5~30 min[59-60]。例如Feng 等[59]将RT、RPA和Cas12a 介导的检测集成在一个试管中,实现了单一温度(40°C)下的快速灵敏RNA 检测,检测结果可在紫外光下直接可视化,或通过便携式荧光读取器进行半定量读数检测。Tsou等[31]开发了一种结合RPA、CRISPR/Cas12a 与LFB 的检测方法,直接靶向血浆进行循环人乳头瘤病毒(human papillomavirus,HPV)DNA 检测,并可以通过肉眼立即读取结果,检测限与PCR 相同,达到0.24 fmol/L,但所需时间更短,在3 h内就可完成。这种方法还可用于监测和追踪其他核酸的感染性和非感染性体液疾病(图2d)。RPA除去可辅助Cas12蛋白实现DNA检测,还可辅助基于CRISPR/Cas13系统的检测,Khan等[61]先利用RPA法对犬细小病毒2型(Canine parvovirus type 2,CPV-2)DNA进行转录与扩增,再利用Cas13a进行检测,可在30 min内达到100 amol/L的检测灵敏度。Cao 等[62]开发了一种结合RPA、CRISPR/Cas13a 与LFB 的SARS-CoV-2检测方法,此外,还设计了一种带有微流控芯片的荧光分析仪,一块芯片可同时检测两个样品,以实现高通量和快速检测。
EXPAR 以含两段重复序列的对称DNA 为模板,目标序列与模板的3'端互补杂交后,在聚合酶的作用下沿模板延伸;延伸出的dsDNA 被切口酶识别并切割后,在DNA 聚合酶的链置换作用下被释放出来,进一步与另一条模板的3'端进行杂交和扩增,形成指数形式的扩增[63]。与其他等温扩增方法相比,EXPAR 具有明显的扩增效率高、速度快的优点。Huang 等[64]建立了一种CRISPR/Cas9触发等温指数扩增反应(CAS-EXPAR)新方法。该方法以CRISPR/Cas9 剪切产生的目的DNA 片段为引物进行循环扩增反应,产生大量DNA 复制产物,并采用实时荧光法进行监测,适用于DNA、RNA 和甲基化DNA 等多种核酸检测。Wang 等[65]建立了一种PAMmer辅助CRISPR/Cas9系统介导的G4-EXPAR (Cas-G4EX) 策略,用于ssRNA 和ssDNA的位点特异性检测。CRISPR/Cas9切割目标ssRNA 或ssDNA 产生的产物片段作为引物激活EXPAR反应,富含G的EXPAR产物与氯化血红素组装形成G-四链(G4/氯化血红素),G4/氯化血红素催化2,2'-联氮-双-3-乙基苯并噻唑啉-6-磺酸(2,2'-azino-bis(3-ethylbenzothiazoline-6-sulfonic acid),ABTS)-H2O2体系产生鲜艳绿色,可实现肉眼分析(图2e)。Hang 等[66]将无逆转录(reverse transcription-free,RTF)-EXPAR 与CRISPR/Cas12a联用,实现了对SARS-CoV-2 RNA 的超灵敏检测,终点荧光读出方式的检测下限为3.77 amol/L(~2 拷贝/μl),智能手机辅助分析系统的检测下限为4.81 amol/L(~3 拷贝/μl)。与上述结合RT-RPA和RT-LAMP 的SARS-CoV-2 检测方法相比,该方法拥有更高的检测灵敏度[44,59]。
2.2 结合高灵敏信号转导技术的核酸检测生物传感器
2.2.1结合电化学技术的核酸检测生物传感器
CRISPR/Cas 体系增强的电化学方法被认为是一类新型的分析生物传感器,由于对各种生物靶标具有基于高选择性亲和力的相互作用,因而得到广泛应用。CRISPR/Cas 体系与电化学的结合通常是将具有电化学活性的底物通过ssDNA 连接到电极上,CRISPR/Cas 工具切割单链DNA,在电化学信号中产生可测量的变化[67]。Xu 等[68]开发了一种CRISPR 增强型电化学DNA (electrochemical DNA,E-DNA)传感器,在不需要核酸扩增的情况下,可实现对细小病毒fmol/L的检测限,创新性地推动了CRISPR 生物传感器的发展(图3a)。Hajian 等[69]发明了一种CRISPR 增强型石墨烯基场效应晶体管 (graphene-based field-effect transistor, gFET), 称为CRISPR-Chip。 这种CRISPR-Chip结合了CRISPR/Cas9的基因靶向能力和gFET 的灵敏检测能力,可在无需核酸扩增的情况下,达到1.7 fmol/L 的检测灵敏度,输出信号可以用手持阅读器测量。Uygun 等[70]利用dCas9-sgRNA 修饰的氧化石墨烯丝网印刷电极(GPHOXE)作为生物识别受体,实现了循环肿瘤DNA(circulating tumor DNA,ctDNA)的免标记检测。在40 s内,dCas9-sgRNA修饰的生物传感器对120 bp 的ctDNA 呈现出良好的线性关系,检出范围为2~20 nmol/L,定量限为1.92 nmol/L。对实际血样进行选择性和重复性研究,回收率大于96%(图3c)。Zamani 等[71]利用CRISPR/Cas12a 与电化学传感器的结合,并通过LAMP 技术扩增病毒基因, 实现了对人免疫缺陷病毒(human immunodeficiency virus,HIV)、HPV-16、HPV-18的检测,对于HPV-16 和HPV-18 可达到1.2×104拷贝/μl的检测灵敏度。Liu等[72]针对无扩增的HPV-16 DNA 设计了一种基于Cas12a 的电化学发光生物传感器,利用Cas12a 的特异性识别和反式切割能力实现特异性增强和信号放大,利用l-甲硫氨酸稳定金纳米团簇(Met-AuNCs)作为高效电化学发光(electrochemiluminescence,ECL) 元件实现ECL信号转换。这种基于Cas12a 的ECL 生物传感器具有较高的选择性,实现了对未稀释人体血液样品HPV-16 的检测。电化学传感器结合靶向RNA 的Cas 酶,可实现对RNA 的高灵敏度检测。Bruch等[73]介绍了一种用于现场检测的CRISPR/Cas13a驱动微流控集成电化学生物传感器,实现了对潜在的肿瘤标志物miRNA-19b 和miRNA-20a 的量化。Cui 等[74]将CRISPR/Cas13a 系统与催化发夹组装(catalytic hairpin assembly,CHA)相结合,开发了一种适用于miRNA-21检测的超灵敏电化学分析方法。在miRNA-21 存在的情况下,它会与Cas13a/crRNA 双链的间隔区杂交,激活CRISPR/Cas13a系统的切割活性,导致CHA引发剂的释放,两个发夹相互杂交形成双链结构,从而使触发器循环,使电化学信号放大百倍。此外,与适配体的联用扩大了基于CRISPR/Cas12a电化学传感器的应用范围,可实现对非核酸的检测[75-77]。
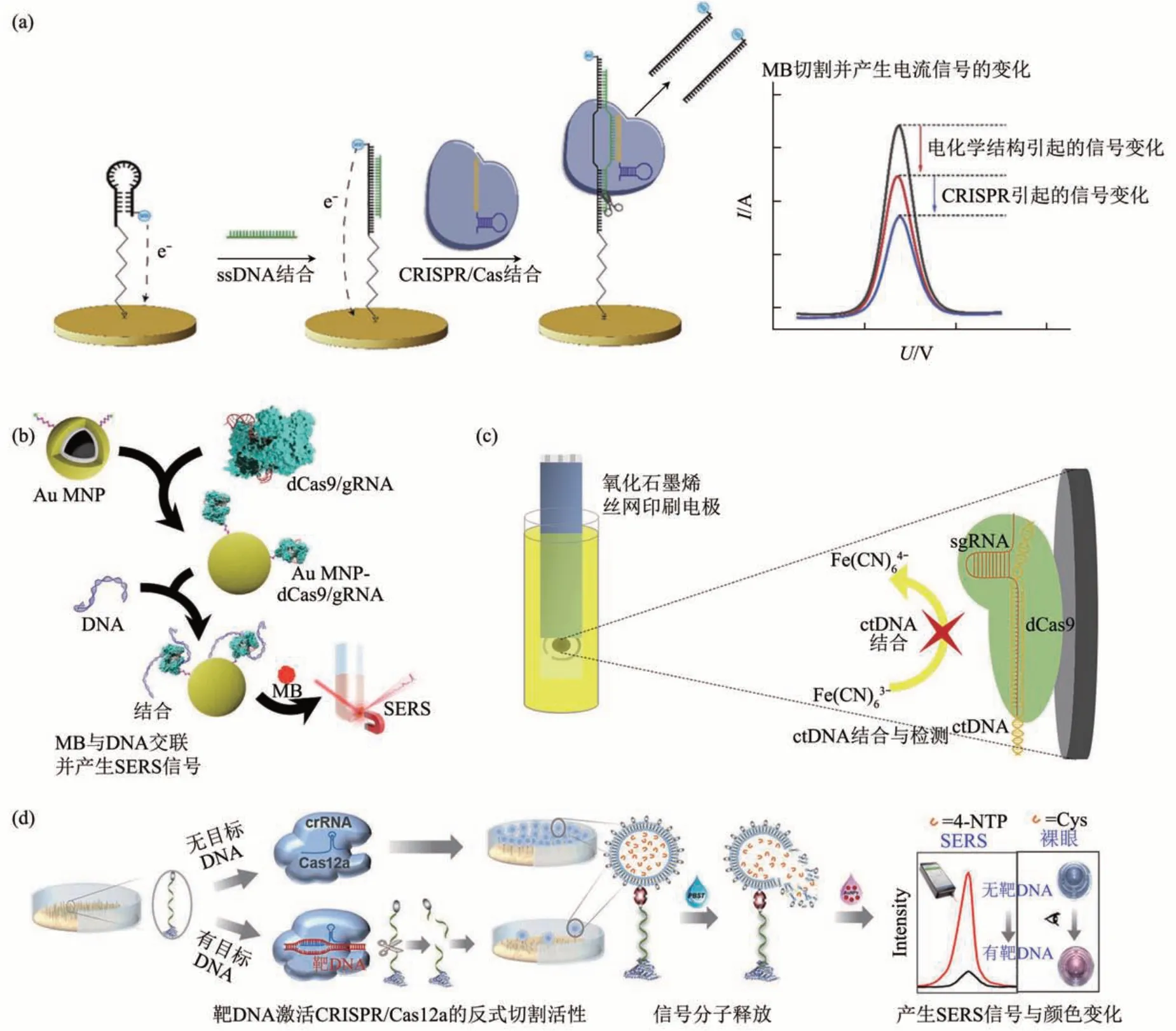
Fig. 3 CRISPR/Cas combined with signal transduction techonology biosensors for nucleic acid detection图3 CRISPR/Cas系统与信号转导技术联用的核酸检测生物传感器
2.2.2结合表面增强拉曼光谱技术的核酸检测生物传感器
表面增强拉曼光谱(surface-enhanced Raman scattering,SERS)是指当一些分子被吸附到某些粗糙的金属(如金、银、铜等)表面上时,它们的拉曼散射强度会增加104~106倍[78]。基于SERS 的生物传感器可以对极少的样品(低至几微升)进行检测并展现出极高的灵敏度(可至单分子),目前文献报道的SERS 与CRISPR/Cas 系统的结合主要是针对病毒及细菌的检测。Kim 等[79]将CRISPR/dCas9 系统与具有SERS 活性的金包磁性纳米粒子(Au coated magnetic nanoparticles,AuMNPs) 结合,开发了一种CRISPR/dCas9 介导的超级细菌检测方法。利用这种方法,可以在不需要扩增和纯化核酸的条件下检测多药耐药金黄色葡萄球菌、鲍曼不动杆菌和肺炎克雷伯菌的基因,并可达到fmol/L级别的检测限。该方法也提示可以通过选择不同的CRISPR/Cas 蛋白和gRNA 实现其他细菌、病毒、癌症和遗传疾病的检测(图3b)。Liang等[80]开发了一种CRISPR/Cas12a结合拉曼换能器的免扩增诊断平台,称为SERS-CRISPR(S-CRISPR)。使用该平台检测从鼻咽拭子标本(n=112)中提取的RNA 提取液中的SARS-CoV-2 衍生核酸,可在30~40 min 内完成。S-CRISPR 的灵敏度和特异性分别达到RT-qPCR 的87.50%和100%。Pang 等[81]结合侧向流动分析(lateral flow assay,LFA)、SERS与CRISPR/Cas12a 可直接无扩增定量检测HIV-1 dsDNA,检测限为0.3 fmol/L,比传统的比色LFA方法低近4个数量级。而且,基于Cas12a的靶点特异性,HIV-1单基因耐药突变(M184V)的识别率最低可达0.01%。Yin 等[82]设计了一种DNA/RNA嵌合发夹探针,并将其引入CRISPR/Cas12a/SERS集成系统中,该系统的SERS探针用AuNPs作为核心,包裹着4-巯基苯甲酸的AuNPs 通过部分互补的DNA 对(DNA1/DNA2)连接在其周围。在靶DNA 存在的情况下,CRISPR/Cas12a 被激活,立即切割发夹探针释放大量RNA,释放的RNA 与DNA1完全互补,导致连接DNA2的周围AuNPs从核心AuNPs表面分离出来,以产生SERS信号的变化,用于超灵敏、高选择性和无扩增的核酸检测。
此外,将SERS的高灵敏度检测能力与核酸扩增技术相结合,可达到更低检测限。比如Liu等[83]提出了一种将CRISPR/Cas12a、LAMP 与SERS 相结合的核酸检测策略,该策略利用CRISPR/Cas12a系统的反式切割活性来捕获负载4-硝基硫苯酚(4-nitrothiophenol,4-NTP)和半胱氨酸两种信号分子的脂质体,可以实现SERS 和肉眼对靶DNA的双重检测,并分别检测到低至100 amol/L 和10 pmol/L 的SERS 和可视化信号(图3d)。Pan等[84]将普鲁士蓝纳米颗粒(prussian blue nanoparticles,PB NPs)修饰的ssDNA 探针固定在微孔板上,LAMP扩增后靶标识别激活的CRISPR/Cas12a 反式切割能够移除部分PB NPs,对剩余的PB NPs 进行碱处理可生成能产生特征拉曼峰的铁氰化物阴离子(Fe(CN)64-)。通过这种方法,能够达到对牛奶DNA 的224 amol/L 检测限。Zhuang等[85]结合CRISPR/Cas12a、SERS 与RPA 扩增技术,设计了一种微流控纸质分析仪(μPAD),简称RPA-Cas12a-μPAD。这种分析仪可实现对鼠伤寒沙门氏菌的高灵敏度检测,能够在45 min 内达到3~4 CFU/ml的检测限。
2.3 利用传感器特殊结构设计提高核酸检测灵敏度
除了上述两种方法,还有一部分研究是通过结合特殊设计的传感器(包括利用信号转化中的特殊结构设计和结合一些非常规的辅助检测设备)以达到很高的检测灵敏度。信号转化中的特殊设计包括利用毛细管、微流控、微阵列等结构放大反应产生的效应,从而增大检测的灵敏度。比如Hass 等[86]设计的全集成微柱聚二甲基硅氧烷CRISPR 检测(IMPACT) 系统用于ASFV DNA 的检测。将CRISPR/Cas12a-crRNA 复合物注入经过表面修饰和探针固定化的全封闭高纵横比微柱通道,这种微柱通道对ssDNA 报告基团具有强结合力,当靶标存在时,Cas12a 酶被激活并切割微柱上的ssDNA报告基团,产生荧光信号的变化,其强度与靶标浓度成线性比例。该系统能够在120 min 内灵敏、准确地检测DNA。对于辅助检测设备的应用,Hu等[87]开发了一种基于DNA 模板化铜纳米粒子(copper nanoparticles,Cu NPs)和CRISPR/Cas9 的免标记检测方法,该方法使用电感耦合等离子体质谱(inductively coupled plasma mass spectrometry,ICPMS)识别靶标poly(AT-TA)处合成的Cu NPs并进行分析。ICPMS具有万亿分之一级的灵敏度、9个数量级的动态范围以及可追溯到初级SI单位的准确定量能力,可实现单碱基突变的快速灵敏检测(图4a)。Katzmeier 等[26]设计了一种基于Cas13a的体外转录和核酸检测低成本微型荧光阅读器,该传感器使用扁平墨盒来最大限度地收集发射光,使用经济高效的硫化镉光敏电阻作为光传感器,并建立了一套校准程序,可实现目标RNA 的序列特异性检测,检测限为3.7 nmol/L(图4b)。利用DNA探针与纳米粒子的特殊交联,并将其与CRISPR/Cas系统的反式切割活性结合,也可以得到令人惊喜的效果,比如Yu等[88]和Choi等[89]用这种模式设计的传感器可分别达到0.34 fmol/L和3.1×10-5U/ml的检测灵敏度。
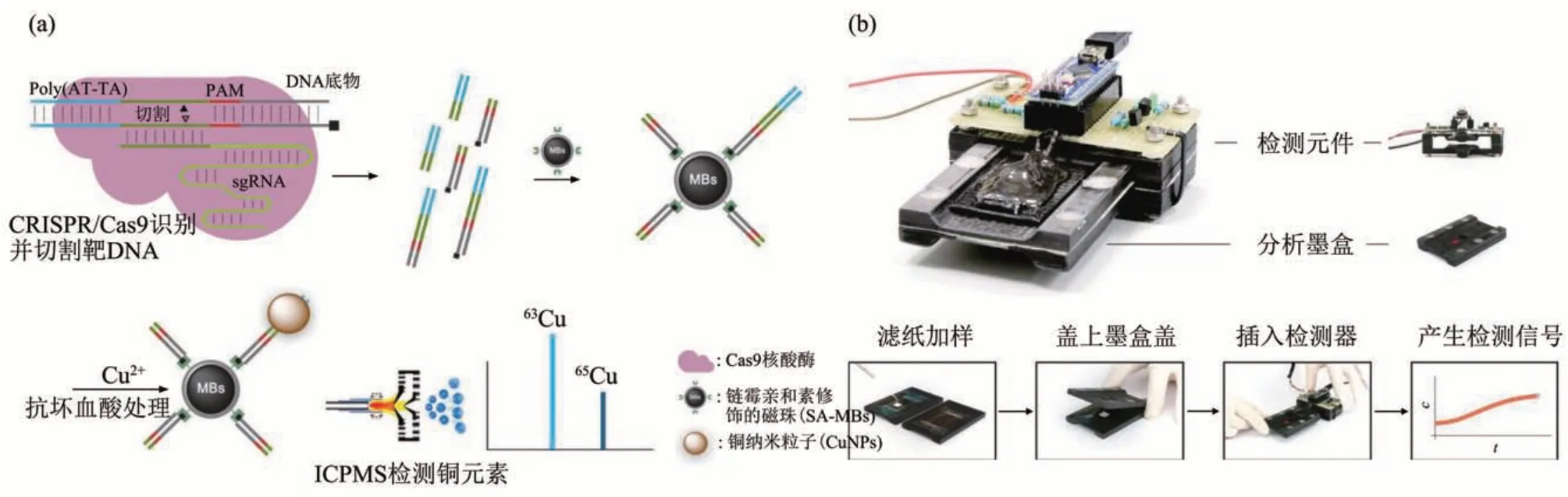
Fig. 4 CRISPR/Cas combined with specially designed sensors for super sensitive nucleic acid detection图4 CRISPR/Cas系统与特殊设计传感器联用以实现超灵敏核酸检测
表3列出了本文中提到的核酸检测生物传感器的具体信息(包括提高灵敏度最主要方式、效应蛋白、检测限、核酸扩增时间、信号输出时间、传感器类型与信号读出系统、靶标类型以及检测样品等)。

Table 3 CRISPR/Cas biosensors based on different technologies表3 结合不同策略的CRISPR/Cas核酸检测生物传感器
3 总结与展望
作为当下最常用的基因编辑工具,CRISPR/Cas系统因其可编程性和高度的特异性被广泛用于新品种培育、基因通路研究、基因药物研制以及核酸检测等领域,并取得了巨大的成功[90]。基于CRISPR/Cas 系统的核酸检测生物传感器具有高灵敏度、高选择性、低成本、快速及操作简单等显著优势,在感染性病原体的核酸检测[44,47,57,80]和蛋白质小分子等非核酸检测[91-93]显示出其巨大潜力。除了本文重点介绍的Cas9、Cas12 和Cas13 蛋白,基于Cas10 的研究也取得了一定的进展。与前3 种系统不同的是,CRISPR/Cas10 的结构是由多个蛋白质亚基结合在crRNA上形成的核酸蛋白复合物,既能降解RNA 又能降解DNA[94]。Santiago-Frangos 等[95]结合RT-LAMP、T7 转录与CRISPR/Cas10,建立了一种靶向SARS-CoV-2 的高灵敏度检测方法,可通过荧光法、比色反应和裸眼检测3种方法观察到实验结果。目前基于CRISPR/Cas 系统的综述多集中在对CRISPR-Cas9、-Cas12、-Cas13 的介绍与应用,但相信随着CRISPR/Cas10优势的不断发掘,会有更多基于其的应用得到发展。
所有的CRISPR/Cas 系统都可以在温和的条件下,在短时间内被编程并用于核酸的高特异性CRISPR/Cas检测,这是基于CRISPR/Cas的核酸检测工具最突出的优势[90]。核酸扩增是核酸检测的首要步骤,在核酸扩增后引入CRISPR/Cas 系统,可以提高检测的特异性[23]。然而在大部分核酸扩增的温度下,CRISPR/Cas系统的活性会大大降低,因而需要分步反应,从而增加了检测的时间和工作量。而CRISPR/Cas 系统与电化学和表面增强拉曼光谱等信号转导策略或与灵敏的检测仪器结合可以省去繁琐的扩增步骤,对核酸进行直接检测,具有快速、高灵敏度、高特异性和低成本等优点。除此之外,基于CRISPR/Cas 的诊断工具还存在一系列挑战:Cas蛋白切割的脱靶效应会导致非靶标序列切割从而产生错误的信号;依赖于PAM 序列的识别导致潜在靶标序列有限,虽通过使用特定设计的引物来引入PAM,可以消除序列限制,但不能用于无扩增检测[29,96];基于CRISPR/Cas的核酸定量检测依赖于复杂的仪器设备从而限制了其广泛应用;Cas蛋白的反式切割导致多路复用困难[97],等等。因此,着重解决以上问题,并与可视化的信号读取方法相结合实现定量及多重检测,CRISPR/Cas系统必将作为核酸检测的有力工具,为人类疾病的诊断事业做出贡献。
