麦卢卡蜂蜜植物源性成分鉴别方法的建立
2023-08-05杨艳歌王迎春刘鸣畅黄文胜吴占文吴亚君
杨艳歌,王迎春,刘鸣畅,牛 娜,黄文胜,吴占文,王 帅,康 婕,吴亚君,
(1.中国检验检疫科学研究院,北京 100176;2.深圳海关食品检验检疫技术中心,广东 深圳 518045;3.陕西省产品质量监督检验研究院,陕西 西安 710048)
蜂蜜一直以抗菌、修复、调养而闻名于世,体外研究和局部使用均已证明蜂蜜对许多病原微生物具有抗菌修复作用,这种抗菌活性是渗透(吸水)、酸度、过氧化氢和植物化学因子(如多酚)等作用的结果[1-4]。少数蜂蜜品种体外抗菌活性高于过氧化氢的存在,这种活性不依赖于蜂蜜中的过氧化氢,被称为非过氧化氢抗菌活性(non-peroxide antibacterial activity,NPA)[5-7]。由新西兰麦卢卡树(Leptospermum scoparium)产生的一种单花蜜——麦卢卡蜂蜜就是这类蜂蜜中最突出的一种,这是由于其花蜜中存在二羟基丙酮(dihydroxyacetone,DHA),可通过非酶脱水形成了杀菌剂甲基乙二醛(methylglyoxal,MGO)的缘故[8-11]。另外,有研究证实麦卢卡蜂蜜还具有抗氧化[12-13]、抗肿瘤以及抑制肿瘤细胞增殖[14-15]、帮助伤口愈合[16]和消炎[17-18]等特性。因此麦卢卡蜂蜜被认为具有较高药用价值而被喻为新西兰的“国宝”,并在全世界范围内受到追捧[19],这也导致其售价远高于其他蜂蜜,并且还在不断攀升,已成为蜂蜜中的奢侈品[20-21]。一些不法分子为谋取暴利,将麦卢卡蜂蜜掺假掺杂卡奴卡或其他蜂蜜,作为单一麦卢卡蜂蜜进行交易[22]。并且据不完全统计,新西兰每年生产约1700 t麦卢卡蜂蜜,然而全球每年的消费量约为1万 t[23],也就是全世界销售的麦卢卡蜂蜜远超过新西兰蜂蜜的产量,因此其真实性也受到全社会的关注[24]。
通过花粉、风味和颜色分析和/或通过电导率测量等传统的蜂蜜纯度测定方法不适用于麦卢卡蜂蜜的真实性鉴别[25],因为通过显微镜观察同时收获的麦卢卡和卡奴卡花粉粒,发现外观几乎相同[26],因此无法通过花粉分析区分这两个品种。然而麦卢卡蜂蜜和卡奴卡蜂蜜的花期相似,在田间也很难区分,而卡奴卡峰蜜既不含DHA[27]也不含MGO[28],也几乎不显示非过氧化物活性[29]。因此,检测麦卢卡蜂蜜中是否含有卡奴卡成分对于确定其纯度非常重要。以往通过检测特征标志物MGO鉴别麦卢卡蜂蜜,以及依赖于MGO的含量对麦卢卡蜂蜜认证定级的方法也不可行[30],因为一方面造假者可以通过直接添加MGO或其前体DHA到卡奴卡蜂蜜中进行掺伪而升级为“麦卢卡”蜂蜜[31],另一方面DHA在长时间的储存和加热过程中会被非酶转化为MGO,而导致MGO增加[17,32]。为此,新西兰农业部(Ministry for Primary Industries,MPI)在2014年发起了“麦卢卡蜂蜜科学计划”项目[25],并确定了用4 种化学物和一个DNA标记物5 种属性综合判别的方式,以区分麦卢卡蜂蜜和非麦卢卡蜂蜜、单花麦卢卡蜂蜜以及多花麦卢卡蜂蜜[33-34]。然而该规定的检测耗时长、成本高,并且其DNA标记物方法未公开检测的引物探针序列,每此实验需要采购其规定的DNA提取试剂盒和实时聚合酶链式反应(real-time polymerase chain reaction,real-time PCR)检测试剂盒,因此开发廉价、快速的麦卢卡蜂蜜鉴定方法具有重要意义[35]。
此外,MPI规定的DNA标记物方法是对蜂蜜花粉沉淀提取DNA进行检测,事实上以往基于DNA区分蜂蜜物种来源的方法一般都是对蜂蜜的花粉沉淀进行DNA提取[36-37]。这是由于研究者一般认为蜂蜜本身不含植物DNA,提取蜂蜜的DNA实际上是蜜蜂采蜜过程中带入的花粉粒DNA[38]。但仅进行此检测很难区分造假者有意将蜂花粉掺入糖浆造假蜂蜜的方式。因此Wu Yajun等[39]建议在对蜂蜜进行植物源分辨时,建议增加蜂蜜上清液的DNA提取方法,从而进行更加客观的判断。
为此本研究开展了以下实验:1)比较市场常见的试剂盒、传统CTAB法以及MPI指定的DNA提取试剂盒对麦卢卡蜂蜜花粉沉淀的DNA提取效果;2)建立麦卢卡蜂蜜上清液DNA提取方法,以弥补以往研究仅对麦卢卡蜂蜜花粉沉淀进行鉴别的不足;3)建立植物内参照、麦卢卡、卡奴卡3 种物种源性成分鉴别的real-time PCR方法,分析方法的特异性、灵敏度与检出限;4)比较本研究与MPI规定的方法对蜂蜜样品的检测效果。通过以上4 个方面的研究,建立了高效提取麦卢卡蜂蜜沉淀和上清液DNA的方法;提供了可快速、灵敏、准确鉴别麦卢卡蜂蜜植物源性成分的方法;并通过对比,证实了建立方法的可行性、准确性与等效性,以期为快速检测麦卢卡蜂蜜的真伪提供技术支撑。
1 材料与方法
1.1 材料与试剂
1.1.1 样品
油菜花粉、柳树花粉、猕猴桃花粉、荞麦花粉、荷花花粉、茶花花粉、玫瑰花粉、虞美人花粉由中国农业科学院蜜蜂研究所提供;洋槐、紫云英、枸杞、荆条、荔枝、龙眼、巨桉、枣、椴树、向日葵、棉花、苹果、橘、芝麻、黄瓜、小麦、桑、蚕豆、冬青、木兰、橄榄、桂花、国槐、甘薯、郁金香、芒果、葡萄、草莓、薰衣草、百合、菊花、杜鹃、水仙、番茄、银杏、松、绿藻样品为本实验室收集;麦卢卡蜂蜜及卡奴卡蜂蜜样品为MPI提供。
1.1.2 试剂
Geneaid蜂蜜基因组DNA提取试剂盒 中国台湾Geneaid技术有限公司;NucleoSpin®Food 德国Macherey-Nagel公司;植物基因组DNA提取试剂盒 天根生化科技(北京)有限公司;Promega Wizard™ Magnetic食品DNA纯化系统 美国Promega公司;ManKanTM蜂蜜real-time PCR试剂盒、ManKan™ DNA标准品、ManKan™蜂蜜阳性质控 新西兰dnature诊断研究有限公司;TaqMan Fast Advanced Master Mix 美国Applied Biosystems公司。
1.1.3 引物探针
从GenBank数据库中检索获得麦卢卡、卡奴卡以及其他常见蜂蜜植物物种的NADH和ITS基因序列。利用Clustal X软件进行多重序列比对,利用Primer Premier 5.0软件在NADH保守区域设计内参照(显花植物通用)引物探针,在ITS种间差异区域分别设计麦卢卡和卡奴卡的特异性引物探针。筛选好的引物和探针信息如表1所示,所有引物探针均在上海英潍捷基生物技术有限公司合成。

表1 麦卢卡蜂蜜植物源检测引物探针信息Table 1 Primers used for the identification of plant-derived ingredients in mānuka honey
1.2 仪器与设备
7500 real-time PCR仪 美国Applied Biosystems 公司;Pico17离心机(离心力≥12000×g)美国Thermo公司;TissueLyser II组织研磨仪 德国QIAGEN 公司;核酸蛋白分析仪 日本岛津公司;涡旋仪 德国 IKA公司;恒温混匀仪、微量移液器 德国Eppendorf 公司;离心管、八连排PCR管 美国Axygen公司;电子天平 德国Sartorius公司;恒温水浴锅 北京六一生物科技有限公司;磁力架 美国Promega公司。
1.3 方法
1.3.1 样品前处理
按照MPI[34]的步骤对蜂蜜样品进行处理,部分步骤稍作改良。将蜂蜜样品50 ℃水浴10 min左右至充分融化,上下颠倒混匀;统一称取1.4 g蜂蜜样品于2 mL离心管中,加入900 μL无菌ddH2O,65 ℃、1200 r/min振荡孵育10 min;15000×g离心5 min,上清液于4 ℃贮存;沉淀部分用1 mL无菌水洗脱,15000×g离心5 min,并重复洗脱一次。
花粉样品采用组织研磨机研磨1 min;水果样品直接粉碎,10000×g离心,取沉淀物部分;其他根茎叶类植物样品液氮速冻采用组织研磨机粉碎。
1.3.2 DNA提取
1.3.2.1 蜂蜜沉淀DNA提取
分别用NucleoSpin食品基因组DNA提取试剂盒、天根植物基因组DNA提取试剂盒、传统的CTAB法,以及MPI推荐的Geneaid蜂蜜基因组DNA提取试剂盒,进行蜂蜜沉淀DNA提取效果比较。Geneaid蜂蜜基因组DNA提取试剂盒的操作步骤按照MPI的文件[34]操作,NucleoSpin食品基因组DNA提取试剂盒、天根植物基因组DNA提取试剂盒、CTAB法统一加入650 μL提取缓冲液,以及10 μL 10 mg/mL蛋白酶K溶液。统一置于65 ℃、800 r/min 恒温混匀仪中温育1~2 h,15000×g离心5 min,转移上层清液至50 mL离心管中。CTAB法其余步骤按照 GB/T 19495.3—2004《转基因产品检测核酸提取纯化方法》CTAB-2方法操作[40],NucleoSpin食品基因组DNA提取试剂盒、天根植物基因组DNA提取试剂盒其余步骤按照各说明书操作,获取的DNA统一用50 μL无菌ddH2O溶解。分别用本研究建立的植物内参照方法和ManKan蜂蜜real-time PCR试剂盒方法进行检测,以ManKanTM蜂蜜质控DNA为阳性,每份DNA重复扩增3 次,比较上述4 种DNA提取方法的扩增效率,-20 ℃保存DNA。
1.3.2.2 蜂蜜上清液DNA提取
取20 mL蜂蜜上清液分装到2 个50 mL离心管中,每管分别加入裂解液A 4 mL、裂解液B 2 mL,充分混匀,静置10 min。加入5 mL沉淀溶液,振荡混匀。加入150 μL磁珠和0.9 倍溶液体积的异丙醇,室温静置1 h,期间不时颠倒,置磁力架上5 min,小心去除水相,其余步骤参照试剂盒说明书,DNA用50 μL无菌ddH2O溶解。用ManKan蜂蜜real-time PCR试剂盒进行DNA扩增效果比较,每份DNA重复扩增3 次,以无菌ddH2O为空白对照,-20 ℃保存DNA。
1.3.2.3 其他植物样品DNA提取
除蜂蜜外的其他植物样品取100~500 mg,均采用Nucleospin食品基因组DNA提取试剂盒提取DNA,用核酸蛋白定量仪测量DNA浓度,-20 ℃保存DNA。
1.3.3 real-time PCR
本研究建立的方法采用25 μL反应体系进行real-time PCR检测:12.5 μLTaqMan Fast Advanced Master Mix,10 μmol/L上下游引物各0.5 μL,10 μmol/L探针0.5 μL,模板DNA 5 μL,用无菌水补至总体积25 μL。反应程序为:50 ℃去除RNA 2 min;95 ℃预变性10 min;95 ℃变性15 s,60 ℃退火1 min,40 个循环。荧光基团设为FAM,猝灭基团设为none,阈值为仪器自动生成。
MPI方法采用其规定ManKan蜂蜜real-time PCR试剂盒[34]进行检测,采用10 μL反应体系:20×Oligo mix 0.5 μL,5×Master mix 2 μL,PCR级别水5.5 μL,模板DNA 2 μL。在real-time PCR仪上进行PCR扩增,反应程序为:95 ℃预变性3 min;96 ℃变性10 s,62 ℃退火30 s,40 个循环,ROX基底荧光校正设为none,内参照、卡奴卡和麦卢卡检测荧光分别设为ROX、VIC和FAM。
1.3.4 引物探针通用性和特异性检测
以47 种植物的DNA(5 ng/μL)进行检测,其中45 种为显花植物:麦卢卡、卡奴卡、油菜、柳树、猕猴桃、荞麦、荷花、茶花、玫瑰、虞美人、洋槐、紫云英、枸杞、荆条、荔枝、龙眼、巨桉、枣、椴树、向日葵、棉花、苹果、橘、芝麻、黄瓜、小麦、桑、蚕豆、冬青、木兰、橄榄、桂花、国槐、甘薯、郁金香、芒果、葡萄、草莓、薰衣草、百合、菊花、杜鹃、水仙、番茄,3 种为非显花植物:银杏、松和绿藻,以无菌水为空白对照,进行real-time PCR扩增,每个样品2 个平行重复。
1.3.5 绝对灵敏度分析
分别将麦卢卡和卡奴卡DNA标准品进行梯度稀释,使其质量浓度分别为100 ng/μL、10 ng/μL、1 ng/μL、100 pg/μL、10 pg/μL、1 pg/μL,共6 个梯度,用筛选好的特异性引物探针进行real-time PCR扩增,每个反应设置3 个平行,标准曲线由仪器自动生成。
1.3.6 检出限分析
将卡奴卡蜂蜜和麦卢卡蜂蜜互掺,分别按照含量比为0.1∶99.9、1∶99、10∶90、50∶50模拟制备混合掺杂蜂蜜样品,混匀后分别提取混合蜂蜜样品上清液和沉淀的DNA,分别以卡奴卡蜂蜜和麦卢卡蜂蜜DNA为阳性,以ddH2O为空白对照,进行real-time PCR扩增检出限分析,每个反应设置3 个平行。
1.3.7 蜂蜜样品检测结果对比
对MPI提供的15 份蜂蜜样品分别采用MPI指定的Geneaid蜂蜜基因组DNA提取试剂盒和NucleoSpin食品基因组DNA提取试剂盒进行蜂蜜沉淀的DNA提取,同时采用Promega Wizard™ Magnetic食品DNA纯化系统对蜂蜜上清液进行DNA提取。采用本研究建立的植物内参照、麦卢卡、卡奴卡物种源性成分real-time PCR检测方法,以及MPI指定的real-time PCR检测试剂盒进行了检测,以ddH2O为空白对照,每个反应设置3 个平行。
1.4 数据统计及图表绘制
将仪器导出检测数据录入在Excel表格中,在Excel中计算平均值和标准偏差,根据数据绘制三线表和柱形图,柱形图用不同图案填充,以重复实验的标准偏差添加误差线。扩增图谱和标准曲线仪器由仪器自动生成并导出。
2 结果与分析
2.1 real-time PCR方法建立
2.1.1 引物探针筛选
为了对蜂蜜样品的DNA进行质量控制,根据常见的蜂蜜为显花植物的特性,设计显花植物通用引物探针,对48 种植物进行real-time PCR通用性检测,结果显示,设计的ndhB基因显花植物引物探针可以对45 种显花植物扩增,扩增Ct值在14~28之间,表明通用性和覆盖性较好;银杏、松和绿藻无扩增,表明对裸子植物和原始植物特异性较好,故依此引物探针作为内参照进行蜂蜜样品DNA提取效果的质控检测,结果如图1A所示。
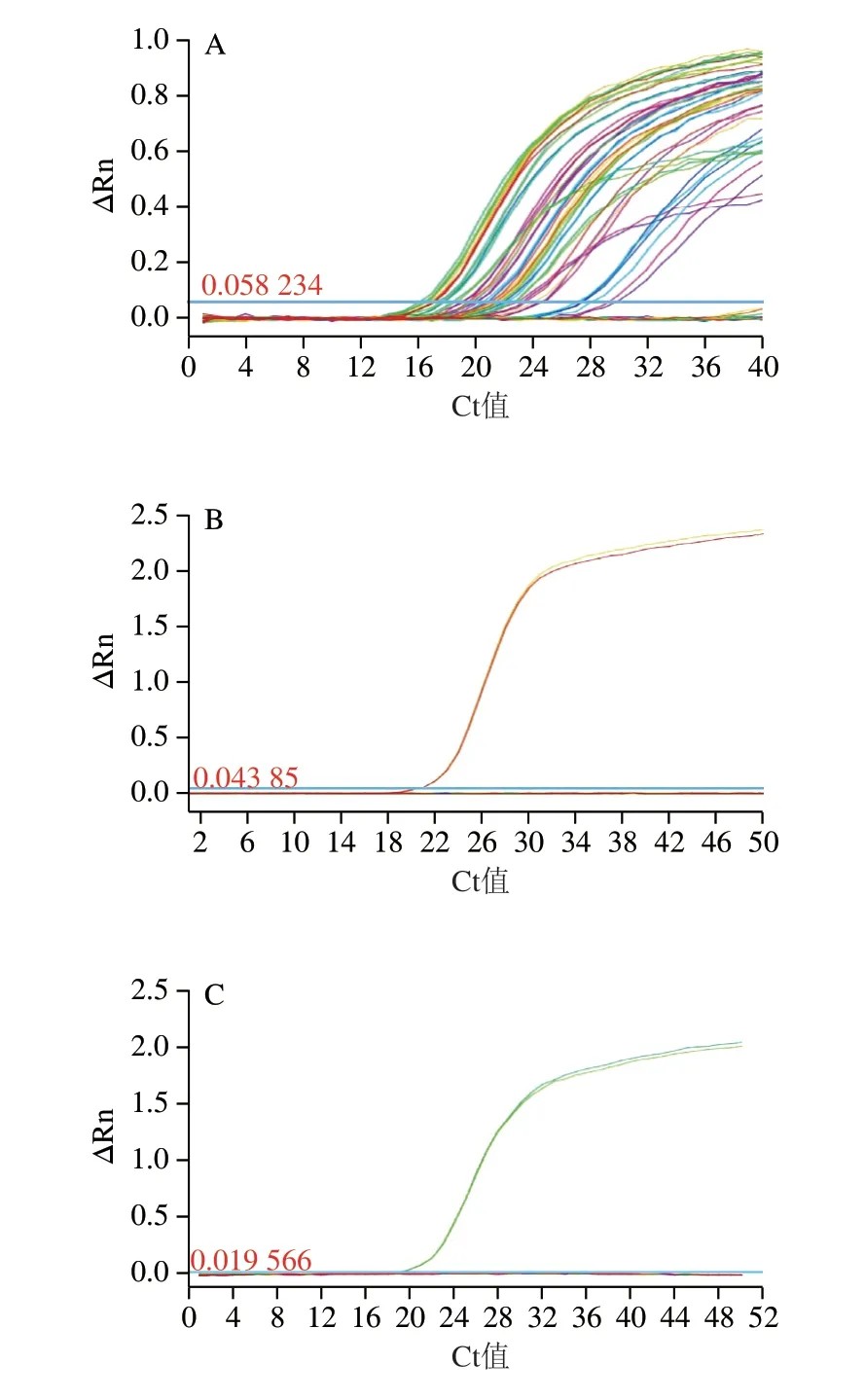
图1 引物探针通用性和特异性分析Fig.1 Generality and specificity of primers and probes
以上述47 种植物为检测对象,分别对设计的麦卢卡和卡奴卡引物探针进行扩增,结果显示这两组引物探针分别仅能对麦卢卡(图1B)和卡奴卡(图1C)进行有效扩增,Ct值分别为18.14±0.02和19.41±0.04,二者之间无交叉扩增,其他植物源成分无非特异扩增,说明设计的两组引物探针的特异性较好,可以进行蜂蜜样品中麦卢卡和卡奴卡成分的检测。
2.1.2 灵敏度分析
将6 个质量浓度梯度的麦卢卡和卡奴卡DNA进行绝对灵敏度检测,结果显示6 个梯度均有明显的扩增,当DNA质量浓度为1 pg/μL时,尚有良好的扩增,此时平均Ct值分别为35.66±0.13、37.02±0.42、36.27±0.29,因此初步确定内参照、麦卢卡和卡奴卡的绝对灵敏度均为1 pg/μL,结果如图2所示。根据仪器自动生成的标准曲线Y内参照=-3.315X+23.662、Y麦卢卡=-3.434X+25.800、Y卡奴卡=-3.880X+23.533,内参照、麦卢卡和卡奴卡引物探针灵敏度检测的扩增效率分别达100.298%、95.507%、81.019%,线性相关系数R2分别为0.999、0.998和0.995,说明线性关系良好,结果可信。因此确定本研究设计的内参照、麦卢卡和卡奴卡引物探针检测的绝对灵敏度最低可达1 pg/μL。与MPI报道的检出限为3 fg/μL,Ct值为36的灵敏度相近。

图2 方法灵敏度检测的扩增曲线Fig.2 Sensitivity analysis of the established method
2.2 DNA提取方法
2.2.1 蜂蜜沉淀DNA提取方法比较
高效的DNA提取方法是保证real-time PCR检测结果准确的前提。实验选用3 种提取方法:CTAB法、NucleoSpin食品基因组DNA提取试剂盒、天根植物基因组DNA提取试剂盒以及传统的CTAB法,与MPI规定的Geneaid蜂蜜基因组DNA提取试剂盒进行麦卢卡蜂蜜花粉沉淀DNA提取效果比较。从操作步骤和时间看,CTAB操作步骤最繁琐、耗时最长,天根、NucleoSpin和Geneaid提取流程相近,步骤较简单、耗时较短,对4 种方法提取流程的比较如图3所示。从扩增效率看,CTAB扩增效果最低,NucleoSpin的扩增效果最好,其次是Geneaid,再次是天根,扩增效果的比较如图4所示。综上,NucleoSpin操作步骤最简便,耗时时间较短,提取效率较Geneaid高,因此对麦卢卡蜂蜜的DNA提取不用局限于MPI的规定,检测过程中选择满足需求的DNA提取方法即可。

图4 不同方法提取的麦卢卡蜂蜜沉淀DNA的扩增效果Fig.4 Amplification efficiency of DNA from mānuka honey sediment by different DNA extraction methods
2.2.2 蜂蜜上清液DNA提取
MPI仅规定了麦卢卡蜂蜜花粉沉淀进行DNA提取的方式,但考虑到造假者有可能添加麦卢卡花粉到非麦卢卡蜂蜜进行冒充,为对这种情况进行鉴别,本研究又针对麦卢卡蜂蜜上清液建立了DNA提取方法。经实验,发现采用Promega WizardTMMagnetic食品DNA纯化系统可对麦卢卡蜂蜜上清液进行DNA提取,并采用该方法对MPI提供的15 份蜂蜜样品上清液进行DNA提取,并与MPI规定的Geneaid蜂蜜基因组DNA提取试剂盒的沉淀效果进行比较,结果发现建立的蜂蜜上清液DNA提取方法可以成功将所有蜂蜜DNA提出,可以满足检测要求,但与沉淀的扩增效果相比,上清液扩增效果略低于沉淀,扩增Ct值在24~36之间,沉淀DNA扩增Ct值在22~32之间,扩增结果如图5所示。
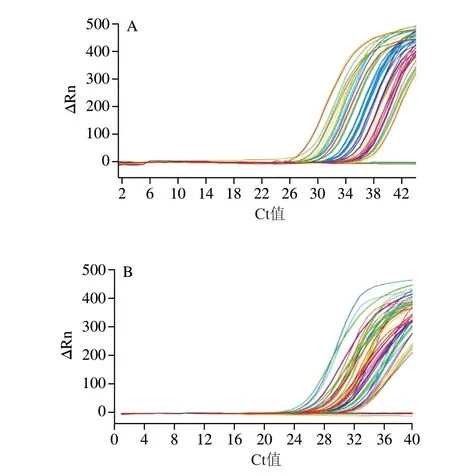
图5 麦卢卡蜂蜜上清液(A)和沉淀(B)DNA扩增效果分析Fig.5 Amplification efficiency of DNA from mānuka honey supernatant (A) and sediment (B)
2.3 蜂蜜实际检出限分析
为分析本研究建立的方法对麦卢卡及卡奴卡蜂蜜检测的实际检出限,分别提取模拟制备的混合蜂蜜样品的上清液和沉淀DNA,并进行real-time PCR扩增,检测结果如图6所示。结果发现无论上清液还是沉淀,本研究建立的内参照、麦卢卡、卡奴卡成分检出限均可达0.1%。
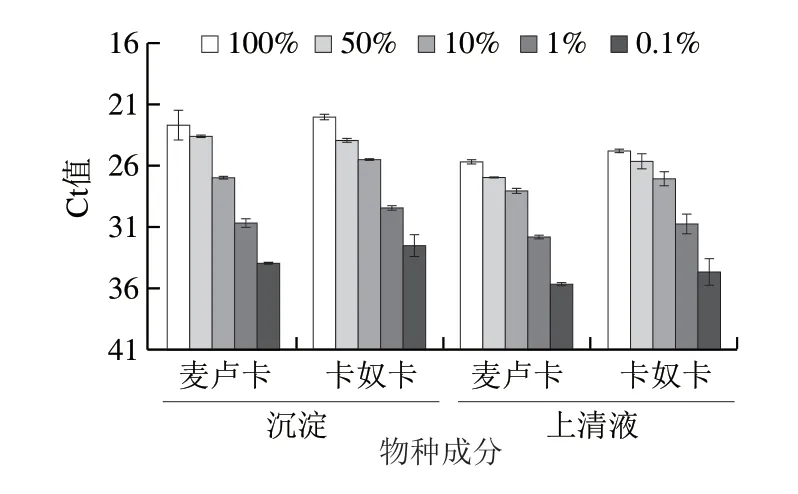
图6 混合麦卢卡蜂蜜检出限分析Fig.6 Detection limits of mixed mānuka honey
2.4 MPI提供蜂蜜样品的检测结果比较
采用本研究建立的方法以及MPI规定的麦卢卡蜂蜜DNA检测方法,对MPI提供的15 份蜂蜜进行DNA提取和扩增效果比较,详情见表2。检测结果显示3 种DNA提取方法均可获得质量良好的DNA,本研究建立的方法以及MPI规定的麦卢卡蜂蜜DNA检测方法检测结果一致,包括以下几种情况:1)3 份检出卡奴卡成分,其中1 份未检出麦卢卡成分,另2 份样品采用MPI方法平均Ct值为33.84±1.20和34.46±2.54,接近于MPI文件要求的Ct≤36.00的检测阈值,但其相对分析误差(relative percent difference,RPD)分别为5%和14.76%,MPI文件规定测试结果RPD应小于5%[22],因此该结果应判为阴性。采用本研究方法,沉淀和上清液均未检出麦卢卡,因此这2 份样品非麦卢卡蜂蜜。2)1 份检出植物成分,但既未检出麦卢卡成分也未检出卡奴卡成分,为其他植物蜂蜜。3)9 份同时检出麦卢卡分和卡奴卡成分,为多花麦卢卡蜂蜜。4)2 份检出麦卢卡分而未检出卡奴卡,应为单花麦卢卡蜂蜜。综上,这15 份样品中,13.33%为麦卢卡单花蜜,66.67%为杂花蜜,13.33%为卡奴卡蜂蜜,6.67%为其他植物蜂蜜。
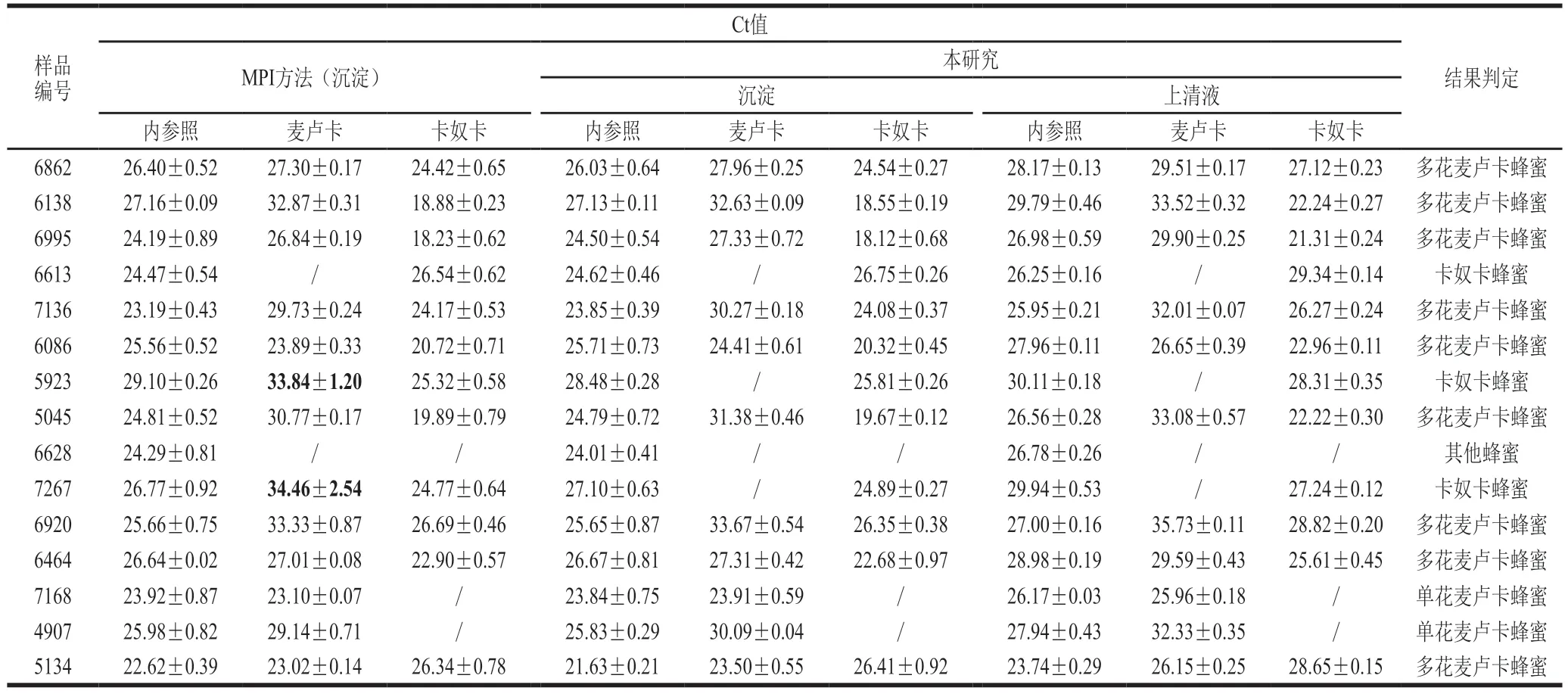
表2 对MPI提供的蜂蜜样品的检测结果分析Table 2 Results of MPI detection of honey samples
3 讨论
3.1 MPI麦卢卡蜂蜜DNA检测方法使用过程中的问题
在参与MPI麦卢卡蜂蜜DNA检测方法验证的过程中,发现一些问题:一是,DNA提取试剂盒和PCR试剂盒均需购买推荐厂商的商品,费用较昂贵,一个PCR扩增试剂盒报价999 美元(不含关税等),且这2 个商品非通用试剂,只能用于麦卢卡蜂蜜检测的,因此通常情况下国内没有现货,每次购买货期较长,影响了检测时效性。二是,未提供内参照、麦卢卡、卡奴卡3 种成分检测的引物探针序列,每次检测都需要购买指定的试剂盒,而无法自行合成,因此在使用的过程中受到诸多限制。三是,仅对蜂蜜花粉沉淀进行的检测,无法分辨以掺入花粉造假蜂蜜的方式。
3.2 麦卢卡蜂蜜DNA提取方法分析
为此,本研究首先比较常见的国产和进口食品DNA提取试剂盒,以及传统CTAB法对麦卢卡蜂蜜的提取效果,并与MPI规定的Geneaid蜂蜜基因组DNA提取试剂盒的提取效果进行比较,结果显示虽然扩增效果NucleoSpin食品基因组DNA提取试剂盒>Geneaid蜂蜜基因组DNA提取试剂盒>天根植物基因组DNA提取试 剂盒>CTAB法,但4 种方法都能提出DNA,即在对麦卢卡蜂蜜检测的过程中并非必须用其指定的或进口的试剂盒,国产试剂盒和传统CTAB法也可满足要求,若想获取高质量浓度的DNA,在使用的过程中可通过采用增加蜂蜜样品量,富集花粉沉淀的方法获取DNA,从而提高检测效率。
同时,本研究对蜂蜜沉淀进行DNA提取的过程中,还对部分步骤进行改良,即增加了对花粉沉淀水洗的过程。这是因为基因组DNA也是糖,将蜂蜜中富集的沉淀增加水洗的过程,可以尽可能去除蜂蜜中的糖分,以避免在DNA提取的过程中造成干扰,从而提高DNA的提取效率。
3.3 蜂蜜上清液DNA提取方法的优势和不足
此外,为谋取利益,造假者可能将麦卢卡花粉添加到非麦卢卡蜂蜜中以假冒麦卢卡蜂蜜,所以仅仅对蜂蜜中的花粉沉淀进行检测尚有不足。为此,本研究还建立了麦卢卡蜂蜜上清液DNA提取方法,并通过对15 份蜂蜜样品的实际检测,证实本方法可以有效提出蜂蜜上清液DNA。但同时研究结果显示,利用磁珠法提取的蜂蜜上清液的DNA扩增效率低于花粉沉淀,推测原因可能是蜂蜜样品中含有较多的糖分,会对磁珠溶液的分散体系造成影响,从而干扰了磁珠对DNA的吸附效率。但蜂蜜上清液DNA提取方法的建立弥补了以往蜂蜜蜜源植物成分溯源时仅对花粉沉淀进行检测的不足,从而能够分辨通过掺入花粉到糖浆中的造假蜂蜜。
4 结论
本研究建立了麦卢卡蜂蜜沉淀和上清液DNA提取方法,并建立了内参照、麦卢卡和卡奴卡植物源性成分real-time PCR检测方法。结果显示,内参照引物探针对显花植物的通用性、覆盖性和特异性较好,可作为内参照进行蜂蜜样品DNA提取的质控检测。麦卢卡和卡奴卡成分鉴别方法的特异性良好、灵敏度高,绝对灵敏度最低可达1 pg/μL,对混合样品的检出限可达0.1%。同时与MPI方法比较,证实了本研究方法的可行性、准确性和等效性。本研究建立的方法不仅可以用于麦卢卡蜂蜜的植物源灵敏、快速、高效的检测,还能鉴别以麦卢卡花粉造假麦卢卡蜂蜜的方式,同时还可解决以往对麦卢卡蜂蜜DNA检测需要依赖MPI官方指定检测方法的问题,为对国境口岸麦卢卡蜂蜜的质量监控提供了良好的技术支撑。
