基于比较基因组学解析植物乳植杆菌ST的功能基因组
2023-08-05杨淑娟周金萍李海燕曹振辉孙志宏林秋叶
杨淑娟,周金萍,李海燕,曹振辉,孙志宏,林秋叶,
(1.内蒙古农业大学 乳品生物技术与工程教育部重点实验室,农业农村部奶制品加工重点实验室,内蒙古自治区乳品生物技术与工程重点实验室,乳酸菌与发酵乳制品省部共建协同创新中心,内蒙古 呼和浩特 010018;2.云南农业大学食品科学技术学院,云南 昆明 650201;3.云南农业大学动物科学技术学院,云南 昆明 650201)
植物乳植杆菌(Lactiplantibacillus plantarum)属于乳植杆菌属,是乳酸菌的一种,其广泛存在于食品(如蔬菜、乳肉制品等)和肠胃道等多种环境中[1],己被人类食用或使用上千年,是一类公认安全的微生物[2]。L.plantarum具备改善肠道菌群平衡、调节免疫代谢、抵抗病原体、降低胆固醇、缓解乳糖不耐症和加强营养成分吸收等益生功能,是益生菌开发的潜力贮备[3-4]。因此,L.plantarum作为潜在益生菌,在食品工业和医疗保健等领域得到广泛开发与应用。
高通量测序技术的快速发展导致对益生菌的研究也上升到基因组学层面,越来越多的菌株完成了全基因组测序[5-6],这种方法在基因组层面上提供了对菌株功能的新认知。比较基因组学能够深入探究菌株的基因组及功能基因[7-8]。Mao Bingyong等[9]利用比较基因组学分析不同来源的L.plantarum基因组差异,发现L.plantarum特异基因数量与其所处的生态位有关。此外,还有研究发现L.plantarumZJ316具有胆盐抗性和黏附于宿主肠壁的相关基因,可在胃肠道存活并定植,维持肠道菌群平衡[10]。L.plantarumDMDL 9010存在降解亚硝酸盐的相关基因,为降低食品中的亚硝酸盐含量提供理论参考[11]。因此,利用比较基因组学对L.plantarum功能基因分析,对其功能开发与应用具有重要意义。
本研究所用L.plantarumST分离自中国云南省德昂酸茶,已完成对L.plantarumST的全基因组测序和基因组组装工作[12]。前期研究发现L.plantarumST对鼠伤寒沙门氏菌和大肠杆菌表现出较强的抗菌活性,在肠道中具有良好的胃肠液耐受能力、较强的黏附和自聚力,是1 株潜在的益生菌株[13]。本研究对L.plantarumST及NCBI已公开的152 株L.plantarum全基因组序列和1 株模式菌株ATCC 14197T基因组序列进行比较基因组学分析,从基因组水平全面分析该菌株的功能基因,同时利用API 50 CHL测定该菌株的碳水化合物利用情况,旨在为使其成为功能性食品的生产菌株或微生态制剂奠定理论基础。
1 材料与方法
1.1 材料与试剂
所用菌株L.plantarumST分离自中国云南德昂族传统食品德昂酸茶中,由云南农业大学提供,GenBank数据库生物保藏号为PRJNA792432。截至2021年10月10日,把NCBI(National Coalition Building Institute,https://www.ncbi.nl m.nih.gov/)Refseq数据库中的152 株L.plantarum基因组完成图全部下载,同时下载模式菌株L.plantarumATCC 14917T基因组完成图。
MRS培养基 广州环凯微生物科技有限公司;API 50 CHL碳水化合物鉴定试剂条 法国生物梅里埃生物公司。
1.2 仪器与设备
电热恒温培养箱 上海一恒科技有限公司;台式高速离心机 德国Eppendorf 公司。
1.3 方法
1.3.1 比较基因组分析
1.3.1.1 平均核苷酸一致性(average nucleotide identity,ANI)计算
154 株L.plantarum的ANI值计算以Goris等[14]方法为参照。再利用TBtools[14]软件绘制ANI聚类热图。
1.3.1.2 泛-核心基因集构建
菌株基因组的基因预测采用Prokka[15]软件,之后利用Roary[16]软件识别核心基因集与泛基因集,编码蛋白氨基酸相似性大于95%是识别核心基因的原则[8]。泛-核心基因趋势图绘制使用软件R(v.4.0.4)进行。
1.3.1.3 系统发育树构建
核心基因序列是通过Roray软件分析后获得,再使用TreeBest软件(http://www.mybiosoftw are.com/treebest)中的邻接法(Neighbor-Joining,NJ)对系统发育树进行构建,并下载NEW文件。系统发育树的可视化使用在线软件iTol完成(https://itol.embl.de/)[8]。
1.3.1.4 基因功能注释
利用子系统技术的快速标注(Rapid Annotation Using Subsystem Technology,RAST)(https://rast.nmpdr.org/)数据库、京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)网站对具有代表性的、已有相关益生特性研究报道的L.plantarum全基因组功能注释。
1.3.1.5 碳水化合物活性酶(carbohydrate-active enzymes,CAZy)注释
将L.plantarum基因组序列上传至在线注释dbCAN(http://bcb.unl.edu/dbCAN2/)进行注释,下载其注释文件。统计L.plantarum基因组信息。
1.3.2 毒力和耐药基因检索分析
将L.plantarum氨基酸序列上传至毒力因子数据库(VirulenceFinder,https://cge.cbs.dtu.dk/services/VirulenceFinder/)进行比对,标准为选择ID的阈值90%、最小长度60%。把L.plantarum氨基酸序列与耐药基因数据库(Comprehensive Antibiotic Research Database,CARD)(http://arpcard.mcmaster.ca)进行比对,耐药基因的确定为比对结果中E值<l×10-10、相似度>50%的蛋白序列。
1.3.3 特有基因和益生基因分析
利用Prokk软件对菌株基因组进行基因预测后,对菌株的特有基因及益生基因进行深入分析。
1.3.4 碳水化合物代谢特性分析
利用API 50CHL碳水化合物鉴定生化试剂条对L.plantarumST碳水化合物代谢情况进行表型研究,并按照试剂盒说明书操作[17]。24、48 h各观察一次并记录结果,以48 h观察到结果作为最终结果。
1.4 数据分析及绘图
利用Perl脚本统计基因组信息。利用TBtools绘制ANI聚类热图。
2 结果与分析
2.1 ANI分析与泛-核心基因构建
ANI是通过比对基因组的同源序列以鉴定菌株亲缘关系[18]。在比较基因组学分析过程中通常认为ANI值大于95%为同一物种,ANI可用于判断基因组间的相似性及评估基因组间多态性程度[19]。本研究对154 株L.plantarum菌株进行ANI计算,并构建聚类热图(图1A)。结果表明,全部菌株之间ANI值均大于98%,表明研究所用菌株均为同一物种即L.plantarum。其中,L.plantarumST和模式菌株L.plantarumATCC 14917T的ANI为99.05%.

图1 L.plantarum ANI值聚类热图(A)与泛基因集和核心 基因集曲线(B)Fig.1 Heatmap of ANI (A) and pan-core gene family curve (B)
通过Prokka和Roary软件进行分析比较菌株间的基因差别,统计核、泛基因个数用大于90%的氨基酸一致性为标准。154 株L.plantarum的泛基因集包括了17378 个基因,其中核心基因1262 个,特有基因6427 个。由图1B可知,随基因组数量的增加,泛基因个数依然表现出增加趋势,而核心基因个数逐渐趋于稳定。这一现象符合开放式基因组[20]的特点,所以推测L.plantarum为开放式基因组。
2.2 系统发育树分析
菌株间遗传进化关系和群体结构可以通过系统发育树反映[21]。为研究L.plantarumST与其余153 株L.plantarum种内遗传进化关系,采用NJ法,以Roary软件识别得到的1262 个核心基因构建系统发育树(图2),Bootstrap值为1000。

图2 基于核心基因序列的系统发育树Fig.2 Phylogenetic tree based on core gene sequences
154 株L.plantarum中包括76 株蔬菜与谷物分离源、14 株乳制品分离源、10 株肉制品分离源、22 株肠道分离源、4 株果蝇分离源及28 株其他分离源。系统发育树结果显示,154 株L.plantarum在进化过程中由于遗传多样性使其被分为两大分支,集中在1 个分支说明亲缘关系更接近。ST处于第II分支中的单独一小分支,与肠道分离株BCC9546遗传距离较近。乳制品分离株集中在第II分支的上半部分,肉制品分离株集中在下半部分,且存在一定的聚集性。可见,乳、肉制品分离株因来源不同而存在差异。此外,呈现明显聚集性有果蝇分离株,刘亚华等[22]探究发现果蝇分离株因为分离环境特别,它对环境的适应会获得有利于其在该环境下生存的环境特异性基因,从而导致其具有一定分离源特异性。
从系统发育树中分支中选取几株代表性、已有相关益生特性研究报道的菌株,作为对照,与L.plantarumST继续进行后续的比较基因组学分析。选取菌株为:JDM1、LIP-1、P8、ZJ316、ZS2058、WCSF1、ST-III、ATCC14197T、KP。
2.3 RAST注释结果
利用RAST 软件对10 株具有良好益生特性的L.plantarum基因组序列进行功能注释,分析ST与其他菌株功能差异。10 株L.plantarum共注释到25 个功能大类。由图3可知,10 株L.plantarum中参与碳水化合物代谢的基因数量最多(21.6%),其次是氨基酸及其衍生物合成与降解(14.62%)和蛋白质代谢(11.87%)相关基因,其中氮代谢(0.21%)相关功能编码基因仅在菌株JDM1、WCFS1、ZS2058中存在。L.plantarumST注释到24 个功能类别。
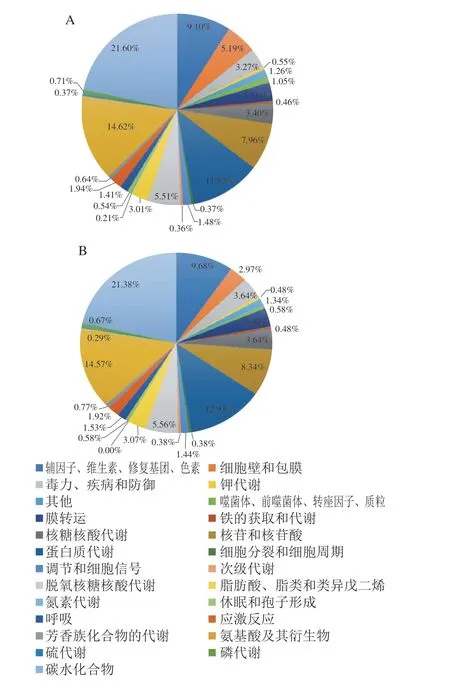
图3 L.plantarum RAST功能注释结果Fig.3 Function annotation of L.plantarum strains based on the RAST database
注释结果表明这些L.plantarum参与碳水化合物代谢的基因数量最多。碳水化合物代谢能力与基因组中存在多种编码、运输糖代谢的基因及相应的调控蛋白有关。因为L.plantarum可以广泛利用多种糖源,所以可以应对更多复杂的环境[23]。很多磷酸转移酶系统(phosphotransferase system,PTS)转运系统存在于L.plantarum中,为机体输送糖类物质是该系统的作用[24]。RAST注释结果显示WCFS1中共注释到57 个PTS相关功能基因,ST共注释到49 个。已有研究报道,L.plantarumWCFS1株包括完整的PTS酶II复合物以及运输系统[25],与WCFS1相比,本研究发现ST也有相同的糖类物质运送系统,从而确保菌株对糖的利用。
2.4 KEGG注释结果
L.plantarumST与另外9 株L.plantarum的氨基酸序列,上传至KEGG数据库进行比对得到注释结果。10 株L.plantarum在KEGG数据库中分别在新陈代谢、细胞过程、遗传信息处理、环境信息处理、生物系统和人类疾病6 大功能37 个通路上得到功能注释,结果如图4所示。
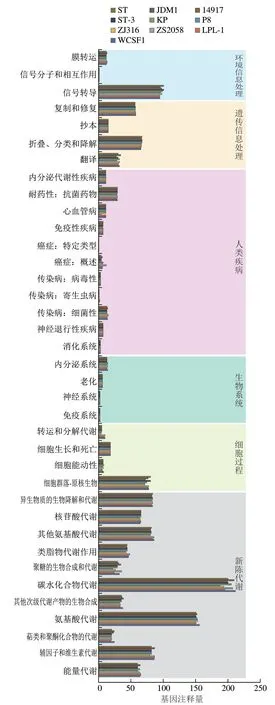
图4 L.plantarum KEGG数据库注释结果Fig.4 KEGG annotation of L.plantarum
分析发现所有菌株的基因功能注释主要分布于代谢通路中,碳水化合物代谢是11 个代谢通路中基因数量最多的,共2008 个基因占代谢通路注释基因的23.2%。氨基酸代谢次之。环境信息处理中最多注释的是信号转导和膜运输,其中信号转导通路主要集中于ABC转运蛋白。多种糖类的代谢通路包含在碳水化合物代谢通路中,L.plantarumST中碳水化合物代谢多达200 个基因注释量,推测L.plantarumST可能有较强的糖类分解及代谢功能,为机体提供能量[6]。
免疫相关基因的通路信息及其通路ID如表1所示。可调控免疫和炎症的通路相关基因,包括过氧化物酶体增生物激活受体(peroxisome proliferator activated receptor,PPAR)和维甲酸诱导基因I(retinoic acid-induced gene I,RIG-I)通路。其中,STGL000154、STGL002738参与PPAR信号通路的调控,PPAR通路与细胞分化、炎性反应和能量代谢等紧密相连。在RIG-I样受体信号通路的调控中基因STGL000333参与,RIG-I样受体是固有免疫的模式识别受体,这个受体可以辨别非自身的病毒RNA,通过激活该受体信号通路最终达到促进细胞因子生成的目的,从而发挥出抗病毒的功效[26],这可 能是L.plantarum具有抗病毒作用的因素。推测L.plantarumST能够帮助宿主抵抗病原菌的侵袭、进而调节肠道菌群的平衡。
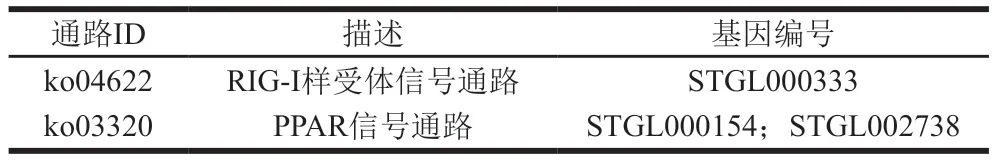
表1 L.plantarum ST基因组免疫调控通路相关基因Table 1 Genes related to genomic immune regulatory pathway in L.plantarum ST
2.5 CAZy注释结果
为了从基因组层面探究L.plantarumST对碳水化合物的利用能力,利用CAZy数据库分析了10 株L.plantarum的CAZy功能基因。5 种CAZy类在这10 株L.plantarum中被注释到,分别为碳水化合物碳结合结构域(carbohydrate-binding modules,CBMs)、糖苷水解酶类(glycoside hydrolases,GHs)、辅模块酶类(auxiliary activities,AAs)、糖苷转移酶类(glycosyl transferases,GTs)、碳水化合物酯酶类(carbohydrate esterases,CEs)。10 株L.plantarum共注释到47 个CAZy基因家族,包括24 个GHs家族、12 个GTs家族、4 个CEs家族、4 个CBMs家族和3 个AAs家族。
由图5可知,ST及另外9 株菌注释到的GHs、GTs含量最为丰富,无显著差异。GHs可水解或重排糖苷键,将多糖水解为单糖,给予菌体能量[27]。GTs催化糖基与其他糖类或非糖物质的基团进行转移,负责糖苷键的形成[28]。其中,GT4在L.plantarum中拷贝数最多(平均约13 个),其次是GT2(平均约11 个)和GH1(平均约10 个)。本研究10 株L.plantarum注释到了24 个GHs,含量丰富。ST的GH家族中含量最多的是GH1、GH13_31,还包括GH170、GH25、GH65、GH73、GH78、GH36等。在纤维素降解的过程中糖苷水解酶类中一些酶类有水解作用,利用关键限速酶β-葡萄糖苷酶能把纤维二糖在内的纤维寡糖分解为单糖[4],故推测L.plantarumST具有良好的降解纤维素潜力。所有L.plantarumGT家族中GT2、GT4含量最多,主要编码蔗糖合酶、纤维素合酶等众多酶类,与蔗糖等二糖以及脂多糖、纤维素等合成有关。CBM家族能够促进催化活性结构域与碳水化合物的结合。由图可知注释到CBM32、CBM34、CBM48,其中CBM 48主要来自糖苷水解酶,多为普鲁兰酶、异淀粉酶、分支酶等。CBM的多样性使其在底物特异识别、促进酶与底物结合及酶稳定性等方面均有重要作用[29]。

图5 L.plantarum CAZy数据库注释结果Fig.5 Annotation results of L.plantarum based on CAZy database
通过碳水化合物注释结果推测L.plantarumST有着较强的水解或重排糖苷键能力,同时可能具有良好的降解纤维素潜力。良好糖苷水解能力为L.plantarumST用于益生菌制品奠定了基础。
2.6 毒力和耐药基因检索分析
安全性评价是判断一株菌是否为益生菌最为关键的一步,一般通过研究菌株的抗生素抗性、有毒代谢物等指标评估乳酸菌的安全性[30]。抗生素滥用严重导致细菌产生耐药性。将L.plantarumST与其他9 株L.plantarum的基因组经毒力因子、耐药基因数据库比对后发现均不存在毒力因子和耐药基因,故认为L.plantarumST是安全的,并且没有潜在的耐药性。
2.7 特有基因和益生基因分析
为进一步挖掘L.plantarumST功能基因同时解析其潜在的益生功能基因,继续对菌株的特有基因和益生基因进行分析。L.plantarumST存在41 个特有基因,除去一些假定蛋白编码基因后,L.plantarumST有3 个特有功能基因,表2为特有功能基因信息,分别为乙二醛还原酶基因(yvgN)、酪氨酸重组酶基因(xerC)、能量耦合因子转运蛋白基因(ecfT)。yvgN是一种加氢酶,对乙二醛进行加氢反应。ecfT是一种转运酶,负责摄入一些维生素及其他微量元素[31]。

表2 154 株L.plantarum中L.plantarum ST特有功能基因Table 2 Unique genes of L.plantarum ST
基于前期对L.plantarumST益生功能的研究[13],使用Prokka软件注释到L.plantarumST含有与益生特性相关的基因,具体信息见表3。群体感应AI-2信号分子合成的最关键蛋白酶是S-核糖同型半胱氨酸裂解酶(LuxS),AI-2 在微生物种内和种间交流起到重要作用,在L.plantarumST中发现了该基因。目前发现在超过80 种细菌中均存在LuxS基因[32-33],细菌许多生理现象离不开LuxS介导的群体感应系统,因为这种系统对细菌有重要的调节作用[34]。L.plantarumST还含有谷胱甘肽合成(garB、gshAB)、核黄素合成(ribBA、ribE、ribF)相关基因,有研究表明[35]谷胱甘肽不仅有助于维持正常的免疫系统功能,同时有整合解毒与抗氧化作用。此外,菌株在胃肠道存活率的增加也与谷胱甘肽有关[36],该物质有助于菌株益生功能的发挥。核黄素(VB2),作为辅酶有助于代谢是其主要功能。NAPPH还原酶类(nfrA),参与脂肪酸、核苷酸和脂类的合成等多种合成代谢反应,该物质起到递氢体的作用在生物体内的化学反应中[37]。L.plantarumST还注释到了黏附因子(atpC)基因,黏附因子有助于细菌在宿主体内的定植因为可使菌株牢固黏附于宿主细胞表面。研究还发现,L.plantarumST存在提高宿主代谢能力(tagE)、乳酸和转运乳酸(pyk、ldh)、细胞壁特殊组分成分(ItaS)和生物活性肽(lmrA)产生相关的基因,可提高菌株益生功能。

表3 L.plantarum ST具有的益生特性相关基因Table 3 Genes related to probiotic characteristics of L.plantarum ST
特有基因和益生基因分析发现,L.plantarumST含有与黏附能力、信号分子传递和抗氧化能力等相关的益生功能基因。因此,本研究从基因组水平揭示L.plantarumST具有益生特性相关基因,认为其是1 株具有潜在益生功能的菌株。
2.8 碳水化合物代谢特性
为了评估L.plantarumST对不同碳源的利用能力,利用API 50 CHL碳水化合物鉴定试剂条对ST的碳水化合物代谢进行研究,挖掘出可被ST利用的碳源,为碳水化合物代谢中的功能基因分析提供表型数据支持,同时为该菌株在之后的应用提供借鉴。
如表4所示,API 50 CHL碳水化合物鉴定试剂条所涉及的49 种碳水化合物中,可被ST利用的共有28 种,划分为单糖类(9)、糖苷类和二糖类(12)、多糖类(1)、醇类(4)、盐类(2)物质五大类。单糖类包括:L-阿拉伯糖、D-核糖、D-半乳糖、D-葡萄糖、D-甘露糖、L-鼠李糖、甲基-α-D-吡喃葡萄糖苷、N-乙酰葡萄糖胺;糖苷类和二糖类包括:熊果苷、水杨苷、苦杏仁苷、七叶灵、D-纤维二糖、D-麦芽糖、D-乳糖、D-蜜二糖、D-蔗糖、D-海藻糖、D-龙胆糖、D-松二糖;多糖类包括:D-松三糖;醇类包括:丙三醇、肌醇、甘露醇、山梨醇;盐类包括:葡萄糖酸钾、5-酮基葡萄糖酸钾。

表4 L.plantarum ST API 50 CHL分析Table 4 Analysis of API 50 CHL in L.plantarum ST
糖发酵实验结果显示,L.plantarumST可代谢不同种类丰富的碳源,包括9 种单糖、12 种糖苷和二糖、1 种多糖及4 种醇类和2 种盐类物质,再次说明了ST具有较强的碳水化合物代谢能力。该结果与本实验KEGG注释以及CAZy分析吻合,利用基因组学和表型分析相结合,发现L.plantarumST拥有较好的产CAZy能力,具备丰富的碳水化合物利用能力,可为开发相关酶制剂提供一定的参考价值。
3 结论
对L.plantarumST结合NCBI已公开的152 株L.plantarum全基因组序列和1 株模式菌株ATCC 14197T的基因组序列进行比较基因组学分析。ANI分析鉴定ST为L.plantarum,系统发育树分析发现L.plantarumST与肠道分离株BCC9546遗传距离较近,乳、肉制品分离株因来源不同而存在差异,分别集中在第II分支的上半部分和下半部分,同时果蝇分离株有明显聚集趋势。RAST和KEGG功能注释发现,L.plantarumST参与碳水化合物代谢的基因数量最多,包含大量的PTS相关基因,还发现PPAR和RIG-1免疫调控通路相关基因。CAZy注释发现,L.plantarumST有较多水解或重排糖苷键的基因,可能具有良好的纤维素降解能力。毒力和耐药注释比对后发现ST不存在毒力因子和耐药基因。特有基因分析发现,L.plantarumST携带特有的与能量转运功能有关功能基因ecfT;益生特性基因显示,L.plantarumST具有与群体感应信号分子AI-2(LuxS)、黏附分子(atpC)和谷胱甘肽(garB、gshAB)合成相关的功能基因。API 50 CHL碳水化合物代谢结果显示,L.plantarumST可代谢单糖类、糖苷和二糖类及多糖类等中的29 种碳源,ST能够利用的碳源非常丰富。综上,本研究从基因组水平分析了L.plantarumST功能基因组特征,结合糖发酵表型结果,认为L.plantarumST是1 株安全且具有潜在益生特性基因的益生菌,为后续L.plantarumST益生功能开发及其生产应用奠定了遗传学基础。
