香石竹斑驳病毒属简并引物RT-PCR检测方法的建立
2023-07-09廖富荣陈红运沈建国方志鹏黄蓬英陈青林玲玲洪钧
廖富荣,陈红运,沈建国,方志鹏,黄蓬英,陈青,林玲玲,洪钧
香石竹斑驳病毒属简并引物RT-PCR检测方法的建立
廖富荣1,陈红运1,沈建国2,方志鹏1,黄蓬英1,陈青1,林玲玲1,洪钧1
1厦门海关技术中心,福建厦门 361026;2福州海关技术中心,福州 350001
【目的】使用简并引物RT-PCR方法并结合序列测定与分析,为甲型香石竹斑驳病毒属()、乙型香石竹斑驳病毒属()和丙型香石竹斑驳病毒属()病毒建立一种快速、灵敏、广谱的检测鉴定方法。【方法】通过基因组序列的多重比对分析,在依赖于RNA的RNA聚合酶(RdRp)基因的保守区域设计1对简并引物Carmo-F2/Carmo-R2,在引物5′端分别加入非互补的富含AT序列(AATAAATCATAA)形成引物Carmo-F2a/Carmo-R2a,测试简并引物RT-PCR方法的广谱性、特异性和灵敏度,并对PCR产物进行测序、序列分析和系统发育分析。利用该方法对来自厦门的扶桑()样品进行病毒检测。【结果】利用Carmo-F2/Carmo-R2和Carmo-F2a/Carmo-R2a两对引物建立的RT-PCR方法,扩增甲型、乙型和丙型香石竹斑驳病毒属病毒的部分RdRp基因,扩增片段分别约为500和550 bp。该方法成功用于检测甲型香石竹斑驳病毒属的香彩雀花碎色病毒(angelonia flower break virus,AnFBV)、小花矮牵牛斑驳病毒(calibrachoa mottle virus,CbMV)、香石竹斑驳病毒(carnation mottle virus,CarMV)、天竺葵花碎色病毒(pelargonium flower break virus,PFBV),乙型香石竹斑驳病毒属的木槿褪绿环斑病毒(hibiscus chlorotic ringspot virus,HCRSV),丙型香石竹斑驳病毒属的甜瓜坏死斑点病毒(melon necrotic spot virus,MNSV),显示出良好的广谱性。特异性检测表明,简并引物Carmo-F2/Carmo-R2和Carmo-F2a/Carmo-R2a在玉米褪绿斑驳病毒(maize chlorotic mottle virus,MCMV;)、天竺葵线纹病毒(pelargonium line pattern virus,PLPV;)、香石竹环斑病毒(carnation ringspot virus,CRSV;),健康的西瓜、甜瓜、南瓜、大豆、豌豆植物未扩增到预期大小的条带。灵敏度检测表明,简并引物Carmo-F2/Carmo-R2最低可检测到10-2稀释液,简并引物Carmo-F2a/Carmo-R2a可检测到10-3稀释液,在5′端加入非互补的富含AT序列可以提高简并引物RT-PCR检测方法的灵敏度。BLAST分析表明,测定的序列均与相应病毒种类具有最高序列一致性。部分RdRp基因氨基酸序列的系统发育分析结果符合目前通读病毒亚科()病毒的分类情况,且可以把病毒鉴定到种的水平。采集13份疑似病毒症状的扶桑样品,均检出木槿褪绿环斑病毒。【结论】基于简并引物Carmo-F2/Carmo-R2和Carmo-F2a/Carmo-R2a的RT-PCR方法,可用于甲型、乙型和丙型香石竹斑驳病毒属病毒的筛查检测,结合序列分析及系统发育分析可进行病毒种类的快速鉴定,并有助于发现新的病毒种类。厦门扶桑植物受到木槿褪绿环斑病毒的侵染。
甲型香石竹斑驳病毒属;乙型香石竹斑驳病毒属;丙型香石竹斑驳病毒属;简并引物;RT-PCR;扶桑;木槿褪绿环斑病毒
0 引言
【研究意义】香石竹斑驳病毒属()又称麝香石竹斑驳病毒属、康乃馨斑点病毒属,为番茄丛矮病毒科()最大的一个病毒属(共有8个病毒属),根据国际病毒分类委员会(ICTV)第九次分类报告,该属有16种确定种(而后增至19种)、11种暂定种,香石竹斑驳病毒(carnation mottle virus,CarMV)为代表种[1]。在我国仅发现CarMV[2]、甜瓜坏死斑点病毒(melon necrotic spot virus,MNSV)[3]、木槿褪绿环斑病毒(hibiscus chlorotic ringspot virus,HCRSV)[4]等病毒,多数病毒还未在我国发生,具有潜在的检疫重要性。因此,建立快速、灵敏的广谱检测方法,对于保护我国农业生产安全和环境生态安全、缩短口岸检疫部门的检测周期均具有重要意义。【前人研究进展】目前,番茄丛矮病毒科的分类已经进行较大调整,由原来的8个病毒属重新划分为3个亚科(、、)、18个病毒属,共96种病毒[5-6]。香石竹斑驳病毒属成员及其暂定种先后被划分为5个病毒属,包括牛膝菊病毒属()、天竺葵线纹病毒属()、甲型香石竹斑驳病毒属()、乙型香石竹斑驳病毒属()和丙型香石竹斑驳病毒属()[7-9]。香石竹斑驳病毒属病毒粒体为等轴对称二十面体,直径约32—35 nm,无包膜,粒体呈圆形,表面粗糙[1,10]。基因组为单分子线形正义ssRNA,长3 879—4 450 nt[1,6]。单分体基因组,含有4个开放阅读框(ORF),RNA的3′末端无Poly(A)结构,5′端可能有一个甲基化的核苷酸帽子结构[1]。该属多数病毒的自然寄主范围较窄,但实验寄主范围较宽。如CarMV自然侵染石竹科植物,但在接种的98种植物中可侵染其中的15科43种植物[11]。该属病毒极易通过机械传播,一些病毒可以通过种子或昆虫种类传播,以及土壤中的油壶菌传播[12-13]。如MNSV不仅可以通过机械传播、还可以通过甜瓜种子[14-15]、黄瓜叶甲(和)[16]、油壶菌(和)[17-18]传播。基于PCR技术的分子检测方法已被广泛应用于植物病毒的检测中。在该属病毒中碎米荠褪绿斑点病毒(cardamine chlorotic fleck virus,CCFV)[19]、CarMV[20-21]、豇豆斑驳病毒(cowpea mottle virus,CPMoV)[13]、HCRSV[22]、日本鸢尾坏死环斑病毒(Japanese iris necrotic ring virus,JINRV)[23]、MNSV[24]、PFBV[25]等均有相应的RT-PCR检测方法。这些特异性RT-PCR检测方法为该属提供了一种快速的分子检测方法,但一次只能用于检测一种特定的病毒。基于简并引物的通用RT-PCR检测方法则可实现多目标检测,即在一次实验中可以检测一个属、科中不同种类的病毒,如双生病毒[26]、马铃薯Y病毒科[27]、马铃薯X病毒属[28]。针对香石竹斑驳病毒属及其相关病毒,Morozov等[29]据依赖于RNA的RNA聚合酶(RNA-dependent RNA polymerase,RdRp)保守区域设计简并引物Carmo-II/Carmo-VI,成功用于检测玉米褪绿斑驳病毒(maize chlorotic mottle virus,MCMV)、TCV、红三叶草坏死花叶病毒(red clover necrotic mosaic virus,RCNMV)、香石竹环斑病毒(carnation ringspot virus,CRSV)、番茄丛矮病毒(tomato bushy stunt virus,TBSV)、PFBV。杨雷亮等[30]根据外壳蛋白基因的保守区域设计了引物CarMV-F1/CarMV-F2,成功检测了8种病毒。此外,美国Agdia公司也有商品化的检测引物在出售,并通过16种病毒的实验验证[31]。【本研究切入点】近年来,随着研究的不断深入,对原香石竹斑驳病毒属病毒的分类进行了较大调整,分类归属趋于完善,并且不断有新的病毒种类被发现。在原属的16种病毒中已有14种病毒的基因组序列均被测定,原暂定种中也已测定6种病毒的基因组序列。因此,根据新的分类情况,研究建立甲型、乙型、丙型香石竹斑驳病毒属病毒的RT-PCR检测方法,可以为该类病毒提供一种快速、灵敏、特异的检测方法,进一步提高检测效率、缩短检测周期、节约检测成本。【拟解决的关键问题】通过病毒基因组序列比对分析、根据保守区域设计简并引物,研究建立甲型、乙型、丙型香石竹斑驳病毒属的广谱RT-PCR检测方法,实现一次试验检测不同种类的病毒;同时,经序列测定、比对及系统发育分析,实现病毒种类的快速鉴定。
1 材料与方法
试验于2021—2022年在厦门海关技术中心植物检疫实验室完成。
1.1 试验材料
供试病毒分离物样品来自各商业公司(表1),健康植物样品来自本实验室,扶桑病叶(褪绿、黄化、斑驳、卷叶等症状)采自厦门湖里区(XM1—XM3)、海沧区(XM4—XM9)、思明区(XM10—XM13)。
1.2 引物设计与合成
从NCBI中下载甲型、乙型、丙型香石竹斑驳病毒属病毒完整的基因组序列(表2),用BioEdit中的ClustalW进行序列多重比对[32-33],根据保守性区域设计简并引物,包括上游引物Carmo-F2和下游引物Carmo-R2,以及引物Carmo-F2a/R2a,由生工生物工程(上海)股份有限公司合成(表3)。
1.3 样品前处理
1.3.1 样品提取缓冲液 样品提取缓冲液为ELISA常规使用的缓冲液。1 L缓冲液中含NaCl 8.0 g、KH2PO40.2 g、Na2HPO41.15 g、KCl 0.2 g、聚乙烯吡咯烷酮((C6H9NO)n,MW 24 000—40 000)20 g,pH 7.4。
1.3.2 样品前处理 冻干粉末:加入1 mL样品提取缓冲液,混匀,取200 μL上清液用于核酸提取。
病叶或其他组织:取50—100 mg病叶或茎秆,用研钵研磨;或放入2 mL离心管中并加入两粒直径4 mm的不锈钢珠,用组织研磨仪研磨(MM400,Retsch GmbH),在振荡频率18次/s下运行1 min。去除不锈钢珠,加入1 mL的样品提取缓冲液,混匀,8 000×离心2 min(或4 ℃冰箱中静置过夜),取200 μL上清液用于核酸提取。
图4为控制系统的主电路,主电路主要包括:总电源回路、主油泵电动机电源回路、控制器电源回路、直流电源回路和伺服电源回路。
1.4 核酸提取
用E.Z.N.A.TM植物RNA提取盒(R6827,Omega Bio-Tek)提取总核酸,略有删减。即:取200 μL的样品上清液,省略去基因组DNA步骤后,根据操作说明提取总核酸,最后分两次加入50 μL经65 ℃预热的DEPC处理水,在10 000×下洗脱核酸。
1.5 RT-PCR方法
采用上海普洛麦格生物产品有限公司的反转录预混液Eastep®RT Master Mix(LS2052)进行反转录。在0.6 mL离心管中加入5×RT Mix 4 μL、RNA模板16 μL,总体积20 μL。反应条件:37 ℃反应30 min(如需要可延长至1 h)、98 ℃灭活5 min。

表1 本研究使用的病毒分离物
*中国检验检疫科学研究院 Chinese Academy of Inspection and Quarantine
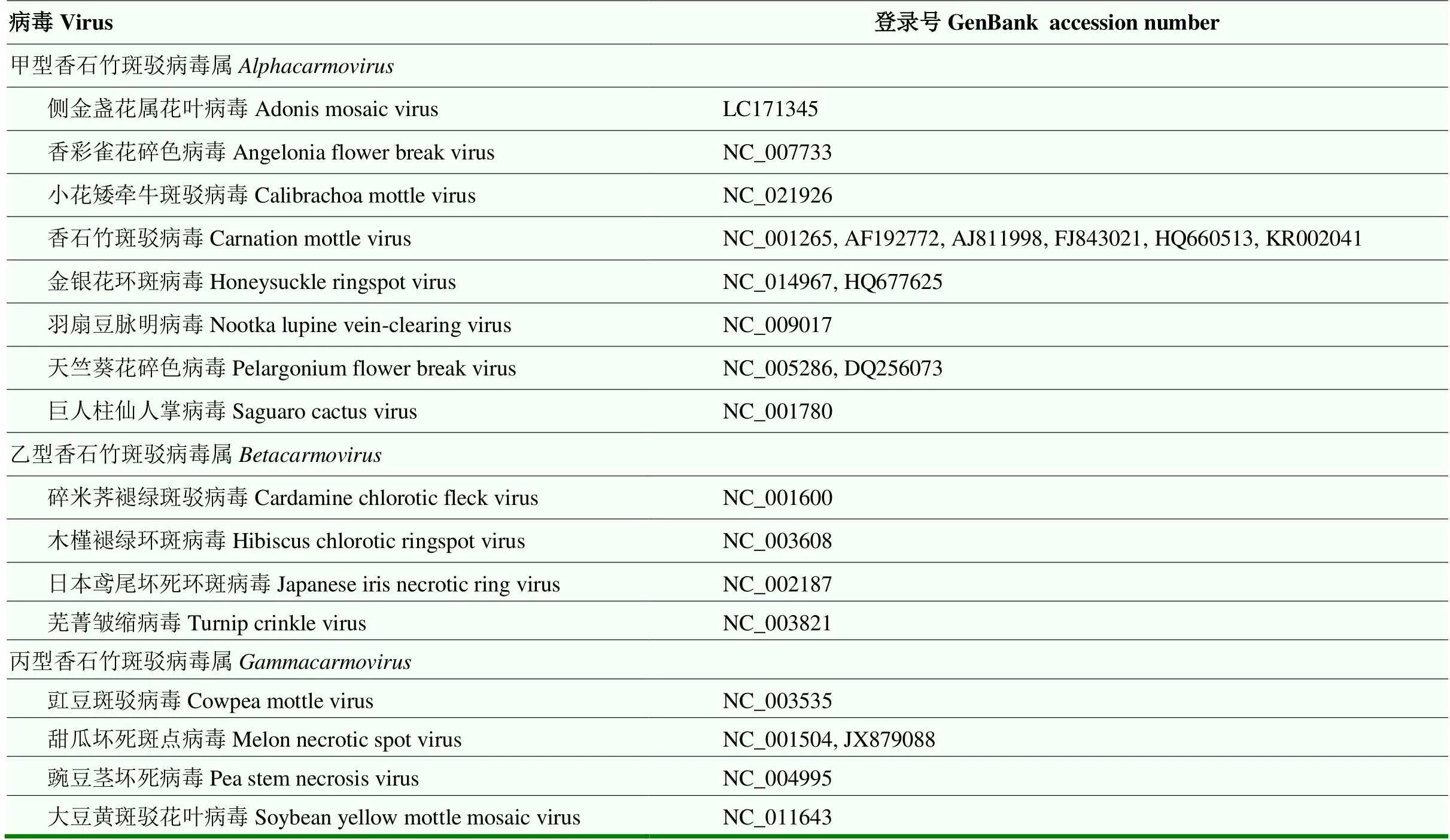
表2 用于引物设计的病毒基因组序列

表3 根据甲型、乙型、丙型香石竹斑驳病毒属病毒基因组序列保守区域设计的简并引物
* R (A,G), Y (C,T), M (A,C), K (G,T), S (G,C) , W (A,T), H (A,T,C), B (G,T,C), V (G,A,C), D (G,A,T), N (A,T,G,C);下划线为非互补的富含AT序列Underlines are the non-complementary 5′ AT-rich sequences
采用北京全式金生物技术有限公司的Easy Taq DNA聚合酶(AP111)进行PCR扩增。10×PCR缓冲液5 μL、dNTP(10 mmol·L-1)1 μL、Taq DNA聚合酶(5 U·μL-1)0.6 μL、上游引物(10 μmol·L-1)4 μL、下游引物(10 μmol·L-1)4 μL、cDNA模板3 μL、无核酸酶水32.4 μL,总体积50 μL。
PCR反应程序:95 ℃预变性3 min;接着94 ℃变性30 s,42 ℃退火30 s,72 ℃延伸50 s,5个循环;然后94 ℃变性30 s,50 ℃退火30 s,72 ℃延伸50 s,35个循环;最后72 ℃延伸7 min。
取5 µL扩增产物在2.0%的琼脂糖凝胶上进行电泳,然后在凝胶成像仪上观察、拍照。
1.6 一步RT-PCR方法
利用南京诺唯赞生物科技有限公司的HiScript®II One Step RT-PCR Kit(P612)进行一步RT-PCR扩增,在PCR管中配制反应液。2×One Step Mix 25 μL、One Step Enzyme Mix 2.5 μL、上游引物(10 μmol·L-1)4 μL、下游引物(10 μmol·L-1)4 μL、RNA模板3 μL、无核酸酶水11.5 μL,总体积50 μL。
反应条件:50 ℃反转录30 min,95 ℃预变性3 min;94 ℃变性30 s,42 ℃退火30 s,72 ℃延伸50 s,5个循环;94 ℃变性30 s,50 ℃退火30 s,72 ℃延伸50 s,35个循环;72 ℃延伸7 min。
1.7 特异性检测
分别提取健康寄主植物(西瓜、甜瓜、南瓜、大豆、豌豆)叶片及其他相关病毒(MCMV、CRSV、PLPV)病叶总RNA,利用所建立的RT-PCR方法进行检测,验证方法的特异性。
1.8 灵敏度检测
把含有HCRSV的总RNA进行10倍系列稀释(即10-1—10-6),然后用建立的方法进行RT-PCR扩增,以检测其灵敏度。
1.9 序列测定与分析
PCR产物由生工生物工程(上海)股份有限公司进行正反向测序。序列拼接后,用BLAST、MEGA X、PhyloSuite等软件进行序列分析和系统发育分析[34-36]。其中,用MAFFT进行多重序列比对分析[37],最大似然法(maximum likelihood,ML)构建系统发育树,自举检验(bootstrap)1 000次[38]。
2 结果
2.1 简并引物设计
通过对16种甲型、乙型、丙型香石竹斑驳病毒属病毒的基因组序列比对分析,根据RdRp基因的保守区域设计上游引物Carmo-F2和下游引物Carmo-R2(图1、表3)。其中,引物Carmo-F2a/R2a为在引物Carmo-F2/R2的5′端分别加入一段非互补的富含AT序列后组成的新引物。这两对引物用于扩增RdRp部分序列,预计扩增长度分别约为500和550 bp。序列比对分析表明,本研究所设计的简并引物Carmo-F2/R2与Morozov等设计的简并引物Carmo-II/VI[29]都是根据两个相同的保守区域设计的、扩增同一区域的RdRp基因,但简并引物的长短及个别碱基上存在差别。通过序列比对分析,发现文献报道的简并引物Carmo-II在中间位置缺少1个碱基,这可能是早期排版错误造成的。根据序列比对结果,对原引物进行修正,形成引物Carmo-II2/VI2(表4)。
2.2 简并引物的广谱性检测
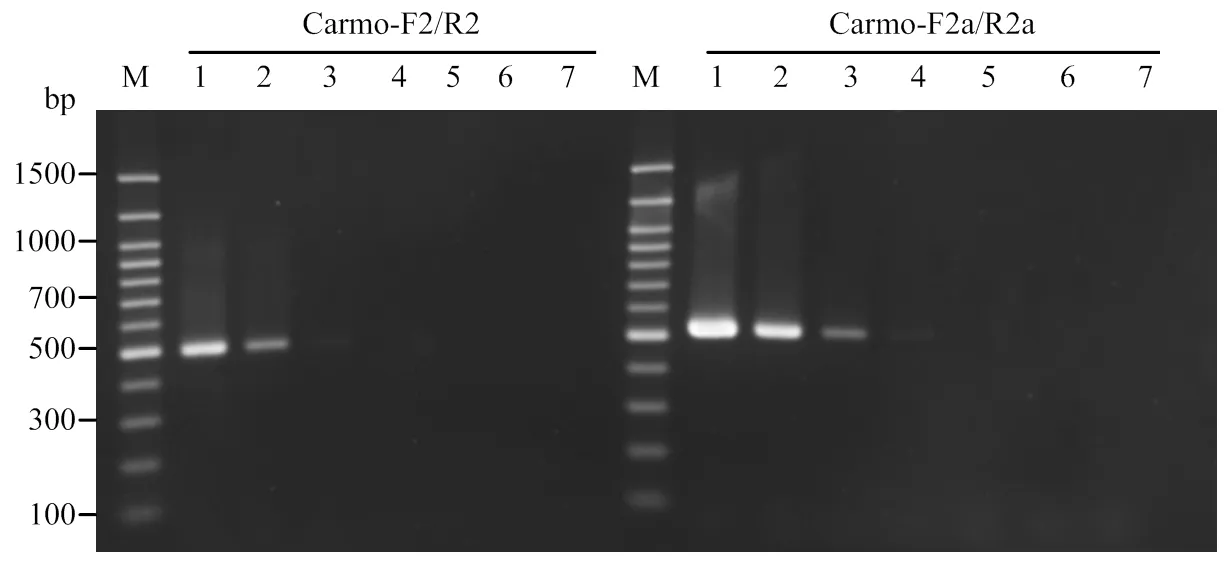
利用简并引物Carmo-F2/R2对7种不同种类的香石竹斑驳病毒属病毒进行RT-PCR扩增。结果表明,7种病毒的11个分离物中,AnFBV的LPC36600(Agdia)、CbMV的LPC83000(Agdia)、HCRSV的LPC12300(Agdia)、PFBV的LPC90000(Agdia)、CbMV的07008PC(Loewe)、PFBV的07073PC(Loewe)、MNSV的07097PC(Loewe)等6种病毒的8个分离物可以扩增到约500 bp的预期大小条带(图2-A)。其中,CarMV的LPC68000(Agdia)分离物还产生约550 bp的非预期大小条带,而07057PC分离物(Loewe)产生的预期大小条带则很弱。PLPV的07094PC(Loewe)可以产生一条约600 bp的条带,但重复性差。
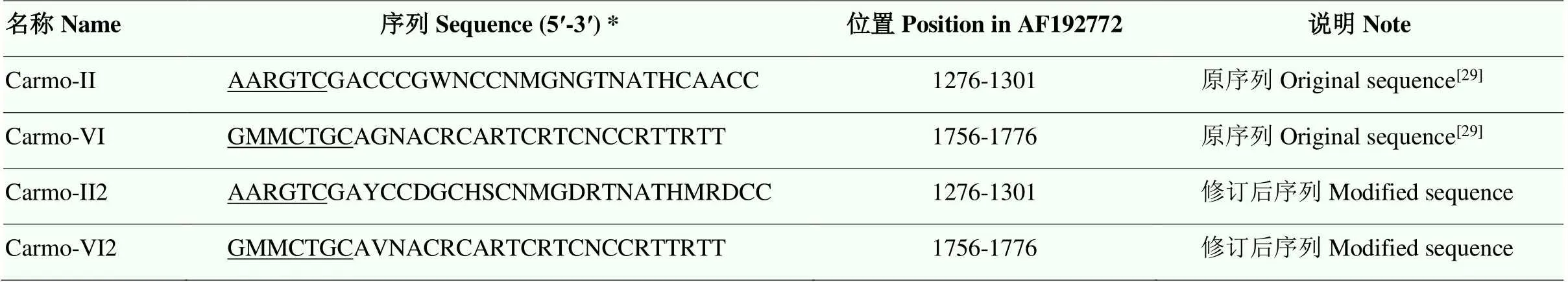
表4 香石竹斑驳病毒属相关病毒的简并引物
* R (A,G), Y (C,T), M (A,C), K (G,T), S (G,C) , W (A,T), H (A,T,C), B (G,T,C), V (G,A,C), D (G,A,T), N (A,T,G,C);下划线为I和I的位置Underlines are the sites ofI andI

图1 用于设计简并引物的香石竹斑驳病毒属保守区域
引物Carmo-F2a/R2a的核心序列与引物Carmo-F2/R2相同,区别在于引物的5′端分别加入一段非互补的富含AT的序列。RT-PCR结果表明,在6种病毒的8个分离物可以扩增到约550 bp的预期大小条带,CarMV的LPC68000(Agdia)分离物还产生一条约400 bp的非预期大小条带,而CarMV的07057PC(Loewe)和PLPV的07094PC(Loewe)分离物产生的条带很弱(图2-B)。该结果与简并引物Carmo-F2/R2的RT-PCR结果类似。
利用一步法试剂进行一步RT-PCR扩增,结果表明也可以扩增到约500 bp的预期条带,但在常规RT-PCR条带比较弱的样品中没有扩增到预期大小条带(图2-C、2-D)。在常规RT-PCR扩增中,CbMV的LPC83000(Agdia)和07008PC(Loewe)分离物均扩增到约400 bp的非特异性条带,而在一步RT-PCR中没有该非特异性条带。因此,该结果初步表明,本研究所使用的一步RT-PCR试剂的检测灵敏度可能比较低,但具有更好的特异性。
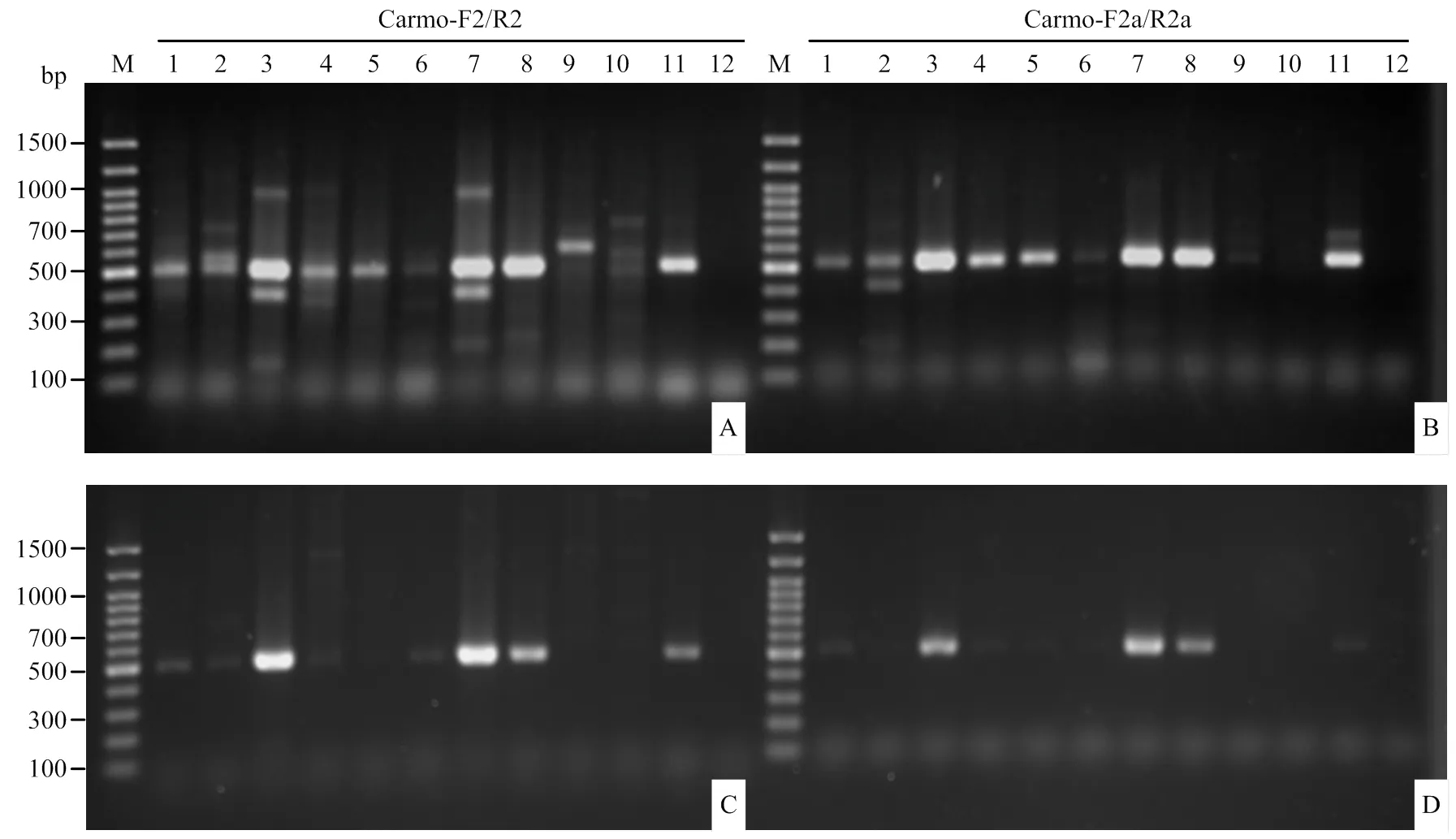
M:100 bp DNA Ladder;1:AnFBV (Agdia LPC36600);2:CarMV (Agdia LPC68000);3:CbMV (Agdia LPC83000);4:HCRSV (Agdia LPC12300);5:PFBV (Agdia LPC90000);6:CarMV (Loewe 07057PC);7:CbMV (Loewe 07008PC);8:PFBV (Loewe 07073PC);9:PLPV (Loewe 07094PC);10:MNSV (Agdia LPC12402);11:MNSV (Loewe 07097PC);12:空白对照Blank control
2.3 特异性检测
利用所建立的方法对健康寄主植物叶片及其他相关病毒进行RT-PCR检测,结果表明,简并引物Carmo-F2/R2及Carmo-F2a/R2a均不能从健康的西瓜、甜瓜、南瓜、大豆、豌豆扩增到与阳性样品大小一致的条带(图3)。此外,在含有MCMV、CRSV、PLPV的阳性样品中,也没有扩增到预期大小条带(部分结果未显示)。因此,该方法具有良好的特异性,在寄主植物中及同科的其他病毒中均未产生非特异性扩增。
M:100 bp DNA Ladder;1:西瓜Watermelon;2:甜瓜melon;3:南瓜Squash;4:大豆Soybean;5:豌豆Pea;6、8:HCRSV阳性样品HCRSV positive sample;7、12:空白对照Blank control;9:MCMV;10:CRSV;11:PLPV
2.4 灵敏度检测
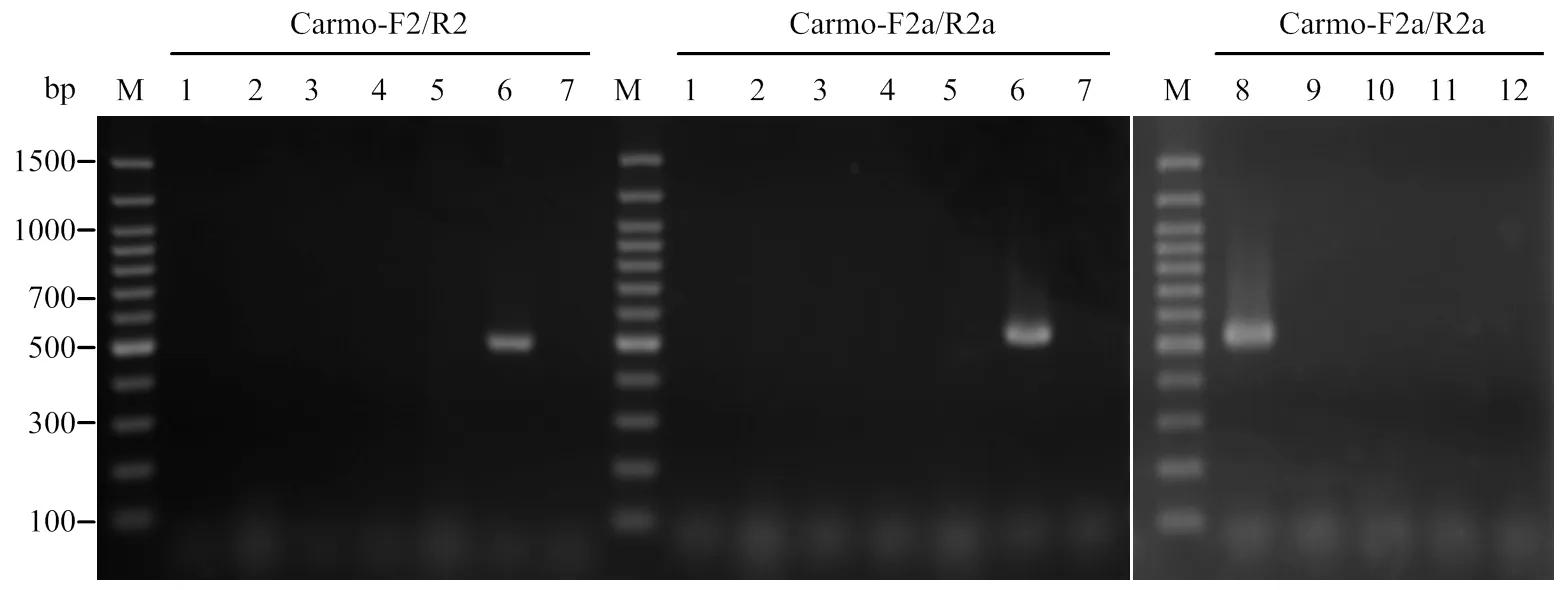
把含有HCRSV的病叶总RNA进行10倍系列稀释,然后用Carmo-F2/R2和Carmo-F2a/R2a分别进行常规RT-PCR和一步RT-PCR检测。常规RT-PCR检测结果表明,当RNA稀释至1 000倍时,简并引物Carmo-F2/R2扩增到条带已经很弱,很难看到明显的条带(约500 bp),而简并引物Carmo-F2a/R2a可以明显扩增到预期大小条带(约550 bp)(图4)。一步RT-PCR检测结果表明,简并引物Carmo-F2/R2和Carmo-F2a/R2a均可从稀释100倍的病叶中扩增到预期大小条带,但稀释至1 000倍时,简并引物Carmo-F2/R2已经基本看不到条带,引物Carmo-F2a/R2a扩增到的条带也比较弱(图5)。因此,无论是一步RT-PCR还是两步RT-PCR,简并引物Carmo-F2/R2和Carmo-F2a/R2a均有良好的灵敏度,可以满足日常检测的需要。在简并引物中引入一段富含AT的序列后,可以提高简并引物RT-PCR方法的检测灵敏度。
M:100 bp DNA Ladder;1—6:10-1—10-6系列稀释serial dilutions;7:空白对照Blank control。图5同The same as Fig. 5
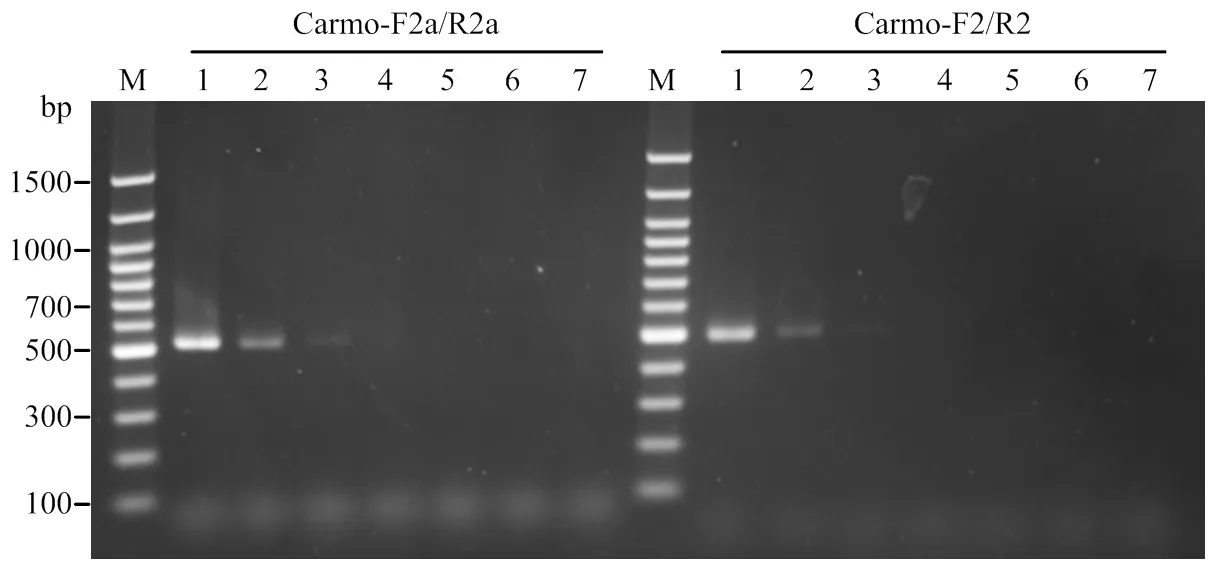
图5 简并引物Carmo-F2/R2和Carmo-F2a/R2a一步RT-PCR的灵敏度检测
2.5 在检测扶桑病毒中的应用
利用所建立的RT-PCR方法,对疑似病毒症状的13份扶桑样品进行检测。结果表明,简并引物Carmo- F2/R2和Carmo-F2a/R2a从扶桑样品中均扩增到约500或550 bp条带(图6,部分结果)。该结果初步表明,扶桑样品中含有香石竹斑驳病毒属病毒。
2.6 序列测定与分析
简并引物Carmo-F2/R2或Carmo-F2a/R2a成功用于PCR产物的序列测定(GenBank登录号为OQ726001 —OQ726008)。其中,CarMV的LPC68000(Agdia)条带比较弱,测序未成功。经序列拼接,去除引物位置序列后,获得458—464 bp序列。BLSATn分析表明,所获得序列均与相应病毒序列具有最高序列一致性(表5),与其他种类的序列一致性则较低。所获得的序列均为RdRp基因序列,为预期的扩增区域。因此,简并引物Carmo-F2/R2或Carmo-F2a/R2a可用于检测不同种类的甲型、乙型和丙型香石竹斑驳病毒属病毒,结合序列分析可用于种类的快速鉴定。
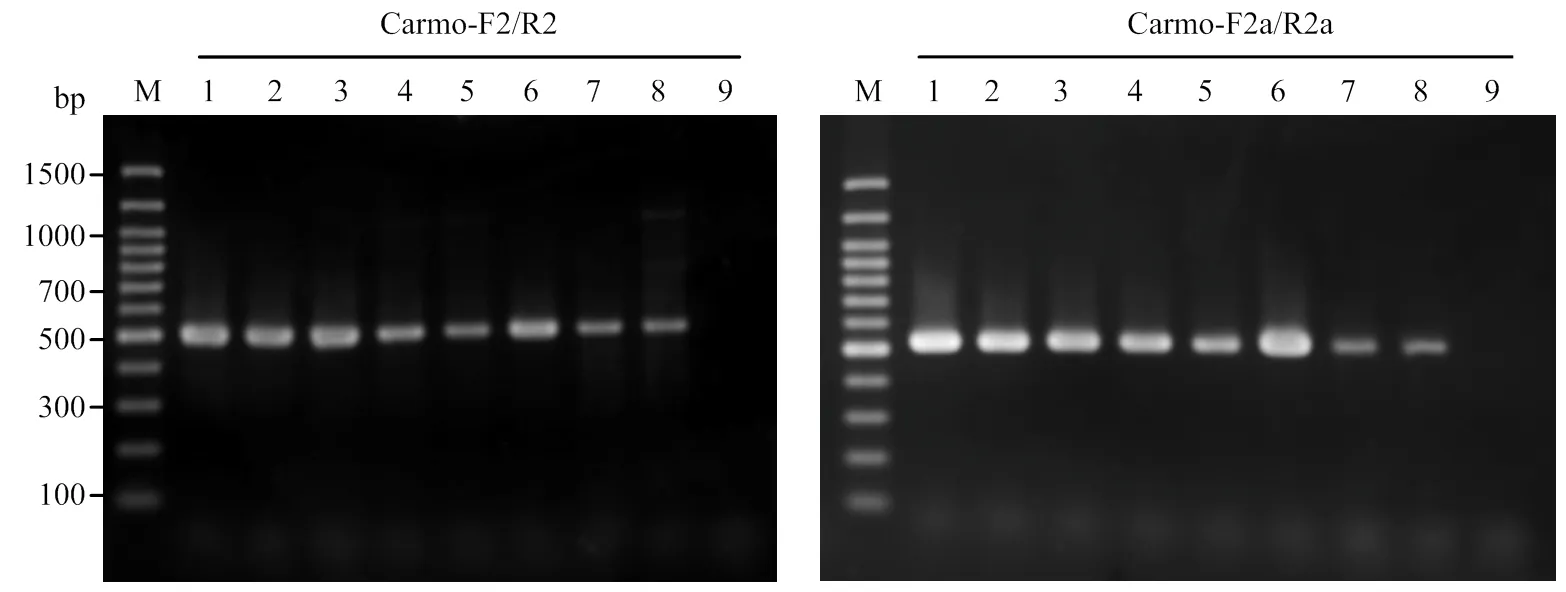
1-3: XM1-XM3; 4-6: XM4-XM6; 7-9: XM7-XM9
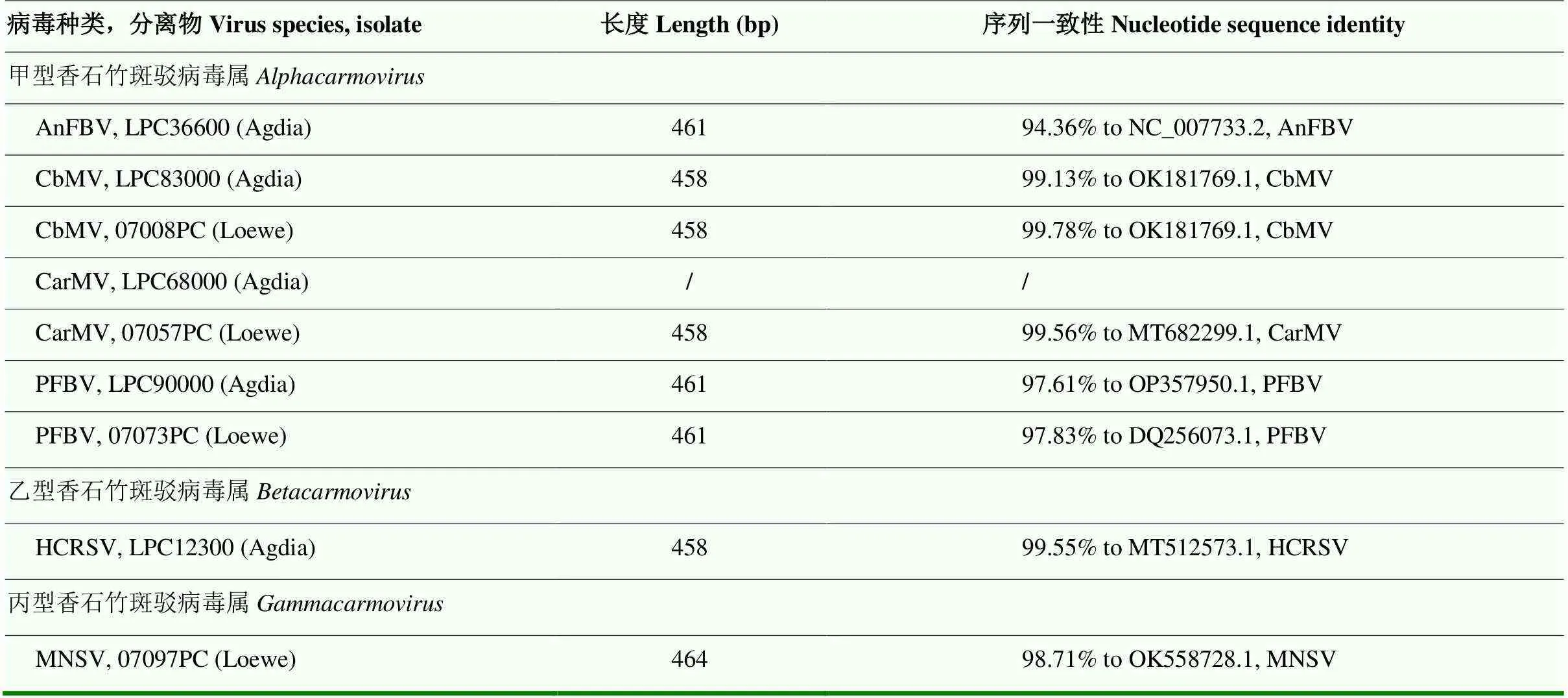
表5 简并引物RT-PCR扩增序列的BLASTn结果
扶桑样品的PCR产物经序列测定后,进行正反向拼接,去除引物位置序列后,获得长458 bp的序列(GenBank登录号为OQ725988—OQ726000)。BLASTn分析表明,不同分离物均与已知的木槿褪绿环斑病毒(hibiscus chlorotic ringspot virus,HCRSV)(MK279671.1、OK636421.1、OP779319.1、KY933060.1、KC876666.1、X86448.2、MT512573.1、DQ392986.1、MN080500.1)具有最高序列一致性(表6)。因此,该结果表明,扶桑上的香石竹斑驳病毒属病毒为HCRSV。
2.7 系统发育分析
对简并引物RT-PCR扩增相应区域的通读病毒亚科()病毒的核苷酸序列和氨基酸序列分别进行系统发育分析。核苷酸序列系统发育分析表明,甲型、乙型香石竹斑驳病毒属、玉米褪绿斑驳病毒属()、番茄丛矮病毒属()均可单独成一分支(图7-A)。在7种天竺葵环斑病毒属()病毒中,除铁线莲褪绿斑驳病毒(clematis chlorotic mottle virus,ClCMV;NC 033777)显示出与乙型香石竹斑驳病毒属具有更近亲缘关系外,其他6种病毒均可单独形成一个分支,显示出更近的亲缘关系。在4种丙型香石竹斑驳病毒属病毒中,大豆黄斑驳花叶病毒(soybean yellow mottle mosaic virus,SYMMV;NC_011643)和豇豆斑驳病毒(cowpea mottle virus,CPMoV;NC_003535),豌豆茎部坏死病毒(pea stem necrosis virus,PSNV;NC_004995)和甜瓜坏死病毒(melon necrotic spot virus,MNSV;JX879088等)可分别形成两个明显的分支,其中SYMMV(NC_011643)和CPMoV(NC_003535)显示出与甲型、乙型香石竹斑驳病毒属和天竺葵环斑病毒属更近的亲缘关系。在本研究所测定AnFBV、CarMV、CbMV、HCRSV、MNSV和PFBV的病毒分离物中,均可与已知相应病毒种类形成独立分支,显示出很近的亲缘关系。在所测定的13个扶桑HCRSV厦门分离物中,均可与已知的HCRSV分离物形成独立分支,显示很近的亲缘关系,该结果进一步证实厦门的扶桑受到HCRSV侵染。因此,利用简并引物RT-PCR扩增区域的核苷酸序列进行系统发育分析,可以对甲型、乙型、丙型香石竹斑驳病毒属病毒进行种类鉴定。

使用MAFFT进行序列比对,最大似然法构建系统发育树。节点上的数值为1000次重复后的自举检验值。比例尺代表每个位点替代。▲:本研究使用的病毒分离物。●:厦门扶桑中检测到的HCRSV

表6 厦门扶桑病毒的BLASTn结果
氨基酸序列系统发育分析表明,通读病毒亚科中各病毒属均可独立形成一个分支(图7-B)。其中,甲型香石竹斑驳病毒属与天竺葵环斑病毒属具有更近的亲缘关系,乙型香石竹斑驳病毒属次之,丙型香石竹斑驳病毒属则与这3个属的亲缘关系较远,与Scheets等[8]、Pérez-Cañamás等[39]分析结果相一致。此外,ClCMV(NC 033777)与其他天竺葵环斑病毒属种类处于同一分支中;4种丙型香石竹斑驳病毒属病毒中形成两个明显的分支,即SYMMV(NC_011643)和CPMoV(NC_003535),PSNV(NC_004995)和MNSV(JX879088等),但这两个分支均处于一个大的分支中。本研究测定的AnFBV、CarMV、CbMV、HCRSV、MNSV和PFBV分离物均可与相应病毒种类形成独立分支,显示出很近的亲缘关系。测定的13个扶桑HCRSV厦门分离物也均可与已知的HCRSV分离物处于同一分支中。因此,与核苷酸序列相比,基于氨基酸序列的系统发育分析更能准确反映各属病毒的分类地位。
3 讨论
3.1 简并引物RT-PCR方法成功用于香石竹斑驳病毒属病毒的检测
简并引物(RT-)PCR方法是一种根据病毒科、属的保守区域设计简并引物,可用于病毒科、属的多目标分子检测方法。多重(RT-)PCR方法也是一种多目标检测方法,这两者的主要区别是:简并引物(RT-)PCR方法针对的目标是分类上同属于一个科、一个属的同类病毒,包括尚未报道的病毒种类;而多重(RT-)PCR方法针对的目标可以是分类上完全不相关的病毒(甚至是真菌、细菌),但检测目标和数量是特定的。本研究根据基因组保守区域设计简并引物,建立的简并引物RT-PCR方法成功用于检测甲型、乙型、丙型香石竹斑驳病毒属不同种类的病毒,但获得的PCR产物浓度有所差异。其中,CarMV的LPC68000(Agdia)分离物,由于PCR产物浓度低,测序并未成功。这种PCR产物的浓度差异是由于病毒的起始浓度差异造成的,还是因为简并引物对不同病毒种类的扩增效率不同而引起的,这还有待于进一步的研究。此外,利用该方法成功用于检测MNSV(07097PC,Loewe)分离物,但多次进行RT-PCR扩增MNSV的LPC12402(Agdia)分离物均未成功,这可能是因为该病叶冻干粉末的保存时间太长而引起病毒降解造成的。
3.2 与已有简并引物RT-PCR方法的比较分析
目前,针对香石竹斑驳病毒属及其相关病毒的简并引物RT-PCR方法不仅已有公开的文献报道,也有商品化的检测引物在出售。Morozov等设计简并引物Carmo-II/VI可以检测同科的其他种类病毒,包括MCMV()、CRSV、RCNMV()和TBSV()[29],而本研究所设计的简并引物Carmo-F2/R2或Carmo-F2a/R2a初步结果表明不能用于检测MCMV、PLPV、CRSV,其检测范围小,但是否可以检测同科的其他病毒还待进一步研究。根据序列比对分析结果,对出现错误的简并引物Carmo-II/VI进行修正,包括增加部分简并碱基,但还需要进一步的试验验证(表4)。杨雷亮等根据外壳蛋白基因设计一对简并引物(CarMV-F1/F2),成功用于8种香石竹斑驳病毒属病毒的检测[30],但在本研究所比对的病毒种类中,未在CP基因区域发现合适的保守区域,这可能是随着用于比对分析的病毒种类增加而导致难以在CP基因区域找到保守区域。与本研究的简并引物相比,Agdia公司的PCR引物不仅在扩增片段大小上存在明显差异,而且其可检测范围更广[31]。但是,由于需要进口,使用该商业化的简并引物还存在一定困难,不仅购置麻烦、到货周期长,而且价格昂贵、使用成本高。因此,本研究建立的方法可以显著降低甲型、乙型和丙型香石竹斑驳病毒属病毒的检测成本。
3.3 在简并引物5′端加入一段非互补序列可以提高RT-PCR检测方法的灵敏度
在PCR引物的5′端加入一段非互补的富含AT的序列(AATAAATCATAA),不仅可以提高实时荧光PCR的荧光信号[40],而且还可以提高普通PCR[41]和多重PCR的灵敏度[42-43]。在简并引物的5′端加入该富含AT的序列,也可以提高简并引物PCR的特异性和灵敏度[44]。本研究表明,在简并引物的5′端加入该富含AT的序列后(Carmo-F2a/R2a),可以提高简并引物RT-PCR的扩增效率,其灵敏度至少可以提高10倍。此外,在简并引物的5′端分别加入一段非互补的测序引物RV-M和M13-47后,也可以提高简并引物RT-PCR的检测灵敏度[45]。因此,在简并引物的5′端加入一段非互补的序列,可以提高简并引物RT-PCR方法的灵敏度。在RT-PCR检测中推荐使用简并引物Carmo-F2a/R2a。
3.4 PCR产物测序峰图分析
同一植物可同时受到不同种类病毒的侵染,包括同一科、属的多种病毒。使用简并引物RT-PCR方法也可以发现同一科、属病毒的复合侵染情况,即当PCR产物直接测序出现杂峰、套峰时,预示着可能存在2种或2种以上同科、属病毒的复合侵染。此时,通过克隆并挑取多个克隆子进行测序,则可明确是否具有多种病毒序列。本研究利用所建立的方法对扶桑样品进行检测,经PCR产物直接测序,并未出现杂峰、套现现象,表明扶桑中没有除HCRSV外的其他甲型、乙型及丙型香石竹斑驳病毒属病毒。扶桑中是否还有其他科、属的病毒,则需要进一步检测确定。
3.5 RdRp基因在种类鉴定中的应用
RdRp基因是番茄丛矮病毒科中属、种的一个重要划分指标。基于完整的RdRp的系统发育分析是划分属的一个重要依据,而RdRp基因的氨基酸序列一致性则是划分种的一个重要依据[1,8]。在甲型、乙型、丙型香石竹斑驳病毒属中划分病毒种的标准为:聚合酶的氨基酸序列一致性小于75%;外壳蛋白的氨基酸序列一致性小于75%[8]。本研究所测定的各种病毒分离物的部分RdRp基因氨基酸序列与已知的相应病毒序列的一致性均大于90%,远高于种的划分标准,证实了测定的序列为相应病毒的序列。进一步系统发育分析表明,与核苷酸序列相比,基于氨基酸序列的系统发育分析更能真实反映出通读病毒亚科中各属病毒的分类地位,各属、各种病毒均可以形成独立分支,该结果与Scheets等[8]、Pérez-Cañamás等[39]的分析结果相一致。因此,利用本研究建立的简并引物RT-PCR方法,并结合序列与系统发育分析,可以实现甲型、乙型、丙型香石竹斑驳病毒属病毒的快速鉴定。
4 结论
4.1 根据保守区域设计简并引物,本研究所建立的RT-PCR检测方法可以用于甲型、乙型和丙型香石竹斑驳病毒属病毒的广谱检测,结合序列及系统发育分析,可以实现病毒种类的快速鉴定。
4.2 在简并引物中加入一段非互补的富含AT序列(AATAAATCATAA)可以提高简并引物RT-PCR检测灵敏度。
4.3 利用本研究所建立的方法对扶桑样品进行检测,初步表明木槿褪绿环斑病毒(HCRSV)在厦门地区的扶桑上普遍发生。
[1] Rochon D, Rubino L, Russo M, Martelli G P, Lommel S.//King A M Q, Adams M J, Carstens E B, Lefkowitz E J. Virus taxonomy: Ninth Report of the International Committee on Taxonomy of Viruses. San Diego: Elsevier, 2012: 1111-1136.
[2] 蔡红, 孔宝华, 吴建宇, 陈海如, 段永嘉. 昆明香石竹斑驳病毒鉴定. 云南农业大学学报, 2001, 16(4): 263-266.
Cai H, Kong B H, WU J Y, CHEN H R, DUAN Y J. Identification of carnation mottle virus on carnation in Kunming. Journal of Yunnan Agricultural University, 2001, 16(4): 263-266. (in Chinese)
[3] 杨世安, 李战彪, 秦碧霞, 谢慧婷, 崔丽贤, 苏琴, 邓铁军, 蔡健和. 广西三种甜瓜病毒分离物的分子检测与鉴定. 植物保护, 2017, 43(3): 83-89.
Yang S A, Li Z B, Qin B X, XIE H T, CUI L X, SU Q, DENG T J, CAI J H. Molecular detection and identification of three viruses isolated from melons in Guangxi. Plant Protection, 2017, 43(3): 83-89. (in Chinese)
[4] Zheng G H, Liao F R, Ye T, ZHANG W Z, MING Y L. First report of hibiscus chlorotic ringspot virus infectingin Fujian Province, China. Plant Disease, 2018, 102(10): 2046.
[5] ICTV. Current ICTV Taxonomy Release. https://ictv.global/taxonomy.
[6] 洪健, 周雪平. ICTV第九次报告以来的植物病毒分类系统. 植物病理学报, 2014, 44(6): 561-572.
HONG J, ZHOU X P. The universal system of plant virus taxonomy since the 9th ICTV report. Acta Phytopathologica Sinica, 2014, 44(6): 561-572. (in Chinese)
[7] Adams M J, King A M Q, Carstens E B. Ratification vote on taxonomic proposals to the International Committee on Taxonomy of Viruses (2013). Archives of Virology, 2013, 158: 2023-2030.
[8] Scheets K, White K A, Rubino L, Martelli g, Rochon D’a. Divide the genusinto three new genera:,, and. http://www.ictvonline.org/ proposals-15/2015.007a-rP.A.v1.split_Carmovirus.pdf.
[9] Scheets K, Jordan R, White K A, Hernández C., a proposed new genus in the family. Archives of Virology, 2015, 160(9): 2385-2393.
[10] 洪健, 李德葆, 周雪平. 植物病毒分类图谱. 北京: 科学出版社, 2001: 130-132.
HONG J, LI D B, ZHOU X P. Taxonomic Atlas of Plant Viruses. Beijing: Science Press, 2001: 130-132. (in Chinese)
[11] Kowalska A. Studies on the properties of carnation mottle virus and carnation ringspot virus isolated in Poland. Phytopathologische Zeitschrift, 1972, 74(4): 329-341.
[12] Sandra N, Tripathi A, Dikshit H K, Mandal B, JAIN R K. Seed transmission of a distinct soybean yellow mottle mosaic virusstrain identified from India in natural and experimental hosts. Virus Research, 2020, 280: 197903.
[13] Gillaspie Jr A G, Mitchell S E, Stuart G W, Bozarth R F. RT-PCR method for detecting cowpea mottle carmovirus ingermplasm. Plant Disease, 1999, 83(7): 639-643.
[14] Gonzalez-Garza R, Gumpf D J, Kishaba A N, Bohn G W. Identification, seed transmission, and host range pathogenicity of a California isolate of melon necrotic spot virus. Phytopathology, 1979, 69(4): 340-345.
[15] Herrera-Vásquez J A, Córdoba-Sellés M C, Cebrián M C, Alfaro-Fernández A, Jordá C. Seed transmission of melon necrotic spot virus and efficacy of seed-disinfection treatments. Plant Pathology, 2009, 58(3): 436-442.
[16] Coudriet D L, Kishaba A N, Carroll J E. Transmission of muskmelon necrotic spot virus in muskmelons by cucumber beetles. Journal of Economic Entomology, 1979, 72(4): 560-561.
[17] Tomlinson J A, Thomas B J. Studies on melon necrotic spot virus disease of cucumber and on the control of the fungus vector (). Annals of Applied Biology, 1986, 108(1): 71-80.
[18] Ohki T, Akita F, Mochizuki T, Kanda A, Sasaya T, Tsuda S. The protruding domain of the coat protein of melon necrotic spot virus is involved in compatibility with and transmission by the fungal vector. Virology, 2010, 402(1): 129-134.
[19] Skotnicki M L, Mo J Q. Detection of cardamine chlorotic fleck carmovirus in soil by the polymerase chain reaction. Australasian Plant Pathology, 1996, 25(1): 18-23.
[20] Alexandre M A V, Duarte L M L, Ramos A F, Harakava R. Identification and molecular characterization of carnation mottle virus Brazilian isolates from carnation. Horticultura Brasileira, 2015, 33(2): 257-260.
[21] Raikhy G, Hallan V, Kulshrestha S, Ram R, Zaidi A A. Multiplex PCR and genome analysis of carnation mottle virus Indian isolate. Current Science, 2006, 90(1): 74-82.
[22] Tang J, Elliott D R, Quinn B D, Clover G R G, Alexander B J R. Occurrence of hibiscus chlorotic ringspot virus inspp. in New Zealand. Plant Disease, 2008, 92(9): 1367.
[23] Wylie S J, Li H, Jones M G K. First report of an isolate of Japanese iris necrotic ring virus from Australia. Australasian Plant Disease Notes, 2012, 7(1): 107-110.
[24] Gosalvez B, Navarro J A, Lorca A, Botella F, Sanchez-Pina M A, Pallas V. Detection of melon necrotic spot virus in water samples and melon plants by molecular methods. Journal of Virological Methods, 2003, 113(2): 87-93.
[25] Franck A, Loebenstein G, Gera A. Use of the reverse transcription-polymerase chain reaction for the detection of pelargonium flower break carmovirus. Journal of Phytopathology, 1997, 145(5/6): 235-238.
[26] Deng D, McGrath P F, Robinson D J, Harrison B D. Detection and differentiation of whitefly-transmitted geminiviruses in plants and vector insects by the polymerase chain reaction with degenerate primers. Annals of Applied Biology, 1994, 125(2): 327-336.
[27] Chen J, Adams M J. A universal PCR primer to detect members of theand its use to examine the taxonomic status of several members of the family. Archives of Virology, 2001, 146(4): 757-766.
[28] Van der Vlugt R A A, Berendsen M. Development of a general potexvirus detection method. European Journal of Plant Pathology, 2002, 108: 367-371.
[29] Morozov S Y, Ryabov E V, Leiser R M,Zavriev S K. Use of highly conserved motifs in plant virus RNA polymerases as the tags for specific detection of carmovirus-related RNA-dependent RNA polymerase genes. Virology, 1995, 207(1): 312-315.
[30] 杨雷亮, 陈定虎, 李明福, 陈洪俊. 香石竹斑驳病毒属和潜隐病毒属的PCR检测方法研究. 检验检疫学刊, 2009, 19(2): 1-3.
Yang L L, Chen D H, Li M F, CHEN H J. Detection ofandwith PCR method. Journal of Inspection and Quarantine, 2009, 19(2): 1-3. (in Chinese)
[31] Agdia. PCR Primer for-. https://orders.agdia. com/agdia-pcr-primer-for-carmo-pcr-94000.
[32] Thompson J D, Higgins D G, Gibson T J. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Research, 1994, 22(22): 4673-4680.
[33] Hall T A. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Series, 1999, 41: 95-98.
[34] BLAST. Basic Local Alignment Search Tool. https://blast.ncbi.nlm. nih.gov/Blast.cgi.
[35] Kumar S, Stecher G, Li M, Knyaz C, Tamura K. MEGA X: molecular evolutionary genetics analysis across computing platforms. Molecular Biology and Evolution, 2018, 35(6): 1547-1549.
[36] Zhang D, Gao F, Jakovlić I, Zou H, Zhang J, Li W X, Wang G T. PhyloSuite: An integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies. Molecular Ecology Resources, 2020, 20(1): 348-355.
[37] Katoh K, Standley D M. MAFFT multiple sequence alignment software version 7: improvements in performance and usability. Molecular Biology and Evolution, 2013, 30(4): 772-780.
[38] Le S Q, Gascuel O. An improved general amino acid replacement matrix. Molecular Biology and Evolution, 2008, 25(7): 1307-1320.
[39] Pérez-Cañamás M, Hernández C. Carmo-like viruses ()//Bamford D H, Zuckerman M. Encyclopedia of Virology, 4th ed, Vol. 3. Academic Press, 2021: 285-292.
[40] Afonina I, Ankoudinova I, Mills A, Lokhov S, Huynh P, Mahoney W. Primers with 5′ flaps improve real-time PCR. Biotechniques, 2007, 43(6): 770, 772, 774.
[41] Arif M, Ochoa-Corona F M. Comparative assessment of 5′ A/T-rich overhang sequences with optimal and sub-optimal primers to increase PCR yields and sensitivity. Molecular Biotechnology, 2013, 55(1): 17-26.
[42] Timmons C, Dobhal S, Fletcher J, MA L M. Primers with 5′ flaps improve the efficiency and sensitivity of multiplex PCR assays for the detection ofandO157: H7. Journal of Food Protection, 2013, 76(4): 668-673.
[43] Larrea-Sarmiento A, Alvarez A M, Stack J P, ARIF M. Synergetic effect of non-complementary 5′ AT-rich sequences on the development of a multiplex TaqMan real-time PCR for specific and robust detection ofandsubsp.. PLoS One, 2019, 14(7): e0218530.
[44] Wei T, Clover G. Use of primers with 5′ non-complementary sequences in RT-PCR for the detection of nepovirus subgroups A and B. Journal of Virological Methods, 2008, 153(1): 16-21.
[45] 叶志红, 廖富荣, 郭木金, 方志鹏, 陈青, 陈红运, 林石明, 林毅. 同时检测豇豆花叶病毒属与蚕豆病毒属病毒的通用RT-PCR检测方法. 中国农业科学, 2015, 48(8): 1527-1537. doi: 10.3864/j.issn. 0578-1752.2015.08.07.
YE Z H, LIAO F R, GUO M J, FANG Z P, CHEN Q, CHEN H Y, LIN S M, LIN Y. A universal RT-PCR method for the simultaneous detection of the viruses in generaand. Scientia Agricultura Sinica, 2015, 48(8): 1527-1537. doi: 10.3864/j.issn.0578- 1752.2015.08.07. (in Chinese)
Development of a degenerate primer RT-PCR assay for detection of
LIAO FuRong1, CHEN HongYun1, SHEN JianGuo2, FANG ZhiPeng1, HUANG PengYing1, CHEN Qing1, LIN LingLing1, HONG Jun1
1Xiamen Customs Technology Center, Xiamen 361026, Fujian;2Technology Center of Fuzhou Customs District, Fuzhou 350001
【Objective】The objective of this study is to establish a rapid, sensitive, and broad-spectrum screening method for simultaneous detection and identification of the genera,, andusing degenerate primer RT-PCR combined with sequence analysis.【Method】Multiplexed analysis of genome sequences was aligned tosearch for suitable conserved regions for the design of the degenerate primers. One pair of degenerate primer Carmo-F2/Carmo-R2 was designed based on the RNA-dependent RNA polymerase (RdRp) gene sequences, and another pair primer Carmo-F2a/Carmo-R2a was formed by adding the non-complementary AT-rich sequences (AATAAATCATAA) to the 5′ end of the degenerate primers. The broad-spectrum, specificity, and sensitivity of RT-PCR method were analyzed. The sequencing, BLASTn analysis and phylogenetic analysis of PCR products were performed. The method was used to screen and detect viruses on Chinese hibiscus () samples from Xiamen, China.【Result】Degenerate primers Carmo-F2/Carmo-R2 and Carmo-F2a/Carmo-R2a were used to amplify partial RdRp gene of the members of genera,, andby RT-PCR. The fragment of approximately 500 and 550 bp was amplified, respectively. The developed RT-PCR assay was successfully used to detect angelonia flower break virus (AnFBV;), calibrachoa mottle virus (CbMV;), carnation mottle virus (CarMV;), and pelargonium flower break virus (PFBV;), hibiscus chlorotic ringspot virus (HCRSV;), melon necrotic spot virus (MNSV;). The specificity test showed that no specific band could be obtained from maize chlorotic mottle virus (MCMV;), pelargonium line pattern virus (PLPV;), carnation ringspot virus (CRSV;), and healthy plants which including watermelon, melon, pumpkin, soybean, and pea. The sensitivity results showed that the primers Carmo-F2/Carmo-R2 could be detected up to 10-2dilution and the primers Carmo-F2a/Carmo-R2a could be detected up to 10-3dilution, which indicated that the non-complementary AT-rich sequences added at the 5′ end of the degenerate primers could increase the sensitivity of degenerate primer RT-PCR. BLAST analysis showed that the sequences determined had the highest sequence consistency with the corresponding virus species. Phylogenetic analysis based on partial amino acid sequences of the RdRp gene showed that it is consistent with the current classification of the subfamily, and the viruses could be identified at the species level. HCRSV was detected in all 13samples with suspected virus symptoms.【Conclusion】The RT-PCR method based on the degenerate primers Carmo-F2/Carmo-R2 and Carmo-F2a/Carmo-R2a can be used for the screening and detection of viruses of the genera,, and, and can be used for rapid identification of virus species in combination with sequence analysis and phylogenetic analysis, and may be used to discover the new virus.plants were infested with HCRSV in Xiamen, China.
;;; degenerate primer; RT-PCR;; hibiscus chlorotic ringspot virus (HCRSV)

10.3864/j.issn.0578-1752.2023.12.005
2023-03-30;
2023-04-18
福建省农业引导性(重点)项目(2019N0029)、厦门海关科研项目(2020XK03)
廖富荣(通信作者),Tel:0592-6806943;E-mail:lfr005@163.com
(责任编辑 岳梅)
