基于16K SNP芯片的小麦株高QTL鉴定及其遗传分析
2023-07-09姚琦馥陈黄鑫周界光马瑞莹邓亮谭陈芯雨宋靖涵吕季娟马建
姚琦馥,陈黄鑫,周界光,马瑞莹,邓亮,谭陈芯雨,宋靖涵,吕季娟,马建
基于16K SNP芯片的小麦株高QTL鉴定及其遗传分析
姚琦馥1,陈黄鑫2,周界光2,马瑞莹3,邓亮3,谭陈芯雨3,宋靖涵4,吕季娟5,马建2
1铜仁学院农林工程与规划学院/贵州省梵净山地区生物多样性保护与利用重点实验室,贵州铜仁 554300;2四川农业大学小麦研究所,成都 611130;3四川农业大学农学院,成都 611130;4北京外国语大学,北京 100089;5四川省种子站,成都 610041
【目的】株高与产量之间关系密切。进一步挖掘具有育种利用价值的小麦株高数量性状位点(quantitative trait loci,QTL),并解析株高QTL对产量相关性状的遗传效应,为分子育种提供理论依据。【方法】以田间自然变异株为母本、小麦品种川农16为父本杂交衍生的F6代重组自交系群体(MC群体)为试验材料,于2020—2022年在四川省温江区、崇州市和雅安市试验基地进行2年5个生态环境点的种植和株高表型鉴定。使用16K SNP芯片所构建的高质量、高密度遗传连锁图谱定位株高性状。同时,利用株高主效QTL侧翼标记的基因型分析其正效应位点对于产量相关性状的遗传效应,评估主效QTL对产量提升的潜力。【结果】定位结果显示,分别在1A、3D、4D、5A和7B染色体共鉴定到8个控制株高的QTL。其中,定位到2个稳定的主效QTL:和,分别解释9.09%—25.56%和3.91%—13.09%的表型变异率,其正效应位点均来源于川农16。加性效应分析发现同时携带和正效应位点株系的株高显著高于仅携带单一正效应位点或没有携带任何正效应位点的株系。相关性分析发现,株高与有效分蘖数之间存在极显著的正相关性,与旗叶宽之间存在显著的负相关性,而与每穗粒数、每穗粒重、千粒重、旗叶长和开花期之间无显著相关性。遗传效应分析发现,正效应位点极显著增加有效分蘖数(56.51%),显著减少每穗粒数(-11.26%)、每穗粒重(-13.04%)、千粒重(-5.47%)和旗叶宽(-2.85%),促进开花期提前(-0.61%)。正效应位点显著增加有效分蘖数(10.57%)、每穗粒重(4.32%)和千粒重(2.92%),延迟开花期(1.07%)。【结论】在5A染色体定位到1个株高主效QTL—,其正效应位点显著提高有效分蘖数、每穗粒重及千粒重,对产量提高可能有积极效应。
小麦;16K SNP芯片;QTL;株高;产量
0 引言
【研究意义】小麦属于世界上三大谷物之一,提高小麦产量对于满足世界粮食需求意义重大[1]。株高作为小麦重要的育种性状,通过影响体内营养分配和抗倒伏性进而影响产量[2]。20世纪60年代利用矮化基因进行的“绿色革命”大幅度提高了小麦粮食产量[3]。进一步发掘适用于育种利用的矮化基因有利于小麦产量的稳步提高。【前人研究进展】小麦株高的遗传基础研究一直受到广泛关注,相关的基因或QTL被国内外学者大量报道[2, 4]。如,小麦首个被克隆的矮秆基因是赤霉素(GA)生长途径的关键因子[5]。Chai等[6]和Xiong等[7]克隆了2D染色体的矮秆基因,并证明其通过影响GA合成相关基因调节赤霉素合成,从而影响株高。具有自激活活性的矮秆基因编码核苷酸结合和富含亮氨酸的重复结构域,能够调节Ⅲ类过氧化物酶活性,不影响GA生物合成或信号转导途径[8]。矮秆基因被定位到7A染色体短臂0.53—1.48 Mb,通过减少节间细胞数降低株高[9]。过表达矮秆基因()降低GA生物活性并引起矮化[10]。矮秆基因被定位于6A染色体短臂144.0—148.3 Mb,对GA敏感[11]。近期研究表明,的候选基因是,与存在遗传和蛋白物理互作[12]。隐性半矮秆基因被定位到3D染色体短臂1.3 Mb的区间[13]。此外,胡文静等[14]以扬麦13和人工合成小麦衍生系C615杂交构建的重组自交系(RIL)为材料,分别在染色体3B、4D和5A检测到3个株高QTL。关攀锋等[15]利用农大3338和京冬6号构建的小麦永久F2群体,共检测到32个株高QTL。廖思敏等[16]以W7268和川育12杂交得到的RIL为材料,定位到,可能为光周期敏感基因。Liu等[17]利用PuBing3228 × Gao8901构建的RIL,鉴定到5个稳定的株高QTL。决定小麦株高表型的基因或QTL通常表现出多效性。如,矮秆基因有效降低了小麦高度,对籽粒产量没有明显的负面影响[7]。矮秆基因对籽粒产量具有显著的负效应[18]。矮秆基因同时影响小麦株高、胚芽鞘长度、穗长、小穗数、穗密度和粒重[11]。李聪等[19]在小麦2D染色体鉴定到一个控制株高和穗长的“一因多效”QTL。此外,理想的小麦株高具有逃避病原菌侵染的机能,对赤霉病抗性也会产生影响[20-21]。【本研究切入点】虽然已有少数矮秆基因被鉴定,但能够被育种利用的位点却不多。不同的基因或QTL可能具有不同的矮化机制,对于其他产量相关性状有着不同影响。发掘和运用优异的矮秆基因并解析其遗传机制,更有利于小麦产量的持续提升。【拟解决的关键问题】本研究以株高较高的多小穗多花自然变异株(,)为母本、小麦品种川农16(CN16)为父本构建RIL(MC)群体,基于16K SNP芯片技术构建高质量、高密度的遗传连锁图谱,对株高性状进行遗传定位。结合产量相关性状,分析株高主效QTL对产量的潜在影响,为分子育种提供理论依据。
1 材料与方法
1.1 试验材料
试验材料为×CN16经单籽粒传法构建的包含198个株系的F6代RIL群体。是在田间发现的自然变异株(变异亲本未知),表现出多小穗和多小花,但株高较高、分蘖较少,生育期较长,难以直接进行育种生产,由四川农业大学小麦研究所鉴定并存于种子库[22]。CN16是四川农业大学小麦研究所选育的小麦品种,具有理想的株型,表现出多分蘖、株高较矮、生育期适中等特点[23]。和CN16的茎节个数无明显差异,而茎节长度显著长于CN16(图1)。

图1 MC群体亲本表型
1.2 试验设计和表型鉴定
对MC群体及其亲本进行2年5个生态环境点的种植:2021年温江(2021WJ)、2021年崇州(2021CZ)、2021年雅安(2021YA)、2022年温江(2022WJ)和2022年崇州(2022CZ)。按照株距0.1 m、行长0.75 m、行间距0.3 m进行单籽粒播种,所有生态环境点均采用2次重复的随机区组设计,田间进行常规管理[22]。于小麦成熟期,对其株高(不包括芒长)、有效分蘖数、每穗粒数、每穗粒重、千粒重、旗叶长、旗叶宽和开花期的表型进行鉴定。每个株系选取至少4株正常生长且较为一致的单株进行测量,取2次重复的平均值作为该株系的表型值。其中,株高、有效分蘖数、千粒重和开花期的最佳线性无偏预测(BLUP)值已被用作相关性分析使用[22]。
1.3 遗传图谱构建和QTL定位
基于Zhou等[22]利用16K SNP芯片(石家庄博瑞迪生物技术有限公司,http://www.molbreeding.com)所构建的遗传连锁图谱,进行株高的遗传定位。利用IciMapping 4.2中的完备区间作图法进行QTL检测,设置参数染色体步长Step=0.1 cM、逐步回归概率PIN=0.001和LOD≥2.5[24-25]。利用IciMapping 4.2进行多环境分析,设置参数染色体步长Step=0.1 cM、逐步回归概率PIN=0.001和LOD≥5。QTL命名方式:+性状名称+研究机构名称+定位群体+染色体编号+QTL序数,其中,“”表示株高、“”表示四川农业大学、“”表示MC群体[26]。利用QTL侧翼标记序列在中国春参考基因组1.0版本(CS RefSeq v1.0)和野生二粒参考基因组2.0版本(WEW_v2.0)中检索其物理位置,并基于CDS序列确定其同源基因[27-29]。
1.4 统计分析
采用IBM SPSS Statistics 20进行株高表型数据的描述分析。运用IciMapping 4.2进行株高表型数据的多环境方差分析(ANOVA)。使用SAS 9.1.3的MIXED进程计算株高的BLUP值。基于广义遗传力(2)公式2=VG/(VG+VE),使用SAS 9.1.3计算株高的2,其中,VG和VE分别表示遗传方差和环境方差。利用Origin 2022进行株高表型数据的图片转化。
2 结果
2.1 表型分析
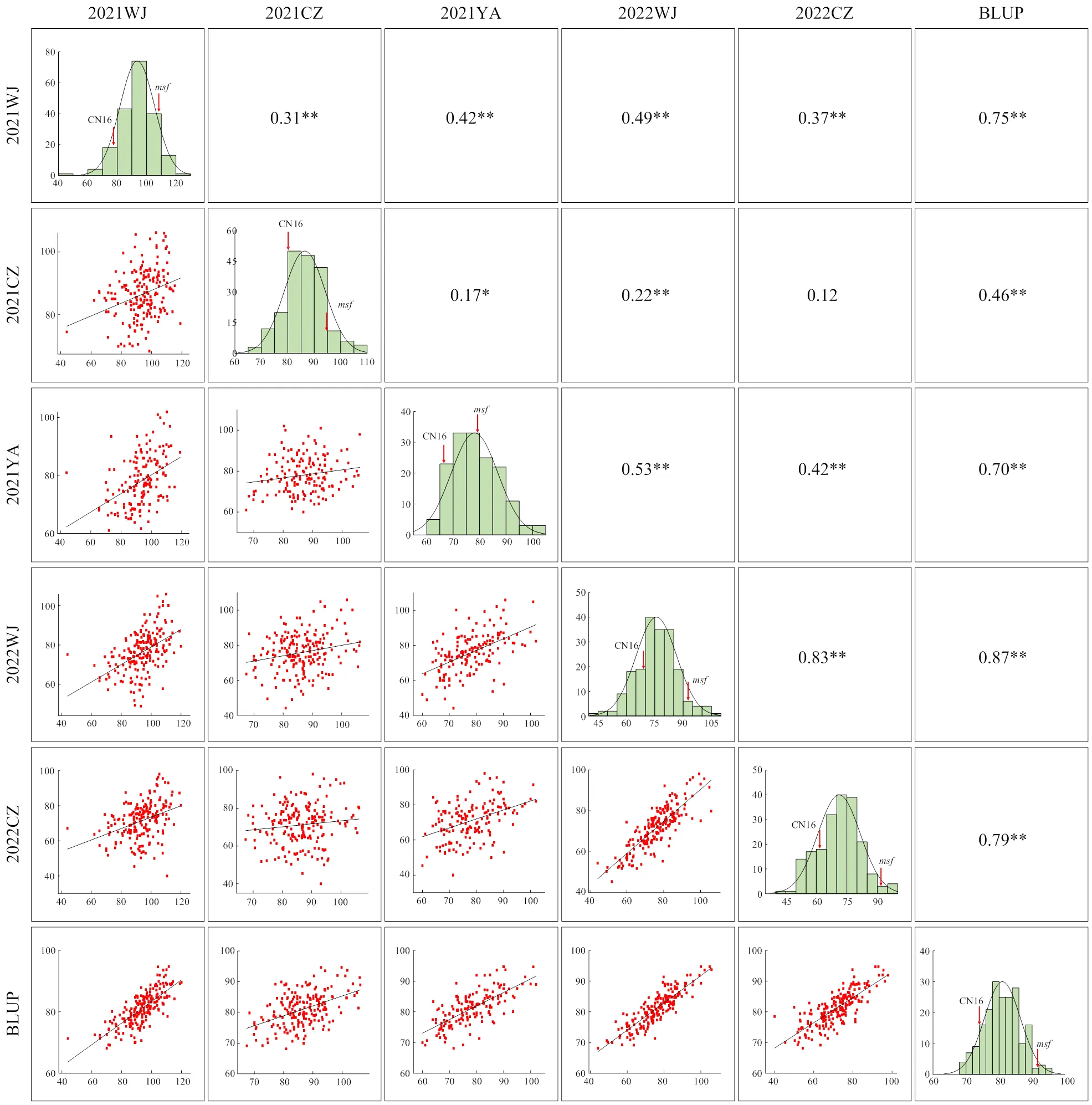
在2年5个生态环境下,对MC群体的亲本和RIL进行株高的表型鉴定。其中,亲本株高表型值为80.0—109.4 cm,亲本CN16株高表型值为65.1—83.6 cm,二者在4个生态环境中存在极显著的差异(表1,<0.01)。RIL株高表型值为40.0—120.0 cm,分布频率呈近似正态分布,具有明显的超亲分离现象(表1和图2)。ANOVA分析表明环境、基因型和环境 × 基因型互作对多环境下的株高有极显著影响(表2)。同时,MC群体株高的2为0.79,表明其受环境因素的影响较小,适合进一步的遗传分析。
2.2 相关性分析
通过对MC群体在不同生态环境的株高表型进行相关性分析,发现其相关系数介于0.12—0.87(图2)。其中,生态环境2021CZ和2022CZ之间不存在显著相关,而生态环境2021CZ和2021YA之间表现出显著的正相关(<0.05),其余均表现出极显著的正相关(<0.01)。

表1 MC群体亲本及其RIL株高的表型分布
WJ:温江;CZ:崇州;YA:雅安;BLUP:最佳线性无偏预测;N:无重复值;*和**:在0.05和0.01水平差异显著。下同
WJ: Wenjiang; CZ: Chongzhou; YA: Ya’an; BLUP: Phenotype values based on the best linear unbiased prediction;N: no duplicate values; * and **: Significant difference at level 0.05 and 0.01. The same as below

表2 MC群体株高的方差分析
基于MC群体株高和产量相关性状的BLUP值,进一步评估表型相关性(表3)。其中,株高和有效分蘖数之间存在极显著的正相关(<0.01),相关系数为0.54。株高和旗叶宽之间存在显著的负相关(<0.05),相关系数为-0.16。株高和每穗粒数、每穗粒重、千粒重、旗叶长和开花期之间无显著相关性。
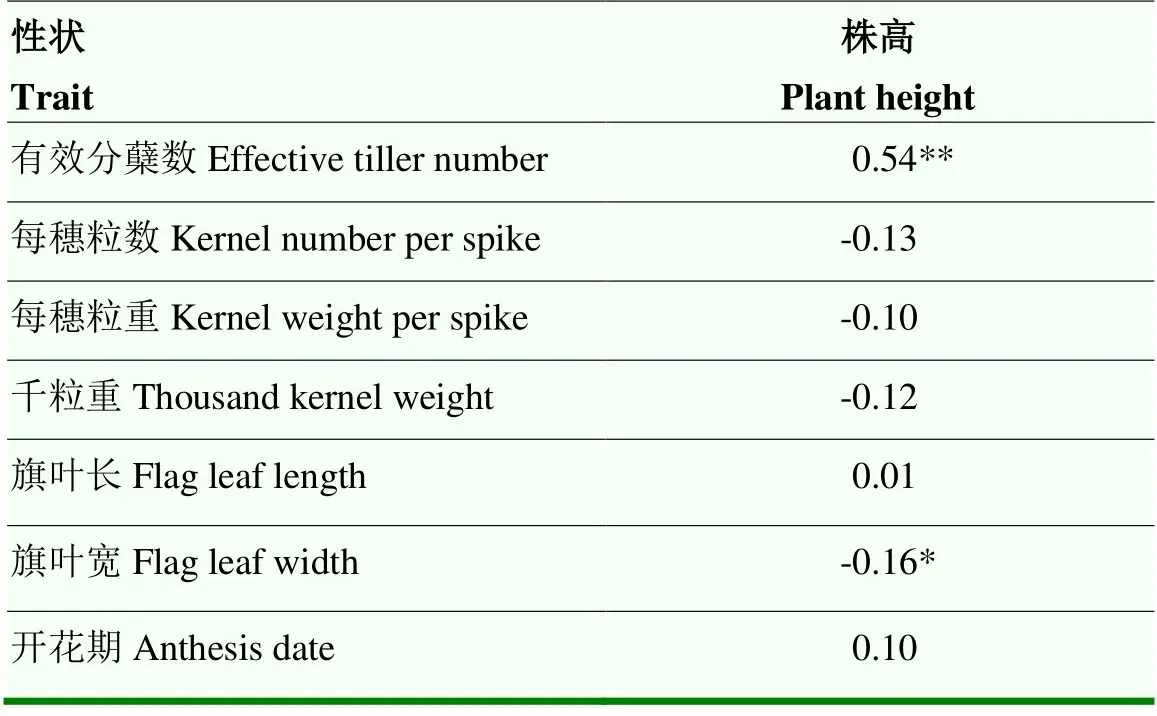
表3 MC群体株高与产量相关性状的相关性
2.3 QTL分析
基于16K SNP芯片所构建的遗传连锁图谱[22],对株高进行遗传定位。该遗传连锁图谱总共包含5 991个标记(总长2 813.26 cM,平均间距为2.45 cM/标记),99.82%标记的物理位置和遗传位置表现出一致性。结合MC群体2年5个生态环境的表型数据,共鉴定到8个QTL,分布在1A、3D(2)、4D、5A和7B(3)染色体,解释3.33%—25.56%的表型变异率(表4)。其中,定位于1A染色体的侧翼标记—,在4个生态环境以及BLUP值中被鉴定,可解释9.09%—25.56%的表型变异率,为主效QTL。定位于5A染色体的侧翼标记—,在2个生态环境以及BLUP值中被鉴定,最高解释13.09%的表型变异率,也为主效QTL。和的正效应位点均来源于CN16。其余6个QTL最高可解释7.02%的表型变异率,仅能在单个生态环境中被鉴定到,均为微效QTL。进一步的多环境QTL分析中可同时检测到和,表明其不易受到环境的影响,为稳定的主效QTL(表5)。
*和**:在0.05和0.01水平差异显著。下同 * and **: Significant difference at level 0.05 and 0.01. The same as below
2.4 株高主效QTL的遗传分析
基于株高主效QTL侧翼标记的基因型,分析其在单个生态环境条件下的遗传效应(图3)。携带正效应位点株系的株高,在除2021CZ之外的所有生态环境中,均极显著高于没有携带正效应位点的株系,增幅为6.91%— 15.24%。携带正效应位点株系的株高,在生态环境2022WJ和2022CZ以及BLUP中,均显著高于没有携带正效应位点的株系,增幅为3.54%—12.12%。
进一步利用MC群体株高的BLUP值,结合株高主效QTL侧翼标记的基因型,对其进行加性效应分析(图4)。MC群体可被划分为四类株系:同时携带和正效应位点的株系;仅携带或正效应位点的株系;没有携带和正效应位点的株系。显著性分析发现同时携带和正效应位点株系的株高显著高于仅携带单一正效应位点或没有携带任何正效应位点的株系。

表4 MC群体株高相关的QTL
遗传位置为LOD峰值处的数值;加性效应为正值表示对应QTL正效应位点来源于亲本,反之来源于亲本CN16
The genetic position is the value at the LOD peak; The positive value of additive effect indicates that the positive allele of corresponding QTL is contributed by, and the negative value of additive effect indicates that the positive allele of corresponding QTL is contributed by CN16

表5 MC群体株高相关的多环境QTL
LOD (A):加性和显性效应的阈值;LOD (AbyE):环境对加性和显性效应影响的阈值;(A):加性和显性效应的表型变异率;(AbyE):环境对加性和显性效应影响的表型变异率
LOD (A): Logarithm of the odds of additive and dominant effects; LOD (AbyE): Logarithm of the odds of the influence of environment on additive and dominant effects;(A): Phenotypic variation explained of additive and dominant effects;(AbyE): Phenotypic variation explained of environmental impact on additive and dominant effects
2.5 株高主效QTL对产量相关性状的影响
利用株高主效QTL侧翼标记的基因型,分析其对产量相关性状可能的影响(图5)。携带正效应位点株系的有效分蘖数(56.51%)极显著增加,每穗粒数(-11.26%)、每穗粒重(-13.04%)、千粒重(-5.47%)和旗叶宽(-2.85%)显著减少,开花期(-0.61%)极显著提前,而旗叶长无显著变化。携带正效应位点株系的有效分蘖数(10.57%)、每穗粒重(4.32%)和千粒重(2.92%)显著增加,开花期(1.07%)显著延迟,而每穗粒数、旗叶长和旗叶宽无显著变化。结果表明,正效应位点对产量影响可能以负效应为主,而正效应位点对产量可能有着积极的影响,更具育种价值。
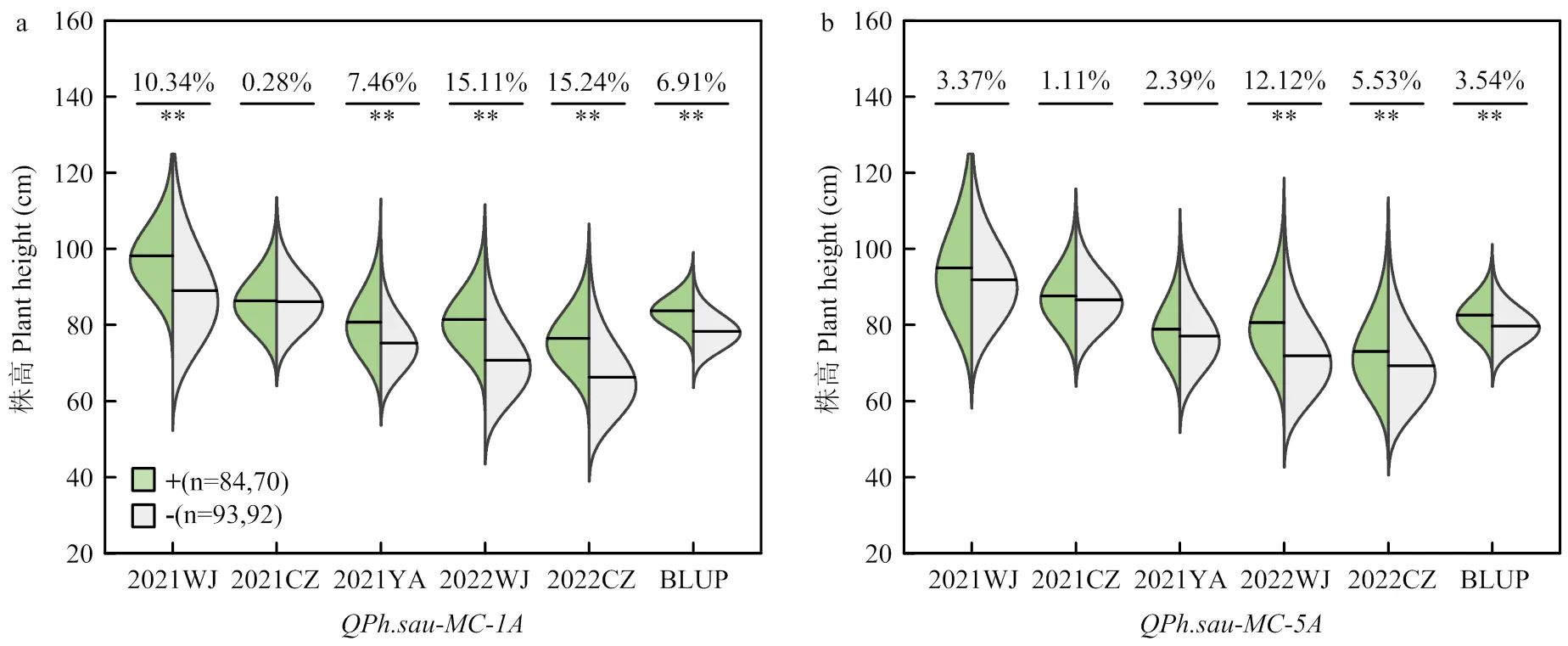
+和−:携带和不携带对应QTL正效应位点的株系;n:株系数。下同

不同小写字母表示差异显著性
3 讨论
3.1 小麦16K SNP芯片用于基因定位的案例
小麦16K SNP芯片基于GenoBaits®技术整合公开发布的重测序和外显子捕获数据,采用液体芯片技术和改进的目标测序系统进行基因分型[30]。该芯片确定了37 669个具有代表性的核心SNP标记,在染色体上分布均匀,并且价格低廉,已被用于小麦种质资源鉴定和遗传图谱构建。如,Qiu等[31]利用小麦16K SNP芯片鉴定近等基因系H962R和H962S的基因组相似程度,并进一步证明白粉病抗性基因对小麦农艺性状没有负面影响。Huang等[32]基于16K SNP芯片在1B、2A、3D和6B染色体检测到4个稳定遗传的条锈病抗性位点,与其紧密连锁的分子标记可用于分子辅助选择育种。Hu等[33]使用16K SNP芯片在小麦Tianmin 668的2B、2A和5A染色体分别检测到3个与白粉病抗性相关的位点、和,这些位点的鉴定有利于Tianmin 668在小麦抗白粉病新品种选育中的应用。然而,少有小麦16K SNP芯片在株高性状遗传分析上的利用。本研究基于16K SNP芯片鉴定到2个稳定的株高QTL(和),进一步证明该芯片在农艺性状遗传定位中的应用潜力,利用其连锁的分子标记可在小麦育种中,无需过高的成本即可选育不同株高类型的品系。
3.2 QTL物理区间比较分析
在CS RefSeq v1.0中检索本研究和前人研究中株高QTL的侧翼标记序列,通过比对其物理位置来确定是否为同一QTL[27-28]。小麦1A染色体暂未有矮秆基因被克隆,但已经有一些QTL被报道[2]。如,Yu等[34]在1A染色体检测到一个控制株高的QTL,定位于标记(6.42 Mb)附近。而本研究中的株高主效QTL定位于1A染色体(1.21—10.06 Mb)。其物理区间存在重叠,它们可能是同一个QTL。前人研究中已经在小麦5A染色体初步确定了2个矮秆基因。与5A染色体标记(558.34 Mb)紧密连锁[35]。被精细定位于5A染色体标记(698.50—698.98 Mb)[36]。此外,位于5A染色体标记_(519.88 Mb)附近[14]。位于5A染色体标记(549.97—558.94 Mb)[16]。位于5A染色体标记(478.64 Mb)附近[37]。而本研究株高主效QTL—位于5A染色体标记(563.83—572.98 Mb),物理距离间隔9.15 Mb。与已发现的株高QTL均无重叠物理区间,可能为新的QTL。考虑不同材料遗传背景差异,仍需对进行候选基因克隆来进一步确定其是否为新的QTL。
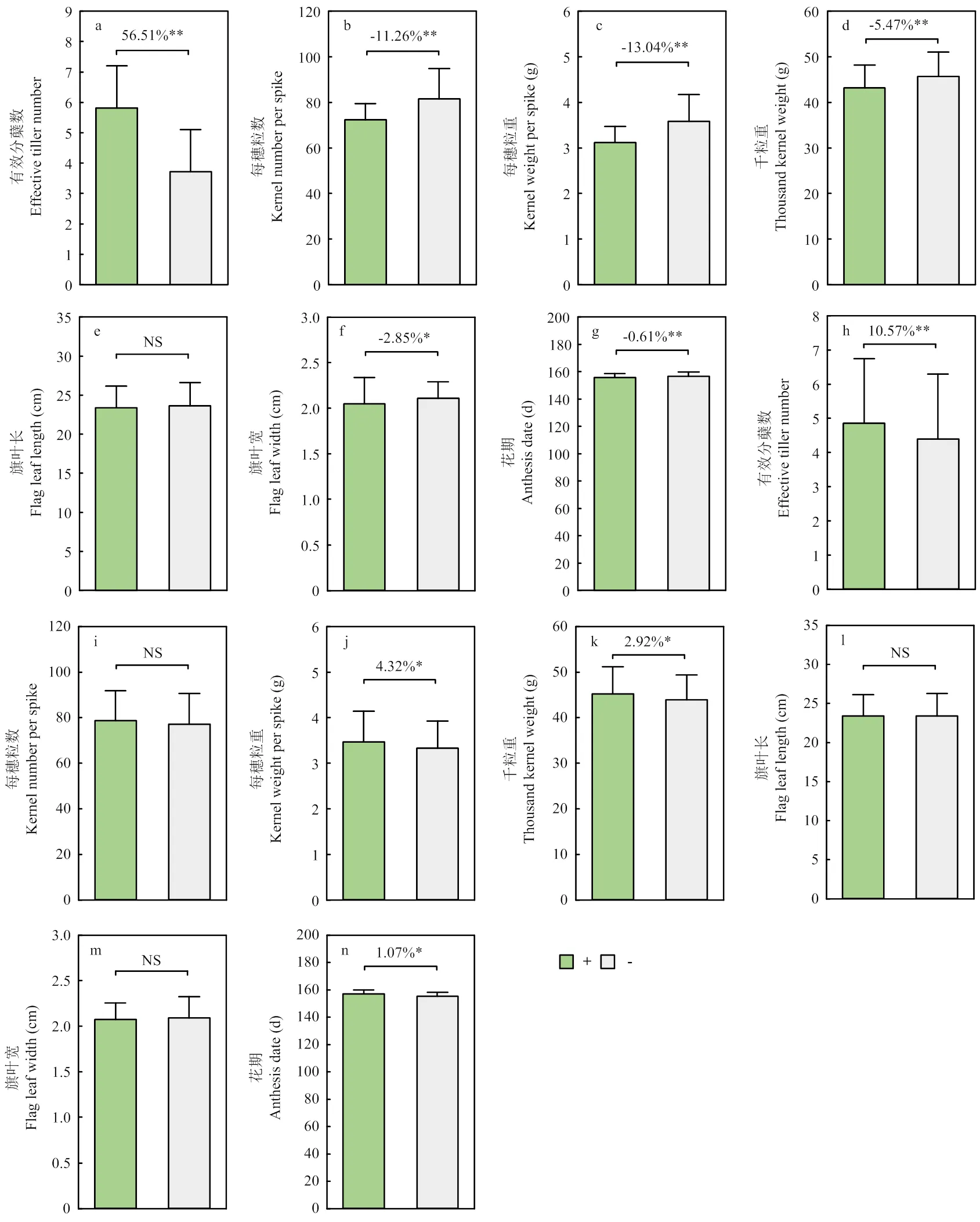
a—g:QPh.sau-MC-1A对产量相关性状的影响,+包含84个株系,−包含93个株系;h—n:QPh.sau-MC-5A对产量相关性状的影响,+包含70个株系,−包含92个株系
为确定和可能的候选基因,对其物理区间的同源基因进行鉴定。在CS RefSeq v1.0的物理区间为1.21—10.06 Mb,对应WEW_v2.0的物理区间为0.21—9.10 Mb,分别包含182和202个高可信度基因,其中97个基因为同源基因。在CS RefSeq v1.0的物理区间为563.83—572.98 Mb,对应WEW_v2.0的物理区间为562.48—568.18 Mb,分别包含115和109个高可信度基因,其中42个基因为同源基因。这些同源基因的鉴定有利于和后续的精细定位和基因克隆。
3.3 株高与其他产量相关性状的关系
株高与产量性状之间的关系在不同背景下表现并不一致。如,陈朝阳等[38]发现株高与抽穗期呈显著负相关,而与有效分蘖数、单株产量呈极显著负相关。赵秋月等[39]发现株高与千粒重、粒长和粒宽呈极显著负相关。Gao等[40]发现株高和千粒重表现出显著的正相关。而MC群体中株高与每穗粒数、每穗粒重和千粒重之间并无相关性,可能由于群体中株高与有效分蘖之间存在显著的正相关,影响到体内营养分配,导致穗部性状并无显著变化。调节小麦株高的矮秆基因,大多为“一因多效”基因,与产量相关性状也高度相关[2, 11]。如,绿色革命基因和,降低株高和提高小麦产量[3]。矮秆基因降低株高和增加有效分蘖数,但推迟抽穗期和成熟期,同时减少穗粒数和粒重[41]。对植株高度有负面影响,对有效分蘖数和籽粒大小有正面影响[42]。Zhang等[37]鉴定的株高主效QTL降低株高的同时降低千粒重和单株产量。本研究和正效应位点均可显著提高有效分蘖数,结合相关性分析,表明其可能是“一因多效”的位点,需要进一步对分蘖性状进行遗传分析来确定。正效应位点显著降低每穗粒数、每穗粒重和千粒重,而正效应位点显著增加每穗粒重和千粒重,暗示该位点在提高作物产量中的利用潜力。和效应差异表明株高和产量性状之间存在复杂的遗传机制,不同的矮秆基因或QTL具有不同的功能机制。需要进一步利用近等基因系等试验来验证可能的育种价值。
4 结论
定位到2个稳定的主效QTL:和。聚合两者正效应位点株系的株高显著高于仅携带单一正效应位点或没有携带任何正效应位点的株系。正效应位点可极显著增加有效分蘖数,显著减少每穗粒数、每穗粒重、千粒重和旗叶宽,促进开花期提前。正效应位点可显著增加有效分蘖数、每穗粒重和千粒重,延迟开花期。
[1] SHEWRY P R. Wheat. Journal of Experimental Botany, 2009, 60(6): 1537-1553.
[2] 吕广德, 靳雪梅, 郭营, 赵岩, 钱兆国, 吴科, 李斯深. 小麦株高分子遗传学研究进展. 植物遗传资源学报, 2021, 22(3): 571-582.
Lü G D, JIN X M, GUO Y, ZHAO Y, QIAN Z G, WU K, LI S S. Advances in molecular genetics of wheat plant height. Journal of Plant Genetic Resources, 2021, 22(3): 571-582. (in Chinese)
[3] KHUSH G S. Green revolution: preparing for the 21st century. Genome, 1999, 42(4): 646-655.
[4] SAINI D K, SRIVASTAVA P, PAL N, GUPTA P K. Meta-QTLs, ortho-meta-QTLs and candidate genes for grain yield and associated traits in wheat (L.). Theoretical and Applied Genetics, 2022, 135(3): 1049-1081.
[5] PENG J R, RICHARDS D E, HARTLEY N M, MURPHY G P, DEVOS K M, FLINTHAM J E, BEALES J, FISH L J, WORLAND A J, PELICA F, SUDHAKAR D, CHRISTOU P, SNAPE J W, GALE M D, HARBERD N P. ‘Green revolution’genes encode mutant gibberellin response modulators. Nature, 1999, 400(6741): 256-261.
[6] CHAI L L, XIN M M, DONG C Q, CHEN Z Y, ZHAI H J, ZHUANG J H, CHENG X J, WANG N J, GENG J, WANG X B, BIAN R L, YAO Y Y, GUO W L, HU Z R, PENG H R, BAI G H, SUN Q X, SU Z Q, LIU J, NI Z F. A natural variation in Ribonuclease H-like gene underliesto confer Green Revolution trait in wheat. Molecular Plant, 2022, 15(3): 377-380.
[7] XIONG H C, ZHOU C Y, FU M Y, GUO H J, XIE Y D, ZHAO L S, GU J Y, ZHAO S R, DING Y P, LI Y T, ZHANG J Z, WANG K, LI X J, LIU L X. Cloning and functional characterization of, a ‘Green Revolution’ replacement gene in wheat. Molecular Plant, 2022, 15(3): 373-376.
[8] BORRILL P, MAGO R, XU T, FORD B, WILLIAMS S J, DERKX A, BOVILL W D, HYLES J, BHATT D, XIA X, MACMILLAN C, WHITE R, BUSS W, MOLN R I, WALKOWIAK S, OLSEN O-A, DOLEŽEL J, POZNIAK C J, SPIELMEYER W. An autoactive NB-LRR gene causesdwarfism in wheat. Proceedings of the National Academy of Sciences of the united states of america, 2022, 119(48): e2209875119.
[9] WANG C, BAO Y J, YAO Q, LONG D, XIAO X, FAN X, KANG H Y, ZENG J, SHA L N, ZHANG H Q, WU D D, ZHOU Y H, ZHOU Q, WANG Y, CHENG Y R. Fine mapping of the reduced height genein tetraploid wheat landrace Jianyangailanmai (L.). Theoretical and Applied Genetics, 2022, 135(10): 3643-3660.
[10] TIAN X L, XIA X C, XU D A, LIU Y Q, XIE L, HASSAN M A, SONG J, LI F J, WANG D S, ZHANG Y, HAO Y F, LI G Y, CHU C C, HE Z H, CAO S H., an ancient variation of, reduces plant height without yield penalty in wheat. The New Phytologist, 2022, 233(2): 738-750.
[11] MO Y, VANZETTI L S, HALE I, SPAGNOLO E J, GUIDOBALDI F, AL-OBOUDI J, ODLE N, PEARCE S, HELGUERA M, DUBCOVSKY J. Identification and characterization of, a locus on chromosome arm 6AS affecting wheat plant height, heading time, and spike development. Theoretical and Applied Genetics, 2018, 131(10): 2021-2035.
[12] ZHANG J L, LI C X, ZHANG W J, ZHANG X Q, MO Y J, TRANQUILLI G E, VANZETTI L S, DUBCOVSKY J. Wheat plant height locusencodes a PLATZ transcription factor that interacts with DELLA (). bioRxiv, 2023.01.05.522836. doi: 0.1101/2023.01.05.522836.
[13] WANG C J, ZHANG L L, XIE Y D, IRSHAD A, GUO H J, GU J Y, ZHAO L S, XIONG H C, ZHAO S R, WANG C S, LIU L X. Agronomic trait analysis and genetic mapping of a new wheat semidwarf gene. Internstional Journal of Molecular Sciences, 2022, 24(1): 583.
[14] 胡文静, 李东升, 裔新, 张春梅, 张勇. 小麦穗部性状和株高的QTL定位及育种标记开发和验证. 作物学报, 2022, 48(6): 1346-1356.
HU W J, LI D S, YI X, ZHANG C M, ZHANG Y. Molecular mapping and validation of quantitative trait loci for spike-related traits and plant height in wheat. Acta Agronomica Sinica, 2022, 48(6): 1346-1356. (in Chinese)
[15] 关攀锋, 逯腊虎, 刘刚, 汪永法, 辛明明, 胡兆荣, 姚颖垠, 倪中福, 孙其信, 彭惠茹. 一个小麦强优势组合的杂种优势遗传基础解析. 科学通报, 2022, 67(26): 3207-3220.
GUAN P F, LU L H, LIU G, WANG Y F, XIN M M, HU Z R, YAO Y Y, NI Z F, SUN Q X, PENG H R. Genetic basis of heterosis in a common wheat cross with strong-heterosis. Chinese Science Bulletin, 2022, 67(26): 3207-3220. (in Chinese)
[16] 廖思敏, 冯波, 徐智斌, 樊小莉, 周强, 纪光思, 刘小凤, 余琴, 王涛. 基于55K SNP芯片检测小麦株高QTL. 应用与环境生物学报, 2022, 28(3): 576-581.
LIAO S M, FENG B, XU Z B, FAN X L, ZHOU Q, JI G S, LIU X F, YU Q, WANG T. Detection of QTLs for plant height in wheat based on the 55K SNP array. Chinese Journal of Applied and Environmental Biology, 2022, 28(3): 576-581. (in Chinese)
[17] LIU H, SHI Z P, MA F F, XU Y F, HAN G H, ZHANG J P, LIU D C, AN D G. Identification and validation of plant height, spike length and spike compactness loci in common wheat (L.). BMC Plant Biology, 2022, 22(1): 568.
[18] REBETZKE G J, ELLIS M H, BONNETT D G, MICKELSON B, CONDON A G, RICHARDS R A. Height reduction and agronomic performance for selected gibberellin-responsive dwarfing genes in bread wheat (L.). Field Crops Research, 2012, 126: 87-96.
[19] 李聪, 马建, 刘航, 丁浦洋, 杨聪聪, 张涵, 秦娜娜, 兰秀锦. 基于小麦55K SNP芯片检测小麦穗长和株高性状QTL. 麦类作物学报, 2019, 39(11): 1284-1292.
LI C, MA J, LIU H, DING P Y, YANG C C, ZHANG H, QIN N N, LAN X J. Detection of QTLs for spike length and plant height in wheat based on 55K SNP array. Journal of Triticeae Crops, 2019, 39(11): 1284-1292. (in Chinese)
[20] 陈士强, 张容, 王建华, 陈刚, 刘建凤, 陈秀兰, 何震天. 小麦赤霉病抗性与株高及穗部性状的相关性研究. 江西农业学报, 2020, 32(6): 23-29.
CHEN S Q, ZHANG R, WANG J H, CHEN G, LIU J F, CHEN X L, HE Z T. Studies on correlation betweenhead blight resistance and plant height, ear traits of wheat. Acta Agriculturae Jiangxi, 2020, 32(6): 23-29. (in Chinese)
[21] GERVAIS L, DEDRYVER F, MORLAIS J Y, BODUSSEAU V, NEGRE S, BILOUS M, GROOS C, TROTTET M. Mapping of quantitative trait loci for field resistance tohead blight in an European winter wheat. Theoretical and Applied Genetics, 2003, 106(6): 961-970.
[22] ZHOU J G, LI W, YANG Y Y, XIE X L, LIU J J, LIU Y L, TANG H, DENG M, XU Q, JIANG Q, CHEN G Y, QI P, JIANG Y F, CHEN G D, HE Y J, REN Y P, TANG L W, GOU L, ZHENG Y L, WEI Y M, MA J. A promising QTLlikely superior tofor wheat spikelet number per spike shows no adverse effects on yield-related traits. bioRxiv, 2023. doi: 10.1101/2023.02.17.528911.
[23] LIU J J, LUO W, QIN N N, DING P Y, ZHANG H, YANG C C, MU Y, TANG H P, LIU Y X, LI W, JIANG Q T, CHEN G Y, WEI Y M, ZHENG Y L, LIU C J, LAN X J, MA J. A 55 K SNP array-based genetic map and its utilization in QTL mapping for productive tiller number in common wheat. Theoretical and Applied Genetics, 2018, 131(11): 2439-2450.
[24] 唐华苹, 陈黄鑫, 李聪, 苟璐璐, 谭翠, 牟杨, 唐力为, 兰秀锦, 魏育明, 马建. 基于55K SNP芯片的普通小麦穗长非条件和条件QTL分析. 中国农业科学, 2022, 55(8): 1492-1502.
TANG H P, CHEN H X, LI C, GOU L L, TAN C, MU Y, TANG L W, LAN X J, WEI Y M, MA J. Unconditional and conditional qtl analysis of wheat spike length in common wheat based on 55K SNP array. Scientia Agricultura Sinica, 2022, 55(8): 1492-1502. (in Chinese)
[25] 陈黄鑫, 李聪, 吴坤燕, 王岳, 牟杨, 唐华苹, 唐力为, 兰秀锦, 马建. 四倍体小麦株高和穗长性状的QTL定位及其遗传效应分析. 麦类作物学报, 2022, 42(7): 799-807.
CHEN H X, LI C, WU K Y, WANG Y, MU Y, TANG H P, TANG L W, LAN X J, MA J. Detection of QTLs for plant height and spike length in tetraploid wheat and analysis of their genetic effect. Journal of Triticeae Crops, 2022, 42(7): 799-807. (in Chinese)
[26] MCINTOSH R, YAMAZAKI Y, DUBCOVSKY J, ROGERS J, MORRIS C, APPELS R. Catalogue of gene symbols for wheat; proceedings of the proceedings of the 12th international wheat genetics symposium, F, 2013. Yokohama Japan.
[27] INTERNATIONAL WHEAT GENOME SEQUENCING CONSORTIUM(IWGSC). Shifting the limits in wheat research and breeding using a fully annotated reference genome. Science, 2018, 361(6403): eaar7191.
[28] MA S W, WANG M, WU J H, GUO W L, CHEN Y M, LI G W, WANG Y P, SHI W M, XIA G M, FU D L, KANG Z S, NI F. WheatOmics: A platform combining multiple omics data to accelerate functional genomics studies in wheat. Molecular Plant, 2021, 14(12): 1965-1968.
[29] ZHU T T, WANG L, RODRIGUEZ J C, DEAL K R, AVNI R, DISTELFELD A, MCGUIRE P E, DVORAK J, LUO M C. Improved genome sequence of wild emmer wheat zavitan with the aid of optical maps. G3 Genes|Genomes|Genetics, 2019, 9(3): 619-624.
[30] GUO Z F, YANG Q N, HUANG F F, ZHENG H J, SANG Z Q, XU Y F, ZHANG C, WU K S, TAO J J, PRASANNA B M, OLSEN M S, WANG Y B, ZHANG J N, XU Y B. Development of high-resolution multiple-SNP arrays for genetic analyses and molecular breeding through genotyping by target sequencing and liquid chip. Plant Communications, 2021, 2(6): 100230.
[31] QIU D, HUANG J, GUO G H, HU J H, LI Y H, ZHANG H J, LIU H W, YANG L, ZHOU Y, YANG B Z, ZHANG Y D, LIU Z Y, LI H J. Thegene has no negative effect on wheat agronomic performance: Evidence from newly established near-isogenic lines. Frontiers in Plant Science, 2022, 13: 918559.
[32] HUANG S, ZHANG Y B, REN H, LI X, ZHANG X, ZHANG Z Y, ZHANG C L, LIU S J, WANG X T, ZENG Q D, WANG Q L, SINGH R P, BHAVANI S, WU J H, HAN D J, KANG Z S. Epistatic interaction effect between chromosome 1BL () and a novel locus on 2AL facilitating resistance to stripe rust in Chinese wheat Changwu 357-9. Theoretical and Applied Genetics, 2022, 135(7): 2501-2513.
[33] HU J H, GEBREMARIAM T G, ZHANG P, QU Y F, QIU D, LIU H W, SHI X H, LI Y H, WU Q H, LUO M, YANG L J, ZHANG H J, YANG L, ZHOU Y, LIU Z Y, WANG B T, LI H J. Resistance to powdery mildew is conferred by different genetic loci at the adult-plant and seedling stages in winter wheat line Tianmin 668. Plant Disease, 2022. doi: 10.1094/PDIS-11-22-2633-RE.
[34] YU M, LIU Z H, YANG B, CHEN H, ZHANG H, HOU D B. The contribution of photosynthesis traits and plant height components to plant height in wheat at the individual quantitative trait locus level. Scientific Reports, 2020, 10(1): 12261.
[35] ELLIS M H, REBETZKE G J, AZANZA F, RICHARDS R A, SPIELMEYER W. Molecular mapping of gibberellin-responsive dwarfing genes in bread wheat. Theoretical and Applied Genetics, 2005, 111(3): 423-430.
[36] SUN L H, YANG W L, LI Y F, SHAN Q Q, YE X B, WANG D Z, YU K, LU W W, XIN P Y, PEI Z, GUO X L, LIU D C, SUN J Z, ZHAN K H, CHU J F, ZHANG A M. A wheat dominant dwarfing line with, which reduces stem cell length and affects gibberellic acid synthesis, is a 5AL terminal deletion line. The Plant Journal, 2019, 97(5): 887-900.
[37] ZHANG N, FAN X L, CUI F, ZHAO C H, ZHANG W, ZHAO X Q, YANG L J, PAN R Q, CHEN M, HAN J, JI J, LIU D C, ZHAO Z W, TONG Y P, ZHANG A M, WANG T, LI J M. Characterization of the temporal and spatial expression of wheat (L.) plant height at the QTL level and their influence on yield-related traits. Theoretical and Applied Genetics, 2017, 130(6): 1235-1252.
[38] 陈朝阳, 易晓余, 熊君, 杨珂, 许珂, 匡成浩, 张志鹏, 丁丽, 蒲至恩, 陈国跃, 李伟. 118个小麦新品系的农艺性状和分子标记评价. 四川农业大学学报, 2018, 36(6): 722-728.
CHEN Z Y, YI X Y, XIONG J, YANG K, XU K, KUANG C H, ZHANG Z P, DING L, PU Z E, CHEN G Y, LI W. Evaluation of agronomic traits and molecular markers in 118 new wheat strains. Journal of Sichuan Agricultural University, 2018, 36(6): 722-728. (in Chinese)
[39] 赵秋月, 张咪咪, 孙清鹏, 韩俊. 小麦微核心种质株高及籽粒性状遗传分析. 安徽农业科学, 2022, 50(7): 37-40, 45.
ZHAO Q Y, ZHANG M M, SUN Q P, HAN J. Genetic analysis of pant height and grain traits of wheat micro-core germplasms. Journal of Anhui Agricultural Sciences, 2022, 50(7): 37-40, 45. (in Chinese)
[40] GAO F F, WEN W E, LIU J D, RASHEED A, YIN G H, XIA X C, WU X X, HE Z H. Genome-wide linkage mapping of qtl for yield components, plant height and yield-related physiological traits in the chinese wheat cross Zhou 8425B/Chinese spring. Frontiers in Plant Science, 2015, 6: 1099.
[41] DAOURA B G, CHEN L, HU Y G. Agronomic traits affected by dwarfing genein common wheat (L.). Australian Journal of Crop Science, 2013, 7(9): 1270-1276.
[42] LU Y, XING L P, XING S J, HU P, CUI C F, ZHANG M Y, XIAO J, WANG H Y, ZHANG R Q, WANG X E, CHEN P D, CAO A Z. Characterization of a putative new semi-dominant reduced height gene,, in wheat (L.). Journal of Genetics and Genomics, 2015, 42(12): 685-698.
QTL Identification and Genetic Analysis of Plant Height in Wheat Based on 16K SNP array
YAO Qifu1, CHEN Huangxin2, ZHOU Jieguang2, MA Ruiying3, Deng Liang3, Tan Chenxinyu3, SONG Jinghan4, Lü Jijuan5, MA Jian
1College of Agroforestry Engineering and Planning, Tongren University/Guizhou Key Laboratory of Biodiversity Conservation and Utilization in the Fanjing Mountain Region, Tongren 554300, Guizhou;2Triticeae Research Institute, Sichuan Agricultural University, Chengdu 611130;3College of Agronomy, Sichuan Agricultural University, Chengdu 611130;4Beijing Foreign Studies University, Beijing 100089;5Sichuan provincial seed station, Chengdu 610041
【Objective】There is a close relationship between plant height (PH) and yield. The aim of this study is to further explore quantitative trait loci (QTL) of PH with breeding value in wheat and analyze the genetic effects of major QTL for PH on other yield related traits toward to providing a theoretical basis for molecular breeding. 【Method】A recombinant inbred line population (MC) derived from a cross between the natural mutantand Chuannong 16 (CN16) was used for QTL analysis. During 2020 to 2022, planting and PH phenotype identification were conducted at five environments in Wenjiang, Chongzhou, and Ya’an of Sichuan Province. The high-quality genetic linkage map constructed using the 16K SNP array was used for QTL mapping of PH. Genotypes of flanking markers of major QTL for PH were used to analyze the genetic effects of positive alleles on yield related traits and evaluate the potentiality of QTL for yield improvement. 【Result】Eight QTL controlling PH were identified on chromosomes 1A, 3D, 4D, 5A, and 7B, respectively. Among them, two stable and major QTL,and, were located, which explained 9.09% to 25.56% and 3.91% to 13.09% of the phenotypic variation rate, respectively. Their positive alleles were all from CN16. The additive effect analysis showed that PH of the lines carrying positive alleles fromandwas significantly higher than that of the lines carrying only a single positive allele or none. Correlation analysis showed that PH has a significantly positive correlation with effective tiller number (ETN), a significantly negative correlation with flag leaf width (FLW), and no significant correlation with kernel number per spike (KNPS), kernel weight per spike (KWPS), thousand kernel weight (TKW), flag leaf length (FLL) and anthesis date (AD). Genetic effects analysis showed that positive allele ofhad a significant effect on improving ETN (56.51%), a significant effect on decreasing KNPS (-11.26%), KWPS (-13.04%), TKW (-5.47%), and FLW (-2.85%), and a significant effect on advancing AD (-0.61%). Positive allele ofhad a significant effect on improving ETN (10.57%), KNPS (4.32%), and TKW (2.92%), and a significant effect on delaying AD (1.07%). 【Conclusion】A major QTLfor PH was mapped on chromosome 5A, and its positive allele significantly increased ETN, KNPS, and TKW, indicating that it may have a positive impact on yield.
wheat; 16K SNP array; QTL; plant height; yield

10.3864/j.issn.0578-1752.2023.12.001
2023-02-14;
2023-03-17
贵州省科技计划基础研究项目(黔科合基础-ZK[2021]一般131)
姚琦馥,E-mail:yaoqifu@126.com。陈黄鑫,E-mail:1252153393@qq.com。姚琦馥和陈黄鑫为同等贡献作者。通信作者马建,E-mail:jianma@sicau.edu.cn。通信作者吕季娟,E-mail:sccdwhljj@163.com
(责任编辑 李莉)
