189份桃种质肉质性状形成相关位点基因型鉴定及组合分析
2023-07-09王朝晖李勇曹珂朱更瑞方伟超陈昌文王新卫吴金龙王力荣
王朝晖,李勇,曹珂,朱更瑞,方伟超,陈昌文,王新卫,吴金龙,王力荣
189份桃种质肉质性状形成相关位点基因型鉴定及组合分析
王朝晖,李勇,曹珂,朱更瑞,方伟超,陈昌文,王新卫,吴金龙,王力荣
中国农业科学院郑州果树研究所,郑州 450009
【目的】通过分子标记与生物信息学手段,鉴定189份桃种质与肉质形成相关的F-M基因座、启动子区转座子插入、编码区9 bp插入的基因型及3个位点组合情况,为深入研究桃果实肉质形成机制及育种亲本选配提供理论基础。【方法】通过PCR扩增、竞争性等位基因特异PCR(kompetitive allele specific PCR,KASP)、高分辨率溶解曲线(high resolution melting analysis,HRM)技术,对F-M基因座倍型、转座子插入、9 bp插入进行检测;同时结合重测序数据利用生物信息学方法进一步验证,从而准确鉴定189份桃种质肉质性状形成相关位点基因型及组合情况。【结果】F-M基因座编码多聚半乳糖醛酸酶(endo-PG)的与是调控桃果实肉质形成的两个重要基因。189份桃种质中,与分别有159份(84%)、99份(52%)。F-M基因座共发现H0、H1、H2、H3 4种单倍型,占比分别为33%、39%、4%、24%。在F-M基因座,不溶质粘核以H3H3、H2H3组合为主,溶质粘核以H0H0、H0H1组合为主,溶质离核以H1H1、H0H1组合为主。发现18份含有硬质基因型的种质,在启动子区含有转座子纯合插入。HRM结果表明,编码区9 bp插入纯合(早熟)有45份种质、插入杂合(中熟)有71份种质、无插入(晚熟)有73份种质,中熟与晚熟种质占比77%;其中6份种质基因型与表型差别较大。不溶质粘核、溶质粘核、溶质离核3种常见肉质类型,在3个位点最多的基因型组合分别是mmffHdHdI、MMffHdHdI、MMFFHdHdL。【结论】通过分子试验与生物信息学相结合进一步证实了F-M基因座存在4种单倍型;发现18份含有硬质基因型的种质;开发了一种用于鉴定桃成熟期的分子标记;鉴定出不同种质在肉质相关位点的基因型组合形式。
桃;肉质;基因型鉴定;基因型组合
0 引言
【研究意义】桃[(L.) Batsch]是蔷薇科李属重要的经济树种,与其他果树相比具有丰富的肉质类型[1]。桃果实肉质是成熟期中果皮薄壁细胞经历一系列生理与生化变化的结果,也是影响消费者评判桃品质的一个重要因素[2]。现阶段育种过程中,对桃果实肉质类型的评判往往以育种者的感官评价和简单的生理指标为主,限制了对肉质性状的精准评价。鉴于此,通过分子试验与生物信息学手段,分析桃基因组中与肉质性状形成的相关位点。从基因型角度评价肉质类型,对构建肉质评价体系、明晰骨干亲本肉质类型及育种亲本选配具有重要指导意义。【前人研究进展】目前,对质量性状控制的肉质类型研究较为透彻,主要分为溶质/不溶质、硬质/非硬质两大类;而数量性状控制的慢溶质以及与成熟期有关的慢成熟类型肉质研究相对较少[3-5]。BAILEY等[6]最早发现溶质(Melting)与不溶质(Non-melting)为一对等位基因控制,并且溶质(M)为显性性状,后续许多研究表明肉质形成主要与细胞壁果胶成分降解有关,其中编码多聚半乳糖醛酸酶()被视为主要候选基因[7]。在桃参考基因组发布后,将定位到第4号染色体上的F-M基因座;同时发现该位点不仅调控溶质与不溶质,还同时控制粘核(Clingstone)与离核(Freestone)性状的形成,是肉质形成的关键位点[8]。GU等[9]提出F-M基因座存在H1、H2、H3三种单倍型,相互组合形成溶质离核、溶质粘核、不溶质粘核3种肉质表型;但NAKANO等[10]研究发现F-M基因座该位点存在M0、M1、M2、M3这4种基础的单倍型,并在4种单倍型基础上共发现了11种亚单倍型。硬质(stony hard)类型肉质由YOSHIDA等[11]首先报道,同时表明该性状由单基因控制。后续研究发现硬质与非硬质主要与乙烯合成不足关系密切,经外源乙烯处理后的结果表明,硬质基因独立于溶质/不溶质基因,且对溶质/不溶质基因具有上位作用[12]。Pan等[13]最早认为上内含子区TC重复与硬质有关;曾文芳等[14]与国外学者TATSUKI等[15]认为启动子区域存在的CACTA型转座子插入与硬质有关;与此同时,孟君仁等[16]基于SNP变异,开发了与硬质有关的KASP分子标记,可用于高效、快速、低成本鉴定硬质基因型。田间育种实践中发现,桃果实成熟期对肉质类型有一定的影响。早熟品种肉质普遍较差、往往没来得及离核就变软[17]。PIRONA等[18]发现第3个外显子上9 bp的插入与桃早熟表型关联;余银梅[19]也利用199份种质在众多成熟期标记中筛选到-indel是唯一可以区分不同成熟期的标记。综上所述,桃果实肉质性状的形成与基因组上3个关键位点有关,涉及众多复杂生理与生化过程。【本研究切入点】桃肉质性状由基因组上多个位点共同决定,尽管已有研究利用分子标记对不同肉质类型分别开展鉴定,但未对F-M基因座、启动子区、编码区indel的3个关键位点同时进行综合分析。【拟解决的关键问题】基于分子试验与重测序数据结合,明确F-M基因座单倍型种类;鉴定189份桃种质中包含F-M基因座、启动子区转座子插入、编码区indel三个肉质性状形成关键位点的基因型及组合情况,为开发肉质性状分子标记、建立不同肉质评价标准及育种利用提供科学指导。
1 材料与方法
试验于2021—2022年在中国农业科学院郑州果树研究所进行。
1.1 材料与取样
采样于2021年9月进行,试验中的189份种质(附表1)均取自国家园艺种质资源库桃种质资源圃。其中147份种质具有重测序数据,在笔者课题组前期研究中已上传至NCBI网站(登录号为PRJNA388029、PRJNA630113)。在材料选择时优先考虑肉质性状明显的种质、名特优的地方种质、肉质类型较为公认的种质、广泛作为育成品种亲本的种质。
1.2 全基因组DNA提取
选取新鲜的嫩叶约30 mg装入2 mL离心管内,放入液氮内充分冷冻。采用CTAB法提取基因组DNA,具体流程参考何雪娇等[20]。提取后,用NanoDrop 1000(Thermo Scientific)紫外分光光度计检测DNA提取质量和浓度,然后用无菌水将DNA浓度稀释到100 ng∙μL-1左右便于后续试验。
1.3 引物设计及PCR扩增
本试验所用到的引物序列详细信息见表1,引物均由上海生工生物工程技术服务有限公司合成,PCR反应酶为南京诺维赞公司高保真Mix。PCR反应总体系为10 μL:Mix 5 μL,上、下游引物各0.4 μL(浓度为10 μmol∙μL-1),ddH2O为3.2 μL,模板为1 μL。PCR扩增仪器为Eppendorf Master cycler,反应程序为:95 ℃ 3 min;95 ℃ 15 s;退火(表1)30 s,72 ℃延伸,共35个循环;72 ℃ 5 min;4 ℃保存。

表1 PCR试验中所用到的引物
1.4 KASP及HRM试验流程
KASP试验流程及引物序列参考孟君仁等[16],仪器为Light Cycler 480(LC480);KASP酶为广州固德生物有限公司的FLU-ARMS for KASP 2×PCR Mix,反应体系为:Mix 5 μL,FAM、HEX引物各0.1 μL,Common引物0.3 μL,ddH2O 5 μL,模板0.5 μL。
高分辨率溶解曲线(HRM)试验引物序列为:NAC-F:TCAGAACTCAGCGGGTTGAT;NAC-R:TGCACTCCCCTTCTACTTTCA。所用酶为北京百瑞极公司的Fast Super Eva Green qPCR Master Mix饱和试剂,反应体系为:Mix 10 μL,上、下游引物各0.4 μL,ddH2O 8.7 μL,模板0.5 μL,反应在Light Cycler 480(LC480)上进行,具体程序参考殷豪等[21]。
1.5 重测序数据分析流程
重测序数据使用FastQC软件(version 0.11.9)[22]质控后,去除接头和低质量序列。通过BWA软件(version 0.7.16a)[23]将质控后的序列比对到桃参考基因组上,使用参数‘bwa mem-M-R’生成sam文件;利用Samtools软件(version 1.14)[24]将sam文件转换并排序生成sort.bam文件,同时构建索引文件。用Picard(version 2.27.1)软件Mark DuplicatesREMOVE_DUPLICATES=true程序,将sort.bam文件去除重复得到sort.rm-dedup.bam文件,建立索引后下载到本地。利用IGV(Integrative Genomics Viewer)软件对F-M基因座、硬质相关的SNP位点和-TE区、-indel进行可视化分析。对于F-M基因座单倍型检测,以Delly(version 0.78)[25]软件默认参数的检测结果为主,同时借助IGV软件[26]可视化sort.rm- dedup.bam文件辅助分析。
2 结果
2.1 溶质/不溶质与粘核/离核基因鉴定结果
桃溶质/不溶质与粘核/离核性状的候选基因位于4号染色体上,分别由()与()控制[11]。通过设计引物,分别扩增与(图1),产物分别为3 066 bp与3 353 bp。
189份种质中159份有(图2-A),基因型与表型一致率93%。其中11份表型为不溶质,但含有;有2份表型为溶质,但不含。189份种质中99份有(图2-B),基因型与表型一致率88%。其中21份表型为粘核,但含有。
2.2 F-M基因座单倍型种类鉴定
桃4号染色体上存在一个大约71 kb(Pp04: 19022760.. 19094035)的F-M基因座,是控制肉质形成的关键位点;该位点存在不同类型结构变异(structure variation,SV),不同SV之间相互组合形成了桃果实的溶质离核、溶质粘核、不溶质粘核3种肉质表型[9]。
本研究通过重测序数据SV鉴定、IGV可视化分析、PCR试验3种方法,对F-M基因座的单倍型种类进行鉴定,结果表明该位点存在4种单倍型(Haplotype),即H0、H1、H2、H3(图3)。桃是二倍体植物,4种单倍型之间可以有10种组合方式。
桃参考基因组(Lovell)是典型的H1单倍型代表,在F-M基因座区域存在、、、、、、、这8个基因;H0单倍型、、、与参考基因组相比只有部分存在,并且在与之间有一个8.8 kb的大片段缺失;H2单倍型在与之间存在一个12.8 kb的缺失,与在此区间之内;H3单倍型在至存在一个70.5 kb的缺失。H0、H2、H3倍型可通过引物进行鉴定(图4);在189份种质中,H0、H1、H2、H3单倍型占比分别为33%、39%、4%和24%(图4)。
2.3 硬质相关YUC11-TE基因型鉴定
本研究首先对硬质相关的一个SNP位点(图5),采用KASP基因分型技术对Pp06: 14090689位点进行基因型鉴定(图6);然后利用重测序数据分析SNP位点、启动子区转座子插入基因型;最后通过PCR试验方法对具有硬质基因型的种质再次验证,189份桃种质硬质基因型详细结果见附表1。
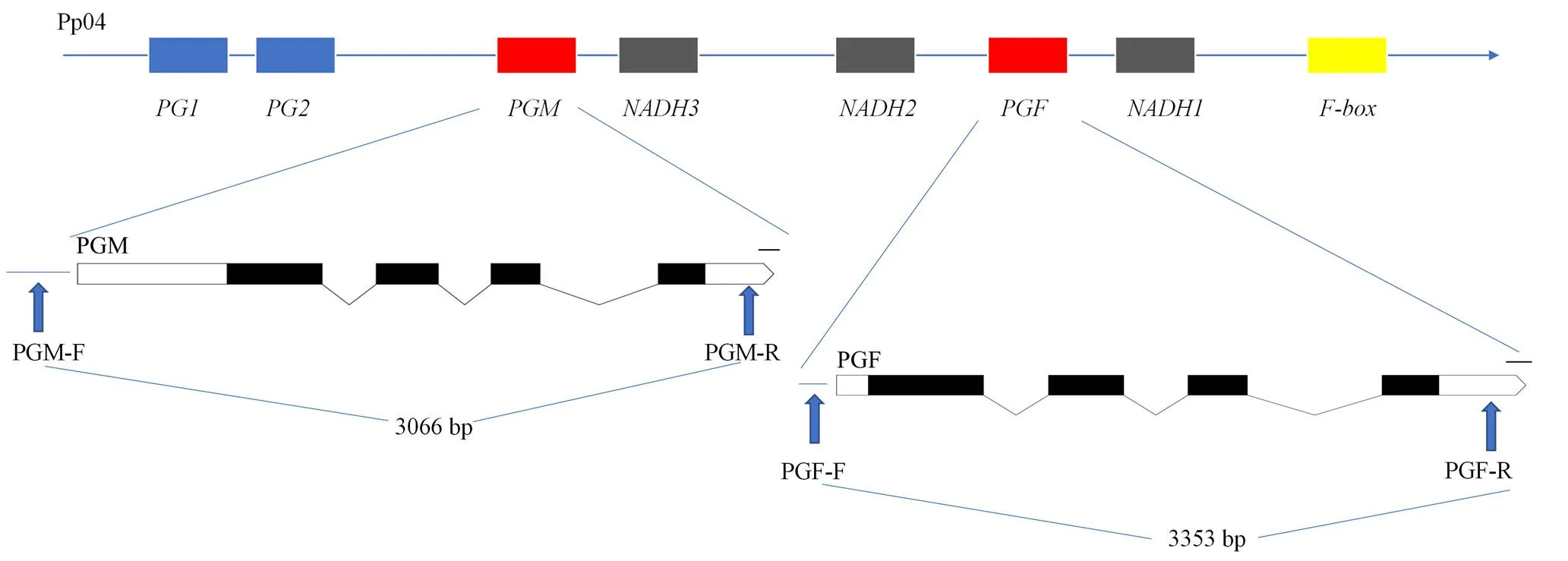
图1 检测PGM与PGF的分子标记

M:Maker 5000;N:空白对照。A:189份种质PGM扩增结果;B:189份种质PGF扩增结果

S:Short,与参考基因组相比该基因不完整,虚线为大片段SV存在位点
KASP结果有28份种质基因型为GG(图6),但重测序数据及PCR试验(图7)表明只有18份种质(附表1)含有转座子纯合插入(hdhd),基因型上为硬质类型;除‘大果黑桃’‘丰白’‘早红不软’外,其余种质均有文献报道[4,14,27-28]为硬质类型。综上,基于Pp06: 14090689位点的SNP标记不能准确鉴定硬质基因型,对于-TE基因型的检测以重测序数据或转座子标记更准确。
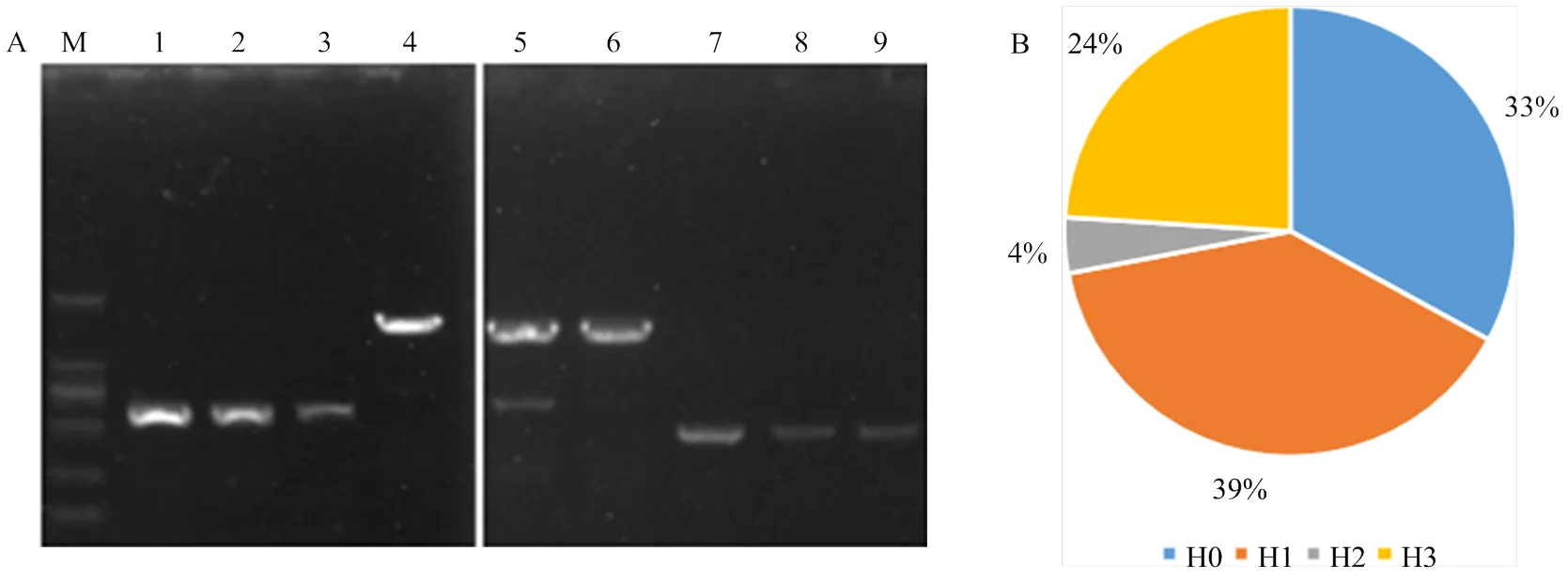
A:Maker 2000;1、2、3泳道为H0单倍型;4、5、6泳道为H2单倍型;7、8、9泳道为H3单倍型。泳道1—6,依次为美锦、西尾金、08-9-107、郑黄2号、NJC83、明星、08-9-106、中油15号、中油16号;B:189份种质在F-M座4种单倍型频数分布

A:YUC11模式图及变异位点;B:重测序数据与硬质相关的SNP变异位点;C:重测序数据YUC11启动区转座子插入位点,红色方框为插入位点存在的clipped reads
2.4 成熟期相关NAC72-indel基因型鉴定
果实成熟涉及呼吸变化、乙烯合成、风味物质产生、颜色变化、果肉软化等非常复杂的过程[29],而果实成熟期是典型的数量性状,开发相应的分子标记存在一定的难度。桃4号染色体上的编码区9 bp变异与早熟表型相关联[19]。本研究对该变异位点(Pp04: 11118143)设计引物,利用HRM方法鉴定不同基因型(图8)。

A:189份种质KASP标记分型结果(部分),绿色、红色、蓝色三角形分别代表基因型GG、GT、TT;B:3种基因型频数分布图
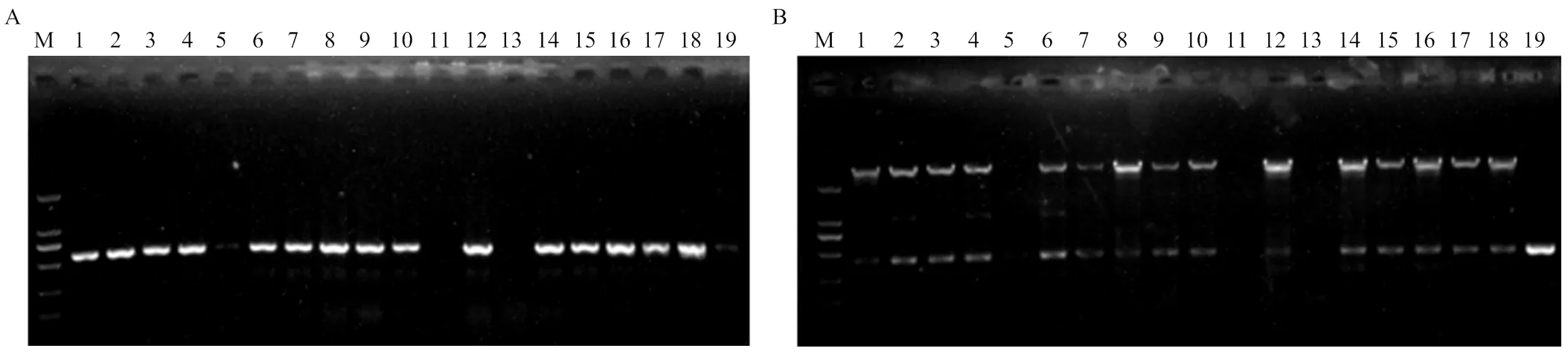
A:扩增出600 bp条带说明含有转座子插入;B:扩增出3 015 bp条带说明含有转座子插入。泳道1—19依次为:08-9-106、美锦、西尾金、霞脆、有明白桃、中油20号、中油15号、京玉、秦王、华玉、空白、白如玉、空白、早玉、中油16号、大果黑桃、丰白、早红不软、阴性对照
在《桃种质资源描述规范和数据标准》中依据果实发育期将成熟期分类,极早熟<60 d、早熟60—90 d、中熟90—120 d、晚熟120—150 d、极晚熟>150 d。HRM结果(附表1)表明,Pp04: 11118143位点插入纯合(早熟)有45份种质、插入杂合(中熟)有71份种质、无插入(晚熟)有73份种质,中熟与晚熟种质占比77%。但是将189份种质成熟期基因型与表型对照时,发现‘五月鲜扁干’‘天津水蜜’‘五月鲜’‘大红袍’‘六月白’‘六月空’6份种质基因型与表型不符。重测序数据与试验结果均显示不含9 bp纯合插入,但表型均为早熟;表明该标记对于成熟期的鉴定能力有限。
2.5 肉质性状相关位点基因型组合分析
F-M基因座组成的肉质分为不溶质粘核、溶质粘核、溶质离核3种类型,尚未报道不溶质离核的肉质类型[5],在本研究189份种质中也并未发现仅含有的不溶质离核类型。通过上文研究可知,肉质形成的F-M基因座、-TE、-indel三个关键位点分别有10、3、3种组合;基因型分别有5、3、3种,本研究中未发现在F-M基因座含有H0H2、H1H2组合的种质(附表1)。
本研究中189份种质的表型评价多达14种,基于GU等[9]与QIAN等[30]发现在F-M基因座仅有与参与肉质的形成,被视作溶质、离核性状的候选基因,因此将189种质分为不溶质粘核、溶质粘核(溶质粘核、软溶质粘核、硬溶质粘核、硬肉粘核、硬质粘核)、溶质离核(粉质离核、绵离核、溶质离核、软溶质半离核、软溶质离核、硬溶质半离核、硬溶质离核、硬肉离核)3大类。
不溶质粘核、溶质粘核、溶质离核都含有成熟期的3种基因型(表2);的3种基因型在3种表型中也都存在,均以HdHd基因型为主,并且在18份含有硬质基因型的种质中F-M基因座的组合类型含有6种、成熟期基因型含有3种(附表1);F-M基因座,在表型为不溶质粘核时以H3H3、H2H3组合为主,在表型为溶质粘核时以H0H0、H0H1组合为主,在表型为溶质离核时以H1H1、H0H1组合为主;在3个位点,3种表型最多的基因型组合依次是mmffHdHdI、MMffHdHdI、MMFFHdHdL。上述结果说明,在肉质形成的3个关键位点中F-M基因座影响最大。
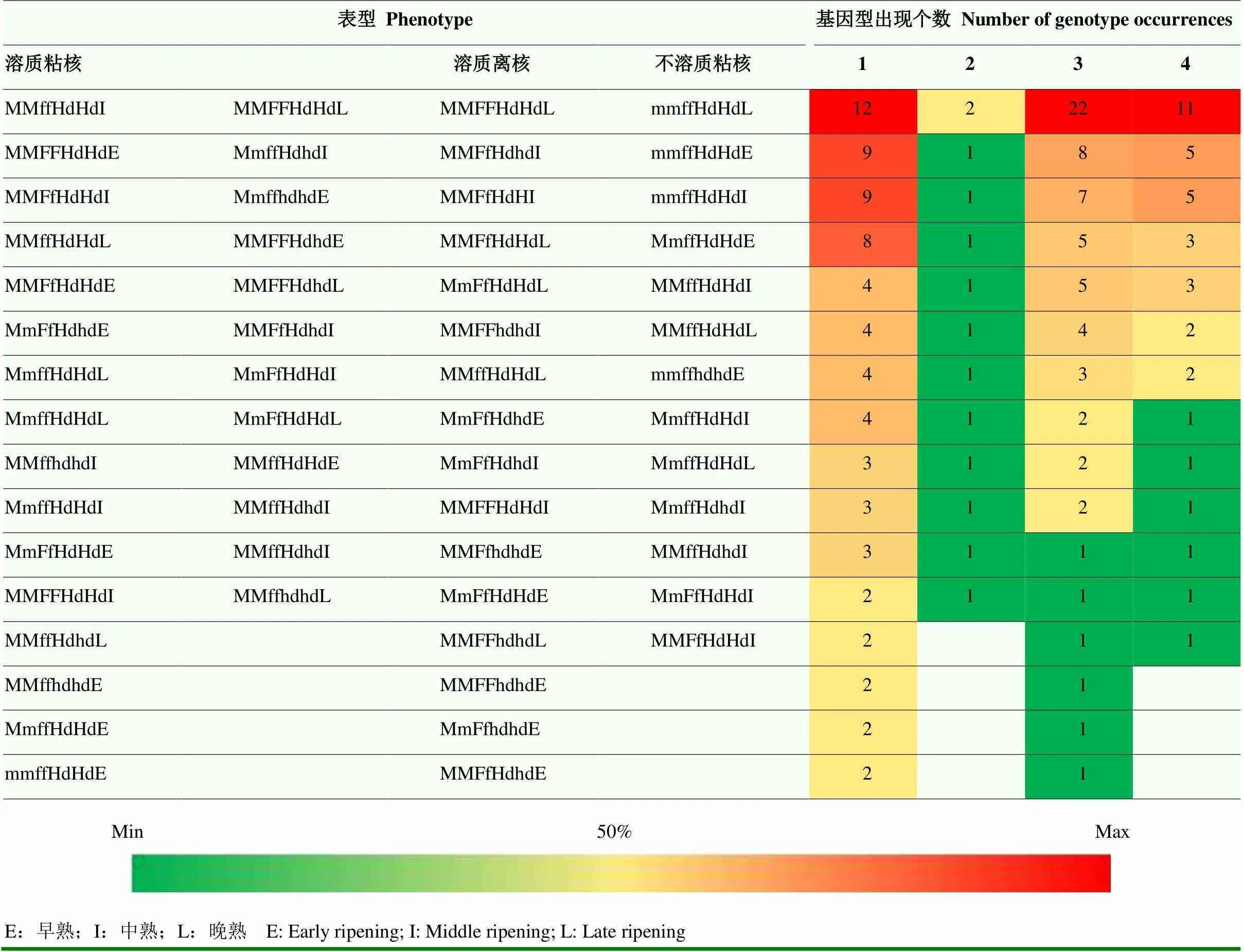
表2 不同肉质表型对应基因型组合

A:189份种质HRM分型结果(部分),绿色、红色、蓝色三角形分别代表基因型0 bp/0 bp、9 bp/9 bp、9 bp/0 bp;B:成熟期3种基因型与表型关系
3 讨论
在众多细胞壁水解酶、植物激素、转录因子等因素共同作用下,果实在成熟和采收后肉质变化具有多样性——变软、变硬、粉质化、纤维化、变脆[31-33]。
3.1 含有多个多聚半乳糖醛酸酶(PG)的F-M基因座是肉质形成关键位点
以番茄为代表的多聚半乳糖醛酸酶调控的呼吸跃变型果实[34-35],草莓为代表的果胶裂解酶调控的非呼吸跃变型果实[31,36],均表明细胞壁水解酶类直接参与肉质形成。内切型多聚半乳糖醛酸酶(Endo-PG)是调控桃果实软化最主要的水解酶,在F-M基因座仅有与参与肉质形成[30]。对189份种质分析发现F-M位点存在4种单倍型,与NAKANO等[10]的研究完全一致,而并非GU等[9]报道的3种;H0、H1、H2、H3单倍型分别对应溶质粘核、溶质离核、溶质粘核、不溶质表型。桃不同倍型组合与肉质表型关系为:溶质粘核H0H0、H0H2、H0H3、H2H2与H2H3;溶质离核H0H1、H1H1、H1H2与H1H3;不溶质H3H3。GU等[9]利用引物分别扩增与鉴定F-M基因组座的倍型,该方法对于基因组上大片段的缺失或基因部分缺失鉴定时存在一定局限性。因此,在4种倍型中,GU等[9]仅鉴定出来3种,与本研究中H1、H2、H3相对应。H0倍型在GU等[9]研究中归为H2之中,本研究正是利用了生物信息学在鉴定基因组上大片段结构变异的优势,从而鉴定出H0倍型。本研究中,‘C17’‘C18’‘中桃金典’‘郑黄2号’‘NJC83’‘明星’‘敦煌冬桃’‘郑黄4号’种质表型为不溶质却含有,但是这些种质在F-M单倍型为H2H2或H2H3,根据NAKANO等[10]与BAILEY等[6]研究结果的可能原因是H2、H3单倍型上的不发挥功能;同时也有研究表明与之间存在相互拮抗作用[17]。综上,在对溶质/不溶质与粘核/离核性状鉴定时应对F-M座进行分析,而不同单倍型功能是否一致、与之间调控关系也需要进一步深入研究。
3.2 乙烯合成受阻是产生硬质桃的原因
植物激素在果实生长发育、成熟过程中均发挥重要作用[32-33,37];果实成熟时呼吸跃变型果实(番茄等)乙烯大量合成[38],而非呼吸跃变型果实(甜瓜等)成熟时脱落酸大量合成[39]。乙烯参与桃果实成熟后软化过程,变异是乙烯合成受阻进而产生硬质桃的原因[4]。相较于溶质/不溶质性状,硬质这一概念提出较晚。因此,在18份具有硬质基因型的种质中有8份种质评价为溶质离核,但F-M基因座显示均含有H1单倍型;可能转座子插入纯合时仅对溶质()有上位作用,或乙烯合成受阻时存在其他途径调控果实软化。
3.3 成熟期与肉质关系较为复杂
在成熟过程中有众多转录因子参与调控肉质、颜色、香气、风味物质的形成:较为常见的NAC家族、MADS家族、ERF家族、NAP家族[40-43]。HUANG等[44]报道了两个MADS家族基因共同调控番茄形成全肉质型表型;MIGICOVSK等[45]研究发现对苹果成熟期、成熟期硬度、储藏后硬度、硬度变化4个表型有一定影响,其中对成熟期影响最大。本研究中189份种质的编码区9 bp插入,除少量种质外基因型与成熟期基本吻合;推测可能为调控桃果实成熟期的主效基因。3个位点基因型组合分析发现,部分早熟品种含有,但表型鉴定被视为粘核;不溶质粘核和溶质粘核的成熟期基因型大多数为中熟,而溶质离核的成熟期基因型大多数为晚熟,可能原因是控制溶质/不溶质的受乙烯调控,而主要受果实发育的影响[17]。因此,成熟期的差异会影响桃果实发育情况,进而间接影响肉质表型。但值得注意的是,即使基因型一致时果实发育期(成熟期)也会有一个月差异,进一步表明成熟期是多基因调控的复杂性状。
3.4 桃肉质由基因组上多个位点共同决定
桃肉质是质量性状与数量性状同时存在的典型性状。基因组上多个位点共同参与肉质的形成,如硬质桃就是由位点和F-M基因座共同决定[5]。本研究对3个位点基因型组合分析时发现溶质粘核的MMFFHdHdE数量仅次于MMffHdHdI数量,也暗示在评价肉质表型时,若未考虑成熟期时可能会存在误差。基于郭健[46]、PAN等[13]与GU等[9]转录组数据表明,在肉质形成的3个关键位点中,位点在果实发育期发挥作用,而位点与F-M基因座主要在成熟期起关键作用。因此,3个位点之间的关系可能为:F-M基因座编码的细胞壁水解酶是肉质形成的直接参与者,位于调控网络的最下游;位点通过控制乙烯合成,进而调控F-M基因座水解酶的活性;位点则决定果实发育程度,影响位点乙烯合成并激活下游F-M基因座水解酶的时间。综上,在对肉质性状进行研究时,应该对参与肉质形成的多个位点综合进行考虑,建议按照成熟期、硬质/非硬质、溶质/不溶质、粘/离核顺序进行表型评价。
桃果实肉质形成过程中伴随着颜色、香气、硬度等一系列变化,这一过程受到内外因素的共同调控,机制非常复杂。除本研究涉及的3个关键位点外,果胶裂解酶[47]、众多转录因子[29]也参与桃果实肉质形成;此外,在转录水平上、蛋白质翻译过程中、蛋白质翻译后也会影响肉质的形成[48]。因此,未来可借助多组学手段揭示肉质形成调控网络,从而深入了解桃果实肉质成熟软化过程中的分子机制。
4 结论
通过分子试验和生物信息学手段,进一步证实了F-M基因座存在4种单倍型,H0、H1单倍型占比较高,H3H3、H2H3是形成不溶质粘核表型的主要组合类型,H0H0、H0H1是形成溶质粘核表型的主要组合类型、H1H1、H0H1是形成溶质离核表型的主要组合类型。进一步补充了鉴定肉质基因型的分子标记;发现18份具有硬质基因型的种质;开发出一种可用于鉴定成熟期的分子标记;不溶质粘核、溶质粘核、溶质离核表型最多的基因型组合分别是mmffHdHdI、MMffHdHdI、MMFFHdHdL。
[1] 王力荣. 中国桃品种改良历史回顾与展望. 果树学报, 2021, 38(12): 2178-2195.
WANG L R. History and prospect of peach breeding in China. Journal of Fruit Science, 2021, 38(12): 2178-2195. (in Chinese)
[2] LI Y, CAO K, LI N, ZHU G R, FANG W C, CHEN C W, WANG X W, GUO J, WANG Q, DING T Y, WANG J, GUAN L, WANG J X, LIU K Z, GUO W W, ARÚS P, HUANG S W, FEI Z, WANG L R. Genomic analyses provide insights into peach local adaptation and responses to climate change. Genome Research, 2021, 31(4): 592-606.
[3] 孟君仁. 桃慢溶质性状调控机制初步研究[D]. 北京: 中国农业科学院, 2021.
MENG J R. Preliminary study on regulation mechanism of slow melting flesh characteristics in peach [D]. Beijing: Chinese Academy of Agricultural Sciences, 2021. (in Chinese)
[4] 牛良, 曾文芳, 潘磊, 孟君仁, 鲁振华, 崔国朝, 王志强. 硬质桃研究现状及展望. 果树学报, 2020, 37(8): 1227-1235.
NIU L, ZENG W F, PAN L, MENG J R, LU Z H, CUI G C, WANG Z Q. Research status and perspective for stony-hard peach. Journal of Fruit Science, 2020, 37(8): 1227-1235. (in Chinese)
[5] 曾文芳, 王志强, 牛良, 潘磊, 丁义峰, 鲁振华, 崔国朝. 桃果实肉质研究进展. 果树学报, 2017, 34(11): 1475-1482.
ZENG W F, WANG Z Q, NIU L, PAN L, DING Y F, LU Z H, CUI G C. Research process on peach fruit flesh texture. Journal of Fruit Science, 2017, 34(11): 1475-1482. (in Chinese)
[6] BAILEY J S, FRENCH A P. The inheritance of certain fruit and foliage characteristics in the peach[R]. Massachusetts Agricultural Experimental Station Bulletin,452,University of Massachusetts,1949.
[7] LESTER D R, SHERMAN W B, ATWELL B J. Endopolygalacturonase and the melting flesh (M) locus in peach. Journal of the American Society for Horticultural Science, 1996, 121(2): 231-235.
[8] DIRLEWANGER E, COSSON P, BOUDEHRI K, RENAUD C, CAPDEVILLE G, TAUZIN Y, LAIGRET F, MOING A. Development of a second-generation genetic linkage map for peach[(L.) Batsch] and characterization of morphological traits affecting flower and fruit. Tree Genetics & Genomes, 2006, 3(1): 1-13.
[9] GU C, WANG L, WANG W, ZHOU H, MA B Q, ZHENG H Y, FANG T, OGUTU C, VIMOLMANGKANG S, HAN Y P. Copy number variation of a gene cluster encoding endopolygalacturonase mediates flesh texture and stone adhesion in peach. Journal of Experimental Botany, 2016, 67(6): 1993-2005.
[10] NAKANO R, KAWAI T, FUKAMATSU Y, AKITA K, WATANABE S, ASANO T, TAKATA D, SATO M, FUKUDA F, USHIJIMA K. Postharvest properties of ultra-late maturing peach cultivars and their attributions to melting flesh (M) locus: Re-evaluation of M locus in association with flesh texture. Frontiers in Plant Science, 2020, 11: 554158.
[11] YOSHIDA M. Genetical studies on the fruit quality of peach varieties. 3. Texture and keeping quality[R]. Bulletin of the Fruit Tree Research Station. Series A. Hiratsuka, 1976.
[12] HAJI T, YAEGAKI H, YAMAGUCHI M. Inheritance and expression of fruit texture melting, non-melting and stony hard in peach. Scientia Horticulturae, 2005, 105(2): 241-248.
[13] PAN L, ZENG W F, NIU L, LU Z H, LIU H, CUI G C, ZHU Y Q, CHU J F, LI W P, FANG W C, CAI Z G, LI G H, WANG Z Q. PpYUC11, a strong candidate gene for the stony hard phenotype in peach (L. Batsch), participates in IAA biosynthesis during fruit ripening. Journal of Experimental Botany, 2015, 66(22): 7031-7044.
[14] 曾文芳, 丁义峰, 潘磊, 王小贝, 牛良, 鲁振华, 崔国朝, 王志强. 桃硬质性状可能源于PpYUC11基因启动子区域CACTA型转座子的插入. 果树学报, 2017, 34(10): 1239-1248.
ZENG W F, DING Y F, PAN L, WANG X B, NIU L, LU Z H, CUI G C, WANG Z Q. A CACTA transposable element in agene promoter is associated with the stony hard phenotype in peach. Journal of Fruit Science, 2017, 34(10): 1239-1248. (in Chinese)
[15] TATSUKI M, SOENO K, SHIMADA Y, SAWAMURA Y, SUESADA Y, YAEGAKI H, SATO A, KAKEI Y, NAKAMURA A, BAI S L, MORIGUCHI T, NAKAJIMA N. Insertion of a transposon-like sequence in the 5’-flanking region of thegene causes the stony hard phenotype. The Plant Journal, 2018, 96(4): 815-827.
[16] 孟君仁, 曾文芳, 邓丽, 潘磊, 鲁振华, 崔国朝, 王志强, 牛良. 桃若干重要性状的KASP分子标记开发与应用. 中国农业科学, 2021, 54(15): 3295-3307.
MENG J R, ZENG W F, DENG L, PAN L, LU Z H, CUI G C, WANG Z Q, NIU L. Development and application of KASP molecular markers of some important traits for peach. Scientia Agricultura Sinica, 2021, 54(15): 3295-3307. (in Chinese)
[17] 孟君仁, 牛良, 邓丽, 潘磊, 鲁振华, 崔国朝, 王志强, 曾文芳. 控制桃粘/离核PG基因的BAC克隆筛选与序列分析. 中国农业科学, 2021, 54(20): 4396-4404. doi: 10.3864/j.issn.0578-1752.2021. 20.013.
MENG J R, NIU L, DENG L, PAN L, LU Z H, CUI G C, WANG Z Q, ZENG W F. Screening and sequence analysis of BAC clone contained PG gene controlling clingstone/freestone characteristic of peach. Scientia Agricultura Sinica, 2021, 54(20): 4396-4404. doi: 10.3864/j.issn.0578-1752.2021.20.013. (in Chinese)
[18] PIRONA R, EDUARDO I, PACHECO I, DA SILVA LINGE C, MICULAN M, VERDE I, TARTARINI S, DONDINI L, PEA G, BASSI D, ROSSINI L. Fine mapping and identification of a candidate gene for a major locus controlling maturity date in peach. BMC Plant Biology, 2013, 13(1): 166.
[19] 余银梅. 桃果实成熟期调控基因、的表达分析及功能初探[D]. 郑州: 河南农业大学, 2018.
YU Y M. Expression analysis and functional exploration ofandrelated to peach fruit maturity date [D]. Zhengzhou: Henan Agricultural University, 2018. (in Chinese)
[20] 何雪娇, 郑涛, 苏金强, 陈振东. 改良CTAB法提取野牡丹科7种植物DNA. 热带农业科学, 2011, 31(10): 73-77.
HE X J, ZHENG T, SU J Q, CHEN Z D. DNA extraction of 7 species plants of Melastomaceae using modified CTAB method. Chinese Journal of Tropical Agriculture, 2011, 31(10): 73-77. (in Chinese)
[21] 殷豪, 王彩虹, 田义轲, 李节法, 王然, 戴洪义. 利用高分辨率熔解曲线(HRM)分析梨微卫星标记. 园艺学报, 2011, 38(8): 1601-1606.
YIN H, WANG C H, TIAN Y K, LI J F, WANG R, DAI H Y. Differentiation of microsatellites in pear by high resolution melting (HRM) analysis. Acta Horticulturae Sinica, 2011, 38(8): 1601-1606. (in Chinese)
[22] WINGETT S W, ANDREWS S. FastQ Screen: A tool for multi-genome mapping and quality control. F1000Research, 2018, 7: 1338.
[23] LI H, DURBIN R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics, 2009, 25(14): 1754-1760.
[24] Li H, Handsaker R E, Wysoker A, FENNELL T, RUAN J, HOMER N, MARTH G, ABECASIS G, DURBIN R. The Sequence Alignment/Map format and SAMtools. Bioinformatics, 2009, 25(16): 2078-2079.
[25] RAUSCH T, ZICHNER T, SCHLATTL A, STÜTZ A M, BENES V, KORBEL J O. DELLY: Structural variant discovery by integrated paired-end and split-read analysis. Bioinformatics, 2012, 28(18): i333-i339.
[26] THORVALDSDÓTTIR H, ROBINSON J T, MESIROV J P. Integrative Genomics Viewer (IGV): High-performance genomics data visualization and exploration. Briefings in Bioinformatics, 2013, 14(2): 178-192.
[27] CIRILLI M, GIOVANNINI D, CIACCIULLI A, CHIOZZOTTO R, GATTOLIN S, ROSSINI L, LIVERANI A, BASSI D. Integrative genomics approaches validate PpYUC11-like as candidate gene for the stony hard trait in peach (L. Batsch). BMC Plant Biology, 2018, 18(1): 1-12.
[28] 韩晴. 桃果肉质地和粘离核性状两个关键PpPG基因的表达和调控关系分析[D]. 北京: 中国农业科学院, 2019.
HAN Q. Expression and regulation relationship analysis of two keygenes related to peach flesh texture and adhension traits [D]. Beijing: Chinese Academy of Agricultural Sciences, 2019. (in Chinese)
[29] SHI Y N, LI B J, SU G Q, ZHANG M X, GRIERSON D, CHEN K S. Transcriptional regulation of fleshy fruit texture. Journal of Integrative Plant Biology, 2022, 64(9): 1649-1672.
[30] QIAN M, ZHANG Y K, YAN X Y, HAN M Y, LI J J, LI F, LI F R, ZHANG D, ZHAO C P. Identification and expression analysis of polygalacturonase family members during peach fruit softening. International Journal of Molecular Sciences, 2016, 17(11): 1933.
[31] ZHANG W W, ZHAO S Q, ZHANG L C, XING Y, JIA W S. Changes in the cell wall during fruit development and ripening in. Plant Physiology and Biochemistry, 2020, 154: 54-65.
[32] ZHU N, ZHAO C N, WEI Y Q, SUN C D, WU D, CHEN K S. Biosynthetic labeling with 3-O-propargylcaffeyl alcohol revealscell-specific patterned lignification in loquat fruits during development and postharvest storage. Horticulture Research, 2021, 8: 61.
[33] 王巍. 生长素应答基因PpIAA5-ARF8对桃果实成熟软化的调控作用[D]. 北京: 北京农学院, 2021.
WANG W. Auxin-responsive gene PpIAA-ARF8 on peach fruit ripening and softing [D]. Beijing: Beijing University of Agriculture, 2021. (in Chinese)
[34] KUMAR R, KHURANA A, SHARMA A K. Role of plant hormones and their interplay in development and ripening of fleshy fruits. Journal of Experimental Botany, 2014, 65(16): 4561-4575.
[35] LIU Y D, TANG M F, LIU M C, SU D D, CHEN J, GAO Y S, BOUZAYEN M, LI Z G. The molecular regulation of ethylene in fruit ripening. Small Methods, 2020, 4(8): 1900485.
[36] ZHOU L L, TANG R K, LI X J, TIAN S P, LI B B, QIN G Z. N6-methyladenosine RNA modification regulates strawberry fruit ripening in an ABA-dependent manner. Genome Biology, 2021, 22(1): 1-32.
[37] BAI Q, HUANG Y, SHEN Y Y. The physiological and molecular mechanism of abscisic acid in regulation of fleshy fruit ripening. Frontiers in Plant Science, 2021, 11: 619953.
[38] ZHU F, JADHAV S S, TOHGE T, SALEM M A, LEE J M, GIOVANNONI J J, CHENG Y J, ALSEEKH S, FERNIE A R. A comparative transcriptomics and eQTL approach identifiesas a tomato fruit ripening regulator. Plant Physiology, 2022, 190(1): 250-266.
[39] WANG J F, TIAN S W, YU Y T, REN Y, GUO S G, ZHANG J, LI M Y, ZHANG H Y, GONG G Y, WANG M, XU Y. Natural variation in the NAC transcription factor NONRIPENING contributes to melon fruit ripening. Journal of Integrative Plant Biology, 2022, 64(7): 1448-1461.
[40] GUO Z H, ZHANG Y J, YAO J L, XIE Z H, ZHANG Y Y, ZHANG S L, GU C. The NAM/ATAF1/2/CUC2transcription factor PpNAC.A59 enhances PpERF.A16 expression to promote ethylene biosynthesis during peach fruit ripening. Horticulture Research, 2021, 8: 209.
[41] 王小贝. 桃ERFs和PpIAA1协同调控桃成熟软化的分子机制研究[D]. 武汉: 华中农业大学, 2019.
WANG X B. Study on the molecular mechanism of synergistic regulation of peach ripening and softening by ERFs and PpIAA1 [D]. Wuhan: Huazhong Agricultural University, 2019. (in Chinese)
[42] 李金金. PpSEP1(SEPALLATA)在不同质地桃果实成熟软化过程中的调控机制[D]. 杨凌: 西北农林科技大学, 2016.
LI J J. Regulatory mechanism of the PpSEP1 (SEPALLATA) gene during fruit ripening and softening in melting and non-melting peachs [D]. Yangling: Northwest A & F University, 2016. (in Chinese)
[43] 李芳. NAP家族成员克隆及其在桃果实发育与成熟期间的表达特性研究[D]. 杨凌: 西北农林科技大学, 2016.
LI F. Cloning and expression characterization of NAP family members during development and ripening of peach [D]. Yangling: Northwest A & F University, 2016. (in Chinese)
[44] HUANG B W, HU G J, WANG K K, FRASSE P, MAZA E, DJARI A, DENG W, PIRRELLO J, BURLAT V, PONS C, GRANELL A, LI Z G, VAN DER REST B, BOUZAYEN M. Interaction of two MADS-box genes leads to growth phenotype divergence of all-flesh type of tomatoes. Nature Communications, 2021, 12: 6892.
[45] MIGICOVSKY Z, YEATS T H, WATTS S, SONG J, FORNEY C F, BURGHER-MACLELLAN K, SOMERS D J, GONG Y H, ZHANG Z Q, VREBALOV J, VAN VELZEN R, GIOVANNONI J G, ROSE J K C, MYLES S. Apple ripening is controlled by a NAC transcription factor. Frontiers in Genetics, 2021, 12: 671300.
[46] 郭健. 基于基因组结构变异发掘桃重要农艺性状关键基因[D]. 武汉: 华中农业大学, 2021.
GUO J. Identification of causal genes responsible for key agronomic traits based on genome structural variations in peach [D]. Wuhan: Huazhong Agricultural University, 2021. (in Chinese)
[47] LI J J, LI F, QIAN M, HAN M Y, LIU H K, ZHANG D, MA J J, ZHAO C P. Characteristics and regulatory pathway of thegene during ripening and softening in peach fruits. Plant Science, 2017, 257: 63-73.
[48] WANG W H, WANG Y Y, CHEN T, QIN G Z, TIAN S P. Current insights into posttranscriptional regulation of fleshy fruit ripening. Plant Physiology, 2022, 190(22): 921-930.
附表1 189份种质肉质表型与基因型鉴定关系
upplementary table 1. Relationship between flesh texture genotype and phenotype
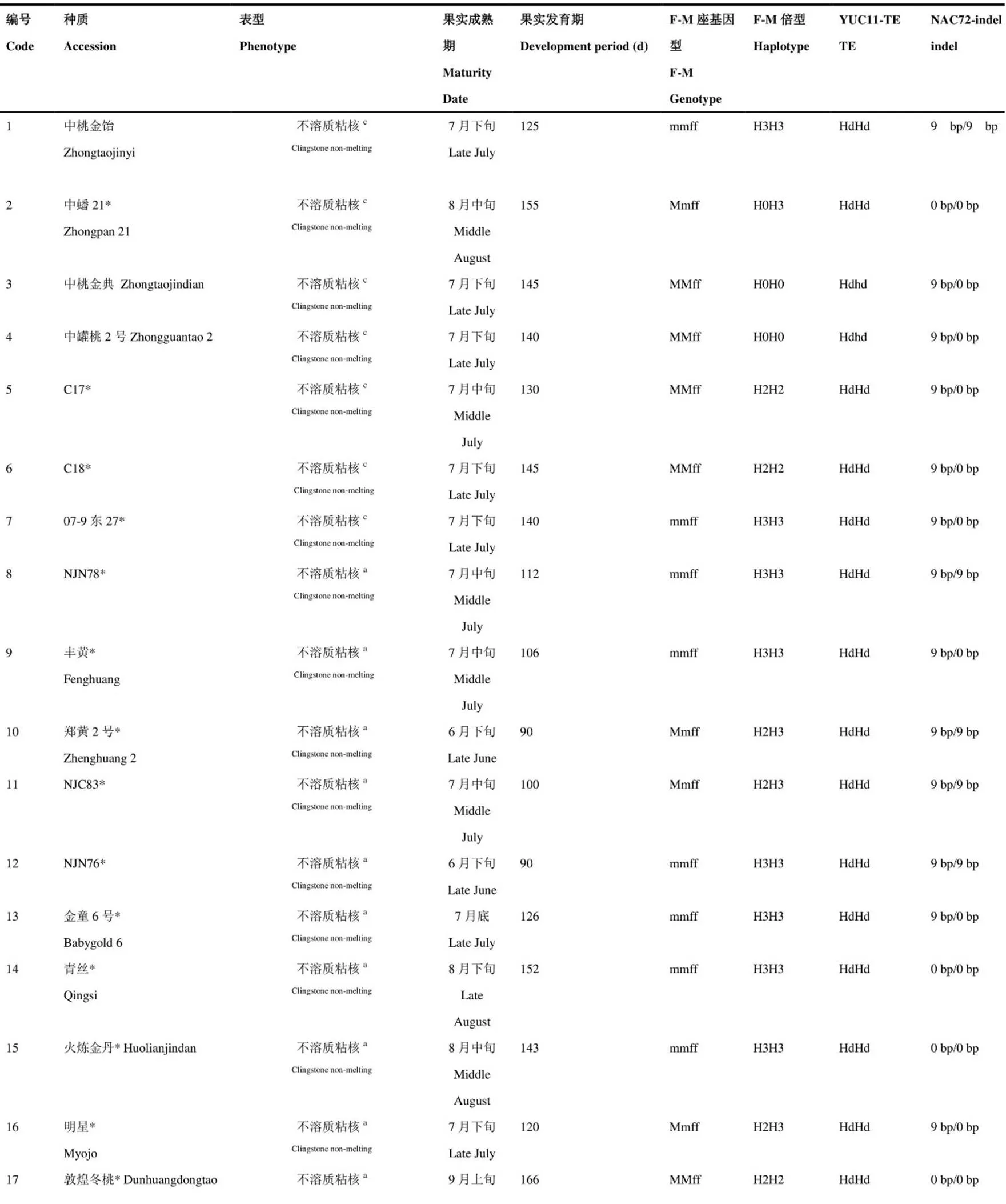
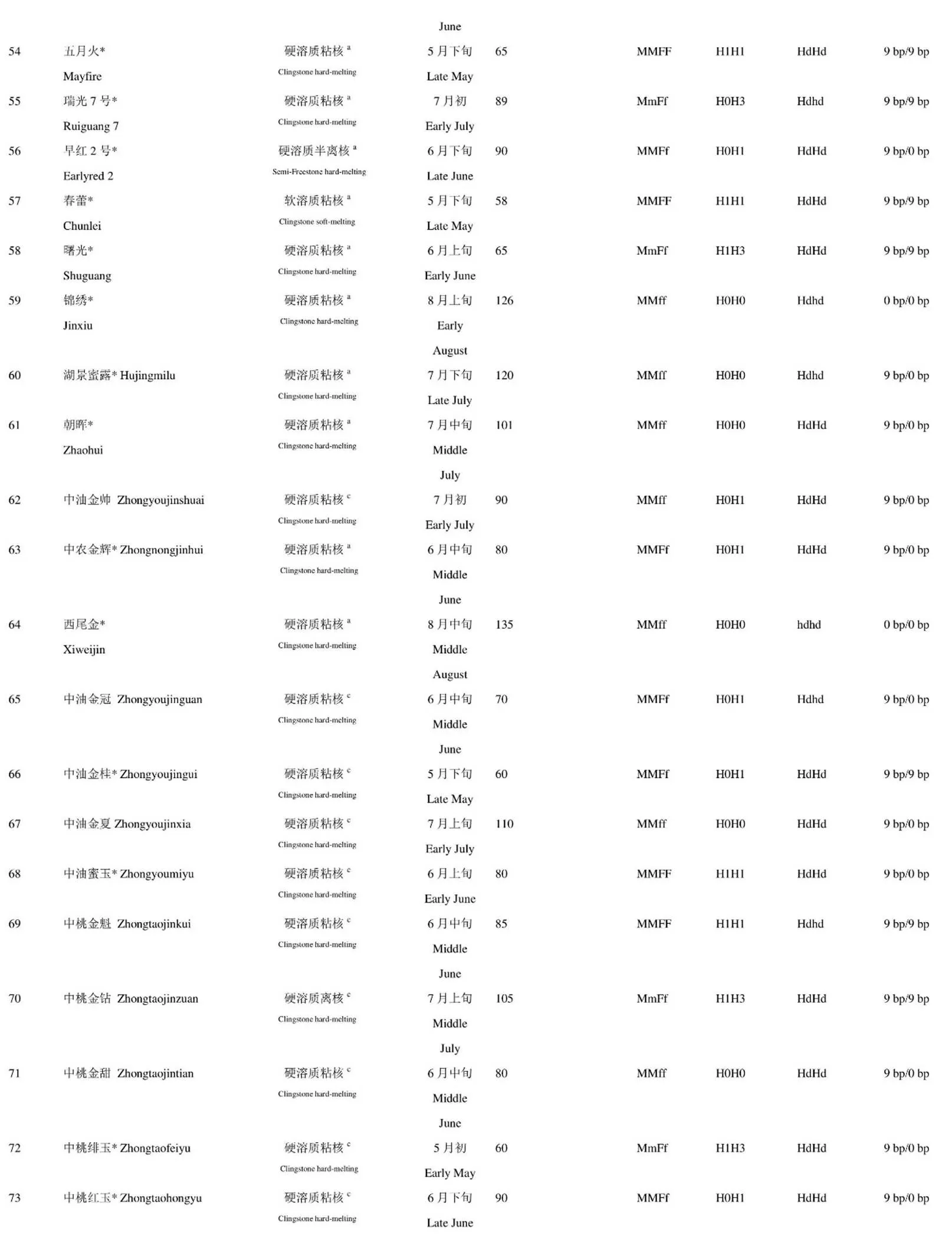
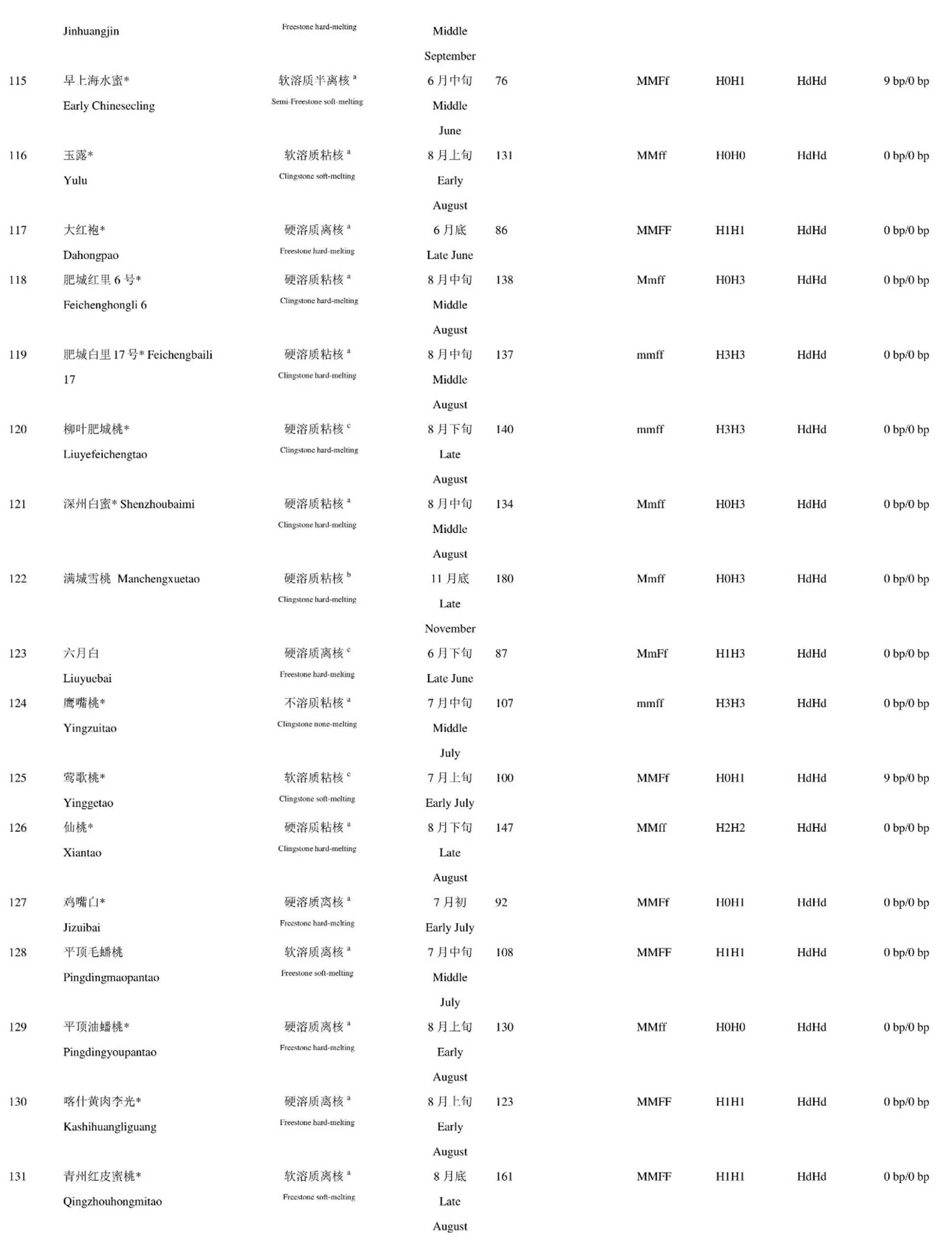

*代表有重测序数据。a:表型评价来源于《中国桃遗传资源》;b:表型来源于文献报道;c:表型来源于田间调查
* indicate have resequencing data. a: Phenotype from; b: Phenotype from literature report; c: Phenotype from field investigation
Genotype Identification and Combination Analysis of Loci Related to the Peach Flesh Texture Trait via 189 Peach Accessions
WANG ZhaoHui, LI Yong, CAO Ke, ZHU GengRui, FANG WeiChao, CHEN ChangWen, WANG XinWei, WU JinLong, WANG LiRong
Zhengzhou Fruit Research Institute, Chinese Academy of Agricultural Sciences, Zhengzhou 450009
【Objective】 The molecular markers and bioinformatics were used to identify the genotypes and combinations of the F-M locus, transposon insertion inpromoter region, and 9 bp insert incoding region by 189 peach germplasms, in order to provide the theoretical foundation for the mechanism of peach flesh formation and the selection of breeding parents.【Method】PCR amplification, KASP, and HRM were used to identify genotypes. Results of loci associated with the formation of flesh trait were further validated using bioinformatics by resequencing data in 189 peach germplasms.【Result】Two genes encoding endopolygalacturonase in F-M locus, designatedand, are associated with peach texture. Through primer amplification, 159 (84%)and 99 (52%)were detected in 189 peach germplasms. Four haplotypes (H0, H1, H2, and H3) were found in F-M locus, while H0 and H1 were the major genotypes. The haplotype combination of non-melting flesh, melting flesh, free-stone melting flesh were H3H3 and H2H3, H0H0 and H0H1, H1H1 and H0H1, respectively. In addition, it was found that 18 germplasms contain homozygous transposon insertion. HRM results demonstrated that 45 germplasms were homozygous insertion (early ripening), 71 germplasms were heterozygous insertion (middle ripening), and 73 germplasms were no insertion (late ripening). But six germplasm genotypes were inconsistent with the phenotypes. The most frequent genotypic combinations at the three loci of non-melting flesh, melting flesh, and free-stone melting flesh were mmffHdHdI, MMffHdHdI, MMFFHdHdL, respectively.【Conclusion】The existence of four haplotypes at the F-M locus further was confirmed, 18 germplasms containing stony hard genotype were identified, a molecular marker for the identification of maturity date was developed, and genotype combinations of different germplasms at flesh-related loci were identified.
peach; flesh texture; genotype identification; genotype combinations

10.3864/j.issn.0578-1752.2023.12.011
2022-08-25;
2022-12-28
国家桃产业技术体系(CARS-30-1-04)、中国农业科学院创新工程项目(Grant CAAS-ASTIP-2022-ZFRI-01)
王朝晖,E-mail:82101221626@caas.cn。通信作者吴金龙,E-mail:wujinlong@caas.cn。通信作者王力荣,E-mail:wanglirong@caas.cn
(责任编辑 赵伶俐)
