辽宁省主要设施农业集散地大棚土壤ARGs与MGEs分布特征分析
2023-07-05黎馨月王百羽
张 珣, 武 轶, 黎馨月, 王百羽, 安 婧, 王 莹
(1. 沈阳大学 环境学院, 辽宁 沈阳 110044;2. 沈阳农业大学 东北粳稻遗传改良与优质高效生产省部共建协同创新中心, 辽宁 沈阳 110866;3. 中国科学院 沈阳应用生态研究所, 辽宁 沈阳 110016)
抗生素主要是由细菌、霉菌或其他微生物产生的次级代谢产物或人工合成的类似物。近年来,兽用抗生素在过度医疗和畜牧业领域的滥用,导致环境中抗生素残留激增、耐药细菌出现。越来越多的抗生素和抗生素抗性基因(antibiotic resistance genes, ARGs)的出现和扩散对抗生素的治疗效果和生态环境的稳定造成了严重危害。
ARGs作为1种新环境污染物于2006年被正式定义[1],近年来受到大众的关注度愈来愈高。土壤中ARGs主要来源于土壤微生物及人或动物的粪便等,其中动物粪便为ARGs进入土壤的主要途径[2-3]。研究表明,钱勋[4]在鸡粪中检出134种ARGs,在猪粪中检出120种ARGs,在牛粪检出80种ARGs;Qian等[5]在猪、鸡、牛等3种畜禽粪便中检测到100余种ARGs;张丹等[6]在鸡粪和牛粪中检测到ARGs和可移动遗传元件(mobile genetic elements, MGEs)。在长期施用畜禽粪便的土壤中ARGs的丰度和多样性会显著增加[7]。Zhou等[8]研究表明,施用鸡粪商业有机肥可显著增加土壤中ARGs的相对丰度;程建华等[9]在养牛场、养鸡场和养猪场的土壤样品中分别检测到27、44和36种ARGs。而土壤中的ARGs可以向水体和农作物中迁移,并随食物链向动物及人类传播[10]。
ARGs通常位于质粒(plasmid)、转座子(transposon)及整合子(integron)等可移动遗传元件MGEs上,可以通过基因水平转移发生迁移,引起抗生素耐药菌的扩散[11]。MGEs主要为整合子、质粒、转座子等,其中整合子位于质粒和染色体上,是ARGs转移的重要分子元件[12]。程建华等[9]研究表明,养殖场土壤中较高的MGEs丰度及ARGs丰度呈显著的相关性(p<0.05);张俊华等[13]研究表明,磺胺类、氯霉素类、氨基糖苷类ARGs和ARGs总和与MGEs均呈极显著正相关。说明MGEs可能促进了ARGs在土壤环境中的迁移扩散。
本文利用超高通量荧光定量PCR仪对辽宁省主要设施农业集散地大棚土壤的ARGs与MGEs进行研究,分析设施农业土壤中ARGs和MGEs的分布特征及相关性。
1 材料与方法
1.1 土壤样品采集
选取辽宁省内的主要设施农业集散地温室大棚作为采样点,每个采样点采集3份重复样品(采样深度为5~10 cm),土壤过2 mm筛并保存于超低温冰箱中,用于后续DNA提取和ARGs的测定。样品的基本信息如表1所示。
1.2 DNA的提取
使用新鲜土壤样品(0.5 g)用于DNA提取,使用土壤DNA基因组提取试剂盒E.Z.N.A.® Soil DNA Kit(美国欧米茄公司生产)从土壤样品中提取DNA,按照说明书的要求进行提取。DNA提取完毕后,使用超微量光度分析仪NanoPhotometerTMP-Class(德国Implen公司生产)测定其DNA的纯度和质量浓度,A260/A280在1.8~2.0之间,表明DNA纯度可进行后续的分析。
1.3 高通量荧光定量PCR(HT-qPCR)定量检测
抗生素抗性基因检测采用超高通量荧光定量PCR系统完成,该系统每次能够并行5 184个纳升级qPCR反应。其中共设置了72对引物,其中包括1对16s rRNA内参引物,8个转座酶(transposon),2个整合子和61个靶向ARGs,实现快速准确地检测各类环境介质中ARGs的多样性和丰度信息。PCR反应混合液先使用纳升级多样品点样仪加入到微孔芯片中,随后进行qPCR反应。反应程序为95℃预变性10 min,然后进行40个循环的扩增阶段({95 ℃,30 s;60 ℃,30 s} 40次循环)。qPCR结果使用仪器的qPCR软件进行自动分析。Ct=31设为检出域,3个重复中有2个及以上检出,偏差< 20%,且满足曲线拟合分析的则判为检出。
1.4 统计分析
采用 Excel分析ARGs和MGEs的组成特征。采用SPSS对ARGs在土壤中迁移传播扩散与MGEs的相关性进行分析。
2 结果与分析
2.1 设施农业土壤ARGs与MGEs的种类和数目
土壤样品中检测到50种ARGs和10种MGEs。50种ARGs为1个氨基糖苷类(aminoglycoside)、2个多重耐药类(multidrug)、4个β-内酰胺酶类(β-lactamase)、4个氯霉素类(chloramphenicol)、13个大环内酯类(MLSB)、6个磺胺类(sulfonamide)、13个四环素类(tetracycline)、7个喹诺酮类(ouinolone)。其中β-内酰胺酶抗生素抗性基因blaTEM、fox5,大环内酯类抗生素抗性基因ermX、ermB、ermF、ermT-02,磺胺类抗生素抗性基因sul2、dfrA1、folA,四环素类抗生素抗性基因tetG-01、tetG-02、tetM-01、tetM-02、tetPA、tetPB-03、tetT和喹诺酮类抗生素抗性基因oprJ、acrA-04的检出率是100%。检测到的10种MGEs可移动基因元件为2种类型整合子和8种类型转座酶,其中整合子intI-1和转座酶tnpA-01、tnpA-05的检出率为100%。
土壤ARGs与MGEs数目分析如图1所示,新民土壤样品X的ARGs和MGEs检测到的数目是最高的,43个ARGs和8个MGEs。辽中土壤样品L检测到的数目是最低的,为33个ARGs和7个MGEs。
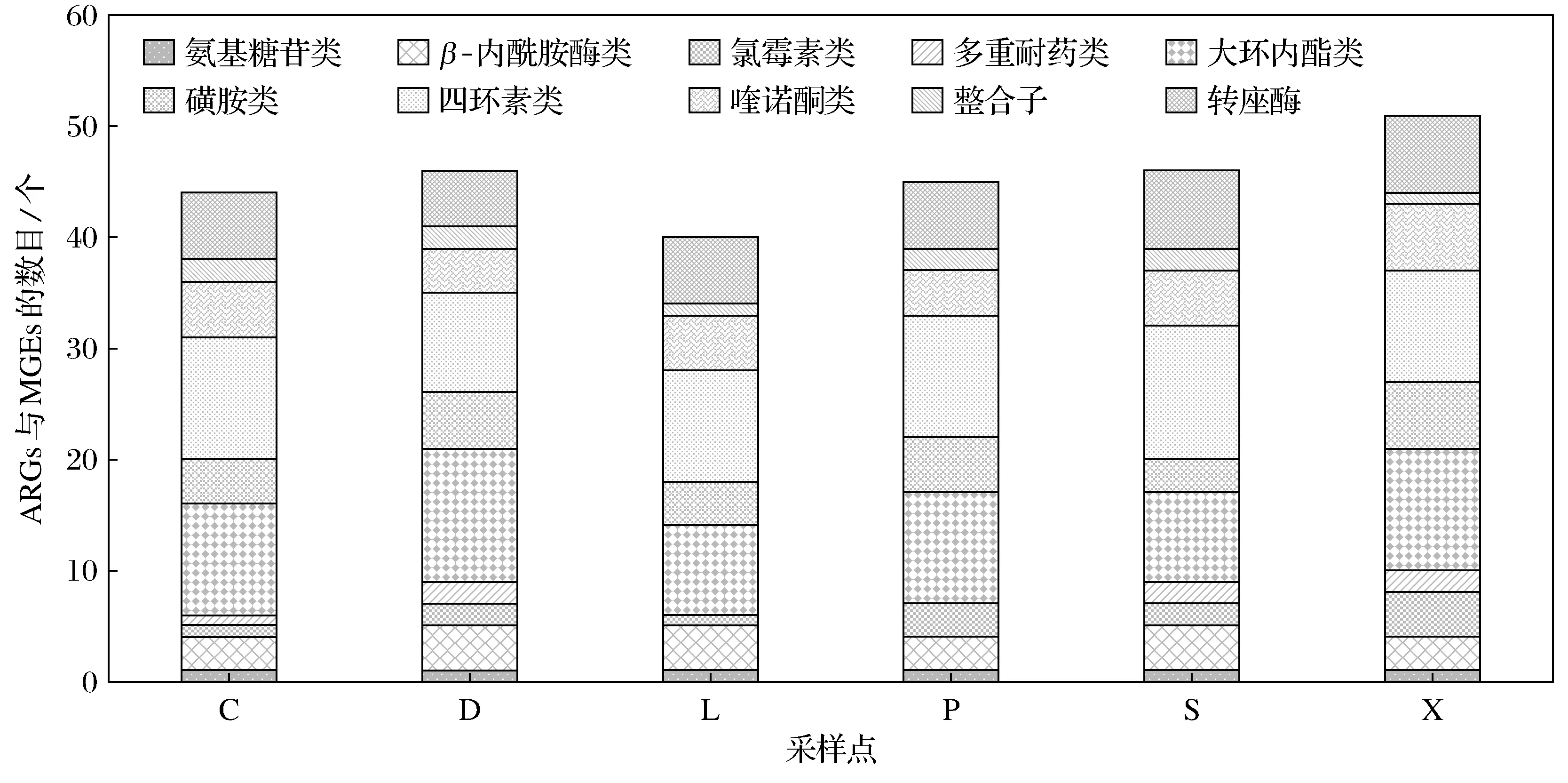
图1 不同采样点土壤中ARGs与MGEs的数目Fig.1 Numbers of ARGs and MGEs in soil at different sampling points
2.2 ARGs和MGEs相对丰度分析
不同采样点的土壤中微生物总量不同,为了减少偏差,将ARGs的浓度与内参基因16S rRNA的浓度进行归一化处理,得到土壤中各个ARGs及MGEs的相对丰度,如表2所示。分析可知,不同的设施农业土壤的ARGs及MGEs的分布特征存在一定差异。ARGs总和相对丰度从大到小为: D、L、C、X、P、S;MGEs总和相对丰度从大到小为:S、C、X、L、D、P。其中庄河土壤样品D的ARGs总和相对丰度为1.75,沈北土壤样品S的ARGs总和相对丰度为1.05×10-1。在6个土壤样品中,喹诺酮类ARGs的相对丰度占比较大,其相对丰度为4.04×10-2~1.68。在土壤样品检测到的MGEs中,整合子的相对丰度为7.85×10-3~1.31×10-2,转座酶的相对丰度为2.83×10-3~1.65×10-1。
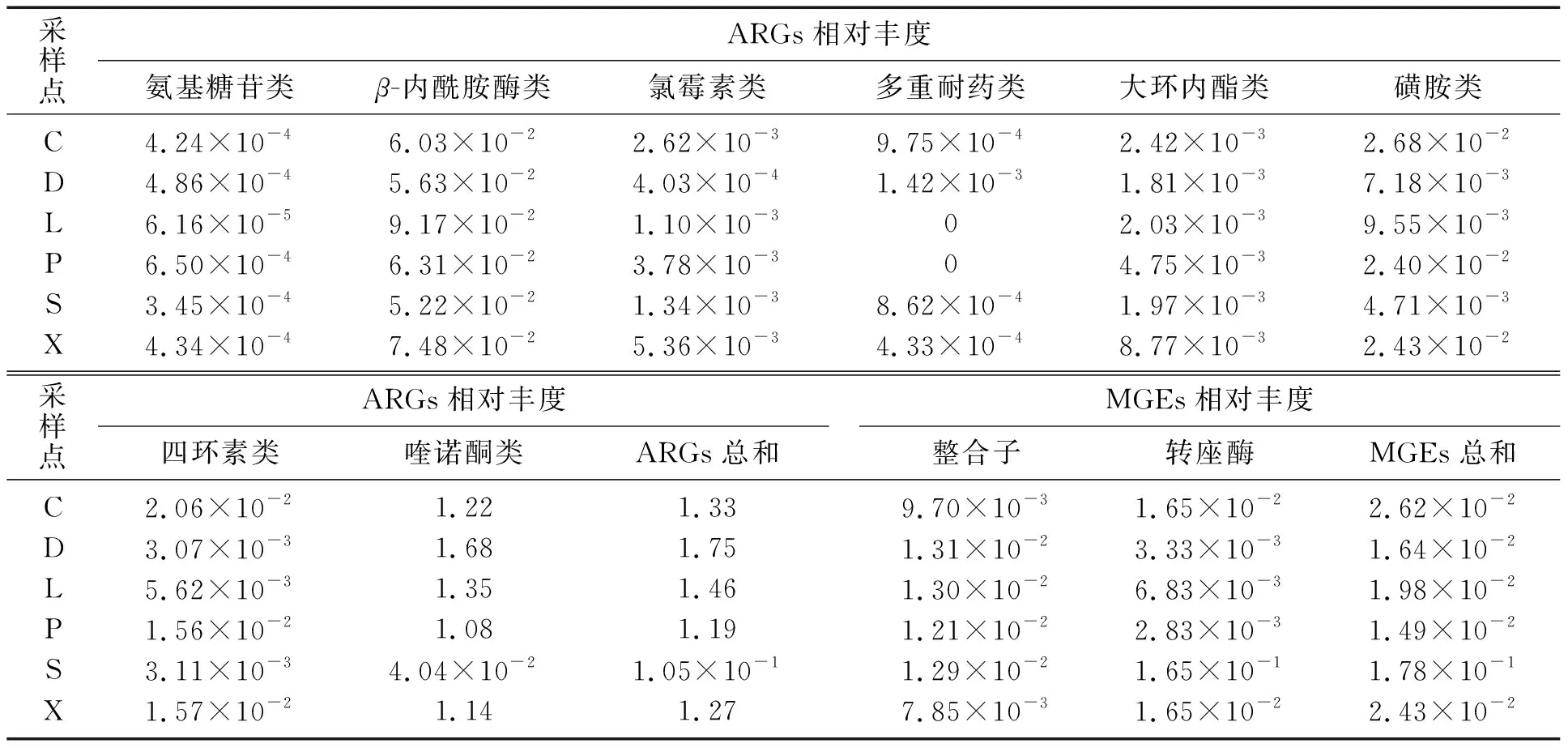
表2 不同采样点土壤中ARGs及MGEs相对丰度Table 2 Relative abundance of ARGs and MGEs in soil at different sampling points
2.3 ARGs和MGEs的抗性机制
通过比较分析不同设施农业土壤ARGs的抗性机制,可以清晰显示出细胞保护(cellular protection)、细胞外排泵(efflux pump)、抗生素失活(antibiotic deactivation)和转座酶移动元件(transposon)、整合酶移动元件(integron)等抗性机制在各采样点的数目,如图2所示。设施农业土壤采样点主要以细胞保护为主要抗性机制,且不同采样点的抗性机制存在不同。新民土壤样品X的抗性机制数量最高,且与其他土壤采样点相比,该地细胞保护、细胞外排泵的数量最高,其数值分别为18、12;辽中土壤样品L的抗性机制数量最少,与其他土壤采样点相比,该地细胞外排泵、整合酶移动元件抗性机制的数量较少,其数值分别为9、1。不同设施农业土壤采样点的抗性机制表现出不同特性,从而反映出不同设施农业土壤中抗生素抗性基因的差异。

图2 不同采样点土壤中抗性机制数目Fig.2 Number analysis of soil resistance in soils at different sampling points
2.4 ARGs与MGEs的相关性分析
对设施农业土壤ARGs和MGEs的相关性进行分析, 如表3所示。土壤中ARGs的相对丰度与MGEs转座酶的相对丰度存在相关性,其中β-内酰胺酶类抗生素抗性基因blaSFO、大环内酯类抗生素抗性基因ermB与转座酶IS613,喹诺酮类抗生素抗性基因oprJ、acrA-04与转座酶tnpA-02,氯霉素类抗生素抗性基因cmlA1-01、大环内酯类抗生素抗性基因ermC、ermX、磺胺类抗生素抗性基因dfrA1、sul1与转座酶tnpA-03,β-内酰胺酶类抗生素抗性基因blaSFO、氯霉素类抗生素抗性基因cfr与转座酶tnpA-04,大环内酯类抗生素抗性基因ermT-01、ermT-02、ermY、四环素类抗生素抗性基因tetL-02与转座酶tnpA-07的相对丰度均呈显著正相关(p<0.05)。此外,四环素类抗生素抗性基因tetO-01与转座酶tnpA-02,四环素类抗生素抗性基因tetX、氯霉素类抗生素抗性基因cmx(A)与转座酶tnpA-03,四环素类抗生素抗性基因tet(34)、喹诺酮类抗生素抗性基因qnrS与转座酶tnpA-04的相对丰度均呈极显著正相关(p<0.01)。说明MGEs可能会促进ARGs在设施农业土壤中的迁移和扩散。

表3 ARGs与MGEs相对丰度的相关性分析Table 3 Correlation analysis of relative abundance of ARGs and MGEs
3 讨 论
环境中抗生素的激增、抗生素抗性基因的扩散对人类和牲畜健康构成了严重威胁。长期施用畜禽粪便有机肥会增加土壤中抗生素耐药菌和ARGs的丰度,并提高ARGs的检出率[14]。研究发现,在施用粪肥的土壤中能检测到ARGs[15-16]和MGEs[17],一畜禽养殖场中检测到10种四环素类ARGs、2种磺胺类ARGs和整合子intl1;Tang等[18]在施用粪肥的土壤中也检测到tetA、tetG、sulI等ARGs。
本研究在6个土壤样品中检测到tetG-02、ermX、ermB、tnpA-05、tnpA-01、intI-1等,且在不同土壤中检测到的ARGs和MGEs数目及相对丰度存在不同。研究发现,沈阳蔬菜土壤中能检测到21种ARGs和6种MGEs,包括14种四环素类、3种磺胺类、3种氯霉素类、1种氨基糖苷类和4种转座子、2种整合子[19];黄福义等[20]在土壤中检测到tetD、mexF、tnpA、tp614等ARGs;曾庆涛[21]在农田土壤中检测到tetG、qnrX、sulI等ARGs。韩婉雪等[22]在土壤中检测到tetW、tetG、sulI等ARGs和intI1,四环素类及磺胺类ARGs的相对丰度为8.10×10-5~1.55×10-1、1.18×10-6~2.94×10-2;Zhou等[23]在农田土壤中检测到四环素类和磺胺类ARGs,相对丰度为10-8~10-2、10-2。
本研究中6个土壤样品中的ARGs与MGEs呈一定的正相关,表明MGEs可能促进ARGs在设施农业土壤中的迁移扩散。何燕等[24]发现稻田土中ARGs和MGEs的丰度呈极显著正相关(p<0.01),7个稻田土中的tetG、acrA-01、mexE等ARGs与intI1(clinic)、intI-1LC等整合子基因呈显著正相关;Yao等[25]研究发现intI与sul1,tnpA03与qnrS、ermA、ermB呈极显著正相关;张俊华等[13]发现MGEs与氨基糖苷类、氯霉素类ARGs呈显著正相关;韩婉雪等[22]研究表明,intI1与tetW、tetG、sulI、sulII呈显著正相关关系。
4 结 论
1) 从辽宁省主要设施农业土壤集散地的6个采样点中检测到50种ARGs和10种MGEs,并且其主要抗性机制为细胞保护。
2) 部分ARGs的相对丰度与MGEs转座酶的相对丰度呈显著正相关(p<0.05)和呈极显著正相关(p<0.01),说明MGEs可能会促进ARGs在设施农业土壤中的迁移和扩散。
