基于转录组的洋葱bHLH转录因子家族鉴定与生物信息学分析
2023-06-30陈微潘美红惠林冲李威亚郇国磊何林玉缪美华陈振泰杨海峰
陈微 潘美红 惠林冲 李威亚 郇国磊 何林玉 缪美华 陈振泰 杨海峰
摘要:bHLH转录因子家族是真核生物中重要的转录因子家族,在植物生长发育、次生代谢和逆境应答中发挥重要作用。本研究利用转录组数据对洋葱基因家族成员进行筛选和鉴定,并对家族成员进行理化性质、保守基序、亚细胞定位、系统发育和蛋白互作生物信息学分析,同时对bHLH基因家族在洋葱幼苗中的表达情况进行分析。结果表明,从洋葱转录组共鉴定到36个bHLH基因,预测编码蛋白质含有的氨基酸数量为127~610个,且均为亲水性蛋白,系统发育分析表明,洋葱bHLH基因家族分为12个亚族,同一亚族的大多数基因具有相似的保守基序,大多数bHLH 基因在洋葱幼苗中表达,不同基因的表达量水平差异较大。本研究结果为进一步研究洋葱bHLH 转录因子家族的生物学功能奠定了基础。
关键词:洋葱;bHLH;转录因子;生物信息学分析
中图分类号:S633.201文献标志码:A
文章编号:1002-1302(2023)05-0011-08
转录因子是真核生物中重要的调节因子,可以与顺式元件相互作用,调节特定基因的表达,来响应环境胁迫。bHLH( basic helix-loop-helix)转录因子家族是植物中最大的转录因子家族之一[1],具有高度保守的 bHLH 结构域,由大约50~60个保守氨基酸组成,包含2个功能区:一个是N端的碱性氨基酸区,长度约为15个氨基酸,另一个是 HLH 区[2]。bHLH 蛋白调节许多生理过程,在黄酮类生物合成[3-4]、信号转导[5]和植物生长发育[6-8]等方面发挥着重要作用。
随着不同物种全基因组和转录组测序的完成,多个物种中鉴定到bHLH 基因家族。目前,已从拟南芥[9]、毛竹[10]、胡萝卜[11]、人参[12]、梨[13]、苹果[14]、葡萄[15]等植物物种鉴定和分析了大量bHLH基因家族。随着 bHLH 蛋白功能的确定,bHLH 的分类不断得到改进。根据序列相似性和进化关系以及结合 DNA的能力,bHLH通常分为从A到F的6个主要组[16-17]。
洋葱是全球范围内种植的蔬菜之一,具有丰富的营养成分,很多国家视其为一种非常重要的蔬菜。迄今为止,bHLH转录因子的研究主要在拟南芥、葡萄、水稻等植物中,在洋葱中未见bHLH转录因子功能及理化性质的报道。本研究利用洋葱转录组测序的数据库,在转录水平上对bHLH基因家族进行鉴定,并对其基本理化性质、保守基序、蛋白互作、系统进化以及基因表达情况等进行生物信息学分析,此外,还筛选到一些可能参与洋葱黄酮生物合成途径的bHLH基因,结果可为洋葱AcbHLH基因功能的研究提供理论参考。
1 材料与方法
1.1 植物材料
本试验所用的材料来自江苏省连云港市农业科学院东辛基地,在苗期,选取长势相近的3株紫皮洋葱和3株黄皮洋葱,选取幼嫩鳞茎的表皮,液氮速冻后,-80 ℃冰箱保存备用,用于转录组测序(转录组数据已发表)。
1.2 数据来源及bHLH基因的鉴定
将测序得到的原始序列进行过滤,去除接头污染、低质量、N比例大于5%的Reads,得到质量较高的Clean Reads,利用Trinity软件对高质量序列进行混合组装,共获到77 474条Unigene[18]。分别从拟南芥数据库 ( https://www.arabidopsis.org)和水稻数据库(http://rapdb.dna.affrc.go.jp/download/irgsp1.html)下载拟南芥bHLH家族蛋白序列和水稻bHLH家族蛋白序列。然后使用HMMER 3.0软件对获取的已知bHLH蛋白家族序列构建隐马尔可夫模型,以此模型搜寻洋葱转录组所有编码蛋白序列,找出洋葱转录组蛋白序列中潜在的bHLH家族序列。使用BLASTP把洋葱转录组所有蛋白的序列比对获取的bHLH家族参考序列,e-value设为1e-10,比对上的序列用作候選的bHLH家族序列。
1.3 bHLH 生物信息学分析
利用在线软件ExPASy-ProtParam(http://web.expasy.org/protparam/)对洋葱bHLH蛋白序列的氨基酸数目、分子量、等电点、不稳定系数等理化性质进行预测。利用MEME软件(http://meme.nbcr.net/meme)分析洋葱bHLH家族的保守基序,保守基序的数目选择15,其他参数均采用默认值。利用ProtComp(http://www.softberry.com/berry.phtml?topic=protcomppl&group=programs &subgroup=proloc)网站对洋葱 bHLH家族成员的亚细胞定位进行预测。
1.4 洋葱bHLH 基因的系统发育
构建方法:用MAFFT进行多序列比对,然后用MEGA6.0软件对鉴定得到的洋葱、拟南芥的bHLH蛋白家族序列构建系统进化树。使用邻接法(neighbor-joining)和P-distance 模型构建进化树,校验参数Bootstrap设置为重复1 000次。
1.5 洋葱bHLH 蛋白功能联系网络
利用 String 蛋白相互作用的数据库(http://string-db.org/)分析洋葱bHLH 蛋白的互作网络,以模式植物拟南芥作为参考物种。
1.6 洋葱bHLH 基因的表达分析
利用洋葱转录组测序数据分析bHLH基因的表达特征。以FPKM 值表示转录本丰度,通过对数据均一化处理,利用Heml 工具做热图。
2 结果与分析
2.1 洋葱bHLH 基因家族的鉴定及理化性质分析
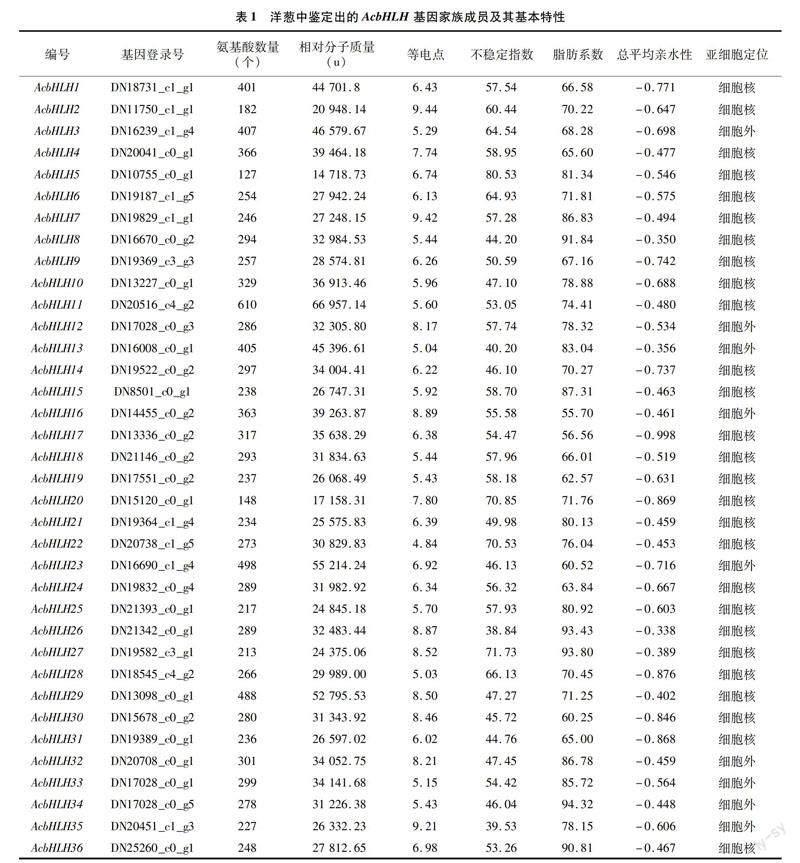
为了从洋葱转录组中获得bHLH家族基因,使用拟南芥和水稻的bHLH蛋白序列进行隐马尔可夫模型和BLASTP搜索,对获得的候选序列利用软件pfamscan和Pfam A数据库对目标序列进行结构域注释,确定将只含有保守结构域bHLH 基序(PF00010)的序列作为最终的bHLH序列,最终获得36个bHLH基因,根据转录组编号顺序将其命名为AcbHLH1~AcbHLH36(表1)。
对洋葱36 个bHLH基因编码蛋白质的基本理化性质进行分析(表1),结果显示,bHLH 基因编码的氨基酸数目为127~610个,平均长度为297个氨基酸,预测其相对分子量大小为14 718.73~66 957.14 u;等电点在4.84~9.44范围内。从整体看,有67%的洋葱bHLH家族蛋白质等电点小于7,等电点处于酸性范围内,蛋白质分子含有丰富的酸性氨基酸。蛋白质疏水性分析结果表明,洋葱中36个bHLH家族蛋白总平均亲水性均小于0,说明该家族蛋白质均属于亲水性蛋白质,但是不同蛋白质之间亲水性存在差异。不稳定指数在38.84~80.53之间,其中94.44%是不稳定蛋白(Ⅱ>40),只有2个属于稳定蛋白(Ⅱ<40),脂肪系数介于55.70~94.32之间。亚细胞定位预测结果显示,AcbHLH1等27个蛋白定位于细胞核,其余的9个定位于细胞外。
2.2 洋葱bHLH蛋白保守基序分析
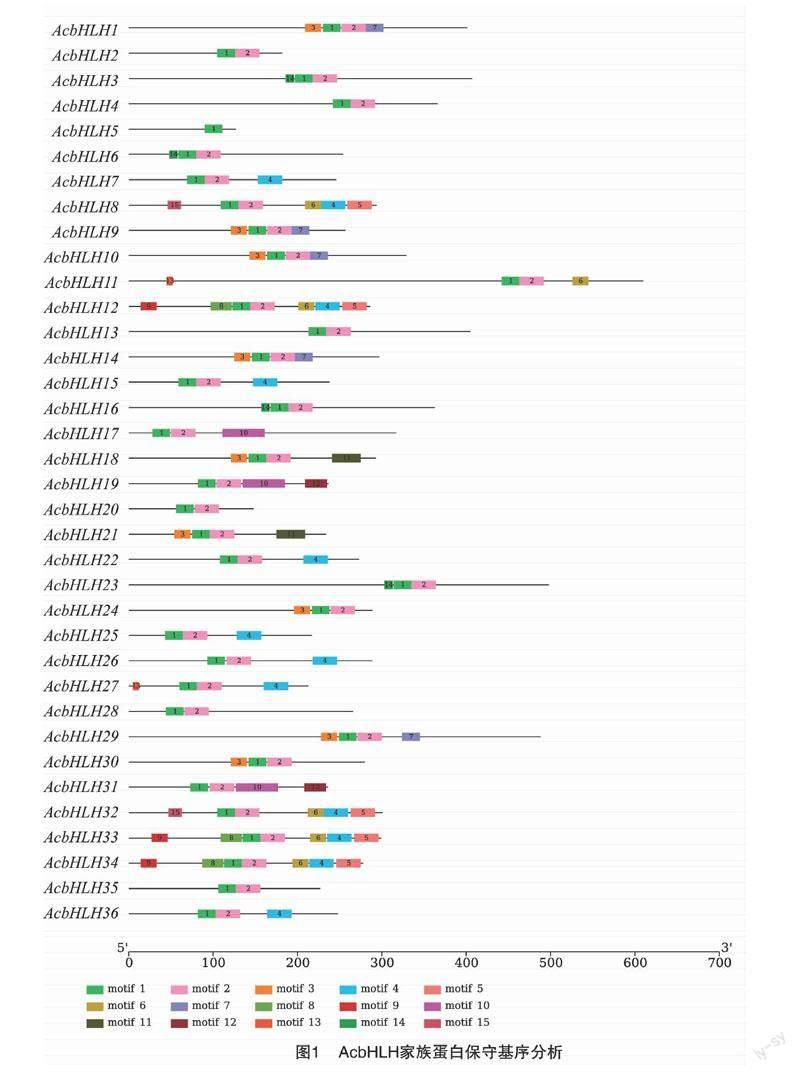
洋葱bHLH蛋白中的保守基序用在线软件MEME分析(图1),结果表明,36个bHLH基因家族成员中保守基序的数目及种类有差异,除AcbHLH5外, 所有的bHLH都含有基序motif 1和motif 2, 而且motif1和motif2 总是相邻,二者共同构成了bHLH 结构域。9个成员含有motif 3,6个成员基序数目和构成一样,都是motif 1、motif 2 和motif 4,AcbHLH8、AcbHLH12、AcbHLH33和AcbHLH34含有7个基序,是基序最多的bHLH蛋白。
2.3 洋葱bHLH 蛋白家族进化关系分析
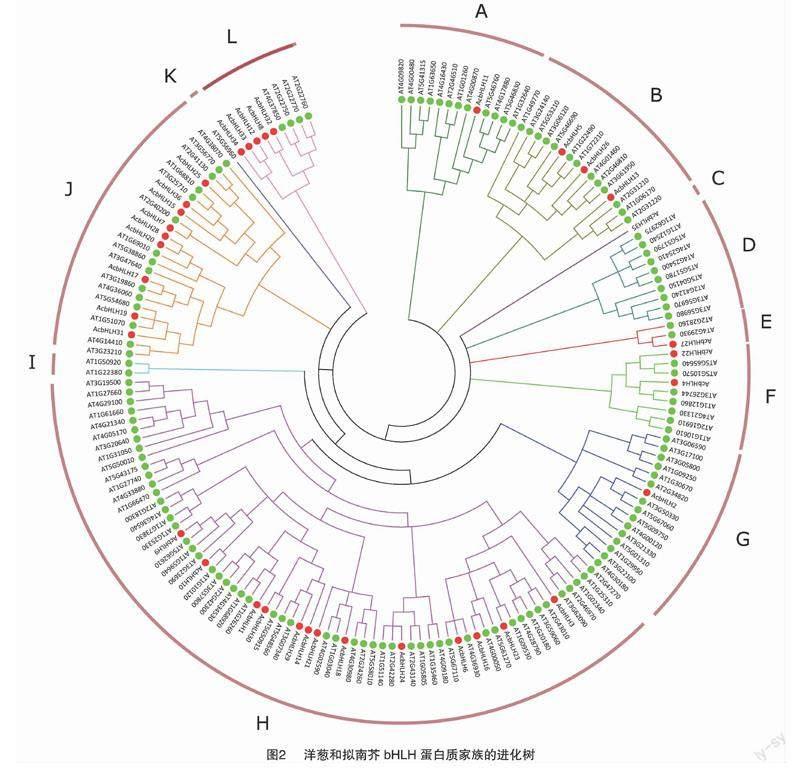
对拟南芥的bHLH蛋白序列和筛选的36条洋葱bHLH蛋白序列构建系统进化树(图2)。根据拟南芥中bHLH 转录因子的分类系统和洋葱bHLH转录因子的进化树分类情况,36个bHLH可以划分为12个亚族,其中数量最多的是H亚族,共13个,J亚族含有的bHLH数量次之,有9个,A、C、E和G亚族仅含有1个bHLH。
2.4 洋葱bHLH蛋白互作关系
利用在线软件STRING,预测洋葱bHLH家族成员在拟南芥中同源蛋白的互作关系(图3、图4)。
结果显示,洋葱bHLH家族成员和多个蛋白相互作用。其中光敏色素互作蛋白PIL5和PIF3共同作用来调节光形态发生。
2.5 洋葱bHLH基因家族表达分析
利用洋葱(紫皮和黄皮)的转录组数据库,找到候选的 36个bHLH基因对应转录本的RPKM 值,通过对RPKM 值取对数值转换,利用Heml 热图软件作热图(图5)。结果显示,36个bHLH基因在紫皮和黄皮洋葱中展现出不同的表达水平,AcbHLH7、AcbHLH32、AcbHLH33、AcbHLH22、AcbHLH6、AcbHLH35、AcbHLH5、AcbHLH19和AcbHLH26在紫皮洋葱中的表达量较高。
3 讨论与结论
作为植物中最大的转录因子家族之一,bHLH基因家族在植物的生长发育、新陈代谢以及应对胁迫方面起着重要的作用。基于全基因组研究的深入,越来越多物种的bHLH基因家族被陆续鉴定出来,其中拟南芥中鉴定出162个bHLH基因[19],水稻中167个,番茄中152个[20],葡萄中发现94个bHLH基因[15],黄花蒿中鉴定出99个bHLH基因[21],桃中有95个bHLH基因[22],陈红霖等从绿豆转录组中鉴定到122个bHLH转录因子[23],Zhu等在龙血树中发现了62个bHLH基因[24]。因为缺乏基因组,目前尚未见洋葱bHLH 基因家族鉴定及其功能分析的报道,本研究利用洋葱幼苗转录组数据库,对洋葱中bHLH基因家族成员进行鉴定,并对AcbHLH基因家族中各蛋白的基本理化性质、保守基序、亚细胞定位、系统进化、基因表達等方面进行了分析,为bHLH基因家族成员在结构和功能以及基因表达调控等方面的研究提供参考。
通过分析鉴定,去除冗余,在洋葱中鉴定出36个bHLH基因家族成员,根据转录组编号顺序,将洋葱36个bHLH基因分别命名为AcbHLH1~AcbHLH36。通过转录组在洋葱中鉴定到的bHLH蛋白数量远远低于拟南芥、水稻和番茄中bHLH蛋白数量,这种情况可能与洋葱基因组数据未获得和转录组取样时期有关,又或者测序组装的条件限制有尚未发现的洋葱bHLH基因家族成员。
洋葱中36个bHLH基因的保守基序利用线软件 MEME进行分析,共找到15个高度保守基序,motif 1和motif 2 几乎存在于所有的AcbHLH 蛋白中,它们总是彼此相邻,共同构成bHLH 结构域,具有相似motif组成的洋葱bHLH基因家族成员在聚类分析中距离较近,推测这些bHLH 保守基序在同一亚家族具有保守性及功能独特性,而其他的保守基序也在每个亚家族发挥着不同的功能。
拟南芥和洋葱bHLH蛋白的系统进化结果表明,36个bHLH划分为12个亚族,而拟南芥、水稻中的bHLH蛋白被划分为21、22个亚族[24-25],由于目前对洋葱bHLH蛋白的功能了解较少,无法更深入的分析其原因。
目前,对洋葱bHLH基因家族的研究远不如拟南芥、水稻等植物深入,bHLH 转录因子可以作为转录激活因子或抑制因子,在代谢和发育过程中起重要作用[26]。在拟南芥中已对大多数鉴定的 bHLH 蛋白进行了功能鉴定。如转录因子PIF3 作为光感受器信号的直接光敏色素反应伙伴,参与控制光调节基因的表达[9]。花青素是类黄酮中最大的一类[27],是最重要的次级代谢产物之一。相关研究表明,MYB 和 WD40 转录因子可以与 bHLH 相互作用,参与花青素和黄酮醇的生物合成。bHLH2 和 bHLH46 在龙血树中可能参与茉莉酸(JA)调节的黄酮类生物合成[28]。本研究中,通过网络共表达发现bHLH 转录因子PIF3与HY5相互作用,HY5在花青素的合成中具有一定的作用,因此,推测PIF3可能在洋葱花青素的合成中起到一定的作用,但具体的作用仍需进一步验证。
由于缺少基因組,目前在洋葱分子生物学方面的研究相对滞后,未见洋葱中bHLH 家族基因的报道,对该家族基因的功能和分类还缺乏系统深入的研究。本研究通过对转录组测序得到的洋葱bHLH家族基因进行鉴定,并对其进行生物信息学、保守基序、系统进化分析以及基因表达分析,为进一步筛选洋葱bHLH家族基因,揭示该家族基因参与植物生长发育的调控机制奠定理论基础。
参考文献:
[1]Hong Y Q,Ahmad N,Tian Y Y,et al. Genome-wide identification,expression analysis,and subcellular localization of Carthamus tinctorius bHLH transcription factors[J]. International Journal of Molecular Sciences,2019,20(12):3044.
[2]Wang J Y,Hu Z Z,Zhao T M,et al. Genome-wide analysis of bHLH transcription factor and involvement in the infection by yellow leaf curl virus in tomato (Solanum lycopersicum)[J]. BMC Genomics,2015,16(1):1-14.
[3]Xu W J,Dubos C,Lepiniec L. Transcriptional control of flavonoid biosynthesis by MYB-bHLH-WDR complexes[J]. Trends in Plant Science,2015,20(3):176-185.
[4]Zhao M,Li J,Zhu L,et al. Identification and characterization of MYB-bHLH-WD40 regulatory complex members controlling anthocyanidin biosynthesis in blueberry fruits development[J]. Genes,2019,10(7):496.
[5]Zhu E G,You C J,Wang S S,et al. The DYT1-interacting proteins bHLH010,bHLH089 and bHLH091 are redundantly required for Arabidopsis anther development and transcriptome[J]. The Plant Journal,2015,83(6):976-990.
[6]Lu R,Zhang J,Liu D,et al. Characterization of bHLH/HLH genes that are involved in brassinosteroid (BR) signaling in fiber development of cotton (Gossypium hirsutum)[J]. BMC Plant Biology,2018,18(1):304.
[7]Xing B C,Yang D F,Yu H Z,et al. Overexpression of SmbHLH10 enhances tanshinones biosynthesis in Salvia miltiorrhiza hairy roots[J]. Plant Science,2018,276:229-238.
[8]Zhu Z G,Liang H L,Chen G P,et al. The bHLH transcription factor SlPRE2 regulates tomato fruit development and modulates plant response to gibberellin[J]. Plant Cell Reports,2019,38(9):1053-1064.
[9]Toledo-Ortiz G,Huq E,Quail P H. The Arabidopsis basic/Helix-loop-Helix transcription factor family[J]. The Plant Cell,2003,15(8):1749-1770.
[10]Cheng X R,Xiong R,Liu H L,et al. Basic helix-loop-helix gene family:genome wide identification,phylogeny,and expression in Moso bamboo[J]. Plant Physiology and Biochemistry,2018,132:104-119.
[11]Chen Y Y,Li M Y,Wu X J,et al. Genome-wide analysis of basic helix-loop-helix family transcription factors and their role in responses to abiotic stress in carrot[J]. Molecular Breeding,2015,35(5):125.
[12]Chu Y,Xiao S M,Su H,et al. Genome-wide characterization and analysis of bHLH transcription factors in Panax ginseng[J]. Acta Pharmaceutica Sinica B,2018,8(4):666-677.
[13]Jin C,Huang X S,Li K Q,et al. Overexpression of a bHLH1 transcription factor of Pyrus ussuriensis confers enhanced cold tolerance and increases expression of stress-responsive genes[J]. Frontiers in Plant Science,2016,7:441.
[14]Mao K,Dong Q L,Li C,et al. Genome wide identification and characterization of apple bHLH transcription factors and expression analysis in response to drought and salt stress[J]. Frontiers in Plant Science,2017,8(12):480.
[15]Wang P F,Su L,Gao H H,et al. Genome-wide characterization of bHLH genes in grape and analysis of their potential relevance to abiotic stress tolerance and secondary metabolite biosynthesis[J]. Frontiers in Plant Science,2018,9:64.
[16]Atchley W R,Fitch W M. A natural classification of the basic helix-loop-helix class of transcription factors[J]. Proceedings of the National Academy of Sciences of the United States of America,1997,94(10):5172-5176.
[17]Simionato E,Ledent V,Richards G,et al. Origin and diversification of the basic helix-loop-helix gene family in metazoans:insights from comparative genomics[J]. BMC Evolutionary Biology,2007,7:33.
[18]陳 微,潘美红,惠林冲,等. 洋葱转录组测序及黄酮类化合物合成相关基因的分析[J/OL].分子植物育种:1-6[2022-10-17].http://kns.cnki.net/kcms/detail/46.1068.S.20210816.1540.010.html.
[19]Carretero-Paulet L,Galstyan A,Roig-Villanova I,et al. Genome-wide classification and evolutionary analysis of the bHLH family of transcription factors in Arabidopsis,poplar,rice,moss,and algae[J]. Plant Physiology,2010,153(3):1398-1412.
[20]Sun H,Fan H J,Ling H Q. Genome-wide identification and characterization of the bHLH gene family in tomato[J]. BMC Genomics,2015,16(1):9.
[21]甘 雨,吴 端,张 栋,等. 黄花蒿bHLH转录因子基因家族鉴定及光调控分析[J]. 中国现代中药,2021,23(3):441-452.
[22]Zhang C H,Feng R C,Ma R J,et al. Genome-wide analysis of basic helix-loop-helix superfamily members in peach[J]. PLoS One,2018,13(4):e0195974.
[23]陈红霖,胡亮亮,王丽侠,等. 绿豆bHLH转录因子家族的鉴定与生物信息学分析[J]. 植物遗传资源学报,2017,18(6):1159-1167.
[24]Zhu J H,Xia D N,Xu J,et al. Identification of the bHLH gene family in Dracaena cambodiana reveals candidate genes involved in flavonoid biosynthesis[J]. Industrial Crops and Products,2020,150:112407.
[25]Heim M A. The basic helix-loop-helix transcription factor family in plants:a genome-wide study of protein structure and functional diversity[J]. Molecular Biology and Evolution,2003,20(5):735-747.
[26]Li X X,Duan X P,Jiang H X,et al. Genome-wide analysis of basic/helix-loop-helix transcription factor family in rice and Arabidopsis[J]. Plant Physiology,2006,141(4):1167-1184.
[27]Feller A,Machemer K,Braun E L,et al. Evolutionary and comparative analysis of MYB and bHLH plant transcription factors[J]. The Plant Journal,2011,66(1):94-116.
[28]Welch C R,Wu Q L,Simon J E. Recent advances in anthocyanin analysis and characterization[J]. Current Analytical Chemistry,2008,4(2):75-101.
收稿日期:2022-04-21
基金项目:江苏省农业科技自主创新资金[编号:CX(20)3164];连云港市财政专项(编号:QNJJ2005)。
作者简介:陈 微(1990—),女,山东菏泽人,硕士,助理研究员,主要从事蔬菜育种和栽培研究。E-mail:1135050599@qq.com。
通信作者:杨海峰,副研究员,主要从事蔬菜育种和栽培研究。E-mail:13775492959@163.com。
