茯苓细胞色素P450基因PcCYP的克隆与序列分析
2023-06-19朱闻君刘和平苏振佳刘金鑫舒少华
唐 静,朱闻君,刘和平,苏振佳,刘金鑫,舒少华*
· 药材与资源•
茯苓细胞色素P450基因的克隆与序列分析
唐 静1,朱闻君2,刘和平1,苏振佳1,刘金鑫1,舒少华1*
1. 华中农业大学植物科学技术学院,湖北 武汉 430070 2. 武汉轻工大学生物与制药工程学院,湖北 武汉 430023
筛选与茯苓生长发育相关的细胞色素P450基因序列,进一步丰富对茯苓生长发育理解。通过分子克隆到1个茯苓细胞色素P450基因,并利用在线工具分析其序列,预测蛋白的理化性质、结构域等分子特征;利用MEGA 5.0分别对氨基酸进行多序列比对和进化关系分析,并采用qRT-PCR方法检测不同发酵培养时间6、8、10 d的相对表达量。从茯苓中克隆到的存在可变性剪切,2个选择性剪切体和CDS区长度分别为1590、1542 bp,分别编码529、513个氨基酸。氨基酸序列分析发现其均具有保守的P450结构域,且具有信号肽和多个磷酸化修饰位点,属于分泌型的不稳定亲水蛋白。氨基酸序列多重比对及系统发育树结果显示PcCYP与担子菌的CYP502距离最近,推测PcCYP和CYP502具有相似功能,即参与调控茯苓子实体的发育。定量PCR结果显示,选择性剪接变体的表达量较低,因此是基因的主要剪接体。与茯苓子实体的生长发育密切相关,为进一步研究在茯苓生长发育、遗传规律及育种等方面的生物学功能奠定基础。
茯苓;细胞色素P450;基因克隆;生物信息学分析;基因表达;生长发育
细胞色素P450(CYP450)广泛分布于生物体中,是一类具有半胱氨酸-亚铁血红素结构的多功能超基因家族[1],主要参与到烷基的羟化、烯基的环氧化、烃基的氧化、氧化性脱氨、脱卤和脱氢、氧化性的碳,碳键断裂及一些还原反应[2-4]。研究表明,在真菌界大约有2500多种CYPs被发现,主要参与多环芳烃、烷烃、甾醇类化合物等初级、次级代谢产物的生物合成过程以及农药降解等方面[5-8]。在许多真菌来源的药物性分子的生物合成中,CYP扮演了重要的角色,如灵芝酸合成途径中CYP5150L8催化羊毛甾醇转化为3-羟基-羊毛甾-8,24-二烯-26-酸(HLDOA)[9];梭链孢烷抗生素的生物合成过程中P450氧化酶HelB1、HelB2、HelB4、HelB3分别催化C-4β的甲基羧基化、C-16羟化、C-20甲基羧基化、C-6羟化和C-7羰基化[10];蕈青霉素合成途径中PaxP催化paspaline转化为13-desoxypaxilline[11];赤霉素合成途径中P450-1催化贝壳杉烯酸转化为GA14[12];单端孢霉烯的合成过程中Tri4催化单端孢霉二烯经由4步加氧反应合成异单端孢霉烯三醇[13];去甲绿胶霉素的生物合成中VidE、VidG、VidR共同作用使4-methyl-fecosterone发生demethoxyviridin反应等[14]。除此之外,细胞色素P450在真菌子实体的形成过程中也具有调控作用,如通过基因敲除方法证实苹果腐烂病菌的缺失可降低产孢量[15];通过表达量分析发现10种强烈表达有利于牛樟芝L.子实体形成[16];利用差异显示方法证实与双孢蘑菇(Large) Sing.子实体形成有关等[17],但关于P450调控子实体形成机制的研究鲜有报道。
茯苓(Schw.) Wolf为多孔菌科药用真菌,是一种药食同源的大宗药材,其主要药用活性成分为茯苓多糖和三萜类化合物[18],但关于其药用成分合成机制的研究较少。另一方面,由于对茯苓子实体的形成缺乏系统的研究,导致其遗传基础研究相对薄弱。本实验基于前期构建茯苓菌核和菌丝的转录组文库中发现的具有差异表达的CYP450超家族基因的全长开放读码框[19],克隆获取了PcCYP1以及选择性剪切体PcCYP2的开放读码框全长序列,并就该基因的结构特征和功能进行初步分析,以期为进一步深入研究基因的功能奠定基础。
1 材料与仪器
1.1 材料
1.1.1 菌株和质粒 茯苓(Schw.) Wolf菌株GIM5.29购买自广东微生物所;大肠杆菌菌株DH5α购自上海唯地生物技术有限公司。
1.1.2 酶和生化试剂 PC82-2×A8 FastHiFi PCR MasterMix、Zero Blunt Quick Ligation Kit均购自北京艾德莱生物科技有限公司;RNAiso Plus试剂购自大连TaKaRa公司;RevertAidTM第一链cDNA合成试剂盒购自Thermo Scientific公司;Gel Extraction Kit购自北京康为世纪生物科技有限公司。PCR引物合成及序列测定由武汉擎科生物技术有限公司完成。其他化学试剂均为进口或国产分析纯。
1.2 仪器
DHP350型恒温培养箱(武汉瑞华有限公司),HNYC-1102CD型振荡摇床(天津欧诺有限公司),DL-CJ-2N型超净工作台(北京东联哈尔有限公司),电泳仪和水平电泳槽(北京六一生物科技有限公司),CFX Connect荧光定量PCR仪、MyCycler普通PCR仪、凝胶成像系统(美国Bio-Rad公司),NanoDrop(美国Thermo Fisher公司),水浴锅(武汉鑫科有限公司),灭菌锅(上海三申有限公司),电子天平和pH计(美国奥豪斯公司),离心机(美国Bio-Rad公司),−80 ℃超低温冰箱(中科美菱有限公司)。
2 方法
2.1 培养基和培养条件
LB、PDA和PDB培养基配方参考《基因工程实验指导(第2版)》。茯苓斜面菌株4 ℃保存,接种于PDA平板培养基上的茯苓菌株在28 ℃下培养,接种于PDB液体培养基中的在28 ℃、150 r/min条件下培养。大肠杆菌DH5α接种于LB培养基在37 ℃培养。
2.2 RNA提取及cDNA合成
收集在PDA平板上培养7 d的茯苓菌丝,在液氮中速冻并充分研磨粉碎后使用TaKaRa公司的RNAiso Plus试剂提取RNA,提取方法参考其说明书进行。并用使用RevertAidTM第1链cDNA合成试剂盒合成第1链cDNA。获得的cDNA测定浓度后−20 ℃保存备用。
2.3 PcCYP基因的克隆
结合报道的茯苓基因组信息(https://www. ncbi.nlm.nih.gov/nuccore/469660165? report= genbank)和本课题组转录本测序结果筛选出在茯苓菌丝和菌核不同生长阶段差异表达的基因,选择其中一条基因,根据其CDS序列设计基因特异性扩增引物PcCYP-F1和PcCYP-R1(表1),以cDNA为模板使用高保真酶进行扩增。PCR反应体系为25 µL 2×A8 PCR MasterMix,1 µL PcCYP-F1(10 µmol/L),1 µL PcCYP-R1(10 µmol/L),1 µL cDNA,总体积50µL。PCR反应程序为:95 ℃预变性5 min;95 ℃变性15 s,58 ℃退火30 s,72 ℃延伸1 min,32个循环;72 ℃延伸10 min。PCR产物经1%琼脂糖凝胶电泳检测后,对符合目的大小的条带进行切胶回收。回收产物与pZERO-Blunt平末端载体连接,转化至DH5α大肠杆菌感受态细胞,并涂布于含有50 μg/mL氨苄抗生素的LB固体培养基上,在37 ℃的培养箱中培养16 h。挑选单克隆进行菌落PCR鉴定,阳性菌株送于公司测序验证。
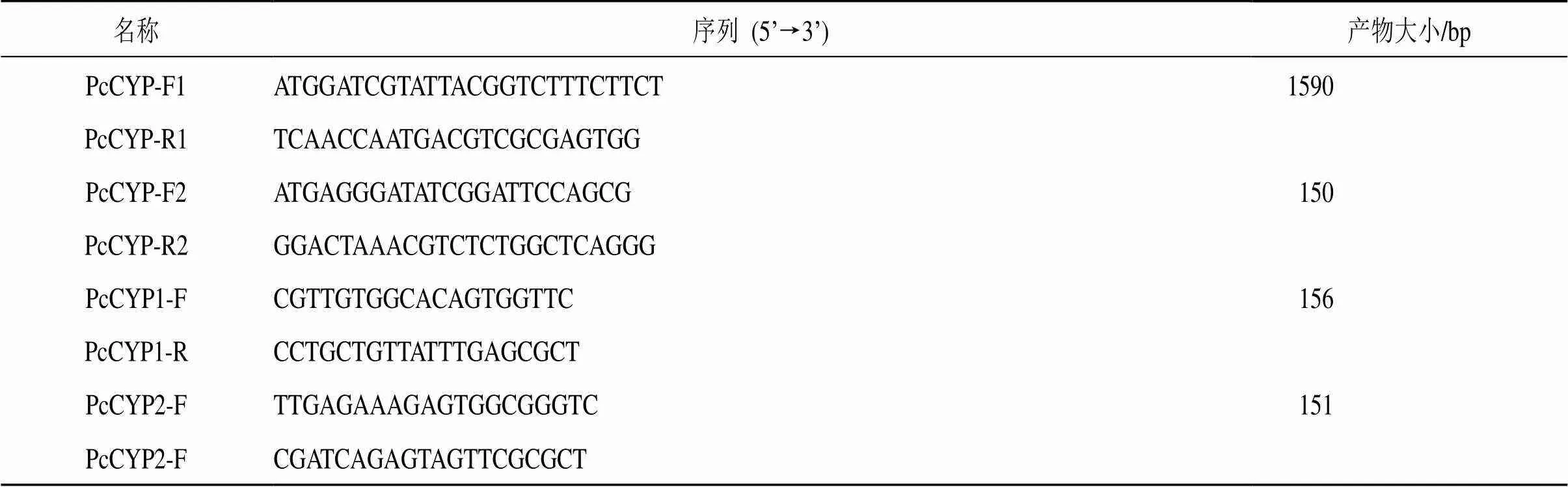
表1 引物序列
2.4 PcCYP基因的序列及系统进化树分析
按照表2中的登录号在NCBI里下载不同真菌物种的CYP450s氨基酸序列与本研究克隆得到的PcCYPs氨基酸序列比对,采用MEGA软件构建系统进化树(neighbor-joining,NJ),bootstrap设置为1000次[20]。

表2 不同真菌细胞色素P450的功能信息及NCBI登录号
2.5 PcCYP基因的基础生物信息学分析
将所得的PcCYP基因序列和氨基酸序列同源性比对:http://www.ncbi.nlm.nih.gov/blast; http:// www.ebi.ac.uk/Tools/clustalw2/index.html;PcCYP基因编码蛋白的基本理化性质利用ExPASy数据库(http://www.expasy.ch/tools/pi_tool.htm)进行分析;利用在线工具TMHMM Server 2.0(http:// www.cbs.dtu.dk/services/TMHMM/)、SignalP 5.0 Server(http://www.cbs.dtu.dk/services/SignalP/)、TargetP 2.0 Server(http://www.cbs.dtu.dk/services/ TargetP/)、NetPhos 3.1 Server(http://www.cbs. dtu.dk/services/NetPhos/)分别进行蛋白质跨膜区、信号肽、亚细胞定位、磷酸化位点分析;利用CDD(https://www.ncbi.nlm.nih.gov/Structure/cdd/ wrpsb. cgi)进行蛋白质保守结构域的分析。
2.6 PcCYP基因不同剪切体的表达量分析
根据PcCYP基因克隆得到的2个不同剪切体()分别设计荧光定量引物PcCYP1-F/PcCYP1-R、PcCYP2-F/PcCYP2-R(表1)。以his3-1为内参基因[21],利用qRT-PCR测定基因不同转录本在液体发酵培养6、8、10 d的菌丝体中的相对表达量。仪器使用CFX96实时定量PCR仪(美国伯乐公司),qRT-PCR实验方法按照康为世纪公司RealSYBR Mixture的实验说明书进行,每个反应3次重复,的相对表达量采用2−∆∆Ct法处理[22]。
3 结果与分析
3.1 茯苓PcCYP基因的克隆
以培养7 d的茯苓菌丝体为材料提取RNA,从电泳检测结果(图1)可知28 S条带的亮度大约是18 S的2倍,表明RNA没有降解,完整性较好,可以用于后续实验。
图1 RNA凝胶电泳图
Fig. 1 RNA quality detection by gel electrophoresis
根据从转录组和基因组信息中获取的基因CDS序列设计特异性引物,以反转录的cDNA为模板进行扩增,得到1个1600 bp左右的特异性条带,结果见图2。回收目的产物,克隆到平末端载体后送测序,测序结果显示基因的CDS有2种序列,其中较长的1条序列长度为1590 bp,编码529个氨基酸,而另1条较短的序列长度为1542 bp,编码513个氨基酸,初步推测基因可能存在选择性剪切。将该基因与由NCBI中茯苓基因组(https://mycocosm. jgi.doe. gov/cgi-bin/dispGene Model?db=Wolco1&id= 131022)进行比对,发现中共含有9个内含子和11个外显子,其中第9个外显子中的48 bp可能存在选择性剪切,当该区域在转录过程中被剪切,就会形成新的转录本即(图3)。为了进一步验证是否存在选择性剪切,在该区段的上下游设计特异性引物PcCYP-F2、PcCYP-R2(表1)进行扩增,PCR产物采用2.5%琼脂糖凝胶电泳检测,结果如图4所示。在100 bp和150 bp附近有2条条带,其中150 bp的基因片段亮度较弱,而100 bp的基因片段亮度较明亮,因此验证了选择性剪切的存在。

图2 PcCYP基因PCR扩增产物1%琼脂糖凝胶电泳检测
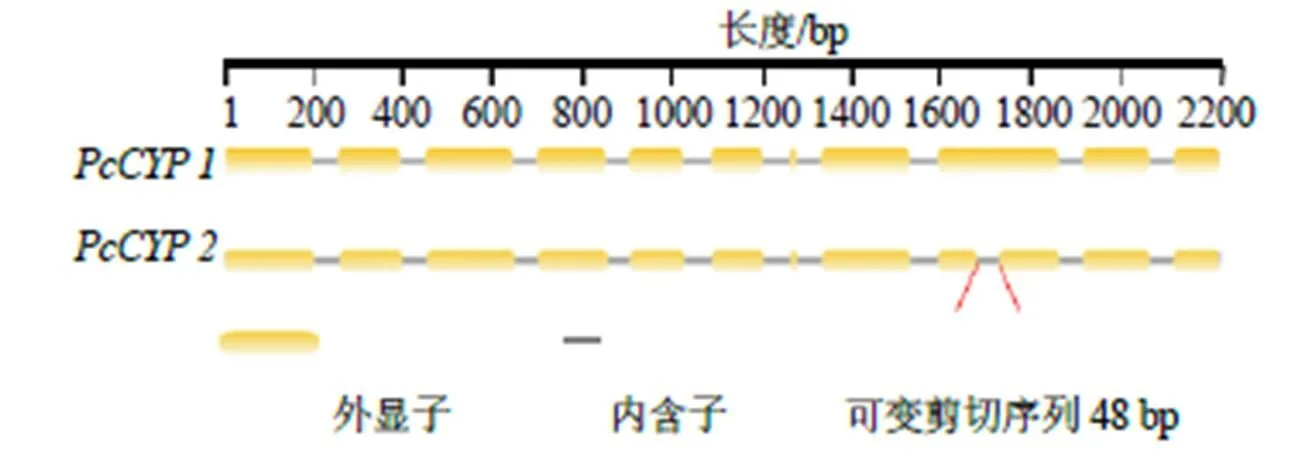
图3 PcCYP基因结构示意图

图4 PcCYP基因选择性剪切片段的2.5%琼脂糖凝胶检测
3.2 PcCYP系统发育分析
为了推测PcCYPs的家族分类以及功能,本研究从真菌细胞色素P450数据库中下载了来自不同物种且功能明确的60条CYPs氨基酸序列,并利用Clustal、Mega软件与PcCYP氨基酸序列进行比对分析和系统进化树的构建。结果如图5所示,来源于不同物种的CYP51、CYP52、CYP55、CYP53、CYP503等分别聚为一支,PcCYP与灰拟鬼伞菌中编码的CYP502比较相近,因此推测CYP可能属于CYP502家族基因。

图5 PcCYP蛋白序列的系统进化分析
3.3 PcCYP生物信息学分析
分别根据和CDS序列,翻译得到相应的2条氨基酸序列。使用ExPASY-ProtParam tool预测其蛋白基本理化性质,结果如表3所示,PcCYP1和PcCYP2相对分子质量分别为59 568.35和57 675.34,参考工具定义,当不稳定系数低于40时预测为稳定蛋白,超过40为不稳定蛋白,平均疏水性值为−2~2,负值表明该蛋白为亲水性蛋白,正值表示为疏水性,因此PcCYP1和PcCYP2均为不稳定亲水蛋白。同时,利用在线工具ProtScale预测蛋白一级结构上的亲疏性,结果(图6)表明大部分区域打分为负值,即表现为亲水性,与表3的结果一致。信号肽预测结果表明第1~17氨基酸为信号肽(图7)。跨膜结构域预测结果(图8)表明PcCYP1和PcCYP2均无跨膜结构。亚细胞定位预测结果表明PcCYP1和PcCYP2均存在于线粒体中。由于蛋白质磷酸化修饰在调节基因转录及改变蛋白质的功能方面具有重要作用,预测结果显示PcCYP1和PcCYP2均存在多个磷酸化位点,其中PcCYP1中包含苏氨酸(T)位点23个、丝氨酸(S)位点22个、酪氨酸(Y)位点11个;而PcCYP2基因由于缺失48 bp,所以预测的磷酸化位点包括苏氨酸(T)位点23个、丝氨酸(S)位点19个、酪氨酸(Y)位点10个(图9)。保守结构域分析如图10所示,PcCYP1的第295~473位和PcCYP2的295~457位氨基酸与CYP450超家族蛋白保守相似,属于CypX多保守域蛋白。此外,PcCYP1、PcCYP2均与pfam00067、PTZ00404具有部分相似位点,而由于选择性剪切基酸序列还与环二肽合成的CYP450(TIGR04538)具有相似位点。因此结合以上结果推测PcCYP1和PcCYP2经过不同的剪切方式翻译成不同的酶可能参与到线粒体中不同的反应途径。
表3 PcCYP理化性质预测
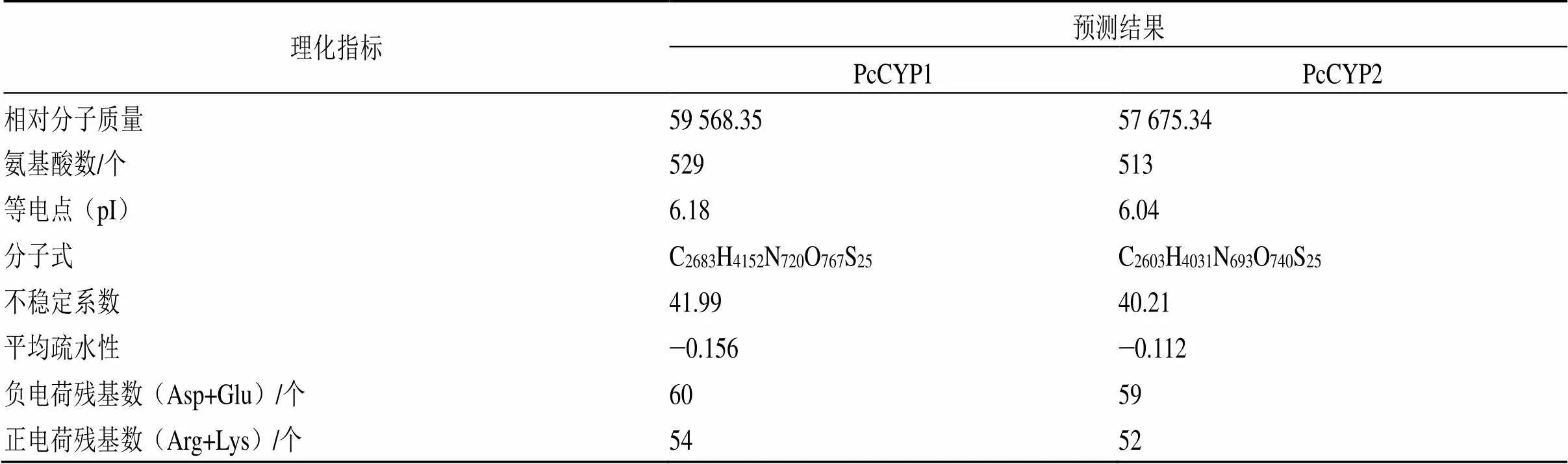
Table 3 Predicted physicochemical properties of PcCYP

图6 PcCYP氨基酸疏水性预测

图7 PcCYP蛋白信号肽预测
3.4 PcCYP1和PcCYP2的表达分析
分别针对和基因设计2对引物进行荧光定量PCR,由于比转录本多1个48 bp的内含子,因此,在此内含子中设计PcCYP1R,定量结果代表转录本的表达量。在和转录本的公共外显子区设计引物(PcCYP2F和PcCYP2R)的定量PCR结果代表2个转录本表达量之和,减去前面定量PCR的结果后,则为转录本真实的表达量。由图11表明,在3个时间段的表达量无显著性差异。在不同发酵培养时间下的表达量具有显著差异,在培养10 d时表达量最高,而在培养8 d时表达量最低。与的表达量相比,在6、8 d时,与的表达量无显著差异,但10 d时表达量显著高于。

图8 PcCYP蛋白跨膜结构域预测

图9 PcCYP磷酸化位点预测

图10 PcCYP蛋白保守结构域分析
4 讨论
细胞色素P450超家族基因广泛存在于生物体内,参与多种内源、外源物质的代谢途径中。随着真菌基因组学技术的发展,目前有399种CYP基因家族在2500种真菌中发现[23]。真菌CYP450分为CYP52、53、54、64等4个集团,各个集团又包含许多家族。而CYP51、56、59、61、67等家族是不归属于以上任何集团,其中CYP51和CYP61的进化较为保守,功能研究较透彻,主要参与到麦角甾醇的代谢途径中[24-25]。

图11 PcCYP1、PcCYP2基因的表达量分析
本研究基于课题组前期筛选出的具有差异表达的基因,选择其中1条基因进行了深入研究。本研究在对茯苓转录本的克隆中首次得到了它的2个转录本:(第9个外显子完整)和(缺失48 bp),编码的蛋白质大小分别为59 560和57 660。通过与60种功能明确的真菌P450氨基酸序列进行比对和系统进化分析可知,PcCYPs与腐生菌灰盖鬼伞菌的CYP502氨基酸序列相似度最大,进化分析也属于同一分枝,同属于伞菌纲。研究表明CYP502蛋白主要参与到的子实体发育过程中,当编码CYP502蛋白的基因发生突变时,子实体的原基菌柄分化受到影响,菌柄细胞伸长能力减弱,菌柄变得短小[26]。因此推测PcCYP可能在茯苓子实体的生长发育过程中具有调节作用,但具体的功能需要进一步的实验验证。进一步对PcCYPs的基本特性分析得出PcCYP1和PcCYP2均属于P450超基因家族酶,且属于分泌型的不稳定的疏水蛋白。亚细胞定位分析发现PcCYP可能位于线粒体中,同时对其进行蛋白质修饰位点分析发现PcCYP具有多个磷酸化修饰位点。研究表明,真菌CYP450蛋白质在其氨基酸序列的氨基端(N端)具有一段疏水性螺旋区域和一段富含脯氨酸的区域,这种特征能够使蛋白很准确地锚定在细胞器(膜)上[27],另一方面,分泌型的疏水蛋白可以通过水溶状态进行亲水/疏水两性分子界面的自我组装,从而在子实体的形成过程中发挥重要作用[28]。蛋白质磷酸化是在蛋白激酶催化作用下,供体分子磷酸基团与蛋白质侧链中含有羟基的氨基酸发生酯化反应的可逆过程,属于翻译后修饰加工过程[29]。蛋白磷酸化修饰可以改变蛋白质的结构、活性及其与其他分子相互作用的能力,是一种动态的生物调节过程,在信号传导、基因表达、细胞分裂等许多生物学过程的调控中起着重要作用[30]。因此,结合PcCYP蛋白系统进化和基本特性分析,本课题组可知PcCYPs有极大可能参与到茯苓子实体的形成过程中,且在对超表达菌株培养过程中,也偶然发现茯苓超表达菌株子实体的产生,但关于基因在茯苓子实体形成过程中的具体作用,这需要后期更深入的基因功能研究加以佐证。另一方面,的这些基本特性增加了其进行外源表达的难度,特别是大肠杆菌表达系统,因为原核生物缺乏翻译后修饰系统,所以CYP450在大肠杆菌中不表达或表达量低且不具有活性,但可以通过优化序列,变该酶的拓扑结构,实现该蛋白在原核系统中的表达[31-32]。
前体mRNA的选择性剪切是真核生物转录后重要的加工环节,通过对外显子和内含子的选择性去除和保留,可以使一个基因产生多个成熟的mRNA,进而翻译成具有不同功能的蛋白质,参与到不同的生物过程[33]。通过qRT-PCR分析基因2种转录本在不同培养时间的表达量可知,的表达量随着培养时间的延长并没有显著变化,的表达量则是先降低再增加,但整体上的表达量是明显高于,因此推测可能是主要的选择性剪切体。选择性剪切是在RNA水平上调控基因表达的机制至之一,一个基因可通过选择性剪切产生多个转录本编码不同结构和功能的蛋白质,它们分别在细胞或个体分化发育不同阶段,在不同的组织, 有各自特异的表达和功能[34]。因此推测PcCYP1和PcCYP2可能与茯苓不同培养时期的组织特异性有关,至于PcCYP的可变剪接受到的调控机制以及它是否参与调控茯苓子实体的生长发育,后期可以通过基因超表达和沉默等方法进行深入的研究。
利益冲突 所有作者均声明不存在利益冲突
[1] Omura T, Sato R. A new cytochrome in liver microsomes [J]., 1962, 237: 1375-1376.
[2] de Montellano P R O, De Voss J J. Oxidizing species in the mechanism of cytochrome P450 [J]., 2002, 19(4): 477-493.
[3] Meunier B, de Visser S P, Shaik S. Mechanism of oxidation reactions catalyzed by cytochrome P450 enzyme [J]., 2004, 35(46): 123.
[4] Sono M, Roach M P, Coulter E D,. Heme-containing oxygenases [J]., 1996, 96(7): 2841-2888.
[5] da Silva M, Cerniglia C E, Pothuluri J V,. Screening filamentous fungi isolated from estuarine sediments for the ability to oxidize polycyclic aromatic hydrocarbons [J]., 2003, 19(4): 399-405.
[6] Hunter A C, Bergin-Simpson H. Distinct metabolic handling of 3beta-hydroxy-17a-oxa--homo-5alpha- androstan-17-one by the filamentous fungusKITA: Evidence in support of steroid/hydroxylase binding hypothesis [J]., 2007, 1771(9): 1254-1261.
[7] Kasai N, Ikushiro S, Hirosue S,. Enzymatic properties of cytochrome P450 catalyzing 3'-hydroxylation of naringenin from the white-rot fungus[J]., 2009, 387(1): 103-108.
[8] Kullman S W, Matsumura F. Metabolic pathways utilized byfor degradation of the cyclodiene pesticide endosulfan [J]., 1996, 62(2): 593-600.
[9] Wang W F, Xiao H, Zhong J J. Biosynthesis of a ganoderic acid inby expressing a cytochrome P450 gene from[J]., 2018, 115(7): 1842-1854.
[10] Mitsuguchi H, Seshime Y, Fujii I,. Biosynthesis of steroidal antibiotic fusidanes: Functional analysis of oxidosqualene cyclase and subsequent tailoring enzymes from[J]., 2009, 131(18): 6402-6411.
[11] Saikia S, Parker E J, Koulman A,. Defining paxilline biosynthesis in: Functional characterization of two cytochrome P450 monooxygenases [J]., 2007, 282(23): 16829-16837.
[12] Malonek S, Rojas M C, Hedden P,. Functional characterization of two cytochrome P450 monooxygenase genes, P450-1 and P450-4, of the gibberellic acid gene cluster in(MP-D) [J]., 2005, 71(3): 1462-1472.
[13] McCormick S P, Alexander N J.encodes a multifunctional oxygenase required for three oxygenation steps [J]., 2007, 53(5): 572-579.
[14] Wang G Q, Chen G D, Qin S Y,. Biosynthetic pathway for furanosteroid demethoxyviridin and identification of an unusual pregnane side-chain cleavage [J]., 2018, 9(1): 1838.
[15] 高明煜, 吴玉星, 朱百涛, 等. 苹果树腐烂病菌细胞色素P450基因Vmcyp5的功能 [J]. 微生物学报, 2018, 58(2): 274-283.
[16] Hsu K H, Lee Y R, Lin Y L,. Cytochrome P450 genes in medicinal mushroomT.T. Chang et W.N. Chou (higher basidiomycetes) are strongly expressed during fruiting body formation [J]., 2011, 13(6): 513-523.
[17] de Groot P W J, Schaap P J, van Griensven L J L D,. Isolation of developmentally regulated genes from the edible mushroom[J].(), 1997, 143(Pt 6): 1993-2001.
[18] 刘惠知, 吴胜莲, 张德元, 等. 茯苓药物成分提取分离及其药用价值研究进展 [J]. 中国食用菌, 2015, 34(6): 1-6.
[19] Shu S H, Chen B, Zhou M C,.to reveal genes related to biosynthesis of triterpenoids [J]., 2013, 8(8): e71350.
[20] Córdova P, Gonzalez A M, Nelson D R,. Characterization of the cytochrome P450 monooxygenase genes (P450ome) from the carotenogenic yeast[J]., 2017, 18(1): 540.
[21] Zhang X, Xu Z C, Xu J,. Selection and validation of reference genes for normalization of quantitative real-time reverse transcription PCR analysis in(Schw.) Wolf (Fuling) [J]., 2016, 11: 8.
[22] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2–ΔΔCTmethod [J]., 2001, 25(4): 402-408.
[23] Ichinose H. Molecular and functional diversity of fungal cytochrome P450s [J]., 2012, 35(6): 833-837.
[24] Nelson D R, Kamataki T, Waxman D J,. The P450 superfamily: Update on new sequences, gene mapping, accession numbers, early trivial names of enzymes, and nomenclature [J]., 1993, 12(1): 1-51.
[25] 冷欣夫, 邱星辉. 细胞色素P450酶系的结构、功能与应用前景 [M]. 北京: 科学出版社, 2001: 26.
[26] Hajime H, Muraguchi Z. A mutation in thegene encoding a cytochrome P450 ofaffects mushroom morphogenesis [J]., 2000, 29(1): 49-59.
[27] Ichinose H, Hatakeyama M, Yamauchi Y. Sequence modifications and heterologous expression of eukaryotic cytochromes P450 in[J]., 2015, 120(3): 268-274.
[28] 孟虎. 茯苓性不亲和模式的初步研究 [D]. 武汉: 华中农业大学, 2012.
[29] Kim J H, Lee J, Oh B,. Prediction of phosphorylation sites using SVMs [J]., 2004, 20(17): 3179-3184.
[30] 姜铮, 王芳, 何湘, 等. 蛋白质磷酸化修饰的研究进展 [J]. 生物技术通讯, 2009, 20(2): 233-237.
[31] 麦婉莹, 洪葵. 真菌细胞色素P450在大肠杆菌中的表达 [J]. 微生物学通报, 2019, 46(5): 1092-1099.
[32] Zelasko S, Palaria A, Das A. Optimizations to achieve high-level expression of cytochrome P450 proteins usingexpression systems [J]., 2013, 92(1): 77-87.
[33] Aaronson Y, Meshorer E. Stem cells: Regulation by alternative splicing [J]., 2013, 498(7453): 176-177.
[34] Nissim-Rafinia M, Kerem B. Splicing regulation as a potential genetic modifier [J]., 2002, 18(3): 123-127.
Cloning, characterization and expression of a novel cytochrome P450 genesin
TANG Jing1, ZHU Wen-jun2, LIU He-ping1, SU Zhen-jia1, LIU Jin-xin1, SHU Shao-hua1
1. College of Plant Science and Technology of Huazhong Agricultural University, Wuhan 430070, China 2. School of Biological and Pharmaceutical Engineering, Wuhan Polytechnic University,Wuhan 430023, China
A cytochrome P450 gene sequence related to the growth and development ofos was screened and reported to further enrich the understanding of the growth and development of.In this study, a cytochorome P450 genewas cloned from, and some bioinformatics tools were used to predict the physical and chemical properties of the protein, as well as the molecular characteristics of the domain; MEGA 5.0 was used to perform multi-sequence alignment and phylogenetic analysis of amino acids. The gene expression levels at 6d, 8d, and 10d of fermentation were quantified by qRT-PCR.The results showed that two transcripts of,andwere founded fromfor the first time. They were 1590 bp and 1542 bp in length and encoded 529 and 513 amino acids, respectively. Sequence analysis revealed that they both had a conserved P450 domain, a signal peptide and multiple phosphorylation sites, which secreted unstable hydrophobins; multiple alignments of amino acid sequences and phylogenetic tree results showed that the PcCYP was similar to CYP502 of the basidiomycete; qPCR results showed that the expression ofis lower than that ofsuggesting thatwas the main transcript ofHydrophobin PcCYP is closely related to the growth and development of the fruit body of, which lays a foundation for further research on the biological functions ofin the growth and development, genetic rule and breeding of.
(Schw.) Wolf; CYP450; gene cloning; bioinformatics analysis; gene expression; growth and development
R286.12
A
0253 - 2670(2023)12 - 3962 - 09
10.7501/j.issn.0253-2670.2023.12.022
2022-12-09
国家自然科学基金面上项目(81872948);名贵中药资源可持续利用能力建设项目(2060302-1907-05)
唐 静(1995—),女,硕士研究生,主要从事茯苓生长发育及次生代谢与调控方面的研究。E-mail: hzautj@163.com
通信作者:舒少华(1979—),男,副教授,主要从事药用植物栽培及次生代谢产物代谢与调控等方面的研究。E-mail: shushaohua@mail.hzau.edu.cn
[责任编辑 时圣明]
