融合语义特征与偏序关系的生物医学文档检索
2023-06-05何伟东杨志豪王治政林鸿飞
何伟东,杨志豪,王治政,林鸿飞
(大连理工大学 计算机科学与技术学院,辽宁 大连 116024)
0 引言
精准医学(Precision Medicine,PM)是整合应用现代科技手段与传统医学方法,系统优化人类疾病防治和健康促进的原理和实践,以高效、安全、经济的健康医疗服务获取个体和社会最大化健康效益的新型健康医疗服务范式[1-2]。该范式侧重于确定最适合个体患者独特属性的治疗方法。
随着精准医学的不断深化,越来越多的科学研究旨在面向重大疾病(如癌症)开展精准知识挖掘与推送。例如,TREC(Text REtrieval Conference)于2017 年提出的TREC-PM 任务可以为患者病例提供最相关的生物医学文章[3]。该任务通常被定义为ad-hoc 式的文档检索模式,即在相对稳定的数据库中为自由查询检索最相关的文档。在精准医学领域,自由查询通常由多个方面内容构成,其中蕴含了对被检索文档的医学信息需求。
如表1 所示,每个查询病例由疾病名称,基因名称和遗传变异类型以及人口统计信息(性别和年龄)组成。

表1 示例查询Table 1 Sample query
基于给定的输入病例,我们需要从候选文档集合中检索出与该查询最为相关的生物医学文档,这涉及文档检索的召回与重排序过程。候选生物医学文档的具体信息如表2 所示,它包含了文档的多方面信息,例如:发布时间、药物表、归一化的MESH 词、标题、文档唯一编号PMID、文档摘要等。
为了有效地检索目标文档,现有的工作大多采用两阶段的方式[4],即先使用召回模型从整个语料库中检索出一部分文档作为候选文档,然后使用更为复杂的排序模型对这些文档作进一步的重排。
第一阶段,即候选生成阶段,现有方法通常使用基于稀疏词袋表示的BM25 等传统检索模式来生成用于重排序的候选集合[5],模型具体可以使用Lucene、Solr、Elasticsearch 等工具实现。但是在生物医学中,医学概念和事件的表达方式千差万别,词汇不匹配是制约医学信息检索的主要问题之一。例如,查询“Tymlos 的潜在副作用是什么?”Tymlos 这种药物是以其品牌名称命名的,相关科学文献可能会更频繁地使用其别名Abaloparatide。
近年来,许多研究者开展了大量的工作来克服这种词汇差异,包括基于相关性反馈的查询扩展、查询词重新加权[6],但是它们都无法解决稀疏性表示对语义特征表征不充分的问题。因此,面向语义信息的密集表示受到了研究者们的关注,它们能够通过捕捉查询的深层语义特征来克服词汇不匹配的问题。基于BERT[7]和RoBERTa 等[8]预训练语言模型高性能密集表示,研究者提出了稠密段落检索器[9],旨在通过微调语言模型对文档进行编码,利用其强大的语义表示能力缓解词汇不匹配问题。
第二阶段,即精排序阶段,现有方法大多使用pointwise(单文档学习)排序方式来学习全局信息,如Subset Ranking[10]、McRank[11]、Prank[12]等。但是这些方法只建模了给定查询与单个文档之间的相关度,只学习到了候选文档和查询的绝对相关性,忽略了候选文档之间的相对关系,即“偏序”关系。因此,研究者引入pairwise(文档对学习)方法以弥补单文档学习方法的不足,如 Ranking SVM[13]、RankBoost[14]、RankNet[15]、GBRank[16]、IR SVM[17]等方法。通常,这些方法将排序问题转为二分类问题,即使用二分类器对文档对进行分类,以此判断两个文档的前后排序,赋予模型学习文档之间偏序关系的能力。但是,以上的方法在精准医学背景下的文档检索任务中面临以下问题:
(1)患者病例的查询文本长度与相关医学文档的长度差异通常很大。一般情况下,给定的患者病例查询与其相关的候选文档在文本长度上存在很大差异,因此在使用预训练语言模型表征“查询”和“文档”时,会出现查询特征的过度平滑的现象,从而导致查询失效。
(2)基于pointwise 或者基于pairwise 的方法仅仅探索了查询与文档之间的单一关系,即全局相关或者偏序相关。而在医学文档检索中,病例查询通常涉及多方面的专业医学信息,因此需要对文档相关性的概念作出更全面的约束,也需要挖掘相关文档内部的顺序关系。
为了解决上述问题,本文提出了一种基于生物医学预训练语言模型(BioBERT)的偏序文档检索方法,如图1 所示。
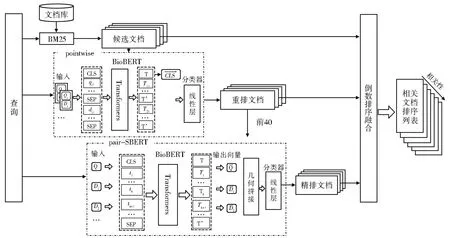
图1 偏序文档检索方法框架图图中分为四个部分,分别是基于BM25的召回模块、基于pointwise的重排模块、基于pairwise的精排模块(pair-SBERT)以及基于RRF(倒数排序融合)的排序融合模块Fig.1 Partial order document retrieval method framework diagramThe figure is divided into four parts, namely the recall module based on BM25, the rearrangement module based on pointwise method,the fine sorting module based on pairwise method (pair-SBERT) and the sorting fusion module based on RRF (reciprocal sorting fusion)
首先,该模型基于BM25 召回部分相关文档,然后使用 BioBERT 对病例查询和相关文档进行编码,采用pointwise 方法学习查询与相关文档的全局关系。其中,模型引入查询和文档的串联拼接来避免查询特征的丢失。随后,该模型引入pairwise 在查询与文档的全局关系中增加相关文档之间的偏序关系。其中,除了使用“查询-文档”对特征的几何拼接外,模型再次引入查询特征来指导文档对内部的偏序特征学习。最后,该模型将第一阶段的BM25 得分,第二阶段的pointwise 得分和pair-SBERT 得分进行融合,得到最终的文档相关度排名。
综上所述,本文的主要贡献如下:
(1)本文探索了领域知识需求更为严格的医学领域查询及相关文档检索研究;
(2)提出了一种融合语义信息与偏序关系的检索方法,除捕捉文档与查询的全局关系以外,该方法还能挖掘相关文档之间的偏序关系;
(3)本文进行了大量经验性实验,验证了本模型在精准医学领域中相关文档检索的有效性。
1 相关工作
1.1 文档检索
现有工作基本上都是基于神经模型进行检索。为了获得高效率,Tang 等[18]设计了一种方法,通过迭代聚类过程模拟每个文档上的查询,并用多个伪查询(即聚类质心)来表示文档。Manotumruksa 等[19]发现查询与文档的拼接顺序会影响排序结果,因此提出了CrossBERT的三元组网络结构,用以挖掘不同方式拼接带来的深层信息。
针对于本文中使用的TREC 精准医疗数据集,许多研究者也做了大量的研究。Akabe等[20]提出的方法基于释义语料库递归地查找释义,扩展源文档,生成释义格(Recursive Paraphrase Lattice),将文档进行扩充来提高检索性能。Qu 等[21]针对于Trec 语料中不同信息类别构建了不同的分类器,再将多个分类器分类结果提供给决策树计算文档相关性。Rybinski等[22]开发了Science 2Cure(S2C)系统,该系统是一个结合了传统倒排索引和神经检索组件的检索系统。
1.2 排序学习
根据样本空间和损失函数的定义方法不同排序学习方法可分为 pointwise、pairwise 和listwise 三类方法,其中pointwise 方法和pairwise 方法最为常用。pointwise 方法将排序任务转化为分类任务或回归任务。徐博等[23]使用手工构建的特征作为排序学习的输入,随着深度学习的发展,庞博等[24]结合深度学习的排序学习方法极大地提高了排序的性能,近年来预训练模型在自然语言处理领域展现出强大的能力,Karpukhin 等[25]结合预训练模型BERT 提出了基于预训练模型的检索模型,通过深层语义信息对文档进行打分。
Pairwise 方法不对文档相关性得分进行学习,而是学习不同文档之间的前后偏序关系。对于每一个文档,pairwise 需要计算它与其他文档的偏序关系,通过拓扑排序将所有偏序关系对排列,将得到最终的排序结果。Pradeep 等[26]基于预训练模型T5[27]构建了一个pairwise 排序模型,实现了一个序列到序列的检索方法。
2 模型
2.1 基于BM25的召回阶段
本文在相关文档召回阶段使用基于词汇级别的BM25 方法,其相关度计算公式如下:
其中Q代表一个查询,qi表示查询中的一个单词,d代表相关文档,Wi表示单词权重。这里使用IDF(inverse document frequency)作为权重,如公式(2)所示。
其中N表示索引中的文档数,dfi表示包含qi的文档个数。
公式(1)中的R(qi,d)表示qi与文档d的相关性,其计算公式如(3)所示。
其中,tfid表示单词qi在文档d中的词频,Ld是文档d的长度,Lave是所有文档的平均长度,k1与b是可调节参数。这里k1=2,b=0.75[19]。
考虑到表2 中所示的文档内容包含了多个不同的字段信息,但这些字段并不都能促进相关文档的检索,因此在该阶段的文档召回时,我们只使用题目、摘要以及MESH 词字段来表示文档。
综合公式(1)-(3),我们可以从海量的备选文档中召回一个数据规模较小的候选文档用于后续的重排步骤。
2.2 基于pointwise的排序模型
对于召回的候选相关文档,我们使用pointwise 方式对其进行重排序。考虑到词汇级匹配难以解决词汇鸿沟的问题,因此在本模块中,我们使用生物医学预训练语言模型(BioBERT) 对查询和候选文档集进行编码,从而获得它们深层的语义特征,以克服查询与候选文档中术语不匹配的问题。
首先,本模块将“查询-文档”对的串联作为编码器的输入,如公式(4)所示
其中qi表示查询中的单词,sj表示文档中的句子。
查询与文档通过公式(4)的方式拼接后送入BioBERT 模型中进行编码,然后使用“CLS”标识符的最后一层表示作为“查询-文档”对的表示,接着经过dropout 层(dp)与分类层(σ)预测查询与文档的相关性,如公式(5)所示:
当对候选文档集进行重排序时,则使用每个“查询-文档”对的相关性得分作为文档重排的依据。因此,使用该模型对召回的候选文档集进行重排,可以得到一个基于深层语义相关性的排序结果。
2.3 pair-SBERT模型
pointwise 重排后的文档集忽略了相关文档之间的相对位置关系,即偏序关系。因此我们提出了一个基于pairwise 的排序模型,即pair-SBERT。受到SentenceBERT 的启发,该模型通过使用查询与文档的几何拼接来捕捉文档间的偏序关系和学习相似文档间的差异信息,从而实现对重排后的文档集进行精排序。该模型的框架如图2 所示。
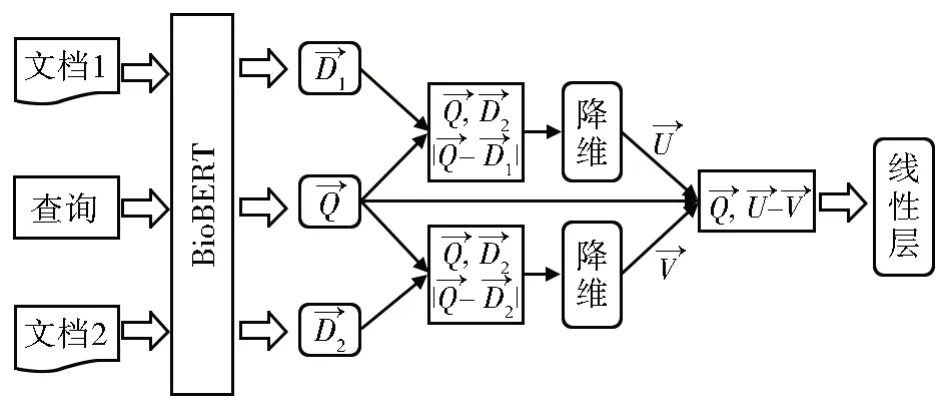
图2 Pair-SBERT 框架图Fig.2 Pair-SBERT framework diagram
对于重排后文档集中的任意文档Di和Dj,模型的输入内容如下所示:
查询:Q,文档i:Di,文档j:Dj。使用BioBERT 分别对Q、Di、Dj编码,输出上述三部分的Last_hidden_state 向量,记为Qlhs、、,如公式(7)-(9)所示:
接下来对三个Last_hidden_state 向量分别进行平均池化(mean_pooling)操作,得到向量i,i和j,以保证后续的向量拼接能够在同一维度上进行。公式如下:
得到查询和文档的向量表示后,该模型使用公式(13)和公式(14)将其进行几何交互,即
经过上述拼接操作后,向量维度会扩大数倍,这给模型计算带来了较大的资源开销,因此我们通过一个线性层对R1和R2进行降维,得到与。
随后,为了保证相关文档间的偏序关系在查询内容的范围之内,模型再次引入查询向量作为全局约束,并对R1和R2进行几何相减,以此引入两个文档之间的相对位置关系,如
分类层的输出包含两个神经元,其中,s0表示文档i排在文档j前边的分数,s1表示文档i排在文档j后边的分数。
训练时,根据s1与s0的差值得出最终的标签0 或者1,再与标准标签计算损失。
在预测时,文档i的分数应该是该文档与其他文档j拼接后得到的两个分数之和,即正序输入文档i排在文档j之前的分数与反向输入文档j排在文档i之后的分数,如公式(18)所示:
其中,D表示全部的候选文档集。
基于pair-SBERT,模型充分挖掘了相关文档的位置信息,得到了基于偏序关系的精排序结果。
2.4 排序结果融合
为了充分利用查询与相关文档的全部序列信息,本文将各个模型的结果进行融合,作为基于查询的最终结果排序。
候选集经过三个模块的打分之后,会得到三个排序序列:BM25 召回的排序S1、pointwise重排的全局排序S2,和使用pair-SBERT 模型精调的排序S3。但是不同模型计算的相关性得分难以直接相加,因为在排序集合S={S1,S2,S3}中,每个序列都是基于不同视角特征对候选集D 中的文档计算相关性得分。因此本文使用reciprocal rank fusion[28]方法融合不同的文档序列,得到最终的相关性文档顺序,如公式(19)所示:
其中s(d) 是排序si中文档d的排名,k为超参数。
3 实验设置
3.1 数据集构建
本文中在TREC-PM 赛道2017-2019 年数据上进行实验。
3.1.1 初始数据集
2017-2019 数据信息如表3 所示,共包含120 个查询与63 387 个带有标签的文档。

表3 数据集统计信息Table 3 Dataset statistics
本文将2017 年与2018 年数据作为训练集,将2019 年作为测试集。
3.1.2 pointwise数据构建
pointwise 模型的输入是一个查询与一个文档,输出是文档的相关性分数。在原始数据集中标签包含0,1,2 三类,为了适应于本方法,将1 与2 归类于relevant,将0 归为irrelevant。除了初始数据集训练数据外,本文还使用了随机采样与难负例采样技术对训练集进行了扩充。对于一个查询,随机采样指从整个数据库中随机获取若干个文档,去掉在初始数据集中出现过的文档,然后将这些文档作为负例加入训练集中。而难负例采样是指通过BM25 算法得到与查询相似度更高的一些文档,去掉在训练集中是正例的文档,剩余的添加到数据集中做负例。正负比例约为1∶10。
3.1.3 pair-SBERT数据构建
pair-SBERT 模型的输入是查询与两个文档,这两个文档具有不同等级的相关度标签。本文根据不同相关度标签的文档分布,随机组合查询与相关文档,并赋予其新的标签。例如,在同一个查询中,标签为0 的文档与标签为1 的文档组合为一条pairwise 输入,并标注1。标签为2 的文档与标签为1 的文档组合为一条pairwise 输入,并标注为0。
3.2 实现细节
(1)在文档召回阶段,设置召回文档数量为1000。
(2)在文档的重排序和精排序阶段,本文使用hunggingface 发布的预训练模型①https://huggingface.co/microsoft/BiomedNLP-PubMedBERT-base-uncased-abstract-fulltext编码源文本。实验中,epochs 设置为5,max_length 设置为512,learning_rate 设置为1×10-5,loss_function 设置为BCEWithLogitsLoss,optimizer 设置为Adam。pointwise 方法设置batchsize 为8。pair-SBERT 方法的batchsize 设置为2。结果融合部分k设置为60。
3.3 对比方法
本小节选取了一些具有代表性的工作进行对比。
本文中设置了两类对比实验,一类是基于查询与相关文档语义的方法,包括BM25,BITEM PM[29],Tree-soft[21],和Science2Cure[22]。
另一类是基于外部知识的方法。Julie[30]使用了BANNER gene tagger 对文档进行扩充,并使用Lexigram 对查询进行扩展。Akabe 等[20]提出的一个使用Recursive Paraphrase Lattice(释义格)的方法,该方法利用了释义语料库扩充了文档。
3.4 评价指标与结果分析
本文采用NDCG@10、Rprec 和p@10 三个指标进行评估。
表4 中“-”表示原论文中并未提及该数据。从表4 中我们可以得出如下结论:首先,与基于查询与相关文档的语义特征的方法相比,本文提出的模型在三个评价指标上取得了最好的结果。因此,在不引入外部知识的情况下,该模型在挖掘深度语义和利用相关文档的偏序关系精调文档方面具有优势。其次,与引入外部知识的方法相比,本文提出的方法优于Julie,但在NDCG 与Rprec 指标上略低于RPL。这主要是因为提出模型没有使用外部资源进行扩展查询,以保证模型的训练效率和较低的资源开销。虽然丢失了全局召回和排序位置的精度,但是模型因不受外部资源的约束,具有更好的可扩展性。最后,本文提出的模型在p@10 指标上比RPL 高了将近0.24,这再次说明了我们的模型取得了更好的相关文档检索精度,在挖掘深度语义和捕捉文档偏序关系方面表现良好。

表4 实验结果Table 4 Experimental results
3.5 消融实验
为了证明本文提出的模型的有效性,针对提出的各个模块进行了消融实验,如表5所示。
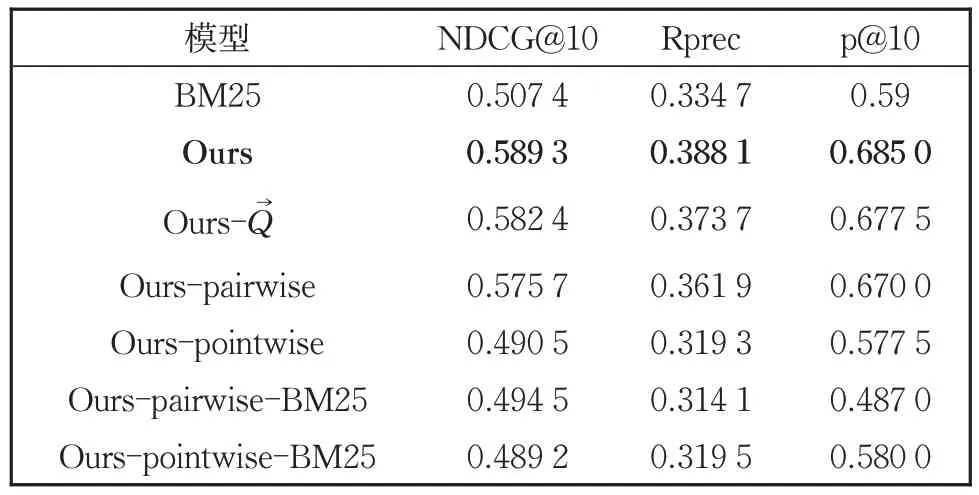
表5 消融实验结果Table 5 Results of ablation experiment
表5 中,Q→表示在pair-SBERT 模型中的拼接查询向量Q→的操作。首先,去掉拼接Q→的操作后(Ours-Q→),我们发现模型结果下降了约0.8%,说明拼接Q→操作是有效的,因为查询能更好的指导相关文档的偏序预测。其次,通过去掉pointwise 模型与pairwise 模型(Ours-pairwise 和Ours-pointwise)的实验结果可以看到,模型性能下降明显,说明这两个模块都有不可替代的功能。最后,通过观察只使用pointwise 或pairwise(Ours-pairwise-BM25 或Ours-pointwise-BM25)模型的结果可以看到,模型性能甚至低于基准方法BM25。
以上结果表明本文提出的排序模型的每一部分都是至关重要的,任何一部分的缺失都会引起整体性能的下降。
3.6 扩展实验
除了上述实验之外,我们还通过复现其他类似任务上的方法并与本文提出的方法进行对比,结果如图3 所示。
图3 中BM25+ATT[18]表示通过查询对文档做Attention,然后加权求和缩减文档的方法,BM25+CBERT[19]表示按照不同顺序拼接查询-文档对的方法,Ptw+SBERT[31]表示使用SentenceBERT 相似度表示的pointwise 方法。
通过图3 可以看出,我们的方法在TRECPM 2019 数据集上取得了最好的结果。这说明在精准医学背景的生物医学文档检索任务中,本文方法更能解决实际问题,是不能简单地通过迁移其他方法来替代的。此外,通过Ptw+SBERT 和BM25+Ptw+SBERT 这两个实验设置可以看出,本文使用的几何拼接方式对于学习文档偏序关系是更有效的。
4 结论与展望
本文提出了一种基于BioBERT 的偏序文档检索方法,解决了当前常用的检索方法因长度差异导致查询特征失效的问题,而且融合pointwise 方法与pairwise 方法能够挖掘出更多有用的文档排序特征,弥补了pointwise 与pairwise 方法在单独使用时不能完全挖掘特征的不足,BioBERT 的引入也在一定程度上改善了模型在医学领域编码的应用。相比于传统的pointwise方法与pairwise 方法,本文提出的方法获得了更好的检索性能。在TREC-PM 的数据集上的实验结果验证了该方法的有效性,它能够学习到邻近文档对的偏序关系,指导模型对相关文档的精排。未来的工作将尝试在文本编码过程中研发更有效的编码方式,加速检索效率,进一步优化检索过程。
