基于高通量测序技术研究原发性胃癌患者胃肠道菌群特征
2023-05-23凌宝殿谢志军张文娟王晓玲王芳胜胡晓梅赖姨梅吴何莉胡龙华
凌宝殿,谢志军,张文娟,王晓玲,王芳胜,胡晓梅,赖姨梅,吴何莉,胡龙华
(1. 赣南医学院第一附属医院检验科;2. 赣南医学院第一附属医院消化内科;3. 赣南医学院第一附属医院病理科,江西 赣州 341000;4. 南昌大学医学院第二附属医院检验科,江西 南昌 330000)
胃癌(Gastric cancer, GC)是全球第五大流行和第四大致命的恶性肿瘤,占所有癌症相关死亡人数的7.7%[1]。人体胃肠道定植大量微生物,微生物之间互相拮抗互为制约,维持相对稳定的微生物微环境,组成一个复杂的生态系统。机体胃肠道中的微生物在多种生理过程中发挥着重要作用,参与了能量代谢、营养吸收、肠道免疫系统的成熟以及防止病原体感染[2],也参与疾病的发生和发展。微生物群的改变可能与癌症有关[3-4]。幽门螺杆菌(Helicobacter pylori)感染与GC 密切相关。研究表明,尽管幽门螺杆菌可能参与了90%以上胃癌的发生,但它在胃炎晚期向癌症发展过程中的作用可能很小[5],这预示着可能存在其他潜在生物菌群[6]在胃癌发展中的作用。以往由于分子生物学技术的局限,很难全面了解人类胃肠道微生物菌群的组成。近年来,随着高通量测序技术的进步,推动了越来越多关于胃微生物群和肠道微生物群与GC 相关性研究。然而,有关胃微生物群和肠道微生物群的多样性与胃癌发生发展的相关性研究较少。本研究拟表征胃癌患者胃黏膜组织和肠道中的微生物群落,并探讨其与胃癌发生的潜在相关性。
1 材料与方法
1.1 研究对象 通过胃镜检查筛选赣南医学院第一附属医院2019年1月—12月疑似胃癌患者52例,最终病理确诊11 例。剔除近期使用抗生素患者5例,转移癌1 例,共收集5 例原发性胃癌患者胃黏膜组织和粪便样本。同时筛选5 例近3 个月内未使用过任何抗菌药物的健康人群作为对照组,收集其胃黏膜组织和粪便样本。共得到20 个样本。纳入标准:(1)初诊胃癌患者;(2)原发性胃癌患者;(3)能同时获得胃黏膜组织和粪便标本;(4)近3个月内未使用过任何抗菌药物和益生菌。排除标准:(1)年龄<18周岁的未成年人,>70周岁的老年人;(2)同时并发其他消化道疾病的患者;(3)既往有放化疗史和胃肠道手术史患者;(4)近2周有胃肠道感染疾病史患者。所有入选者本人或其家属知情同意并签署知情同意书,本研究经医院伦理委员会批准。
1.2 胃黏膜组织和粪便标本采集 使用标准胃镜钳获得1~2 mm 的胃黏膜活检样本置于无菌EP 管中;使用粪便收集管患者自我收集50~100 mg 粪便样本。样本立即转移到实验室,储存在-80 ℃冰箱保存,待进一步处理。
1.3 DNA 提取和PCR 扩增 采用CTAB 或SDS 方法对样品基因组进行提取,使用琼脂糖凝胶电泳检测提取出DNA 纯度和浓度;随后取适量DNA 于离心管中,并用无菌水稀释至1 ng·µL-1。以稀释后的基因组DNA 为模板,根据扩增区域选择使用包含barcode 的特异性引物,采用New England Biolabs 公司的Phusion®High-Fidelity PCR Master Mix with GC Buffer 和高效高保真酶进行PCR,以确保扩增效率和准确性。PCR 产物用2%浓度的琼脂糖凝胶电泳进行检测,并根据PCR 产物浓度进行等量混样,充分混匀后使用2%浓度的琼脂糖凝胶电泳再次进行检测,对目的条带使用Qiagen 公司提供的胶回收试剂 盒 进 行 回 收。使 用NEBNext®Ultra ™ IIDNA Library Prep Kit 建库试剂盒进行文库构建,将构建好的文库进行Qubit 和Q-PCR 定量;待文库合格后,使用NovaSeq6000进行测序。
1.4 肠道菌群的高通量测序 样本委托北京诺禾致源科技股份有限公司进行高通量测序,根据barcode 序列和PCR 扩增引物序列获得的数据拆分出各样本数据,截去barcode 和引物序列后使用FLASH 软件对样本的reads 进行拼接, 获得原始标签(Raw Tags)。随后使用fastp 软件对获得的Raw Tags 进行质控,得到高质量测序标签(Clean Tags)。最后使用Usearch 软件将Clean Tags 与数据库进行比对以检测嵌合体并进行去除,从而得到最终的有效数据(Effective Tags)。使用QIIME2 软件中的DADA2 模块或deblur 进行降噪,并过滤掉丰度<5的序列,从而获得最终的ASVs 以及特征表。随后,使用QIIME2软件中的classify-sklearn模块将得到的ASVs与数据库比对得到每个ASV的物种信息。
1.5 统计学方 法 使用QIIME2 软 件计算Observed_Otus、Shannon、Simpson、Chao1、Goods_Coverage、Dominance 和Pielou_E 指数,并绘制稀释曲线(Rarefaction Curve)和物种累积箱形图(Species Accumulation Boxplot)。同时对alpha 多样性的组间差异进行分析。使用QIIME2 软件计算Unifrac 距离,并使用R 软件绘制PCA、主坐标分析(PCoA)和NMDS 降维图。其中,PCA 和PCoA 调用R 软件中的ade4 和ggplot2 包。随 后,调 用QIIME2 软 件 中 的adonis 和anosim 函数分析组间群落结构差异显著性。最后,使用线性判别效应分析(LEfSe)或R软件完成组间显著差异性物种分析。其中,LEfSe 分析通过LEfSe 软件来完成,默认设置LDA score 阈值为4。MetaStat 分析则使用R 软件对两个比较组在门、纲、目、科、属和种6 个分类水平上进行差异性检验筛选出组间显著差异性物种;而t检验同样使用R软件来实现各个分类水平上的物种显著差异性分析。P<0.05为差异有统计学意义。
2 结果
2.1 两组胃黏膜和粪便标本细菌的OTU分析 根据聚类从20 个样本中总共获得3 069 个OTU,其中胃黏膜和粪便样本共享OTU 557 个,胃癌组黏膜(WANM)和对照组黏膜(ZCNM)样本共享OTU 1 049个,胃癌组粪便(WAFB)和对照组粪便(ZCFB)样本共享OTU 815 个(图1)。随着测序深度的增加,胃黏膜和粪便样本菌群稀释曲线趋于平坦(图2),表明实测数据合理,基本达到测序深度。
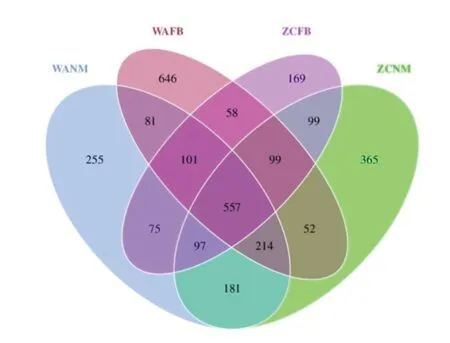
图1 胃癌组黏膜(WANM)、对照组黏膜(ZCNM)、胃癌组粪便(WAFB)、对照组粪便(ZCFB)菌群OTU分析韦恩图

图2 胃黏膜和粪便样本菌群稀释曲线
2.2 两组胃黏膜和粪便菌群ɑ多样性分析 ɑ多样性显示,胃癌组黏膜(WANM)、对照组黏膜(ZCNM)、胃 癌 组 粪 便(WAFB)和 对 照 组 粪 便(ZCFB)组间Dominance、Shannon、Chao1、Simpson、Pielou_e、Observed_otus指标比较差异均无统计学意义(P>0.05)(表1)。
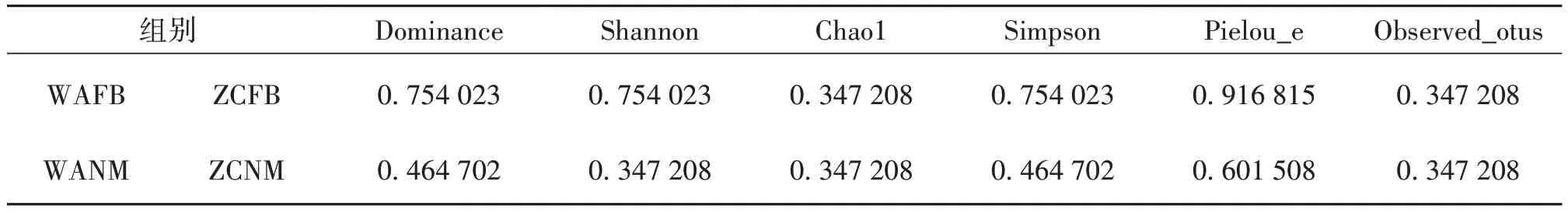
表1 两组胃黏膜和粪便菌群α 多样性指数差异分析(P-value)
2.3 两组胃黏膜和粪便菌群β 多样性分析 本研究基于非加权Unifrac 的PCoA 法分析胃癌组和对照组胃黏膜和粪便样本的微生物结构差异,结果显示,胃癌组胃黏膜和粪便菌群结构与对照组存在显著差异(P<0.05),胃癌组胃黏膜和粪便样本之间物种丰度和构成较为分散,而对照组之间的关系则更近(图3)。
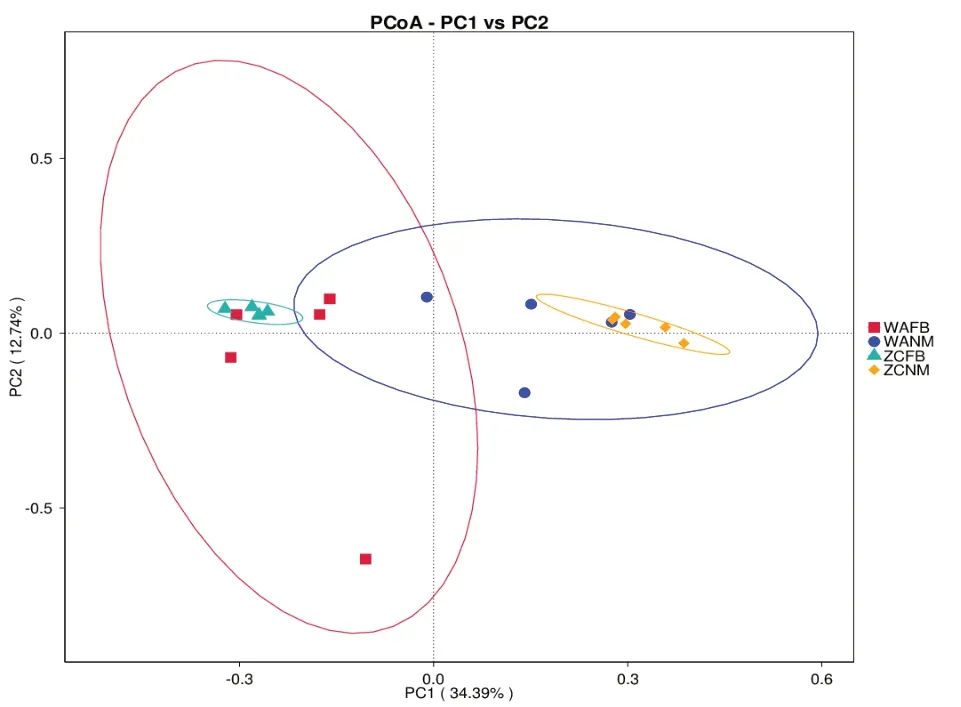
图3 两组胃黏膜和粪便菌群坐标分析(PCoA)分析图
2.4 两组群落结构差异显著性分析 在细菌分类学的门水平上,胃癌组黏膜(WANM)和对照组黏膜(ZCNM)菌群主要由6 个门组成,包括厚壁菌门(Firmicutes)、变形杆菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、弯曲杆菌门(Campylobacter)和古细菌(Archaea)。相比对照组黏 膜(ZCNM),胃 癌 组 黏 膜(WANM)Archaea、Bacteroidetes丰度降低,而Firmicutes、Campylobacter丰度增加。胃癌组粪便(WAFB)和对照组粪便(ZCFB)菌群主要由4 个门组成,包括Firmicutes、Proteobacteria、Actinobacteria和Bacteroidetes,其 中Firmicutes和Proteobacteria明显占优势,占总数的65%以上。相比对照组粪便(ZCFB),胃癌组粪便(WAFB)Firmicutes、Actinobacteria丰 度 降 低,而Proteobacteria、Bacteroidetes、Campylobacter丰度增加(图4)。
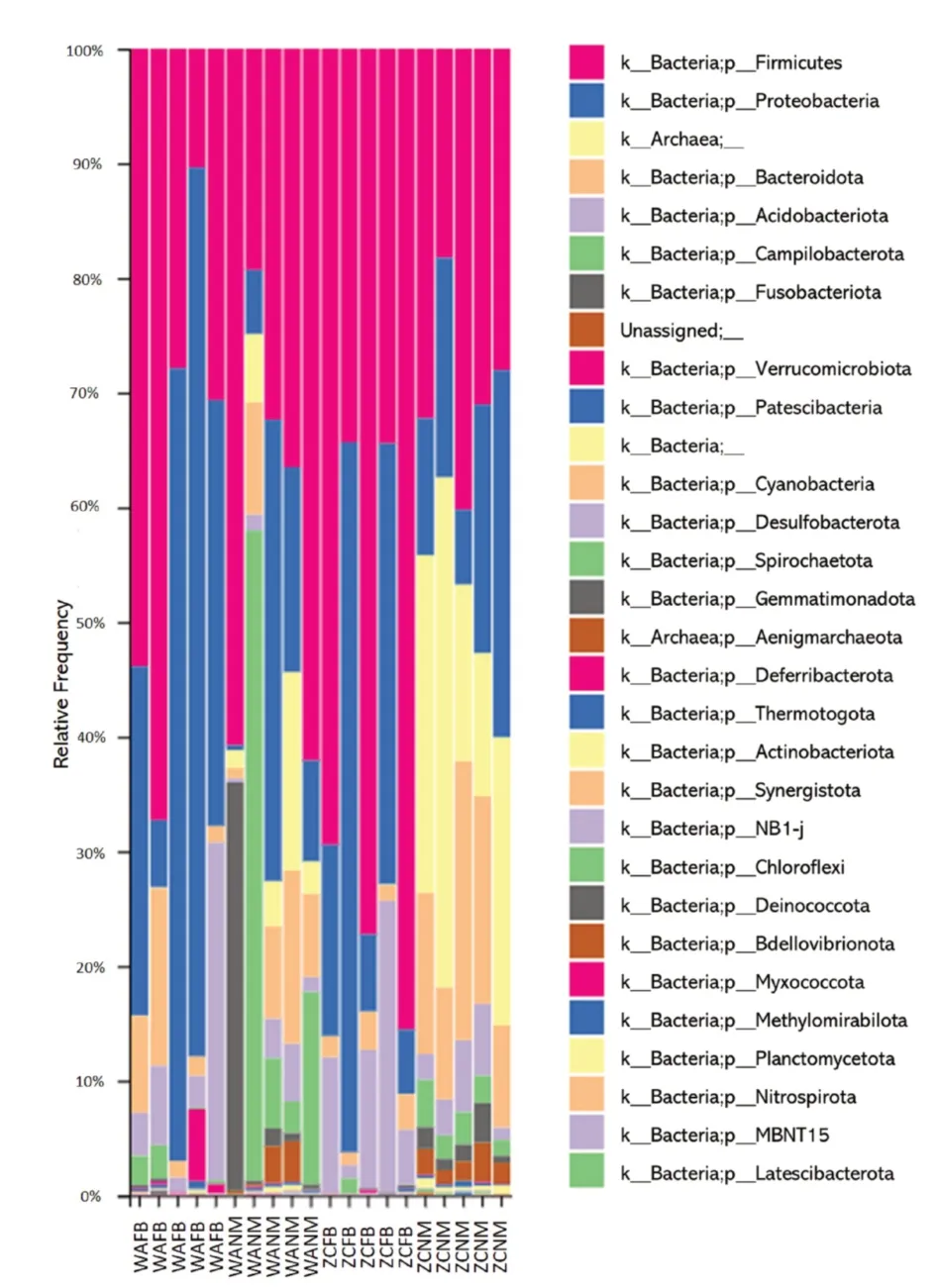
图4 两组细菌群落结构差异分析图
2.5 两组胃黏膜和粪便菌群LEfSe 差异分析 使用LEfSe差异分析表明,细菌系统发育型在4组样本之间存在显著差异(图5A)。LDA 值分布柱状图中展示了LDA 评分≥4的物种,即认为组间差异有统计学意义的生物标志物,柱状图的长度代表差异物种的影响大小。当LDA 评分的绝对值≥4 时,4 组间有39个物种差异显著。其中对照组黏膜(ZCNM)富集9 种,即Archaea、Bacteroidetes、普 雷 沃 菌 属(Prevotellaceae)、巴斯德菌属(Pasteurellales)、放线杆菌属(Actinobacillus)、肠炎弧菌(Parahaemolyticus)、伯克霍尔德菌属(Bukholderiales)、异普雷沃菌属(Alloprevotella)、奈瑟菌科(Neisseraceae);胃癌组黏膜(WANM)富集5 种,即Helicobacteriota、梭杆菌属(Fusobacteriia)、纤毛菌属(Leptotrichia)、赫尔曼埃希菌(Haemonadales)、韦荣球菌(Veronococcus)。对照组粪便(ZCFB)富集5 种,即梭菌属(Clostridia)、颤螺旋菌目(Oscillospirales)、毛螺菌目(Lachnospirales)、双歧杆菌目(Bifidobacteriales)、布劳特氏菌属(Blautia);胃癌组粪便(WAFB)富集2 种,即肠杆菌目(Enterobacterales)、大 肠 埃 希 菌- 志 贺 菌 属(Escherichia-Shigella)(图5B)。在属水平上,胃癌组大肠埃希菌-志贺菌属(Escherichia-Shigella)丰度高于对照组,而拟杆菌属(Bacteroides)等益生菌则低于对照组。
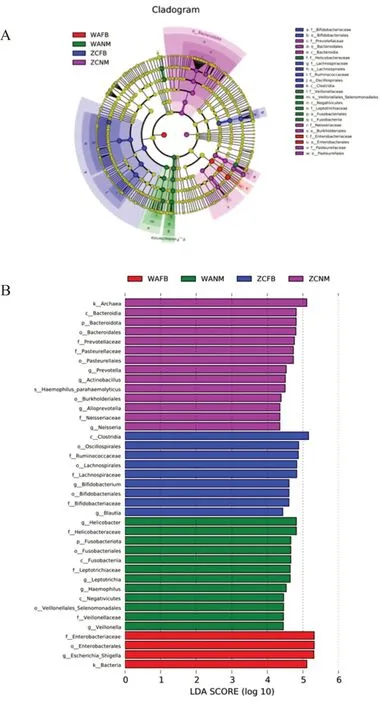
图5 4组菌群分布的富集偏差图
2.6 KEGG 功能预测 采用PICRUSt2 软件基于KEGG 数据库对胃肠道菌群进行功能预测,共发现35个代谢通路有显著差异(图6)。
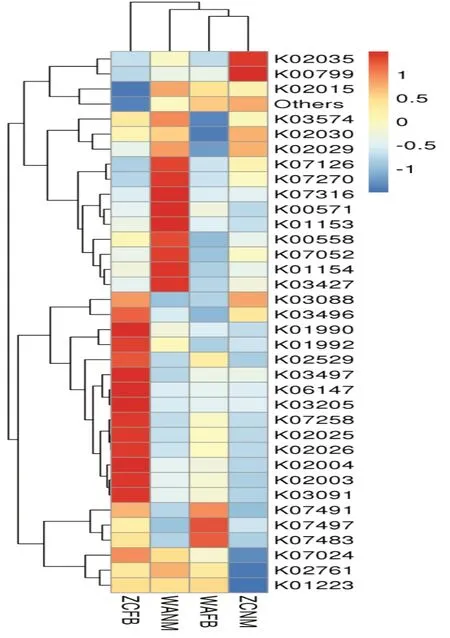
图6 PICRUSt2 基于细菌16S rRNA 基因序列的胃黏膜相关微生物组功能
3 讨论
胃癌是一种多因素疾病,肿瘤微环境的改变是胃癌发生、发展和转移的必要条件。作为肿瘤微环境的一部分,胃微生物群和肠道微生物群中的细菌可以通过产生促肿瘤代谢物、DNA 损伤、抑制抗肿瘤免疫和激活致癌信号通路来促进胃肿瘤的发展。大多数研究发现,与正常对照组相比,胃癌患者的微生物群多样性存在差异[7-8]。许多肠道和胃微生物被认为是结直肠癌和胃癌的致癌物[9-11],或是增加患者对癌症的免疫治疗反应的益生菌[12]。然而,关于疾病进展与胃和肠道微生物群多样性之间关系的研究结果不一。
幽门螺杆菌感染被认为是胃癌发生的主要危险因素,该菌具有多种与胃癌相关的毒力因子[13],如空泡毒素、细胞毒素相关基因A和外膜蛋白,亦可过度表达如IL-1β、IL-8、IL-17 和TNF-α 等促炎细胞因子,引起慢性胃炎,进而发展为胃萎缩、肠化生,最终进展为胃肠腺癌[14]。幽门螺杆菌感染和酸分泌的损害,改变了胃微生物群的组成,促进了胃内其他细菌的定植。有研究发现,胃癌患者乳酸菌属的比例升高[8],乳球菌和乳杆菌属可以产生乳酸,而乳酸可以作为肿瘤生长和血管生成的能量来源,从而促进肿瘤进展。
本研究发现,胃癌组和对照组之间胃黏膜和粪便样本优化序列数量差异无统计学意义,但胃癌组粪便(WAFB)OTU 数明显高于对照组,表明胃癌组肠道菌群物种丰富程度大于对照组,这与李慧珍等[15]报道一致。ɑ 多样性显示,两组人群胃黏膜和肠道菌群的Dominance、Shannon、Chao1、Simpson 指标比较差异均无统计学意义,这与YOUSSEF O等[16]、COSTEA P I 等[17]研究结果一致,即人体肠道微生物受饮食、疾病、治疗干预等因素影响,可能会出现相应菌群丰度及多样性的暂时性变化。但胃癌组粪便和胃癌组黏膜组内物种多样性中位数均高于对照组,这表明胃癌患者肠道菌群和胃黏膜菌群多样性较高。这与聂蓬等[18]、HU Y L 等[19]研究一致,但亦有不一致的研究结果[20-22],可能由于地域因素对胃肠道菌群的影响所致。β 多样性显示,胃癌组胃黏膜和粪便样本之间物种丰度和构成较为分散,而对照组之间的关系则更近。这表明胃癌中微生物群落的结构在系统发育上是多样化的,与研究报道一致[23-24]。
主坐标分析显示,胃癌组与对照组菌群结构存在差异。线性判别效应分析结果显示,在门水平上,胃黏膜菌群的丰度差异主要集中在厚壁菌门、变形杆菌门、拟杆菌门、放线菌门、弯曲杆菌门、古细菌,肠道菌群的丰度差异主要集中在厚壁菌门、变形杆菌门、放线菌门和拟杆菌门,与COSTEA P I等[17]研究结果一致。其中拟杆菌门和变形菌门为具有显著差异的标志性菌群,与LIANG W R 等[21]研究结果一致。在属水平上,胃癌组大肠埃希菌—志贺菌属丰度显著高于对照组,而拟杆菌属等益生菌则显著低于对照组,差异均有统计学意义(线性判别分析值=4,P<0.05),是属水平上的标志性物种。与李宁宁等[20]、黄鑫等[25]研究一致。提示志贺氏杆菌的富集可能预测GC 的发生。京都基因和基因组百科全书结果显示,胃癌患者与健康对照组间的菌群代谢存在差异。
然而,尽管大多数研究表明与健康人群相比,胃癌患者的微生物群发生了改变,但微生物群中不同细菌在特定微生境中的作用仍不清楚。在胃癌发生过程中,需要进一步研究确定胃癌细菌的微生物群和宿主间的相互作用。
