甘蓝DELLA基因家族鉴定及其mRNA在嫁接体中的运输分析
2023-03-03李必元赵彦婷岳智臣雷娟利胡齐赞陶鹏
李必元,赵彦婷,岳智臣,雷娟利,胡齐赞,陶鹏
研究报告
甘蓝DELLA基因家族鉴定及其mRNA在嫁接体中的运输分析
李必元,赵彦婷,岳智臣,雷娟利,胡齐赞,陶鹏
浙江省农业科学院蔬菜研究所,杭州 310021
DELLA基因家族参与植物激素信号转导通路的调控,其中()mRNA还是植物体内长距离运输的信号分子。在全基因组范围内鉴定甘蓝(var.) DELLA基因家族成员并分析mRNA运输特性,可为甘蓝DELLA基因家族的开发应用提供基础数据。本研究利用甘蓝基因组数据和转录组数据,在甘蓝中鉴定了5个DELLA基因家族成员(、、、和),但甘蓝基因组缺失了基因。采用劈接法将甘蓝(自交系G27)接穗和菜心(L. ssp.var.Tsen et Lee)(四九菜心)砧木嫁接在一起,构建异源嫁接体。对砧木菜心花序轴和对应位置的实生苗菜心花序轴(对照)取样进行转录组测序。在甘蓝/菜心异源嫁接体的砧木菜心花序轴的转录组测序文库中分别鉴定到8、9、3、5、1个来自甘蓝、、、和的外源read。甘蓝DELLA家族基因mRNA运输并没有提高砧木菜心中DELLA家族基因的转录表达水平。相关性分析显示,甘蓝DELLA家族基因mRNA运输效率与其自身的序列和接穗甘蓝中的DELLA家族基因的转录表达水平相关。本研究为深入探究甘蓝DELLA家族基因mRNA运输的分子机制奠定了基础。
甘蓝;菜心;嫁接;DELLA基因;mRNA运输
植物的生长发育受到多种内源、外源激素和外部环境的影响,而DELLA蛋白是多种激素信号和环境信号的主要整合者。因其蛋白N端含有高度保守的DELLA基序,而被命名为DELLA基因家族[1,2]。DELLA和VHYNP基序控制DELLA蛋白与GA-insensitive dwarf 1 (GID1)的结合能力以及DELLA蛋白的水解[3]。DELLA蛋白的C端有一个保守的GRAS功能结构域,具有调控DELLA蛋白活性的功能。DELLA蛋白可与ABA INSENSITIVE 3 (ABI3)、ABI5、AUXIN RESPONSE FACTOR 6、PHYTOCHROME INTERACTING FACTORs等转录因子相互作用,参与调控多种植物激素的信号通路[4,5]。多种植物的DELLA 基因家族成员已被鉴定,如模式植物拟南芥DELLA基因家族包含()、()、()、()和()共5个成员[6,7]。DELLA家族基因参与调控植物的生长发育和抗逆响应等;此外,通过基因工程手段改造DELLA家族基因,在多个经济作物中实现了植株矮化和产量提高[8]。
DELLA基因家族成员mRNA是长距离运输信号,可在植物体中进行长距离运输,并调控植物的生长发育[9]。嫁接是一种人工营养繁殖技术,可用于分析接穗和砧木之间的物质运输[10,11],为研究DELLA家族基因mRNA运输提供了技术支撑。通过构建嫁接体,发现茶海棠(Redh. var. pingyiensis)mRNA可在嫁接体中进行运输[12]。mRNA可以从伴细胞移动到筛管中,并且其翻译的蛋白产物保留在其运输位置[13]。研究还发现拟南芥()mRNA的编码序列和3′UTR的特定基序和序列介导了mRNA在嫁接体中的长距离运输[14]。结球甘蓝(下文均简称甘蓝)是十字花科芸薹属甘蓝种中的一个变种,在我国蔬菜出口贸易以及周年供应中占有极其重要的地位。当前,对甘蓝DELLA基因家族成员及其mRNA运输特性并不清楚,影响了甘蓝品种改良的应用。目前,菜心DELLA基因家族成员已被鉴定[15],为利用甘蓝/菜心异源嫁接体研究甘蓝DELLA家族基因mRNA运输提供了基础数据。本研究在甘蓝全基因组范围内鉴定了5个DELLA基因家族成员;通过构建甘蓝/菜心异源嫁接体,发现甘蓝DELLA家族5个基因的mRNA均可从接穗运输到砧木菜心中,并比较分析了它们的运输效率。本研究为后期开展甘蓝DELLA家族基因mRNA运输分子机制研究提供了基础数据。
1 材料和方法
1.1 嫁接体的构建和转录组测序
2016年8月下旬在浙江省农业科学院玻璃温室中(经度120.2°,纬度30.3°)播种四九菜心,一周后播种甘蓝G27,生长40 d后,采用劈接法将甘蓝G27的茎尖(营养生长阶段)分别嫁接到甘蓝G27的茎(营养生长阶段)和四九菜心花序轴(生殖生长阶段)上,构建甘蓝/甘蓝同源嫁接体和甘蓝/菜心异源嫁接体。另外,保留一部分菜心实生苗作为对照。嫁接生长30 d后,接穗甘蓝为莲座状,而砧木菜心和实生苗菜心均为抽薹开花状态。取甘蓝/菜心嫁接的砧木菜心花序轴和相同位置的菜心实生苗的花序轴进行转录组测序;在甘蓝/甘蓝同源嫁接体生长30 d后,在接穗上取长约2 cm的甘蓝茎尖进行转录组测序,为后续的DELLA基因家族成员mRNA运输效率与其在接穗中的转录表达量之间的相关性分析提供数据。上述样品均取样3次作为生物学重复。由北京百迈客公司完成RNA提取,通过Nanodrop检测RNA的纯度和Agilent 2100检测RNA的完整性。构建文库后,使用Qubit2.0进行初步定量,使用Agilent 2100对文库的插入片段大小进行检测。使用qPCR方法对文库的有效浓度进行准确定量(文库有效浓度> 2 nmol/L),完成库检。库检合格后,不同文库按照目标下机数据量进行建池,在Illumina HiSeq平台进行测序,转录组测序的read长为150 bp。甘蓝的转录组数据参考Braol JZS V 2.0基因组,菜心的数据参考Brara Chiifu V 1.5基因组分别进行序列比对,基因的转录表达水平基于FPKM算法获得。
1.2 甘蓝DELLA基因家族成员鉴定
使用拟南芥DELLA基因家族成员(、、、和)的序列[6,7],在甘蓝基因组数据库(Braol JZS V 2.0) (http://brassicadb.cn/#/)进行BLASTn检索(检索覆盖率>95%、序列一致率>75%),在甘蓝全基因组中鉴定DELLA基因家族成员。基于拟南芥中的同源基因,分别命名为、、、和。使用ProtParam tool (https://web.expasy.org/protparam/)分析甘蓝DELLA家族蛋白的氨基酸数量、分子量和等电点(pI)。参考甘蓝DELLA基因家族成员的序列,利用甘蓝/甘蓝嫁接体的接穗甘蓝茎尖的转录组测序文库中的reads,拼接获得甘蓝G27的DELLA基因家族成员的编码序列和3′UTR序列。参考白菜DELLA基因的序列,利用菜心实生苗花序轴的转录组测序文库中的reads,拼接获得四九菜心的DELLA基因家族成员的编码序列和3′UTR。
1.3 DELLA基因家族系统进化分析
使用拟南芥DELLA基因家族在Ensembl Plants(http://plants.ensembl.org/)检索可获得动物、真菌以及植物中DELLA基因家族成员的获得和丢失情况,了解DELLA的进化历程。下载十字花科植物DELLA基因的编码序列,使用MEGA7,采用最大似然法(maximum likelihood method,ML) Kimura 2-parameter 模型,Bootstrap值设置为1000,构建ML系统树。
1.4 mRNA运输分析
使用ClustalX比对甘蓝DELLA基因和菜心DELLA基因的序列,在甘蓝每个DELLA基因的编码区上筛选3段长为28 bp的特异序列作为检索序列(表1),使用UltraEdit文本编辑器在甘蓝/菜心异源嫁接体砧木花序轴的3个生物学重复的转录组测序文库(G1、G2和G3)中过滤获得外源的甘蓝DELLA基因的read。在菜心实生苗花序轴的3个生物学重复的转录组测序文库(C1、C2和C3)筛选外源read。对初步获得的甘蓝read进行验证,排除重复的read,对余下的read使用ClustalX进行序列比对。参考文献[16],在一条read上存在2个以上的差异碱基,可认定为运输的read。

表1 在菜心转录组测序文库中筛选外源甘蓝DELLA基因read的检索序列
1.5 DELLA家族基因mRNA运输效率相关性分析
拟南芥DELLA基因家族成员() mRNA可进行长距离运输,研究显示的运输效率与其部分编码序列和3′UTR序列密切相关[14]。使用甘蓝DELLA基因家族成员序列分别与拟南芥(998~1771)进行序列比对,计算它们与(998~1771)序列的一致率。设置甘蓝DELLA基因家族每个成员mRNA运输的read数量作为运输效率,并使用SPSS(V26.0)与上述的序列一致率进行双变量皮尔逊相关性分析。比较运输效率与甘蓝/甘蓝嫁接体的接穗甘蓝中DELLA家族基因转录表达量的双变量皮尔逊相关性。
2 结果与分析
2.1 甘蓝DELLA基因家族鉴定
通过BLAST分析,本研究在甘蓝全基因组范围内鉴定到5个DELLA基因家族成员,未鉴定到。拟南芥包含1个基因,甘蓝中存在2个基因,分别定位于甘蓝的亚基因组LF和MF2中。芸薹属植物的祖先在进化中发生过基因组三倍化,但甘蓝DELLA基因家族成员的数量并未增加。甘蓝DELLA家族蛋白的氨基酸数量在507~576之间,分子量介于55.8~62.8 kDa,等电点在4.73~5.52之间。甘蓝DELLA家族基因不存在内含子(表2)。
2.2 DELLA基因系统进化分析
为了解DELLA基因家族的起源与进化,本研究分析了DELLA基因家族的分布情况。结果显示,动物、真菌和藻类植物(红藻、绿藻和轮藻)中均不存在DELLA基因。DELLA基因广泛存在于地钱()、小立碗藓()、江南卷柏()以及开花植物中(图1)。
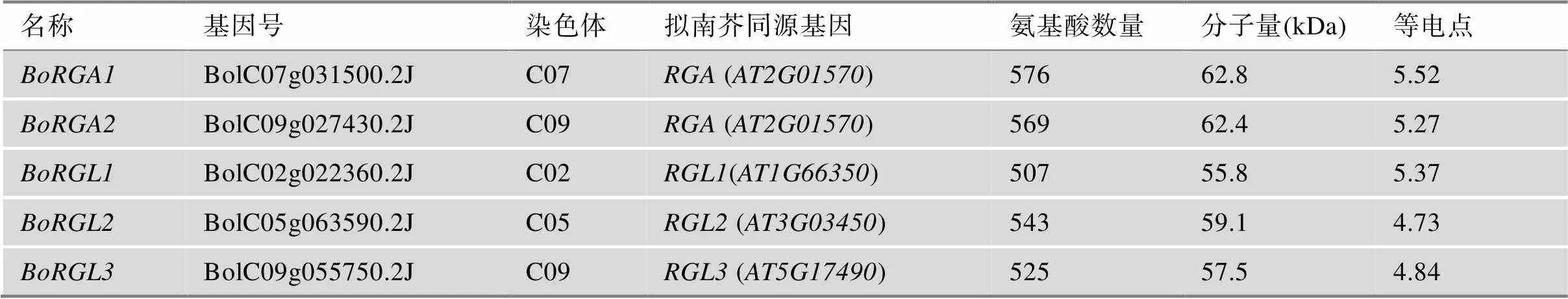
表2 甘蓝中DELLA家族基因对应的同源基因及其蛋白属性
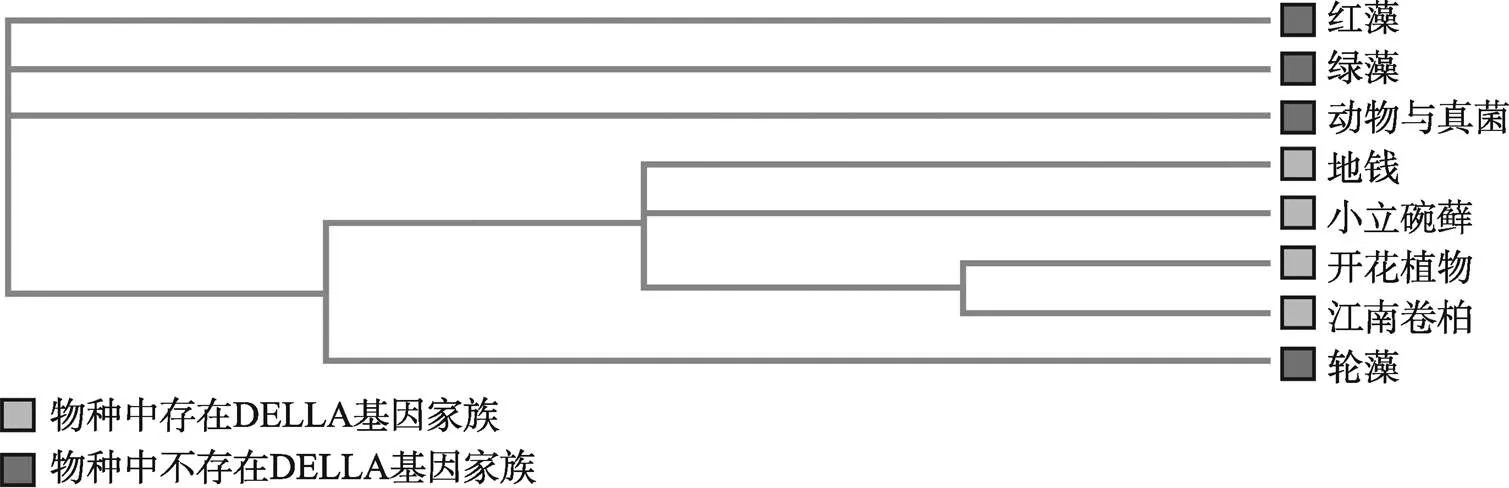
图1 DELLA基因在不同物种中的分布
为分析DELLA基因家族在十字花科植物中的系统发育关系,本文构建了ML系统进化树。结果显示,不仅在甘蓝中缺失基因,芸薹属其他物种(白菜、菜心、芥菜、黑芥以及甘蓝型油菜)均缺失基因。该结果暗示基因在芸薹属祖先中已经丢失,定位在LF亚基因组的和MF2亚基因组的是通过进化过程中的基因组三倍化形成的。亚麻荠()在进化过程中也发生过三倍化,其每种类型的DELLA基因家族成员的数量均是拟南芥中对应直系同源基因数量的3倍。十字花科植物中,基因和基因聚类在同一分支,亲缘关系较近。、和聚类在另一分支,但和之间的亲缘关系更近(图2)。
2.3 甘蓝和菜心DELLA基因在嫁接体中的转录表达分析
本研究构建了甘蓝/菜心异源嫁接体,并分别对甘蓝/菜心异源嫁接体的砧木花序轴以及菜心实生苗的花序轴进行了转录组测序。在甘蓝/菜心异源嫁接体中,和的表达量是、、表达量的6倍以上,的表达量是、表达量的8倍以上。、、、在菜心实生苗花序轴中的表达量与在甘蓝/菜心异源嫁接体中菜心花序轴中的表达量相比,差异不明显。相比于菜心实生苗花序轴中的表达,在甘蓝/菜心异源嫁接体的花序轴中的表达量更高(图3)。
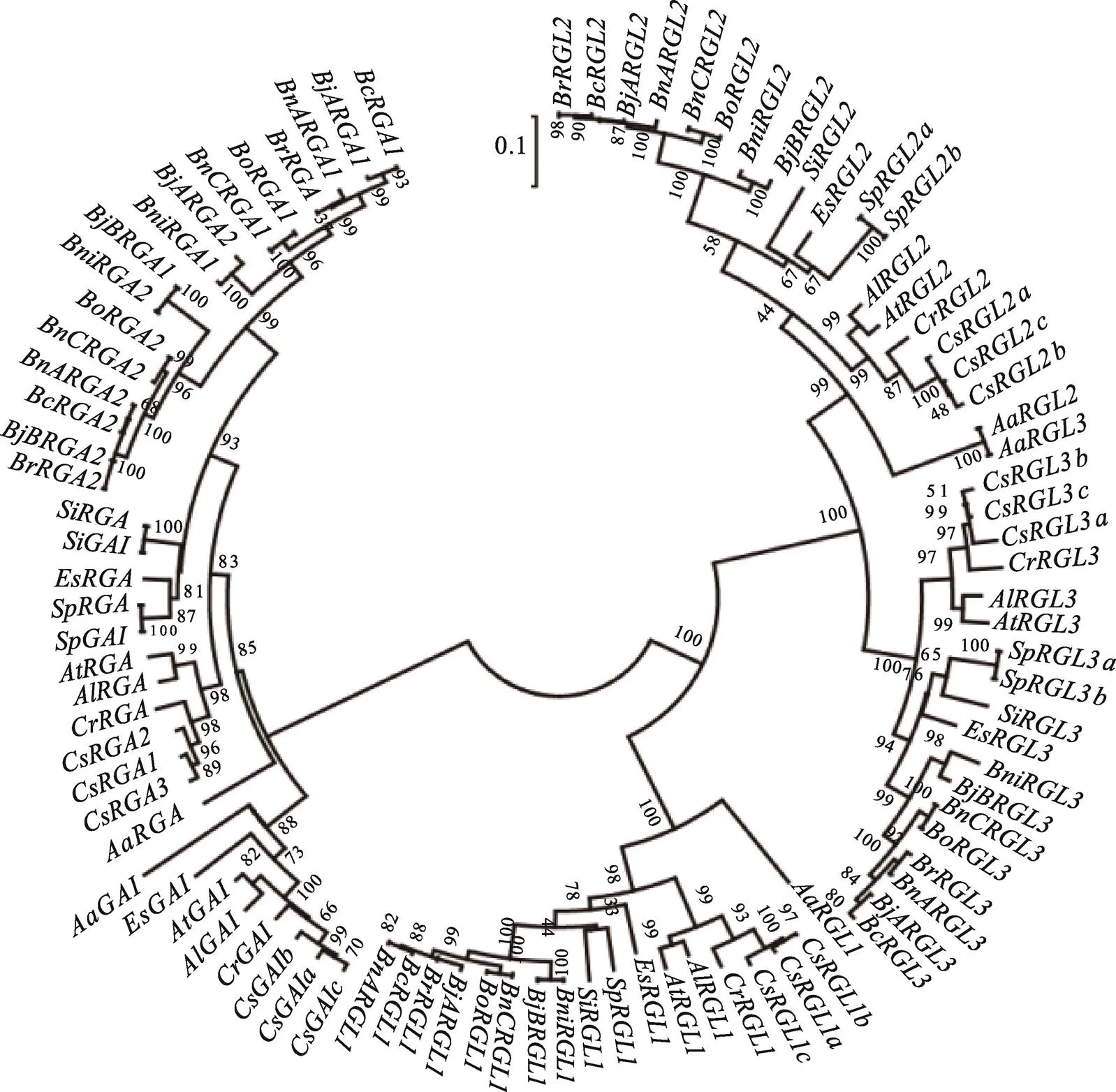
图2 十字花科植物DELLA基因系统发育树
Aa:阿拉伯岩芥;Al:琴叶拟南芥;At:拟南芥;Bc:菜心;Bni:黑芥;Bo:甘蓝;Br:白菜;BjA:芥菜A基因组;BjB:芥菜B基因组;BnA:甘蓝A基因组;BnC:甘蓝C基因组;Si:水蒜芥;Es:盐芥;Cs:亚麻荠;Cr:荠菜;Sp:条叶盐芥。
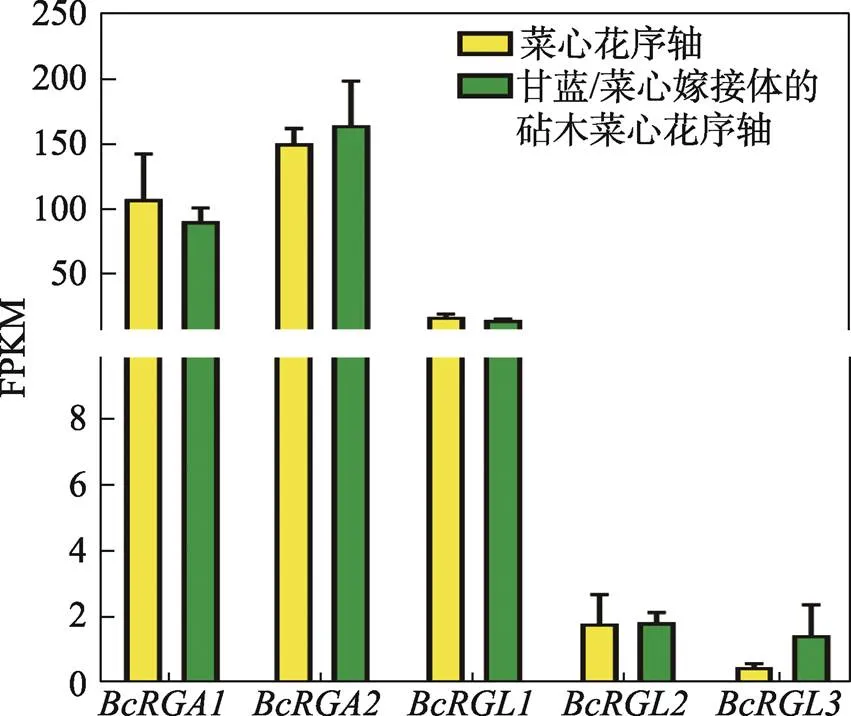
图3 DELLA基因在甘蓝/菜心异源嫁接体的菜心花序轴以及菜心实生苗对应位置的花序轴中的转录表达
2.4 甘蓝DELLA基因在异源嫁接体中的mRNA运输分析
甘蓝和菜心属于不同的物种,两者在基因序列上存在差异。通过构建甘蓝/菜心异源嫁接体,并对其进行转录组测序,可利用种间差异序列来鉴定长距离运输及外源的mRNA read (图4A)。通过比对甘蓝和菜心的DELLA基因序列,在甘蓝每个DELLA基因编码区内选择了3段特异性序列作为检索序列(表1)。使用上述的检索序列在菜心实生苗花序轴的转录组测序文库(C1、C2和C3)进行筛选,未获得外源的甘蓝DELLA基因的read,证明了上述检索序列的可靠性。利用上述验证后的检索序列在甘蓝/菜心异源嫁接体的菜心花序轴转录组文库中(G1、G2和G3)中进行检索,在3个检索位点(;)和3个菜心转录组测序文库中(G1、G2和G3;G1、G2和G3)鉴定到分别来自甘蓝和的8、9条read。在2个检索位点(和;和)和2个菜心转录组测序文库中(G1和G3;G2和G3)鉴定到分别来自和的3、5条外源read。在位点和G2文库中鉴定到来自的1条外源read(图4B)。上述的每条read长度为150 bp。每条read与相应的菜心和甘蓝的DELLA基因进行序列比对(图5),结果显示在砧木菜心花序轴中筛选的read在序列上与甘蓝同源,证明了接穗甘蓝中的DELLA基因的mRNA发生了运输。
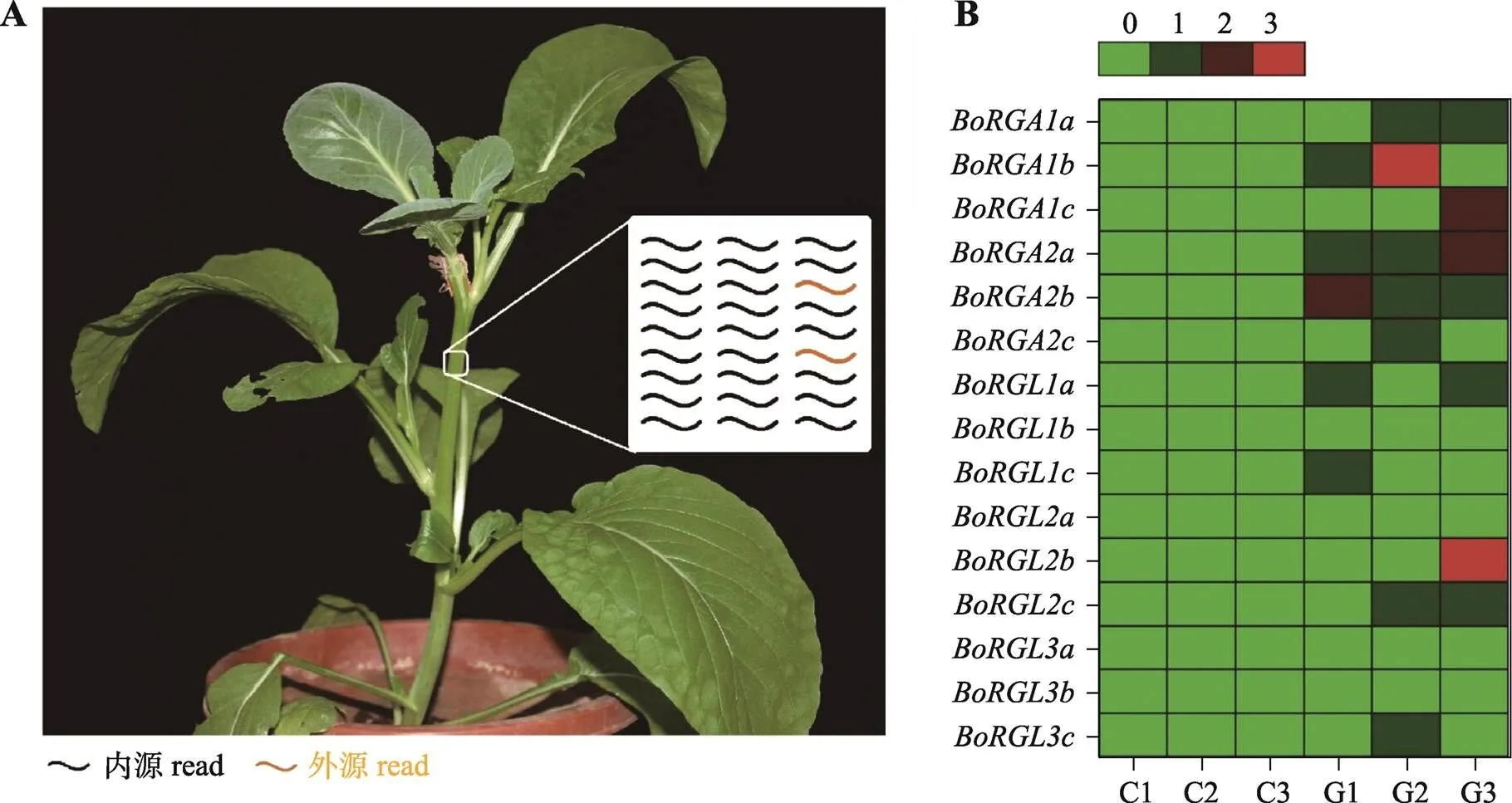
图4 利用异源嫁接体分析DELLA家族基因mRNA运输
A:甘蓝/菜心异源嫁接体及其转录组测序文库示意图;B:不同检索序列在菜心花序轴转录组测序文库(C1、C2和C3)和在甘蓝/菜心异源嫁接体的菜心花序轴的转录组文库(G1、G2和G3)中筛选结果。
2.5 甘蓝DELLA基因运输效率分析
为系统评价DELLA基因家族每个成员的mRNA运输效率,本研究分析了3个检索位点、3个文库的检测情况以及运输read的总数。结果显示,和在每个检测位点、每个文库中都能找到外源的和的read。而和只能在部分位点和部分文库中找到运输的read。基于检索位点的覆盖度、文库中重复性以及运输read的总数,显示和的mRNA运输效率最高。
研究显示拟南芥的mRNA运输效率与其部分编码区和3′UTR的序列密切相关。由于甘蓝DELLA基因家族5个成员的mRNA在3′UTR的长度上有差异,本研究选择(998~1771)作为参考序列,比较分析了甘蓝DELLA基因家族成员与(998~1771)的序列一致率,结果显示、与(998~1771)的序列一致率在81%以上。而、、与(998~1771)的序列一致率介于64.2%~65.1%之间(表3)。
为找出影响DELLA基因家族成员mRNA运输效率的相关因素,以运输read的数量作为其mRNA运输效率的指标,分别与序列的一致率、甘蓝/甘蓝嫁接体的接穗甘蓝茎尖中的表达量进行双变量相关性分析。结果显示,DELLA基因家族成员的mRNA运输效率与序列一致率(与(998~1771)比对)呈正显著相关(皮尔逊相关性系数=0.889,=0.04<0.05)。甘蓝DELLA基因的mRNA运输效率与接穗甘蓝中DELLA基因的转录表达量也呈正显著相关(皮尔逊相关性系数=0.928,=0.02<0.05)。

图5 运输的read与相应的甘蓝和菜心DELLA基因序列比对
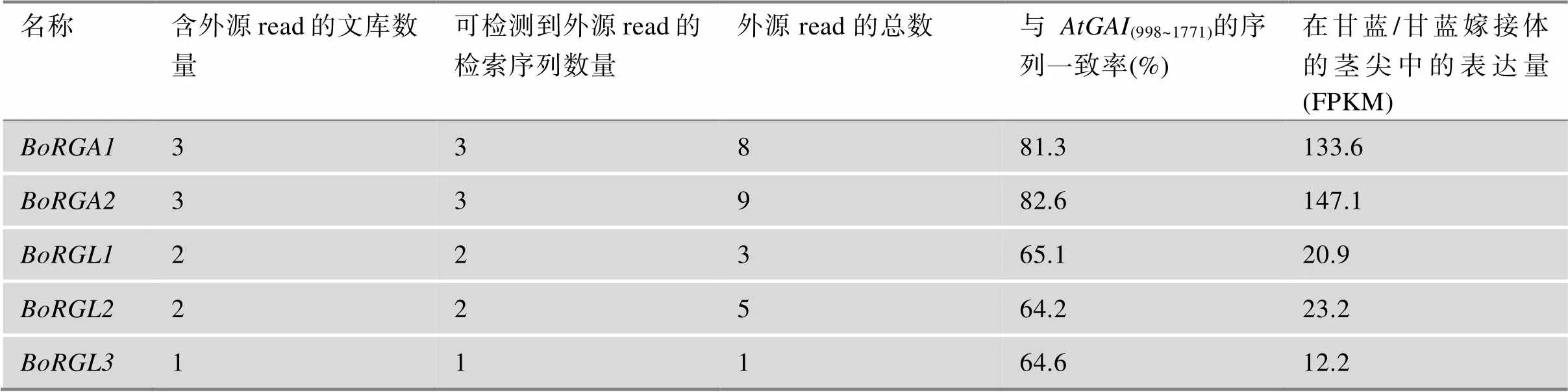
表3 甘蓝DELLA家族基因mRNA运输效率分析
3 讨论
GA是调控植物生长发育的一类激素,而DELLA家族基因是GA代谢通路中的关键基因。因此,在动物和真菌中均未检测到DELLA基因,表明DELLA基因家族是植物所特有的,但不是所有植物中均有DELLA基因(如红藻和绿藻)(图1)。轮藻具有类似根、茎、叶的分化,但也没有DELLA基因。在苔藓植物地钱、小立碗藓以及蕨类植物江南卷柏中开始出现DELLA基因,之后广泛分布于开花植物中(图1)[17]。研究表明,DELLA蛋白参与调控植物茎、叶、花的生长发育以及种子萌发与休眠[18]。DELLA基因家族的出现可能与植物形态特征的进化密切相关。
本研究通过构建异源嫁接体和转录组测序,在砧木菜心花序轴中鉴定到来自于甘蓝DELLA基因家族的read,并且甘蓝DELLA基因家族成员mRNA运输效率存在差异,其中和的mRNA运输效率明显高于、、(表3)。研究认为,基因的mRNA运输效率与其mRNA的表达量和序列结构均有关[14,16]。拟南芥基因mRNA可进行长距离运输,其运输效率与其部分编码序列和3′UTR序列有关[14]。甘蓝中不存在基因,但甘蓝DELLA基因家族成员与序列相似,且均发生了mRNA运输,暗示甘蓝DELLA家族基因序列可能与mRNA运输效率有关。相比于、和,和序列与的序列相似度更高(表3)。相关性分析显示甘蓝DELLA基因家族成员的mRNA运输效率与其序列一致率(和(998~1771)相比)相关,但仍需要更多研究才能确定两者之间的因果关系。研究发现,某些mRNA (如DMC1:tRNA)形成三叶草结构,并富集在韧皮部中,在接穗和砧木之间进行运输[19,20]。有研究认为基因的mRNA运输效率与其自身的转录表达量有关[16]。本研究结果显示接穗甘蓝DELLA基因和在甘蓝茎尖的表达量明显高于、和(图3),而DELLA基因的mRNA运输效率与之相对应。
在甘蓝/菜心异源嫁接体中,尽管接穗甘蓝向砧木菜心花序轴运输了DELLA基因mRNA,但并没有提高砧木菜心DELLA基因的转录表达量(图3)。推测与甘蓝DELLA基因mRNA运输到砧木菜心的数量太少有关。另一方面,菜心砧木中的DELLA基因的mRNA也有可能向上运输到了接穗甘蓝中,从而形成一种动态平衡。本研究鉴定了甘蓝DELLA基因家族成员,并分析它们的mRNA运输特征,为理解甘蓝DELLA基因家族的生物学功能和甘蓝的遗传改良提供了参考依据。
[1] Fleet CM, Sun TP. A DELLAcate balance: the role of gibberellin in plant morphogenesis., 2005, 8(1): 77–85.
[2] Sun TP. Gibberellin-GID1-DELLA: a pivotal regulatory module for plant growth and development., 2010, 154(2): 567–570.
[3] Murase K, Hirano Y, Sun TP, Hakoshima T. Gibberellin- induced DELLA recognition by the gibberellin receptor GID1., 2008, 456(7221): 459–463.
[4] Wang PF, Zhang QQ, Chen YC, Zhao YX, Ren FS, Shi HM, Wu XY. Comprehensive identification and analysis of DELLA genes throughout the plant kingdom.,2020, 20(1): 372.
[5] Zou LP, Pan C, Wang MX, Cui L, Han BY. Progress on the mechanism of hormones regulating plant flower formation., 2020, 42(8): 739–751.
邹礼平, 潘铖, 王梦馨, 崔林, 韩宝瑜. 激素调控植物成花机理研究进展. 遗传, 2020, 42(8): 739–751.
[6] Peng JR, Carol P, Richards DE, King KE, Cowling RJ, Murphy GP, Harberd NP. Thegene defines a signaling pathway that negatively regulates gibberellin responses., 1997, 11(23): 3194– 3205.
[7] Sánchez-Fernández R, Ardiles-Díaz W, Van Montagu M, Inzé D, May MJ. Cloning of a novel-like gene, a putative member of the VHIID-domain transcription factor family., 1998, 49(326): 1609–1610.
[8] Liu Q, Wu K, Harberd NP, Fu XD. Green revolution DELLAs: from translational reinitiation to future sustainable agriculture., 2021, 14(4): 547–549.
[9] Haywood V, Yu TS, Huang NC, Lucas WJ. Phloem long- distance trafficking ofRNA regulates leaf development., 2005, 42(1): 49–68.
[10] Thomas HR, Frank MH. Connecting the pieces: uncovering the molecular basis for long-distance communication through plant grafting., 2019, 223(2): 582–589.
[11] Yu NN, Cao LW, Yuan L, Zhi X, Chen YQ, Gan SS, Chen LP. Maintenance of grafting-induced epigenetic variations in the asexual progeny ofandchimera., 2018, 96(1): 22–38.
[12] Wang SS, Liu ZZ, Sun C, Shi QH, Yao YX, You CX, Hao YJ. Functional characterization of the applegene through ectopic expression and grafting experiments in tomatoes., 2012, 169(3): 303–310.
[13] Xu HY, Iwashiro R, Li TZ, Harada T. Long-distance transport ofmRNA in., 2013, 13: 165.
[14] Huang NC, Yu TS. The sequences ofRNA constitute the motifs that are necessary and sufficient for RNA long-distance trafficking., 2009, 59(6): 921–929.
[15] Guan HL, Huang XM, Zhu YN, Xie BX, Liu HC, Song SW, Hao YW, Chen RY. Identification ofgenes and key stage for GA sensitivity in bolting and flowering of flowering Chinese Cabbage., 2021, 22(22): 12092.
[16] Yang YZ, Mao LY, Jittayasothorn Y, Kang Y, Jiao C, Fei ZJ, Zhong GY. Messenger RNA exchange between scions and rootstocks in grafted grapevines., 2015, 15(1): 1–14.
[17] Hernández-García J, Briones-Moreno A, Blázquez MA. Origin and evolution of gibberellin signaling and metabolism in plants., 2021, 109: 46–54.
[18] Zhao CL, Wang X, Chen JL, Chen H, Wang L, Lai ZX, Liu SC. Progress in research on plant DELLA family proteins., 2020, 26(5): 1299–1308.
赵春丽, 王晓, 陈家兰, 陈何, 王乐, 赖钟雄, 刘生财. 植物DELLA蛋白家族研究进展. 应用与环境生物学报, 2020, 26(5): 1299–1308.
[19] Zhang WN, Thieme CJ, Kollwig G, Apelt F, Yang L, Winter N, Andresen N, Walther D, Kragler F. tRNA-related sequences trigger systemic mRNA transport in plants., 2016, 28(6): 1237–1249.
[20] Wang T, Li XJ, Zhang XJ, Wang Q, Liu WQ, Lu XH, Gao SL, Liu ZX, Liu MS, Gao LH, Zhang WN. RNA motifs and modification involve in RNA long-distance transport in plants., 2021, 9: 651278.
Identification of DELLA gene family in head cabbage and analysis of mRNA transport in the heterograft
Biyuan Li, Yanting Zhao, Zhichen Yue, Juanli Lei, Qizan Hu, Peng Tao
DELLA gene family is involved in the regulation of signal transduction of plant hormones. mRNAs of(), the member of DELLA gene family, are also signaling molecules of long-distance transport in plants. Genome-wide identification and mRNA transport analysis of the members of DELLA gene family in head cabbage (var.) can provide basic data for their application in head cabbage. In this study, five members of DELLA gene family (,,,, and) were identified in head cabbage using genome and transcriptome data. However, head cabbage lacked agene in its genome. The scion (head cabbage, inbred line G27) and the rootstock Chinese flowering cabbage (L. ssp.var.Tsen et Lee) (sijiucaixin) were cleft-grafted together to produce the heterograft. Inflorescence stem of the rootstock and the corresponding inflorescence stem in Chinese flowering cabbage seedlings (as controls) were purified and analyzed with transcriptome sequencing. The total of 8, 9, 3, 5, and 1 exogenous read(s), derived respectively from,,,, and, were identified in the transcriptomes of the rootstocks. Nevertheless, mRNA transport of DELLA family genes from scion to rootstock did not increase the transcriptional level of the members of DELLA gene family in the rootstocks. Correlation analysis suggested that mRNA transport efficiency of the DELLA family genes was correlated with the sequence and the transcriptional level of the respective DELLA gene in the scion (head cabbage). This study lays the foundation for further investigation on the molecular mechanism of mRNA transport of the members of DELLA gene family in head cabbage.
head cabbage; Chinese flowering cabbage; grafting; DELLA genes; mRNA transport
2022-10-18;
2022-12-31;
2023-02-06
浙江省自然科学基金项目(编号:LY21C150006),浙江省农业新品种选育重大科技专项(编号:2021C02065-4-4)和国家自然科学基金项目(编号:31601746)资助[Supported by the Natural Science Foundation of Zhejiang Province (No.LY21C150006), the Grand Science and Technology Special Project of Zhejiang Province (No.2021C02065-4-4), and the National Natural Science Foundation of China (No.31601746)]
李必元,学士,副研究员,研究方向:十字花科蔬菜作物遗传育种。E-mail: 16061944@qq.com
陶鹏,博士,副研究员,研究方向:十字花科蔬菜作物分子遗传学。E-mail: taopeng-84@163.com
10.16288/j.yczz.22-330
(责任编委: 许勇)
