应用RNA重测序分析低硫条件下大豆基因表达谱
2023-01-12吴志医张玉娥喻德跃
王 慧 吴志医 张玉娥 喻德跃
应用RNA重测序分析低硫条件下大豆基因表达谱
王 慧 吴志医 张玉娥 喻德跃*
南京农业大学/ 国家大豆改良中心/ 作物遗传与种质创新国家重点实验室/ 农业农村部大豆生物学与遗传育种重点实验室, 江苏南京 210095
大豆是重要的粮油兼用作物, 其硫利用的研究不足。本研究评价了云梦六月花叶和沁阳大豆对低硫的耐性, 以这2个品种为材料, 利用RNA重测序技术分析了对照(+S)和缺硫(–S)水平下根和叶中的表达谱。结果表明, 云梦六月花叶对低硫表现为耐性, 沁阳大豆对低硫表现为敏感。表达谱分析在云梦六月花叶和沁阳大豆的叶中分别鉴定到9064个和9795个低硫响应的差异表达基因, 根中分别鉴定到3185个和5006个差异表达基因。KEGG富集分析发现, 2个材料叶中有9个共有途径, 仅植物MAPK信号途径富集更多的上调表达基因。2个材料根中有18个共有途径, 其中9个途径在2个材料的中对低硫的响应一致, 4个途径包含更多的上调表达基因, 5个途径包含更多的下调表达基因。在其余9个途径中, 云梦六月花叶包含更多的上调表达基因。大豆硫酸根转运蛋白基因对硫酸根的吸收和转运非常重要, 在表达谱中鉴定到27个硫酸根转运蛋白基因, 分属4个亚组, 亚组1、2、4的基因多受低硫诱导, 亚组3的基因对低硫的响应较为复杂。基于富集分析结果, 本研究从植物MAPK信号途径中克隆了一个受低硫诱导的基因, 通过转化大豆毛状根证明该基因参与大豆硫利用的调控。本研究结果为深入探索大豆硫利用效率的遗传机理奠定了基础, 为大豆耐低硫育种提供了候选基因。
大豆; 低硫; RNA重测序; 硫酸根转运蛋白; EIN3/EIL
大豆是重要的粮油兼用作物, 为人类和其他动物提供优质的蛋白质和脂肪。硫是大豆生长和发育必须的营养元素, 是氨基酸、叶绿素、硫苷脂、维生素、辅酶等有机物的组成成分, 在多个生理过程中具有重要作用。和其他植物一样, 大豆中的硫元素主要来自于土壤中的硫酸盐, 与氮、磷、钾相比, 大豆硫利用的遗传研究较少。
在大豆上, 硫利用的遗传研究集中在发掘和鉴定硫酸根吸收、转运和还原的蛋白编码基因上。植物从土壤中吸收硫酸根依赖于硫酸根转运蛋白[1], 大豆基因组中有28个硫酸根转运蛋白编码基因, 其中基因强烈响应低硫诱导, 在烟草中过量表达增加植株的生物量、千粒重、半胱氨酸含量、叶片中的可溶性总蛋白含量等[2]。进入细胞的硫酸根不能被植物直接利用, 先活化为5'-磷酸腺苷硫酸(APS), 这个过程由三磷酸腺苷硫酸化酶(ATP sulfurylase, ATPS)催化[1]。大豆基因组中有4个ATPS编码基因,基因与拟南芥、马铃薯的基因高度同源, 在根中表达量丰富, 在种子中随着种子的发育过程表达量和酶活性降低[3]。随后APS又在腺苷酰硫酸还原酶(adenosine 5’-phosphosulfate reductase, APSR)催化下产生亚硫酸盐[1], 大豆APSR编码基因有3个, 其中基因在大豆的根、叶、花、种子和根瘤中都有表达, 在种子发育早期基因表达量和酶活性都较高, 同样随着种子发育进程逐渐降低, 在根中该基因的表达量受硫、磷饥饿的诱导[4]。亚硫酸盐再在铁盐还原蛋白和亚硫酸盐还原酶(sulfite reductase, SIR)的作用下进一步还原成二阶亚硫酸盐, 在丝氨酸乙酰转移酶(serine acetyltransferase, SATase/SERAT)和O-乙酰胆碱(硫醇)裂解酶(O-acetylserine sulfhydrylase, OASS/ OAS-TL)作用下, 硫酸盐整合进碳骨架生成第1个含硫有机物Cys[1]。大豆的SATase/SERAT和OASS/ OAS-TL编码基因较多, 分别有8个和6个[5], 其中能拯救大肠杆菌基因突变体的表型, 在大豆种子发育过程中低水平表达, 叶片和种子中酶活性较低[6]。6个不同的OASS基因在不同时期种子中高表达[7],能弥补大肠杆菌半胱氨酸营养突变体的功能[8], 在大豆中过表达基因增加含Cys蛋白质的含量, 但造成植株矮化, 产量降低[5]。可见, 大豆硫利用的遗传研究基础是比较薄弱的, 单个基因的功能是有限的, 所以有必要开展全基因组水平上的遗传研究, 探索大豆硫利用的调控网络, 发掘和鉴定更多的硫利用关键基因。
在前期研究的基础上, 本研究以2个大豆地方品种为材料, 通过室内水培试验评价了低硫对苗期大豆的影响, 利用RNA重测序(RNA-seq)分析了2个材料根和叶中的低硫响应的基因表达谱, KEGG富集分析探索低硫调控的代谢途径, 分析了不同的硫酸根转运蛋白基因低硫响应的表达规律, 对植物MAPK信号途径中基因进行了功能的初步研究。本研究结果为深入探索大豆硫利用效率的遗传机理奠定了基础, 也为大豆耐低硫育种提供了候选基因。
1 材料与方法
1.1 材料种植、表型测定和取样
利用室内水培试验评价云梦六月花叶和沁阳大豆对低硫的耐性。该试验在人工气候室内进行, 光照时间为16 h, 温度为26~28℃。设置缺硫和对照2个处理, 5个生物学重复, 以1/2浓度的霍格兰(Hoagland)完全营养液为对照(约1 mmol L–1SO42–), 以无硫酸根的霍格兰营养液为缺硫处理, 即用MgCl2、ZnCl2、CuCl2分别代替1/2浓度霍格兰完全营养液中的MgSO4、ZnSO4·7H2O、CuSO4·5H2O, 2种营养液的具体配方参见陈燕宁等[9]。经过精心挑选的大豆种子播种在蛭石中育苗, 4 d后移苗至水培箱中培养4 d, 此时第1对真叶完全展开, 去掉子叶进行低硫诱导处理, 营养液每2 d更换1次, 处理10 d后测量表型。用叶绿素仪(Soil and Plant Analyzer Development 502)测量倒一位完全展开叶的叶绿素含量(SPAD值), 测量子叶节到茎顶点的距离记为株高, 测量子叶节到根尖的距离记为根长。从子叶节把植株截断分为地上部和地下部(根), 分别称量地上部和地下部(根)的鲜重(g), 然后装入牛皮纸袋中, 放入预热好的烘箱内105℃杀青30 min, 75℃烘干至恒重, 称量得到地上部干重(g)和地下部(根)干重(g)。
采用同样方法处理云梦六月花叶和沁阳大豆10 d, 处理结束后根和叶分别取样, 3个生物学重复, 共24个样品。样品置于液氮中速冻, 用于后续RNA重测序、总RNA提取和cDNA反转录。
1.2 总RNA提取、cDNA反转录和qRT-PCR
根据美国英杰生命技术有限公司的TRIzol RNA分离试剂盒说明书提取大豆根和叶的总RNA。利用南京诺唯赞生物科技股份有限公司的两步法RT-PCR试剂盒(HiScript II Q RT SuperMix for qPCR (+gDNA eraser))反转录cDNA。(GenBank登录号为AY907703)作为内参基因。参照ChamQ SYBR qPCR Master Mix (Low ROX Premixed) (诺唯赞)试剂盒说明书, 配制qRT-PCR反应体系, 反应过程在ABI 7500 实时定量PCR系统(Applied Biosystems, 美国)中进行。用2–ΔΔCT方法计算基因的相对表达量[10]。采用T测验进行基因表达量的显著性分析。利用Primer- Premier 5软件(Premier Biosoft Interpairs,美国)设计所有引物, 由北京擎科生物科技有限公司合成, 引物序列见表1。

表1 引物序列
1.3 RNA重测序和数据分析
由北京博奥晶典生物技术有限公司完成样本RNA的重测序和组装, 以大豆Williams 82 (Wm82.a2.v1)基因组序列为参考。StringTie软件用于基因表达水平的定量和标准化, 标准化方法采用FPKM法(fragments per kilobases per million mapped reads)[11]。基因FPKM值为0时认为该基因没有被检测到。DESeq2软件包用于分析差异表达基因, 以不同硫水平下基因表达量改变倍数的超过2或小于0.5 (对数(log2)转换值超过1或小于–1), 且值小于0.05的基因被认定为差异表达基因(differentially expressed gene, DEG)。
利用NCBI数据库(http://www.ncbi.nlm.nih. gov/)、GO数据库(http://www.geneontology.org/)和KEGG数据库(https://www.kegg.jp/)对基因功能进行注释。利用在线软件KOBAS 3.0 (http://kobas.cbi. pku.edu.cn/index.php)对差异表达基因进行KEGG富集分析[12]。KEGG富集分析的参考基因组为大豆, 以矫正值(FDR值)小于0.05为显著富集。
MEGA 6.0 用于构建基因家族的聚类树[13], 在线软件Morpheus (https://software.broadinstitute.org/ morpheus/)绘制基因表达的热图, R软件的ggplot2数据包绘制KEGG途径热图。
1.4 GmEIL1基因的克隆与组织表达分析
以的mRNA序列为模板, 设计特异引物(表1), 利用PCR反应从大豆根的cDNA中克隆基因。取自然条件下种植的云梦六月花叶的根、茎、叶、花、荚和种子, 液氮速冻, 提取总RNA, 反转录cDNA, 利用qRT-PCR分析基因在不同组织中的表达水平。总RNA提取、cDNA反转录和qRT-PCR分析同1.2。
1.5 GmEIL1基因转化毛状根创制与硫酸根含量测定
根据南京诺唯赞生物科技股份有限公司的ClonExpress一步法克隆试剂盒说明书把全长cDNA连入表达载体pMDC83, 构建基因过表达重组载体OE-GmEIL1。设计引物克隆编码区620 bp片段, 利用Gateway技术将该片段连入RNA干扰载体pB7GWIWG2 (II), 构建基因RNA干扰重组载体Ri-GmEIL1。构建过表达重组载体和RNA干扰重组载体的引物见表1。空载pMDC83 (OE-EV)和pB7GWIWG2 (II) (Ri-EV)分别作为过表达和RNA干扰的对照。4个载体通过冻融法分别转入发根农杆菌K599菌株[14], 根据Kereszt等[15]描述的方法侵染大豆Jack子叶节诱发毛状根。当毛状根长到5~10 cm时, 从靠近毛状根诱发处下端剪掉幼苗的根, 得到大豆嵌合体。
将嵌合体转入完全营养液中适应培养3 d, 再分别转入低硫和对照营养液中处理, 营养液配方、处理方法、生长环境同1.1。处理结束后取样, 从子叶节把大豆嵌合体的地上部和根分开, 少量的毛状根用于总RNA的提取、cDNA反转录(同1.2)及qRT- PCR分析(同1.3), 验证阳性毛状根。其余样品烘干研磨成粉, 称取0.1 g转移到消煮管中, 加入5 mL HNO3, 在微波消解系统(Milestone Ethos)中150℃消煮30 min, 取出后冷却到50℃以下开盖, 用超纯水定容至50 mL, 然后用等离子体发射光谱仪(Perkin Elmer Optima 2100DV ICP-OES system)测定样品中的硫酸根含量(mg g–1)。
2 结果与分析
2.1 在不同硫水平条件下评价大豆对低硫的耐性
利用室内水培试验鉴定云梦六月花叶和沁阳大豆2个大豆地方品种对低硫的表现发现, 云梦六月花叶对低硫处理表现为耐性, 沁阳大豆对低硫处理表现为敏感(图1-A)。在低硫条件下, 2个材料的叶绿素含量和地上部鲜重都比对照条件下显著降低。在其他性状上2个材料对低硫的反应不同, 与对照相比, 沁阳大豆根长显著增加, 根鲜重、地上部鲜重、根干重显著降低, 而云梦六月花叶在这些性状上与对照相比没有显著差异(图1-B)。

图1 云梦六月花叶和沁阳大豆的低硫表型鉴定
+S和-S分别表示对照和缺硫处理。YM: 云梦六月花叶; QY: 沁阳大豆; *、**、***分别表示在0.05、0.01、0.001水平显著差异。
+S and-S represent the treatments of control and low sulfate, respectively. YM: Yunmengliuyuehuaye; QY: Qinyangdadou. *, **, and *** mean significant difference at the 0.05, 0.01, and 0.001 probability levels, respectively.
2.2 大豆响应低硫的差异表达基因
采集低硫和对照处理的云梦六月花叶和沁阳大豆的叶和根, 3个生物学重复, 构建24个转录组重测序文库, 进行转录组分析。测序数据经过质控过滤, 每个文库包含约3.2亿个高质量片段(clean read), 测序片段在参考基因组上的平均图谱定位率超过90%, 每个文库鉴定的基因数超过40,000个, 除个别样本外, 同一样本的3个生物学重复间基因表达量的相关性超过90%。用qRT-PCR方法对其中21个差异表达基因的表达量进行分析, 结果与表达谱数据高度一致(附图1)。这些结果说明该转录组数据质量较高。
转录组分析发现, 2个材料叶中比根中鉴定到更多的差异表达基因。在云梦六月花叶和沁阳大豆的叶中分别鉴定到9064个和9795个差异表达基因, 根中分别鉴定到3185个和5006个差异表达基因(图2)。尽管2个材料叶中差异表达基因数目接近, 但在上调和下调基因的数目上差别较大, 低硫敏感材料沁阳大豆叶中上调和下调基因的数目接近, 而低硫耐性材料云梦六月花叶的叶中上调表达的基因数目(3097个)远低于下调表达的基因数(5968个), 也低于沁阳大豆叶中上调表达的基因数目(5175个)。2个材料根中上调基因的数目都超过了下调基因的数目, 但与沁阳大豆相比, 云梦六月花叶的根中上调和下调基因的数目都较少。

图2 云梦六月花叶和沁阳大豆的叶和根中的差异表达基因数
L和R分别表示叶和根。YM: 云梦六月花叶; QY: 沁阳大豆。
L and R represent leaves and roots, respectively. YM: Yunmengliuyuehuaye; QY: Qinyangdadou.
2.3 低硫重塑大豆的代谢网络
KEGG富集分析显示, 在云梦六月花叶的叶和根中分别富集到22个和23个途径(图3-A, B), 在沁阳大豆的叶和根中分别富集到18个和26个途径(图3-C, D), 2个材料叶中共同富集了9个途径(图3-A, C), 根中共同富集了18个途径(图3-B, D)。
在9个叶共同富集途径中, 3个途径与光合相关, 分别是光合作用途径(photosynthesis)、光合作用-外周捕光(天线)色素蛋白途径(photosynthesis - antenna proteins)、卟啉-叶绿素代谢途径(porphyrin and chlorophyll metabolism) (图3-A, C)。分析各途径富集基因对低硫的响应发现, 几乎所有富集在核糖体途径、光合作用途径、光合作用-外周捕光(天线)色素蛋白途径的基因和超过80%的富集在卟啉-叶绿素代谢途径的基因在低硫条件下都下调表达(图4-A, B)。可见低硫胁迫对大豆光合作用的影响非常严重。除这3个途径外, 2个材料在核糖体途径(ribosome)、氧化磷酸化途径(oxidative phosphorylation)、代谢途径(metabolic pathways)和RNA聚合酶途径(RNA polymerase pathways)也富集了更多的下调表达基因(图4-A, B)。植物MAPK信号途径(MAPK signaling pathway-plant)是唯一一个在2个材料中富集较多上调表达基因的途径。在次生代谢途径(biosynthesis of secondary metabolites)中, 2个材料的差异表达基因对低硫的效应不同, 耐低硫材料云梦六月花叶富集了更多的下调表达基因(74%) (图4-A), 低硫敏感材料沁阳大豆富集了更多的上调表达基因(53%) (图4-B)。
与叶相比, 根中响应的基因数目虽然较少, 但在2个材料根中共有的低硫响应途径却较多(图3-B, D和图4-C, D), 其中9个途径在2个材料中对低硫的响应一致(图4-C, D)。脂肪酸降解途径(fatty acid degradation)、硫代谢途径(sulfur metabolism)、光合途径(photosynthesis)、角质-木栓质-蜡质生物合成途径(cutin, suberine and wax biosynthesis pathways)中富集了更多的上调差异表达基因, 而核糖体途径(ribosome), 丙氨酸-精氨酸-谷氨酸代谢途径(alanine, aspartate and glutamate metabolism)、氨基酸合成途径(biosynthesis of amino acids)、糖酵解/糖原生成途径(glycolysis/gluconeogenesis)、半胱氨酸和甲硫氨酸代谢途径(cysteine and methionine metabolism pathways)富集了更多的下调差异表达基因(图4-C, D)。其他9个共有途径在2个材料中对低硫的响应不同, 耐低硫材料云梦六月花叶在这些途径中富集了更多的上调表达基因(图4-C), 低硫敏感材料沁阳大豆在这些途径中富集了更多的下调表达基因(图4-D), 这些途径包括苯丙烷生物合成途径(phenylpropanoid biosynthesis)、光合生物的碳固定途径(carbon fixation in photosynthetic organisms)、次生代谢途径(biosynthesis of secondary metabolites)、碳代谢途径(carbon metabolism)、磷酸戊糖途径(pentose phosphate pathway)、甘氨酸-丝氨酸-苏氨酸代谢途径(glycine, serine and threonine metabolism)等(图4-C, D)。

(图3)
L和R分别表示叶和根。YM: 云梦六月花叶; QY: 沁阳大豆。
A: L-YM; B: R-YM; C: L-QY; D: R-QY. L and R represent leaves and roots, respectively. YM: Yunmengliuyuehuaye; QY: Qinyangdadou.
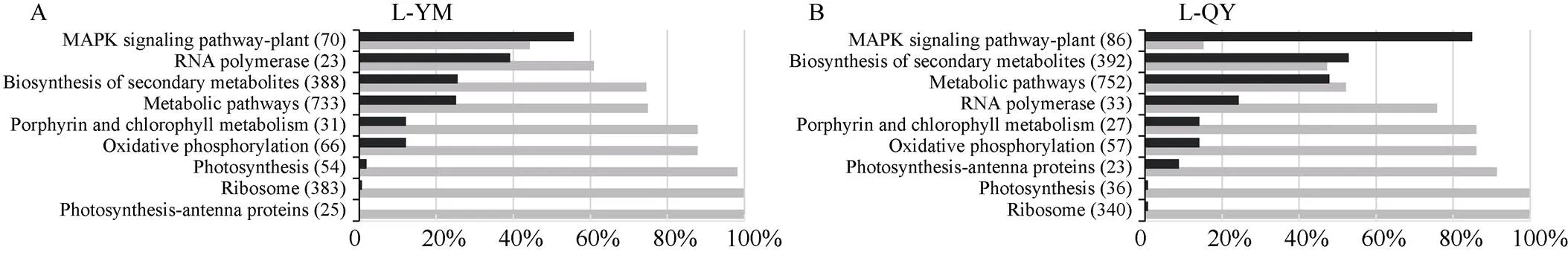
(图4)
富集在该途径的差异表达基因数列在括号内。L和R分别表示叶和根。YM: 云梦六月花叶; QY: 沁阳大豆。
The number of DEGs enriched in the pathway is list in bracket. L and R represent leaves and roots, respectively. YM: Yunmengliuyuehuaye; QY: Qinyangdadou.
2.4 不同的大豆硫酸根转运蛋白对低硫的响应不同
硫酸根转运蛋白在植物硫吸收和转运中起到重要作用。利用转录组数据, 本研究又分析了硫酸根转运蛋白编码基因的表达, 共鉴定到27个硫酸根转运蛋白基因, 根据Ding等[2]报道, 这些硫酸根转运蛋白基因分属4个亚组, 亚组1和2中分别鉴定到6个基因, 亚组 3中鉴定到13个基因, 是4个亚组中包含基因最多的一个亚组, 亚组 4中鉴定到2个(图5)。从图5可以看出, 不同亚组的硫酸根转运蛋白基因对低硫的响应是不同的, 亚组1、2、4中的基因多受低硫诱导上调表达, 其中()、()、()、()在根中受到低硫强烈诱导(图6),()、()、()、()、()在叶中受到低硫强烈诱导(图5)。亚组3中的基因对低硫的响应和其他3个亚组差别较大, 更加多变(图5)。其中有7个基因仅在一个材料或一个组织中响应低硫处理, 只有基因()在2个材料根和叶中都上调表达,其余5个基因没有响应低硫处理, 而且该亚组几乎包括了所有受低硫抑制的硫酸根转运蛋白基因。
进一步分析27个硫酸根转运蛋白基因在两个材料间的差异表达发现, 12个基因表达量在2个材料的根或叶中存在差异, 其中一半来自亚组3 (附表1)。多数根中差异表达基因在耐性材料云梦六月花叶中的表达量低于其在低硫敏感材料沁阳大豆中的表达量,而叶中差异表达基因的表现与根中相反, 在云梦六月花叶中的表达量都高于在沁阳大豆中的表达量(附表1)。
2.5 GmEIL1基因参与调控大豆硫代谢
由KEGG富集分析发现, 植物MAPK信号途径是2个材料叶中唯一一个包含较多上调基因的途径(图4-A, B), 富集在该途径的30%的差异表达基因属于乙烯信号途径(云梦六月花叶: 21/70; 沁阳大豆: 24/86), 其中包括乙烯钝感蛋白EIN3/EIL基因(图6)。该基因家族较小, 大豆中有12个成员, 5个响应低硫处理, 其中高度同源的基因和受低硫强烈诱导(图6-B), 2个基因预测蛋白与烟草NtEIL2和拟南芥AtEIL1聚在一个分支(图6-A)。qRT-PCR结果进一步确认和受低硫的诱导(附图1)。本研究选择了基因继续进行功能研究, 将其命名为。
组织分析表明,基因在大豆叶中的表达量最高, 在茎中的表达量最低, 在根、花和荚中的表达量相近(图7)。从云梦六月花叶的根cDNA中克隆了基因, 其编码区全长1866 bp, 编码621个氨基酸, 假定蛋白序列含有EIN3保守结构域。

图5 硫酸根转运蛋白基因的表达谱
数值为对照和处理间差异表达基因改变倍数的对数(log2)转换值, 灰色空格表示没有显著差异。L和R分别表示叶和根。YM: 云梦六月花叶; QY: 沁阳大豆。
The number shows the log2-transformed fold change value of DEGs between the control and low-sulfur treatments. The gray blank represents no significant. L and R represent leaves and roots, respectively. YM: Yunmengliuyuehuaye; QY: Qinyangdadou.

图6 EILs蛋白序列的系统进化树(A)与大豆EIL基因的表达热图(B)
数字为对照和处理间差异表达基因改变倍数的对数(log2)转换值, 灰色空格表示没有显著差异。L和R分别表示叶和根。YM: 云梦六月花叶; QY: 沁阳大豆。
The number shows the log2-transformed fold change value of DEGs between the control and low-sulfur treatments. The gray blank represents no significant. L and R represent leaves and roots, respectively. YM: Yunmengliuyuehuaye; QY: Qinyangdadou.
构建基因过表达和干扰载体, 通过遗传转化获得转基因毛状根大豆嵌合体。与各自的对照(OE-EV, RI-EV)相比, 基因在过表达毛根(OE- GmEIL1)中上调表达, 在干扰毛根(RI-GmEIL1)中下调表达(图8-A)。本研究测定了4个转化嵌合体株系地上部和毛根中硫酸根含量。结果表明, 基因过表达嵌合体地上部硫酸根含量在不同硫水平下与空载对照都没有显著差异, 基因RNA干扰嵌合体地上部的硫酸根含量在低硫条件下显著低于空载对照, 在正常硫水平下与空载对照没有显著差异(图8-B)。说明根中过表达基因对地上部硫酸根含量没有影响, 但在低硫条件下降低根中该基因的表达, 地上部硫酸根含量也降低(图8-B)。过表达嵌合体毛根中硫酸根含量在正常硫水平下显著高于对照, 在低硫条件下与对照没有显著差异, 干扰嵌合体毛根中硫酸根含量在2种硫水平下都显著低于对照。说明在硫充足水平下, 根中过表达基因能增加毛根中硫酸根的含量, 在硫充足和缺失条件下, 毛根中的硫酸根含量都随基因表达量的降低而显著降低(图8-C)。说明,基因参与调控大豆的硫代谢, 且对硫饥饿条件下大豆植株内硫酸根的吸收和转运是必不可少的。
在乙烯信号途径中,位于/的下游。在转录组中有5个ERF1基因被低硫胁迫强烈诱导, 本研究进一步检测了这些基因在大豆转基因毛根中的表达。5个基因中只有基因在低硫条件下的干扰毛根中下调表达(图8-D)。说明基因()可能在介导的大豆硫饥饿反应中发挥作用。
3 讨论
本研究对2个低硫表现不同的大豆材料进行了不同硫水平间的差异转录组分析, 鉴定到大量响应低硫的基因(图2), 与根相比, 2个材料叶中响应的基因更多, 共同富集9个KEGG途径, 3个途径与光合作用相关, 根中对低硫响应的差异表达基因虽然较少, 但富集到更多共有KEGG途径(图3和图4), 涉及光合作用相关、硫代谢相关、碳代谢相关、氨基酸代谢相关、脂肪酸代谢相关等途径。根和叶共同富集到4个途径, 分别是光合作用途径、核糖体途径、次生代谢途径和代谢途径, 其中光合作用途径的基因在根和叶中的表现差别较大, 在叶中大部分差异表达基因在为下调表达, 而根中的差异表达基因多为上调表达(图4)。同样, 低硫处理的拟南芥在叶中响应的基因比根中更多, 涉及硫代谢、碳代谢和光合作用相关的基因[16]。可见, 硫元素对植物的生长发育是非常重要的, 缺硫重塑了植物的代谢网络, 而且植物叶和根对低硫响应的遗传基础是不同的。
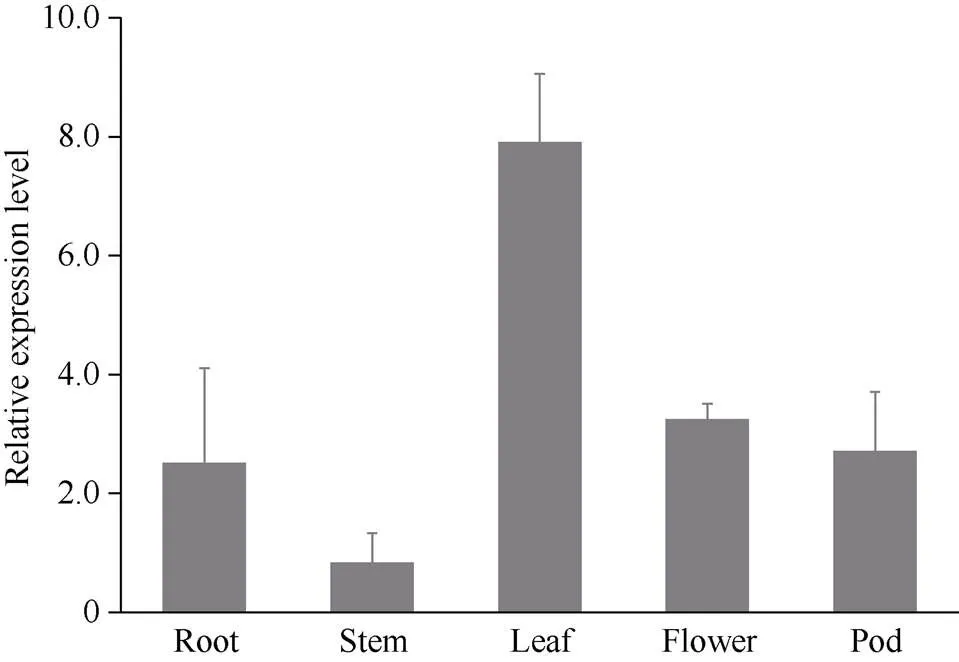
图7 GmNIL1的组织表达

图8 大豆转基因嵌合体中GmEIL1基因表达量(A)、地上部和毛根中硫酸根含量(B和C)和ERF1(Glyma.13G123100)基因的表达量(D)
+S: 对照;-S: 缺硫。OE-EV、OE-EIL1、RI-EV、RI-EIL1分别表示转过表达空载、基因过表达、干扰空载、基因干扰的转基因嵌合体。*、**、***分别表示在0.05、0.01、0.001水平显著差异。
+S: control;-S: low sulfate. OE-EV, OE-EIL1, RI-EV, and RI-EIL1 stand fortransgenic soybean chimera containing gene expression empty vector of gene expression,gene overexpression vector, empty vector of RNAi, andgene RNAi vector, respectively. *, **, and *** mean significant difference at the 0.05, 0.01, and 0.001 probability levels, respectively.
光合作用途径和核糖体途径是2个材料根和叶共有的途径, 包含较多差异表达基因(图4和图5)。核糖体途径的差异表达基因编码核糖体大、小亚基。在其他植物上, 核糖体基因不仅参与蛋白质的合成, 而且在植物生长发育、应答非生物胁迫、营养平衡等方面都发挥作用[17-21]。本课题组已报道过大豆核糖体大亚基编码基因参与调控大豆的硫利用, 在低硫环境下促进大豆对硫元素的吸收, 增加大豆地上部和地下部的干物质积累[9]。在本研究中该基因响应低硫处理(结果未给出), 这也暗示了其他响应低硫处理的核糖体基因可能在大豆硫元素利用中发挥作用, 值得进一步研究。光合作用途径的差异表达基因编码光系统I (PSI)、II (PSII)的蛋白、细胞色素b6/f复合体亚基、光合作用电子传递过程的蛋白、ATP酶等。与核糖体编码亚基基因一样, 光合作用途径基因在多种植物中也参与非生物胁迫的调控, 比如在马铃薯中, PSII外周蛋白PsbO基因突变体对非生物胁迫表现出较高的耐性, 在高盐、重金属、干旱环境下, 突变体的叶绿素含量、株高、叶片数目、块茎产量均高于对照, 突变体内H2O2积累较少, 活性氧清除酶的活性增强[22]。在甘薯中, PSII外周蛋白IbPsbP受非生物胁迫诱导上调表达, 与调控环境胁迫蛋白IbOr (Ipomoea batatas Orange protein)互作, 保护其不被热胁迫变性[23]。在拟南芥中, NADPH脱氢酶调控植物对生物胁迫和非生物胁迫的防御, 而植株体内NADPH脱氢酶的水平依赖于PSII外周蛋白PQL (PsbQ-like proteins, PQL)基因的正常表达[24]。本研究中这些响应低硫处理的光合作用途径基因有可能和其他植物中的基因一样参与调控大豆对非生物胁迫的反应, 特别是对低硫的胁迫反应, 可以作为候选基因进一步研究。
植物MAPK信号途径是2个材料叶中唯一一个包含较多上调表达基因的共有途径(图4-A, B), 该途径在植物应答各种胁迫中有重要作用, 其中EIN3/EIL是一个较小的乙烯钝感转录因子家族, 在多种植物中都有鉴定。拟南芥中有6个成员, 其中和在硫酸根吸收和转运中发挥作用, 参与调控硫饥饿反应,不受低硫诱导, 但突变体丧失硫饥饿反应[25-26]。烟草中有5个成员, 其中在硫饥饿条件下激活硫饥饿响应基因的表达[27]。本研究从大豆转录组中鉴定到12个EIL基因, 对基因进行功能研究发现,基因对硫饥饿条件下大豆硫代谢调控是必须的(图8), 该基因可作为候选基因进行育种利用研究。除基因外, 还有4个大豆基因响应低硫处理,与拟南芥同源,与拟南芥、、及烟草同源(图6), 此外, 植物MAPK信号途径中还有很多响应低硫诱导的基因(图4), 这些基因有可能和基因一样在大豆应答低硫胁迫反应中发挥作用, 有待作为候选基因进行深入研究。
植物硫酸根转运蛋白对于硫酸根的吸收和转运非常重要, 到目前为止对大豆硫酸根转运蛋白基因的研究十分有限。本研究利用转录组数据分析了27个硫酸根转运蛋白基因低硫响应的表达谱, 亚组1、2、4的基因多受低硫诱导上调表达(图5), 其中基因已进行功能研究, 该基因在根中强烈响应低硫诱导, 在烟草中过量表达可以提高不同硫水平下转基因植株的生物量、硫酸根吸收、硫元素含量等[2]。除基因外,、、、在根中同样受到低硫的强烈诱导, 上调水平都高于基因(图5), 这些基因可能在大豆硫酸根吸收和转运中发挥作用, 可以作为候选基因进一步研究其功能和利用潜力。在4个亚组中, 亚组3包含的硫酸根转运蛋白基因最多, 但该组基因对低硫响应与其他亚组基因不同, 仅有少数基因受低硫诱导, 多数基因受低硫抑制或者没有响应低硫处理(图5)。大豆亚组3基因的功能研究未见报道, 但其他植物上该亚组基因功能研究的报道较多。拟南芥的在发育的种子中尤其是胚的周围表达, 可能与硫酸根向胚中转运有关[28],基因定位在叶绿体中, 负责把硫酸根运输到叶绿体[29],增强从土壤中吸收硫酸盐的功能,基因发生突变会限制低硫条件下硫酸盐从根部向地上部的运输[30]。水稻中的表达与硫饥饿条件下总硫含量负相关[31],无论在缺硫还在硫充足的环境下都与植株的硫元素含量无关, 而是作为关键基因控制磷的吸收[32]。这些结果都说明亚组3基因的功能复杂, 在大豆中该亚组的基因是否参与以及如何参与硫酸根运输与再分配有待研究。
4 结论
本研究通过RNA重测序技术分析2个大豆材料不同水平下的转录组发现, 大豆根和叶对低硫的响应不同, 叶中鉴定到更多的低硫响应基因, 而根中富集到更多的KEGG途径。KEGG富集分析发现, 缺硫重塑了大豆多个代谢途径, 包括硫代谢相关、氨基酸代谢相关、光合作用相关、碳代谢相关、脂肪酸代谢相关等途径。对27个大豆硫酸根转运蛋白基因的表达谱分析发现, 亚组1、2、4的基因多受低硫诱导, 亚组3的基因对低硫的响应多变。通过转化大豆毛状根证明低硫诱导上调表达基因参与大豆硫利用的调控。总之, 本研究揭示了全基因组水平上大豆应答低硫胁迫的转录组, 发掘大量的硫利用候选基因, 初步证明基因参与大豆硫利用的调控, 为深入探索大豆硫利用效率的遗传机理奠定基础, 为大豆耐低硫育种提供了候选基因。

附图1 21个差异表达基因的qRT-PCR与转录组重测序结果
Fig. S1 qRT-PCR and RNA-seq results of 21 DEGs
log2(FC): YM和QY间差异表达基因改变倍数的对数(log2)转换值。L和R分别表示叶和根。YM: 云梦六月花叶; QY: 沁阳大豆。
log2(FC): log2-transformed fold change value of DEGs between the control and low-sulfur treatments. L and R represent leaves and roots, respectively. YM: Yunmengliuyuehuaye; QY: Qinyangdadou.

附表1 2个大豆材料间差异表达的硫酸根转运蛋白基因
数值表示YM和QY间差异表达基因改变倍数的对数(log2)转换值, 正、负值分别代表上调和下调。—: 没有差异。L和R分别表示叶和根。YM: 云梦六月花叶; QY: 沁阳大豆。
The numbers show the log2-transformed fold change value of DEGs between YM and QY. The positive and negative values represent up and down, respectively. —: no different. L and R represent leaves and roots. YM: Yunmengliuyuehuaye; QY: Qinyangdadou.
[1] Li Q, Gao Y, Yang A. Sulfur homeostasis in plants., 2020, 21: 8926.
[2] Ding Y, Zhou X, Zuo L, Wang H, Yu D. Identification and functional characterization of the sulfate transporter genein soybean., 2016, 17: 373.
[3] Phartiyal P, Kim W S, Cahoon R E, Jez J M, Krishnan H B. Soybean ATP sulfurylase, a homodimeric enzyme involved in sulfur assimilation, is abundantly expressed in roots and induced by cold treatment., 2006, 450: 20–29.
[4] Phartiyal P, Kim W S, Cahoon R E, Jez J M, Krishnan H B. The role of 5’-adenylylsulfate reductase in the sulfur assimilation pathway of soybean: molecular cloning, kinetic characterization, and gene expression., 2008, 69: 356–364.
[5] Krishnana H B, Jez J M. Review: the promise and limits for enhancing sulfur-containing amino acid content of soybean seed., 2018, 272: 14–21.
[6] Chronis D, Krishnan H B. Sulfur assimilation in soybean ([L.] Merr.): molecular cloning and characterization of a cytosolic isoform of serine acetyltransferase., 2004, 218: 417–426.
[7] Zhang C, Meng Q, Zhang M, Huang F, Gai J, Yu D. Characterization of O-acetylserine (thiol) lyase-encoding genes reveals their distinct but cooperative expression in cysteine synthesis of soybean [(L.) Merr.]., 2008, 26: 277–291.
[8] Chronis D, Krishnan H B. Sulfur assimilation in soybean: molecular cloning and characterization of O-acetylserine (thiol) lyase (cysteine synthase)., 2003, 43: 1819–1827.
[9] 陈燕宁, 吴志医, 元文杰, 阚贵珍, 黄方, 喻德跃, 王慧. 核糖体基因对大豆低硫耐性的调控作用研究. 大豆科学, 2020, 39: 518–526.
Chen Y N, Wu Z Y, Yuan W J, Kan G Z, Huang F, Yu D Y, Wang H. Research on the regulation effect of ribosomal geneon low sulfur tolerance in soybean., 2020, 39: 518–526 (in Chinese with English abstract).
[10] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2–ΔΔCTmethod., 2001, 25: 402–408.
[11] Pertea M, Pertea G M, Antonescu C M, Chang T C, Mendell J T, Salzberg S L. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads., 2015, 33: 290–295.
[12] Xie C, Mao X, Huang J, Ding Y, Wu J M, Dong S, Kong L, Gao G, Li C Y, Wei L P. KOBAS 2.0: a web server for annotation and identification of enriched pathways and diseases., 2011, 39: W316–W322.
[13] Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6: molecular evolutionary genetics analysis version 6.0., 2013, 30: 2725–2729.
[14] Holsters M, de Waele D, Depicker A, Messens E, van Montagu M, Schell J. Transfection and transformation of., 1978, 163: 181–187.
[15] Kereszt A, Li D X, Indrasumunar A, Nguyen C D T, Nontachaiyapoom S, Kinkema M, Gresshoff P M.-mediated transformation of soybean to study root biology., 2007, 2: 948–952.
[16] Hirai M Y, Fujiwara T, Awazuhara M, Kimura T, Noji M, Saito K. Global expression profiling of sulfur-starvedby DNA macroarray reveals the role of O-acetyl-L-serine as a general regulator of gene expression in response to sulfur nutrition., 2003, 33: 651–663.
[17] Luo A, Zhan H, Zhang X, Du H, Zhang Y, Peng X. Cytoplasmic ribosomal protein L14B is essential for fertilization in., 2020, 292: 110394.
[18] Lin D, Jiang Q, Zheng K, Chen S, Zhou H, Gong X, Xu J, Teng S, Dong Y. Mutation of the ricegene encoding plastid ribosomal protein L21 causes chloroplast developmental defects and seedling death., 2015, 17: 599–607.
[19] Ludwig A, Tenhaken R. Suppression of the ribosomalgene reveals a novel mechanism for stress adaptation in soybean., 2001, 212: 792–798.
[20] Kim K Y, Park S W, Chung Y S, Chung C H, Kim J I, Lee J H. Molecular cloning of low-temperature-inducible ribosomal proteins from soybean., 2004, 55: 1153–1155.
[21] Dong X, Duan S, Wang H B, Jin H L. Plastid ribosomal proteinis involved in photosynthesis and the response to C/N balance in., 2020, 62: 1418–1432.
[22] Gururani M A, Upadhyaya C P, Strasser R J, Yu J W, Park S W. Evaluation of abiotic stress tolerance in transgenic potato plants with reduced expression of PSII manganese stabilizing protein., 2013, 198: 7–16.
[23] Kang L, Kim H S, Kwon Y S, Ke Q, Ji C Y, Park S, Lee H, Deng X, Kwak S. IbOr regulates photosynthesis under heat stress by stabilizing IbPsbP in sweet potato., 2017, 8: 989.
[24] Yabuta S, Ifuku K, Takabayashi A, Ishihara S, Ido K, Ishikawa N, Endo T, Sato F. Three PsbQ-like proteins are required for the function of the chloroplast NAD(P)H dehydrogenase complex in., 2018, 51: 866–876.
[25] Dietzen C, Koprivova A, Whitcomb S J, Langen G, Jobe T O, Hoefgen R, Kopriva S. The transcription factorparticipates in the regulation of sulfur-deficiency response., 2020, 84: 2120–2136.
[26] Maruyama-Nakashita A, Nakamura Y, Tohge T, Saito K, Takahashi H.is a central transcriptional regulator of plant sulfur response and metabolism., 2006, 18: 3235–3251.
[27] Wawrzyńska A, Lewandowska M, Sirko A.directly regulates expression of at least one tobacco gene induced by sulphur starvation., 2010, 61: 889–900.
[28] Zuber H, Davidian J C, Aubert G, Aimé D, Belghazi M, Lugan R, Heintz D, Wirtz M, Hell R, Thompson R, Gallardo K.The seed composition ofmutants for the group 3 sulfate transporters indicates a role in sulfate translocation within developing seeds., 2010, 154: 913–926.
[29] Cao M J, Wang Z, Wirtz M, Hell R, Oliver D J, Xiang C B. The chloroplast-localized sulfate transporteraffects ABA biosynthesis in., 2013, 73: 607–616.
[30] Kataoka T, Hayashi N, Yamaya T, Takahashi H. Root-to-shoot transport of sulfate in: evidence for the role ofas a component of low affinity sulfate transport system in the root vasculature., 2004, 136: 4198–4204.
[31] Zhao H, Frank T, Tan Y, Zhou C, Jabnoune M, Arpat A B, Cui H, Huang J, He Z, Poirier Y, Engel K H, Shu Q. Disruption ofreduces phytate and phosphorus concentrations and alters the metabolite profile in rice grains., 2016, 211: 926–939.
[32] Yamaji N, Takemoto Y, Miyaji T, Mitani-Ueno N, Yoshida K T, Ma J F. Reducing phosphorus accumulation in rice grains with an impaired transporter in the node., 2017, 541: 92–95.
Transcriptional expression profiling of soybean genes under sulfur-starved conditions by RNA-seq
WANG Hui, WU Zhi-Yi, ZHANG Yu-E, and YU De-Yue*
National Center for Soybean Improvement / National Key Laboratory of Crop Genetics and Germplasm Enhancement / Key Laboratory of Biology and Genetic Improvement of Soybean, Ministry of Agriculture and Rural Affairs / Nanjing Agricultural University, Nanjing 210095, Jiangsu, China
Soybean is an important grain and oil crop. Few researches focused on soybean sulfur utilization. Here, the tolerance of Yunmengliuyuehuaye (YM) and Qinyangdadou (QY) to low sulfur were evaluated. The gene expression profiles of roots and leaves of the two materials under the control (+S) and sulfur deficiency (-S) environments were analyzedRNA resequencing. The results showed that YM was tolerance to low sulfur and QY was susceptibility to low sulfur. 9064 and 9795 differentially expressed genes (DEGs) were identified in leaves of YM and QY, and 3185 and 5006 DEGs were identified in roots of YM and QY, respectively. KEGG enrichment revealed that nine pathways were common in the two material leaves, of which MAPK signaling pathway—plant specially enriched more upregulated expressed genes. There were 18 common pathways in both roots. Nine of them responded consistently to low sulfur in YM and QY, of which four contained more up-regulated genes and five contained more downregulated genes. In the remaining nine pathways, YM contained more upregulated expressed genes.Soybean sulfate transporter genes were important for the absorption and transportation of sulfate. In the transcriptome, 27 soybean sulfate transporter genes were identified. These genes belonged to 4 subgroups respectively. Most of the genes in subgroups 1, 2, and 4 were induced by low sulfur, and the genes in subgroup 3 responded to low sulfur in a complex way. KEGG showed that the upregulated gene of(ethylene-insensitive 3-like) in the MAPK signaling pathway-plant was clone. The gene was involved in the regulation of soybean sulfur utilization in the soybean chimeras with transgenic hairy root. These results provide a basis for deeply exploring the genetic mechanism of soybean sulfur utilization efficiency and candidate genes for soybean tolerance to low sulfate breeding in the future.
soybean; low sulfur; RNA-seq; sulfate transporter; EIN3/EIL
10.3724/SP.J.1006.2023.24004
本研究由国家自然科学基金项目(32072080)资助。
This study was supported by the National Natural Science Foundation of China (32072080).
通信作者(Corresponding author):喻德跃, E-mail: dyyu@njau.edu.cn
E-mail: wanghui0@njau.edu.cn
2022-01-04;
2022-03-25;
(网络出版日期): 2022-04-20.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220418.1423.016.html
