利用高密度Bin遗传图谱定位水稻抽穗期QTL
2023-01-12梁文化赵春芳魏晓东周丽慧王才林张亚东
赵 凌 梁文化 赵春芳 魏晓东 周丽慧 姚 姝 王才林 张亚东
利用高密度Bin遗传图谱定位水稻抽穗期QTL
赵 凌 梁文化 赵春芳 魏晓东 周丽慧 姚 姝 王才林 张亚东*
江苏省农业科学院粮食作物研究所/ 国家水稻改良中心南京分中心 / 国家耐盐碱水稻技术创新中心华东中心 / 江苏省优质水稻工程技术研究中心, 江苏南京 210014
挖掘新的控制水稻抽穗期相关位点和候选基因, 对抽穗期的遗传机制研究和品种改良具有重要的意义。利用抽穗期存在明显差异的粳稻TD70和籼稻Kasalath杂交衍生的包含186个家系的重组自交系群体, 构建了基于深度重测序的高密度Bin遗传图谱, 图谱共包含12,328个Bin标记。RIL群体及亲本2018年和2021年正季种植于江苏省南京市江苏省农业科学院。以家系从播种到抽穗所经历的天数作为抽穗期表型值, 使用IciMapping软件3.4版的完备区间作图法, 对控制水稻抽穗期的QTL进行鉴定。2年共检测到15个抽穗期的QTL, 分布在3号、6号、7号、8号、10号和12号染色体上, LOD值为2.58~10.68, 其中7个QTL和已知抽穗期QTL的位置存在重叠或者部分重叠。共有4个QTL在2年均检测到, 表现出较强的稳定性。对2年重复鉴定到的4个QTL区间进行基因功能注释和亲本间序列分析, 共发现7个注释有功能, 且在2个亲本间编码区存在非同义突变的基因。根据候选基因SNP的类型对RIL群体家系进行基因等位型分类和效应分析, 发现4个基因其不同等位型的RIL家系在抽穗期上存在显著或者极显著差异, 推测可能为候选基因, 可用于后续水稻抽穗期的分子机制研究。
水稻; 重组自交系; 高密度Bin图谱; 抽穗期; QTL
水稻是我国主要粮食作物, 对于保障粮食安全具有重要的作用。培育高产、优质多抗的水稻品种是保障粮食安全的重要举措。抽穗期(heading date, HD)指水稻从种子萌发到抽穗的时间, 是水稻重要农艺性状之一, 不但影响生育期, 而且与产量、品质及抗逆性等紧密相关, 是决定水稻种植地区及季节适应性的关键性状[1-2]。解析水稻抽穗期的相关基因及其分子调控机制, 对于培育和应用高产水稻品种、保障国家粮食安全和经济发展具有重要意义。
水稻抽穗期受基因型与环境, 如光照和温度等共同影响, 具体表现为感光性、感温性和基本营养生长性的相互作用[3]。内在遗传发育机制和外在环境因素的共同作用使得水稻抽穗期的遗传机制异常复杂。作为典型的短日照喜温型植物, 不仅短日照可以加快水稻生长发育速度, 适当的高温也能促进营养生长向生殖生长提早转变, 从而缩短抽穗期[4]。
目前研究表明抽穗期是由主效基因和微效基因共同控制的数量性状, 现已报道的水稻抽穗期相关数量性状位点有618个, 在12条染色体上均有分布。已有、、、、、和等50多个抽穗期相关基因被成功克隆[5-9]。按照功能和作用机制, 抽穗期基因主要分为成花素类基因、光敏色素类基因、开花集成基因、促进开花基因和抑制开花基因等[3]。这些基因共同组成了1个至少包括光周期和生物钟、染色质相关、激素等3个通径的复杂分子网络[7,10]。最早通过图位克隆方法鉴定的抽穗期基因参与保守的光周期遗传调控通路, 是水稻抽穗期的重要调控基因[11-13]。10号染色体上基因源自非洲栽培稻, 编码的B型反应调节子促进水稻短日照下提早抽穗, 是水稻成花素的重要调控基因[12]。目前对通路和通路等调控水稻抽穗期均有较深入的研究[3,14-16]。
水稻抽穗期调控机制一直是分子生物学家和育种家关注和研究的热点, 虽然目前定位到的抽穗期QTL很多, 但大多数QTL受环境影响较大, 且很多QTL定位区间比较大, 功能得到验证和克隆的QTL有限, 在水稻育种中成功应用的也很少, 对各通路调控抽穗期的机制研究也亟待深入。通过抽穗期QTL定位和基因鉴定及功能研究揭示调控机制, 对于实现水稻抽穗期的分子育种, 保证水稻稳产与高产具有重要的意义。
Bin图谱是基于SSR/RFLP标记的传统遗传图谱后的新一代遗传图谱, 以连续SNP标记作为染色体重组事件的最小单位(recombination bin), 判断子代每个bin的来源, 从而构建出Bin遗传图谱[17-18]。Bin图谱基于高通量测序构建, SNP标记分布广泛、多样性高、数量大, 而且构建时间短、精确度高, 在水稻复杂数量性状研究中具有广泛的应用前景[19]。
水稻品种间抽穗期存在着巨大的差异[20], 已经定位和克隆的相关QTL和基因并不能完全解释抽穗期的遗传及变异, 其分子机制还需要更加深入地研究。本研究以抽穗期存在显著差异的粳稻TD70和籼稻Kasalath衍生的重组自交系群体(RIL)为作图群体, 利用构建的包括12,328个Bin标记的高密度连锁图谱, 定位和分析抽穗期QTL, 寻找一些新的位点, 以期为水稻抽穗期相关基因的鉴定提供基础。
1 材料与方法
1.1 试验材料
以一个杂交系谱为天鹅谷///9520//(72-496/苏御糯)的粳稻品系TD70和籼稻品种Kasalath为亲本配制组合。2005年夏从F1单株收取自交种子, 以单粒传方法构建成由186个家系组成的RIL群体[21]。基因型调查为F10世代, 表型调查为F9和F12世代。
1.2 田间试验及抽穗期记载
于2018年和2021年, 在江苏省农业科学院粮食作物研究所试验田种植RIL群体的186个家系及2个亲本, 随机区组设计。每年均于5月10日播种, 6月10日移栽, 每个家系种植4行, 每行7株, 单本栽插, 行株距为25.0 cm × 16.7 cm。移栽后至收获不施肥, 搁田后田间保持干干湿湿至收获, 其他田间管理按常规栽培管理方法。以每家系小区内50%单株始穗日期为该家系的抽穗日期。以家系从播种到抽穗所经历天数的均值作为该家系的抽穗期表型值。
1.3 连锁图谱构建与QTL定位
对粳稻TD70与籼稻Kasalath杂交衍生RILs群体的186个家系及亲本进行全基因组重测序和基于SNP的Binmap连锁图谱构建, 构建的高密度遗传图谱含有12,328个Bin标记, 各染色体Bin标记数在763~1367个, 标记间平均物理距离为30.26 kb[21]。利用QTL IciMapping软件3.4版, 采用完备区间作图法(inclusive composite interval mapping, ICIM)在全基因组范围内进行扫描[22-23], 扫描步长为1.0 cM, 以LOD 2.5作为阈值检测控制抽穗期QTL的存在, QTL的命名方法参照McCouch等[24]规则。
1.4 数据的统计分析
在Microsoft Excel 2016中处理表型数据和作图、绘制频次分布图以及方差分析。应用Prism (Version 8.0.1)软件绘制箱式图。
2 结果与分析
2.1 RIL群体及其亲本的抽穗时间表型特征
2018年和2021年RIL群体及其双亲抽穗期的表型变异见图1。亲本TD70抽穗期分别为106 d和99 d, 而Kasalath的抽穗期为84 d和89 d, 2年不同环境下TD70的抽穗期均极显著长于Kasalath (<0.01)。
2018年RIL群体的抽穗期均值为98.6 d, 抽穗期最长的家系达到117 d, 最短的家系为85 d (图1)。2021年RIL群体的抽穗期均值为96.5 d, 抽穗期最长的家系为111 d, 最短的家系为84 d (表1), 群体中抽穗期长度存在明显的超亲现象。2年RIL群体的抽穗期分布的峰度和偏度绝对值都小于1, 表明群体的抽穗期呈连续性正态分布, 表现出数量性状的遗传特点(图1), 可以对其进行QTL定位分析。
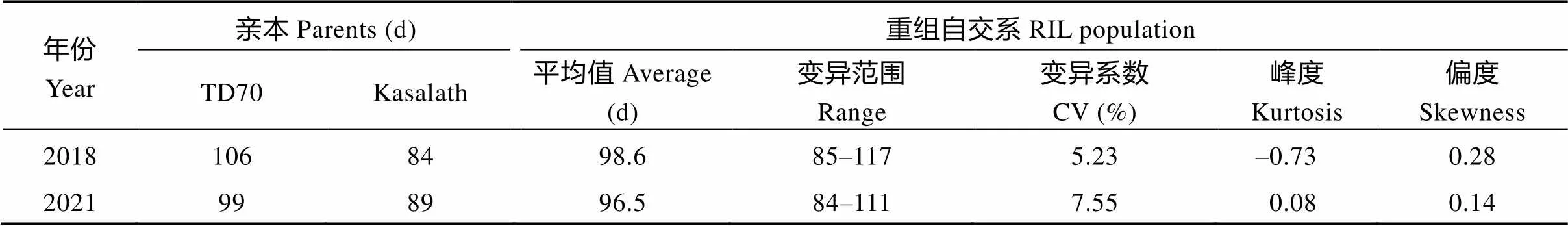
表1 亲本与重组自交系群体的抽穗天数
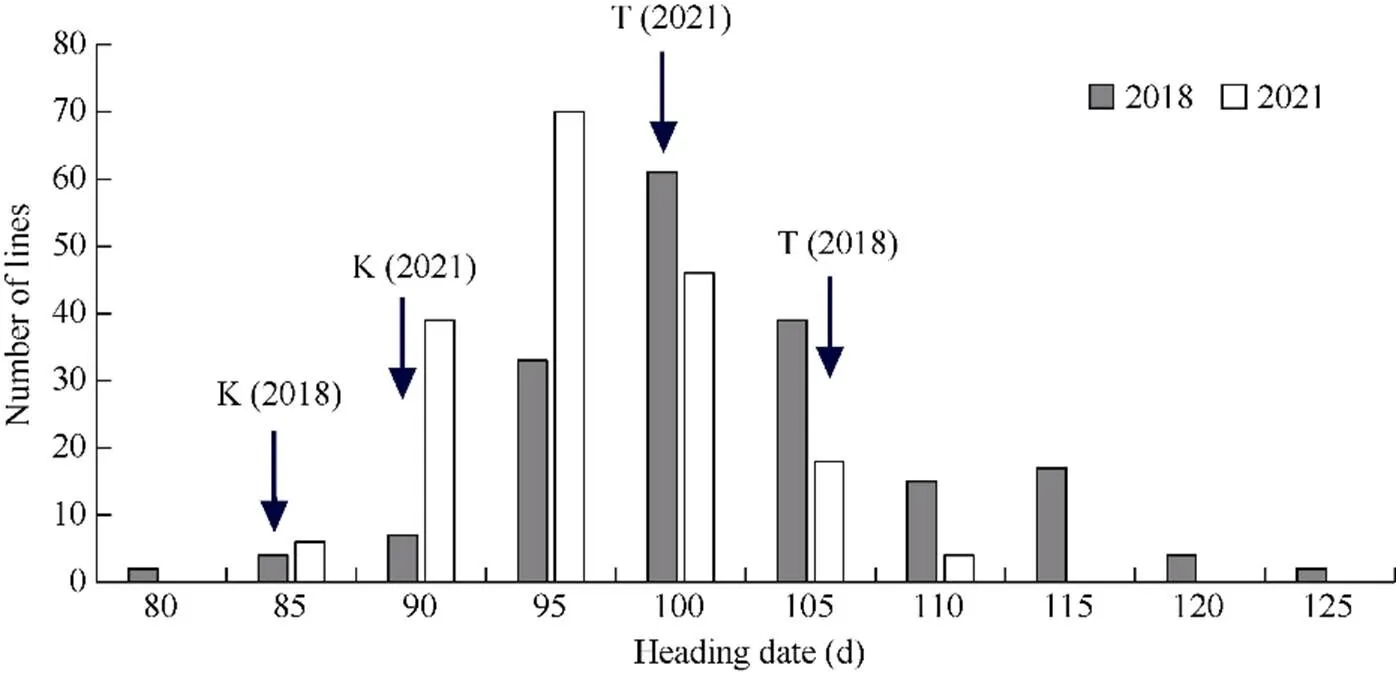
图1 重测序RIL群体抽穗期分布
K: Kasalath; T: TD70.
2.2 抽穗期QTL检测
对RIL群体进行抽穗期QTL检测, 2年共检测到15个QTL, 分布在3号、6号、7号、8号、10号和12号染色体上(表2和图2)。其中, 2018年检测到8个QTL, LOD值介于2.58~10.68, 贡献率介于3.29%~14.73%。2021年检测到7个QTL, 分别位于7号、8号、10号和12号染色体, LOD值介于3.06~8.21, 贡献率3.92%~12.58%。7号染色体的、8号染色体的、10号染色体的和都在2年被检测到。
位于7号染色体上14.50~14.54 Mb, 2年的LOD均大于6, 表现出较强的稳定性。在2018年可解释11.77%的表型变异, 来自于TD70的等位基因可使抽穗期延长2.66 d。在2021年检测到LOD值为8.21, 可解释12.58%的表型变异。来自于TD70的等位基因可使抽穗期延长1.81 d。
2年都检测到的位点位于8号染色体上1.73~1.82 Mb, 在2018年可解释3.97%的表型变异, 在2021年可解释3.92%的表型变异, 来自于TD70的等位基因可使抽穗期分别延长1.56 d和1.01 d。和也是2年均检测到位点, 位于10号染色体的3~4 Mb之间, 2年LOD值都在4~6之间, 来自于TD70的等位基因可使抽穗期延长。
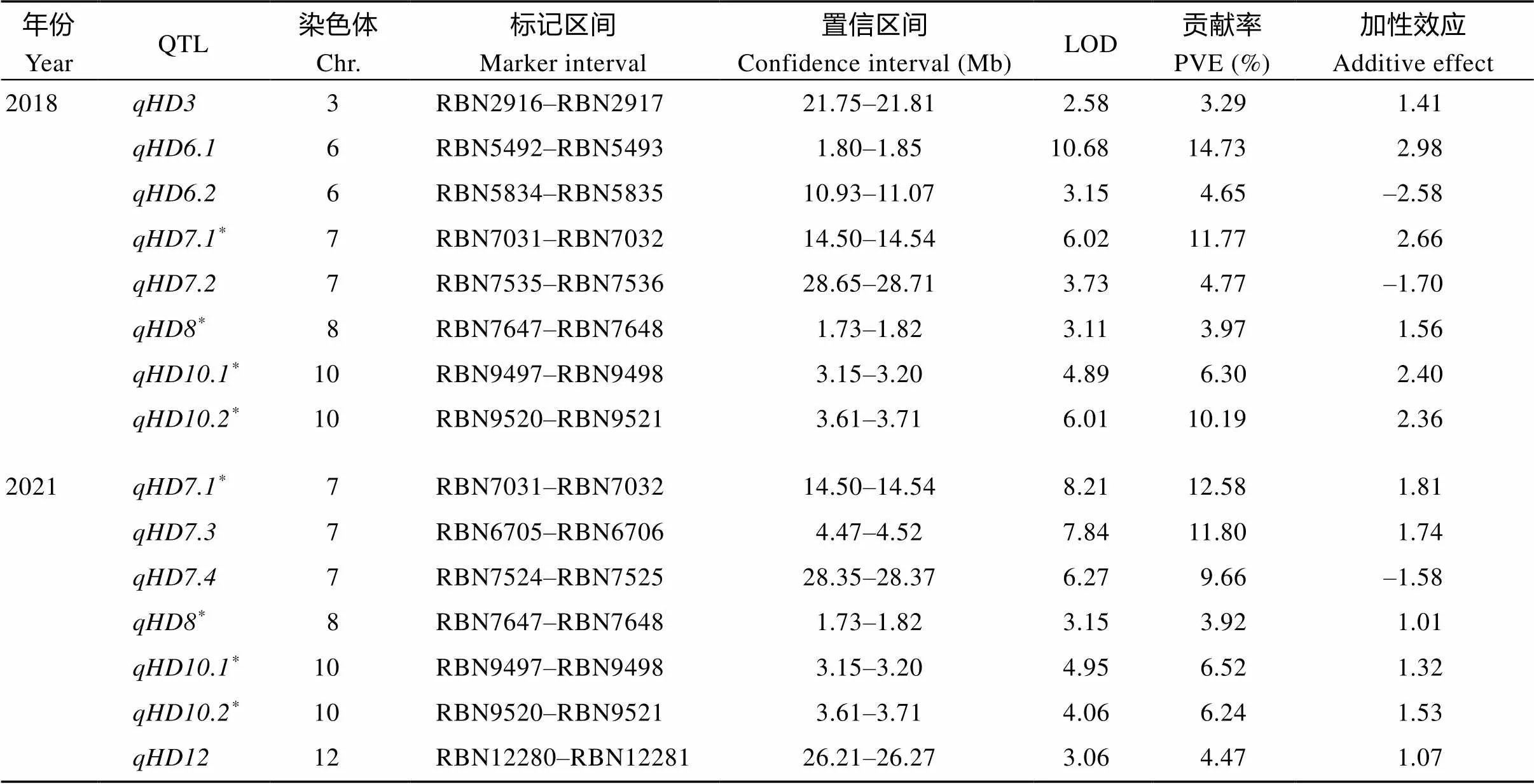
表2 检测到控制抽穗期的QTL
加性效应值为正值, 表明增效等位基因来源于TD70。*: 在2年间重复检测到的QTL。
Positive additive effect showed that source of favorable allele came from TD70.*: the QTL was detected in both two years.
除了2年均检测到的4个QTL外, 2018年在3号染色体21.75~21.81 Mb区间检测到, 2021年在12号染色体26.21~26.27 Mb区间检测到,均为来自于TD70的等位基因可使抽穗期延长。
2018年在6号染色体检测到2个影响抽穗期的QTL, 分别位于1.8 Mb和11 Mb左右, 其中的LOD值达到10.68, 是所有检测到QTL中LOD值最大的位点, 解释14.73%的表型变异, 增效基因来自于TD70。的LOD值3.15, 解释4.65%的表型变异, 增效等位基因来自于Kasalath。
7号染色体上除2年均检测到的外, 2018年检测到, 2021年检测到和。的LOD值3.73,和的LOD值为7左右。和的增效等位基因来自于Kasalath,的增效等位基因来自于TD70。
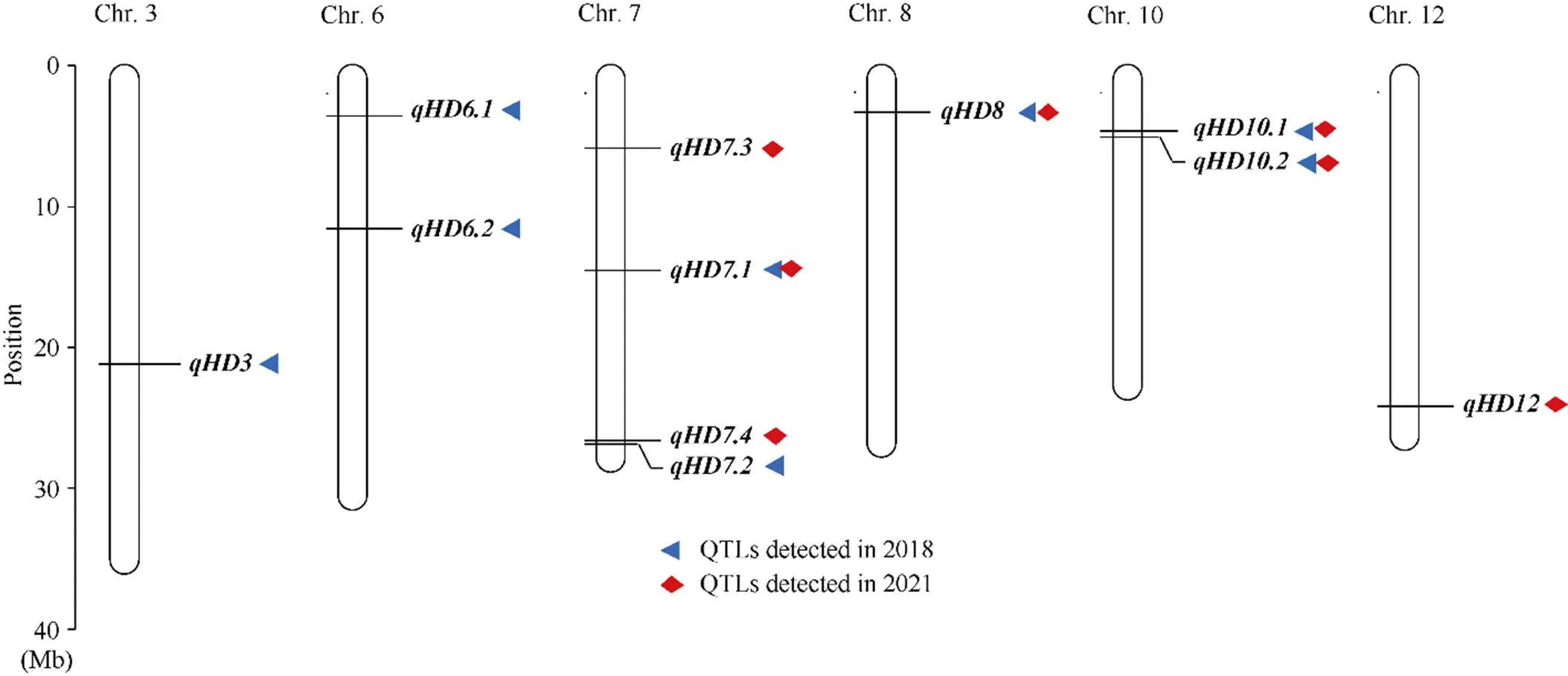
图2 检测到抽穗期QTL的染色体分布图
2.3 候选基因的筛选
利用水稻注释数据库(Rice Annotation Project Database, https://rapdb.dna.affrc.go.jp)对2年重复检测到的影响抽穗期的4个QTL,、和区间内的基因进行功能注释(表3)。位于7号染色体上14.50~14.54区域内, 经过查询水稻注释数据库, 该区间内共有5个注释基因。序列分析发现除外, 其他4个基因的编码区在2个亲本间不存在非同义突变。全长4358 bp编码的蛋白注释为甲硫氨酸氨基肽酶家族成员。TD70和Kasalath的编码区存在5处错义突变: CDS+372 bp (T/C)、CDS+1849 bp (G/A)、CDS+1850 bp (A/C)、CDS+2205 bp (G/A)和CDS+2459 bp (C/T), 编码的氨基酸发生了变化(Gln/Arg、Ala/Val、Leu/Trp、Arg/Trp和Ser/Asn) (图3-A)。位于8号染色体上1.8 Mb, 在水稻注释数据库查询发现该区间内共有8个注释基因, 除不转录蛋白、假定蛋白等基因外, 还有5个注释有功能的基因, 分别编码半乳糖基转移酶家族、五肽、细胞色素P450羟化酶、LTP家族蛋白前体和3-磷酸甘油酰基转移酶。其中基因和的编码区在亲本间存在非同义突变。编码区存在1个移码突变和5个错义突变,编码区存在3个错义突变,编码区存在7个错义突变、3个移码突变和2个碱基缺失(图3- B~D)。
区间内共有3个注释基因, 除去不转录蛋白、假定蛋白外, 还有1个注释有功能的基因, 编码OsWAK受体类似蛋白激酶。编码区在亲本间存在4个错义突变, 分别为CDS+405 bp (A/C)、CDS+4665 bp (T/A)、CDS+4816 bp (A/C)、CDS+4970 bp (T/C), 导致编码的氨基酸发生了变化(Tyr/Asp、Thr/Ser、Asp/Glu和Asn/Ser) (图3-E)。
位于10号染色体上3.61~3.71区域内, 在水稻注释数据库查询发现该区间共有6个注释基因, 其中3个基因和注释有功能, 分别编码丝裂原活化蛋白激酶、钙依赖性蛋白激酶和查尔酮合酶。编码区的核苷酸序列在2个亲本间不存在非同义突变。编码区在亲本间存在1个错义突变CDS+414 bp (A/T), 编码的氨基酸发生了Phe/Tyr变化。编码区在亲本间存在1个错义突变和1个移码错义突变, 分别发生在CDS+109 bp (A/G)、CDS+1174 bp (T/GC) (图3-F~G)。
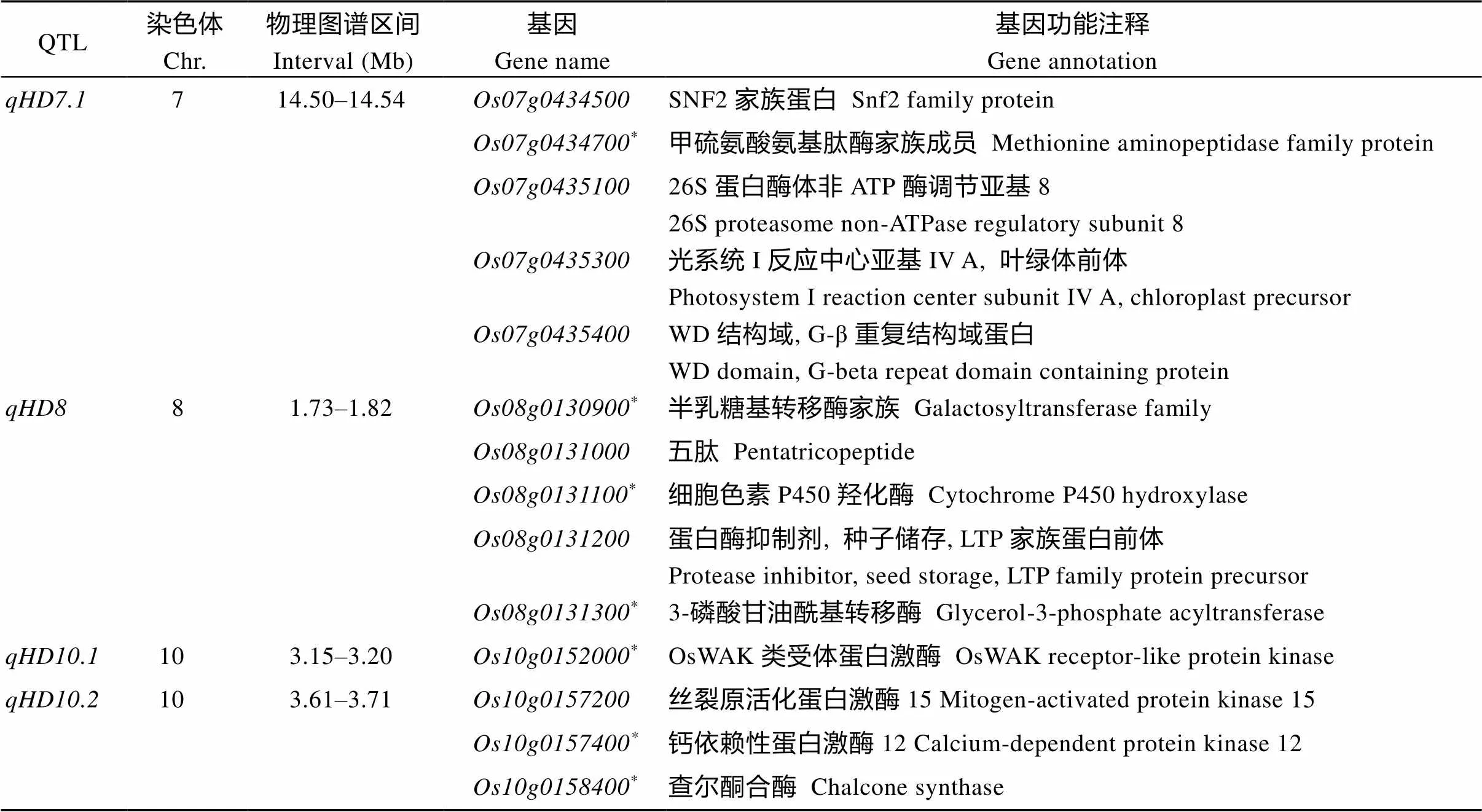
表3 重复检测到QTL区间内基因的注释
*: 基因编码区在亲本间存在非同义突变。
*: there are non-synonymous mutations in the coding regions of genes between the parents.
在2年重复检测到的4个QTL区间内, 经过基因注释查询和序列分析, 确定了7个注释有功能、且编码区在2个亲本间存在非同义突变的基因, 非常有可能为抽穗期候选基因。根据这些基因SNP的类型对RIL群体的家系进行等位型分类, SNP位点核苷酸为亲本TD70型的命名为HapA, 为Kasalath型命名为HapB。通过方差分析检测HapA和HapB等位型对群体家系抽穗期的效应, 结果发现其中4个基因:、、和00, 其不同等位型RIL家系间抽穗期存在显著或者极显著差异, 推测其为最有可能的候选基因(图4)。的候选基因编码甲硫氨酸氨基肽酶,的候选基因和编码半乳糖基转移酶及细胞色素P450羟化酶。的候选基因编码钙依赖性蛋白激酶, 具体的生物学功能有待进一步研究。
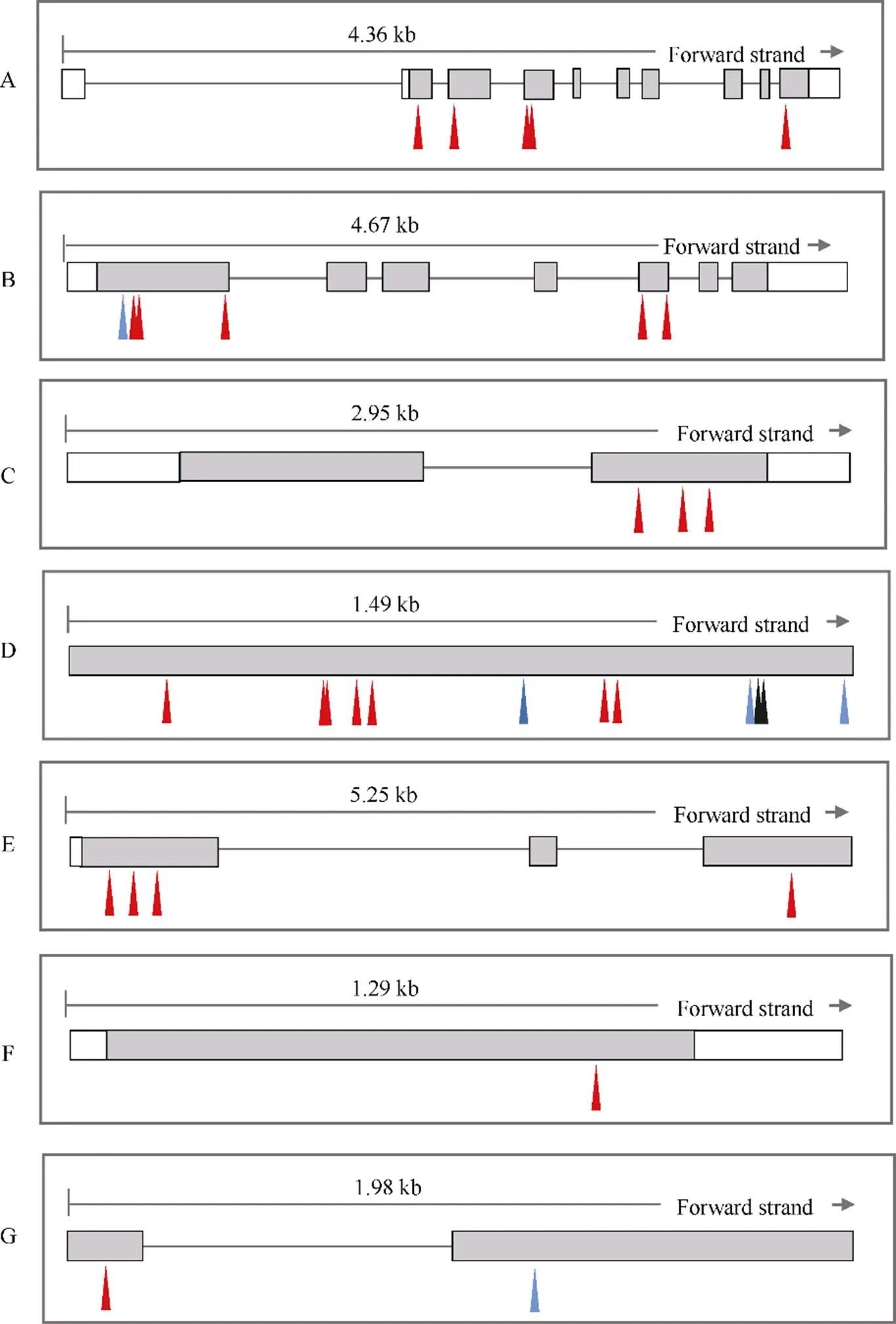
图3 TD70与Kasalath之间候选基因的结构和非同义突变
黑色框: 外显子; 灰色部分: 编码序列; 红色箭头: 错义突变; 蓝色箭头: 移码突变; 黑色箭头: 缺失。
A:; B:; C:; D:; E:; F:00; G:. Frames with black lines: exon; Grey boxes: protein coding sequence; Red arrow: missense mutation; Blue arrow: frameshift variant: Black arrow: indel.
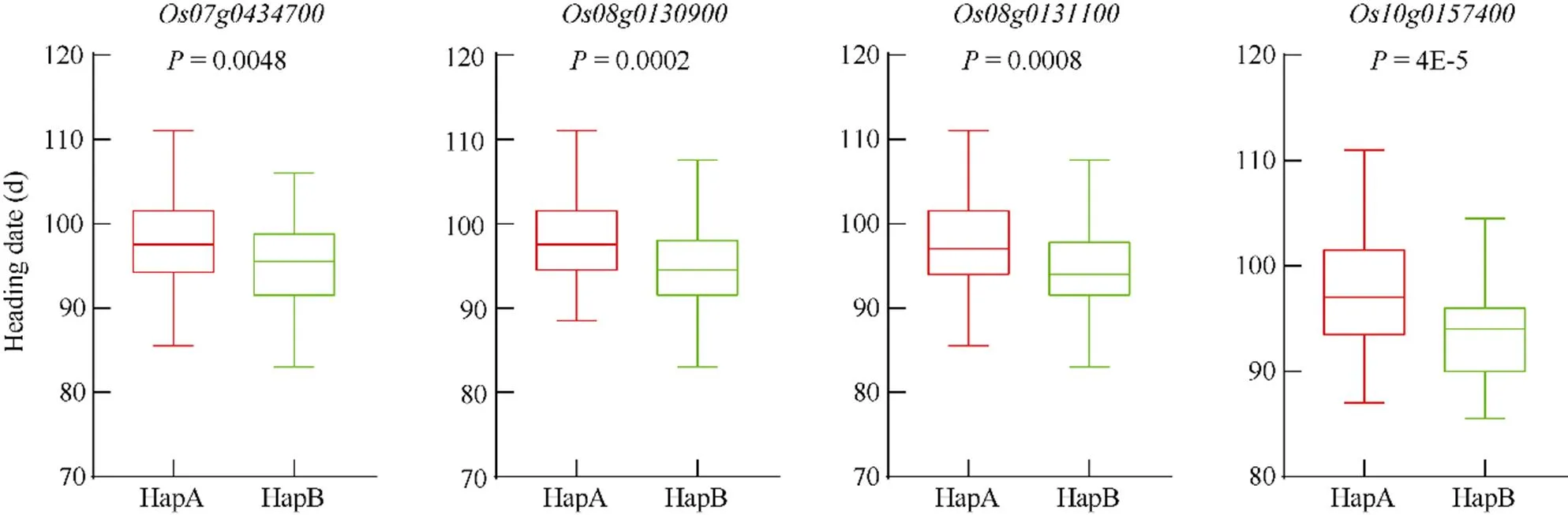
图4 候选基因不同等位型对RIL群体抽穗期的影响
SNP位点核苷酸为TD70型的命名为HapA, 为Kasalath型命名为HapB。
HapA: the SNP of candidate genes are the same as TD70; HapB: the SNP of candidate genes are the same as Kasalath.
3 讨论
3.1 Bin遗传图谱定位水稻复杂性状QTL的优势
Bin图谱是基于高通量测序技术构建的新一代遗传图谱, 与传统遗传图谱相比, 不仅耗时短, 可准确提供物理位置, 而且标记密度高, 遗传分析和QTL定位更加准确, 可用于QTL的精细定位。近年来研究者利用高密度Bin图谱, 成功定位了抽穗期、产量等多个复杂性状[9,25-28]。本研究定位使用的Bin遗传图谱包含12,328个Bin标记, 标记间平均物理距离30.26 kb, 标记整体分布达到精细作图的要求, 可直接从定位区间筛选候选基因。应用本群体已经成功定位了粒型、抽穗期剑叶叶绿素含量等性状的QTL, 并鉴定到了候选基因[21]。在利用传统遗传图谱进行QTL定位时, 当LOD值超过阈值的一段区域内存在多个峰值时, 多个峰值间很难区分开, 往往导致微效位点被遮盖[10]。本研究在位置相邻的区域定位到了控制抽穗期的QTL, 如和分别位于10号染色体3.15~3.20 Mb和3.61~3.71 Mb。董骥驰等利用02428和玉针香的高密度Bin图谱进行水稻抽穗期QTL定位, 也发现定位的抽穗期QTL之间存在这种位置相邻的现象[25]。利用Bin图谱进行QTL检测可将位置临近的QTL有效分离, 测序得到的SNP标记还可以应用于分子育种, 在水稻复杂性状的定位、候选基因鉴定和标记辅助选择方面具有非常大的应用前景。
此外, 比较本研究2年定位结果发现, 在表型贡献率值相近的情况下, QTL的加性效应2018年大于2021年, 推测是不同环境对QTL效应的影响。同时还存在QTL贡献率较小, 而加性效应稍大的现象, 可能是由于位点附近的基因型存在奇异分离, 导致计算加性效应时出现了偏差。
3.2 本研究与已知抽穗期位点/基因的比较
作为重要的农艺性状, 抽穗期一直是水稻遗传和分子生物学研究的热点。根据Gramene (http://en- sembl.gramene.org/Oryza_sativa/Info/Index)网站公布的数据, 现已报道的水稻抽穗期相关基因与数量性状位点已有618个, 这和水稻抽穗期复杂的调控机制有密切的关系[5,16]。
本研究应用籼粳交RIL群体构建高密度Bin遗传图谱, 2年共所检测到15个控制抽穗期的QTL, 通过遗传图谱和物理图谱的对比(http:/www.gramene. org), 发现一些位点和已知抽穗期位点间有着区间的重叠(表4)。如本研究检测到的位于3号染色体21.75~21.81 Mb, 在该区域Moncada等[29]利用Caiapo/野生稻的BC2F2群体在19.41~22.34 Mb检测到抽穗期QTL, LOD值3.91。Lin等[30]利用Nipponbare/Kasalath的BC3F2NIL群体, 在6号染色体检测到控制抽穗期的2个位点分别位于1.64~3.08 Mb和6.82~11.68 Mb, 均覆盖了我们检测到的和。
在2年均能检测到的位点附近, Yu等[32]利用Zhenshan 97/Ming 63的F2:3家系也发现了抽穗期QTL, 位于7号染色体7.23~16.87 Mb, 区间较大, 表型贡献率为19.2%, LOD值为8.1, 覆盖了我们的位点。此外, 利用IR64/Azucena的DH群体、Sasanishiki/Habataki的BCIL群体, 分别在7号染色体4.5 Mb左右区间定位到了抽穗期QTL, 和本研究定位到的位置重合。
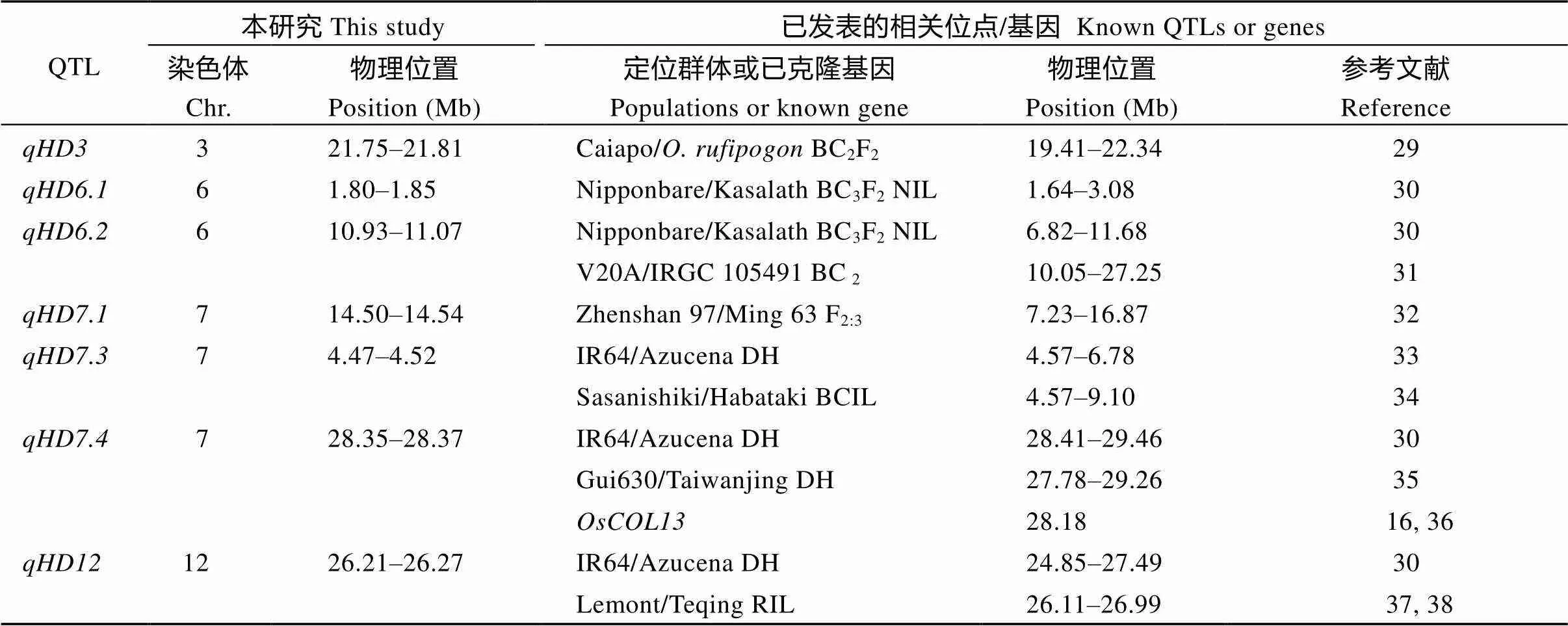
表4 本研究定位的QTL和已知抽穗期相关位点和基因的位置比较
在所在区域, 除了Li等利用IR64/ Azucena的DH群体定位到抽穗期QTL外, Zhou等[35]利用Gui 630/Taiwanjing的DH群体也定位到了, 加性效应值为4.53。在所在区域, 除了Li等利用IR64/Azucena的DH群体定位到了相应的抽穗期QTL, Mei等[37-38]利用Lemont/Teqing的DH群体定位到了一个抽穗期QTL, 加性效应值为-2.4, LOD值为4.14。Sheng等[36]分离的调控抽穗期基因位于7号染色体28.18 Mb, 临近编码一个转录激活因子, 通过抑制的表达抑制成花基因和在长日照和短日照条件下均负调控水稻开花。从本研究定位结果与已知抽穗期QTL的比较可以发现, 存在染色体区域重叠的位点来自于Caiapo/野生稻的BC2F2群体、IR64/Azucena的DH群体、Nipponbare/Kasalath的BC3F2NIL群体、Zhenshan 97/Ming 63的F2:3家系、V20A/IRGC 105491的BC2群体、Sasanishiki/ Habataki BCIL群体、Gui630/Taiwanjing的DH群体和Lemont/Teqing的RIL群体。分析这些群体的亲本可以发现, Caiapo/野生稻的BC2F2群体、V20A /IRGC 105491 BC2群体中的父本IRGC 105491为野生稻, Azucena是陆稻品种[39]。明恢63由IR30和圭630 (Gui630)杂交育成, IR30是国际水稻研究所选育的籼型常规水稻, 也具有野生稻血统[40]。Lemont是来源于美国的热带常规粳稻。本研究所用的亲本之一Kasalath是起源于印度的籼型常规水稻, 亚种类型为AUS。亲本类型都较为古老可能是本研究和其他已知抽穗期QTL存在位置重叠的原因。
4 结论
本研究检测到15个控制水稻抽穗期的QTL。对其中4个2年重复检测到的QTL区间内基因功能注释查询、序列比较和不同等位型效应分析, 进一步明确了4个可能的抽穗期候选基因,、、和, 分别编码水稻甲硫氨酸氨基肽酶、半乳糖基转移酶、细胞色素P450羟化酶和依赖钙的蛋白激酶。候选基因的具体生物学功能有待于进一步研究。
[1] 郭梁, 张振华, 庄杰云. 水稻抽穗期QTL及其与产量性状遗传控制的关系. 中国水稻科学, 2012, 26: 235–245.
Guo L, Zhang Z H, Zhuang J Y. Quantitative trait loci for heading date and their relationship with the genetic control of yield traits in rice ()., 2012, 26: 235–245 (in Chinese with English abstract).
[2] Zhang J, Zhou X, Yan W, Zhang Z Y, Lu L, Han Z M, Zhao H, Liu H Y, Song P, Hu Y, Shen G J He Q, Guo S B, Gao S P, Wang G W, Xing Y Z. Combinations of the,andgenes largely define the eco-geographical adaptation and yield potential ofcultivated rice., 2015,208: 1056–1066.
[3] 杨德卫, 陈壬杰, 程朝平, 郑向华, 叶宁, 叶新福, 黄凤凰. 水稻抽穗期基因的鉴定与遗传调控网络研究进展. 分子植物育种, 2019, 17: 4656–4660.
Yang D W, Chen R J, Cheng C P, Zheng X H, Ye N, Ye X F, Huang F H. The progress of gene identification and genetic regulation mechanism for heading date in rice (L.)., 2019, 17: 4656–4660 (in Chinese with English abstract).
[4] 胡时开, 苏岩, 叶卫军,郭龙彪. 水稻抽穗期遗传与分子调控机制研究进展. 中国水稻科学, 2012, 26: 373–382.
Hu S K, Su Y, Ye W J, Guo L B. Advances in genetic analysis and molecular regulation mechanism of heading date in rice (L.)., 2012, 26: 373–382 (in Chinese with English abstract).
[5] Wei H, Wang X L, Xu H, Wang L. Molecular basis of heading date control in rice., 2020, 1: 219–232.
[6] Yang D W, Cheng C P, Zheng X H, Ye X F, Ye N, Huang F H. Identification and fine mapping of a major QTL,, that plays pleiotropic roles in regulating the heading date in rice., 2020, 40: 1–12.
[7] 蒋丹, 洪广成, 陈倩, 刘石锋, 秦小健. 水稻抽穗期分子调控研究进展. 分子植物育种, 2019, 17: 7071–7077.
Jiang D, Hong G C, Chen Q, Liu S F, Qin X J. Research progress in molecular regulation of heading date in rice ()., 2019, 17: 7071–7077 (in Chinese with English abstract).
[8] Xu Z P, Chen Z A, Wang R X, Miao Y X, Gao H L, Tang S Z, Zhang H G, Liu Q Q. Characterization and fine-mapping of, a minor quantitative locus that affects heading date under long-day conditions in rice (L.)., 2020, 40: 521–532.
[9] 龚晓平, 杨正林, 赵芳明, 钟秉强, 凌英华, 何光华. 一个水稻抽穗期主基因()的遗传分析及分子定位. 作物学报, 2007, 33: 1906–1909.
Gong X P, Yang Z L, Zhao F M, Zhong B Q, Ling Y H, He G H. Genetic analysis and molecular mapping of a dominant heading period gene()., 2007, 33: 1906–1909 (in Chinese with English abstract).
[10] 王玉博, 王悦, 刘雄, 唐文帮. 水稻光周期调控开花的研究进展. 中国水稻科学, 2021, 35: 207–224.
Wang Y B, Wang Y, Liu X, Tang W B. Research progress of photoperiod regulation in rice flowering., 2021, 35: 207–224 (in Chinese with English abstract).
[11] Yano M, Katayose Y, Ashikari M, Yamanouchi U, Monna L, Fuse T, Baba T, Yamamoto K, Umehara Y, Nagamura Y, Sasakia T., a major photoperiod sensitivity quantitative trait locus in rice, is closely related to theflowering time gene., 2000, 12: 2473–2484.
[12] Doi K, Izawa T, Fuse T, Yamanouchi U, Kubo T, Shimatani Z, Yano M, Yoshimura A., a B-type response regulator in rice, confers short-day promotion of flowering and controls FT-like gene expression independently of., 2004, 18: 926–936.
[13] Fujino K. Days to heading, controlled by the heading date genes,and, limits rice yield-related traits in Hokkaido, Japan.2020, 70: 277–282.
[14] Zhang B, Liu H Y, Qi F X, Zhang Z Y, Li Q P, Han Z M, Xing Y Z. Genetic interactions among,,andcontribute to large variation in heading date in rice., 2019, 12: 48.
[15] Zong W, Ren D, Huang M H, Sun K L, Feng J L, Zhao J, Xiao D D, Xie W B, Liu S Q, Zhang H, Qiu R, Tang W J, Yang R Q, Chen H Y, Xie X R, Chen L T, Liu Y G, Guo J X. Strong photo period sensitivity is controlled by cooperation and competition among,andin rice heading., 2021, 229: 1635–1649.
[16] Zhou S R, Zhu S S, Cui S, Hou H G, Wu H Q, Hao B Y, Cai L, Xu Z, Liu L L, Jiang L, Wang H Y, Wan J M. Transcriptional and post-transcriptional regulation of heading date in rice., 2021, 230: 943–956.
[17] 唐立群, 肖层林, 王伟平. SNP分子标记的研究及其应用进展. 中国农学通报, 2012, 28(12): 154–158.
Tang L Q, Xiao C L, Wang W P. Research and application progress of SNP markers., 2012, 28(12): 154–158 (in Chinese with English abstract).
[18] He Q, Zhi H, Tang S, Xing L, Wang S Y, Wang H G, Zhang A Y, Li Y H, Gao M, Zhang H J, Chen G Q, Dai S T, Li J X, Yang J J, Liu H F, Zhang W, Jia Y C, Li S J, Liu J R, Qiao Z J, Guo E H, Jia G Q, Liu J, Diao X M. QTL mapping for foxtail millet plant height in multi-environment using an ultra-high density bin map., 2021, 134: 557–572.
[19] Han Z M, Hu G, Liu H, Liang F M, Yang L, Zhao H, Zhang Q H, Li Z X, Zhang Q F, Xing Y Z. Bin-based genome wide association analyses improve power and resolution in QTL mapping and identify favorable alleles from multiple parents in a four-way MAGIC rice population., 2020, 133: 59–71.
[20] 魏祥进, 徐俊锋, 江玲, 王洪俊, 周振玲, 翟虎渠, 万建民. 我国水稻主栽品种抽穗期多样性的遗传分析. 作物学报, 2012, 38: 10–22.
Wei X J, Xu J F, Jiang L, Wang H J, Zhou Z L, Zhai H Q, Wan J M. Genetic analysis for the diversity of heading date of cultivated rice in China., 2012, 38: 10–22 (in Chinese with English abstract).
[21] 张亚东, 梁文化, 赫磊, 赵春芳, 朱镇, 陈涛, 赵庆勇, 赵凌, 姚姝, 周丽慧, 路凯, 王才林. 水稻RIL群体高密度遗传图谱构建及粒型QTL定位. 中国农业科学, 2021, 54: 5163–5176.
Zhang Y D, Liang W H, He L, Zhao C F, Zhu Z, Chen T, Zhao Q Y, Zhao L, Yao S, Zhou L H, Lu K, Wang C L. Construction of high-density genetic map and QTL analysis of grain shape in rice RIL population., 2021, 54: 5163–5176 (in Chinese with English abstract).
[22] Meng L, Li H H, Zhang L Y, Wang J K. QTL IciMapping: integrated software for genetic linkage map construction and quantitative trait locus mapping in bi-parental populations., 2015, 3: 269–283.
[23] 王建康. 数量性状基因的完备区间作图方法. 作物学报, 2009, 35: 239–245.
Wang j k. Inclusive composite interval mapping of quantitative trait genes., 2009, 35: 239–245 (in Chinese with English abstract).
[24] McCouch S R, Cho Y G, Yano M, Paul E, Blinstrub M, Morishima H. Report on QTL nomenclature., 1997, 14: 11–13.
[25] 董骥驰, 杨靖, 郭涛, 陈立凯, 陈志强, 王慧. 基于高密度Bin图谱的水稻抽穗期QTL定位. 作物学报, 2018, 44: 938–946.
Dong J C, Yang J, Guo T, Chen L K, Chen Z Q, Wang H. QTL mapping for heading date in rice using high-density Bin map., 2018, 44: 938–946 (in Chinese with English abstract).
[26] 李冬秀, 杨靖, 孙凯, 李丹丹, 杨瑰丽, 郭涛, 王慧, 陈志强. 基于高密度遗传图谱定位新的水稻抽穗期QTLs. 西北农林科技大学学报, 2020, 48(8): 44–49.
Li D X, Yang Q, Sun K, Li D D, Yang G L, Guo T, Wang H, Chen Z Q. Mapping new rice heading date QTLs based on high-density genetic map.(Nat Sci Edn), 2020, 48(8): 44–49 (in Chinese with English abstract).
[27] Zhang M, Zhou Z P, Chen Y Y, Cao Y R, Deng C W, Xue P, Zhan X D, Cheng S H, Cao L Y, Zhang Y X. Finding new addictive QTL for yield traits based on a high-density genetic map in hybrid rice., 2021, 93: 105–115.
[28] Li X K, Wu L, Wang J H, Sun J, Xia X H, Geng X, Wang X H, Xu Z J, Xu Q. Genome sequencing of rice subspecies and genetic analysis of recombinant lines reveals regional yield- and quality-associated loci., 2018, 16: 102.
[29] Moncada P, Martinez C P, Borrero J, Chatel M, Gauch H, Guimaraes E, Tohme J, McCouch S R. Quantitative trait loci for yield and yield components in an×BC2F2population evaluated in an upland environment., 2001, 102: 41–42.
[30] Lin H X, Yamamoto T, Sasaki T, Yano M. Characterization and detection of epistatic interactions of 3 QTLs,,, and, controlling heading date in rice using nearly isogenic lines., 2000, 101: 1021–1028.
[31] Xiao J H, Li J, Grandillo S, Ahn S N, Yuan L, Tanksley S D, McCouch S R. Identification of trait improving quantitative trait loci alleles from a wild rice relative,., 1998, 150: 899–909.
[32] Yu S B, Li J X, Xu C G, Tan Y F, Li X H, Zhang Q. Identification of quantitative trait loci and epistatic interactions for plant height and heading date in rice., 2002, 104: 619–625.
[33] Li Z K, Yu S B, Lafitte H R, Huang N, Courtois B, Hittalmani S, Vijayakumar C H, Liu G F, Wang G C, Shashidhar H E, Zhuang J Y, Zheng K L, Singh V P, Sidhu J S, Srivantaneeyakul S, Khush G S. QTL × environment interactions in rice: I. Heading date and plant height.2003, 108: 141–153.
[34] Nagata K, Shimizu H, Terao T. Quantitative trait loci for nonstructural carbohydrate accumulation in leaf sheaths and culms of rice (L.) and their effects on grain filling., 2002, 52: 275–283.
[35] Zhou Y, Li W, Wu W, Chen Q, Mao D, Worland A J. Genetic dissection of heading time and its components in rice., 2001, 102: 1236–1242.
[36] Sheng P K, Wu F Q, Tan J J, Zhang H, Ma W W, Chen L P, Wang J, Wang J, Zhu S S, Guo X P, Wang J L, Zhang X, Cheng Z J, Bao Y Q, Wu C Y, Liu X M, Wan J M. A CONSTANS-like transcriptional activator,, functions as a negative regulator of flowering down stream ofand upstream ofin rice., 2016,92: 209–222
[37] Mei H W, Luo L J, Ying C S, Wang Y P, Yu X Q, Guo L B, Paterson A H, Li Z K. Gene actions of QTLs affecting several agronomic traits resolved in a recombinant inbred rice population and two testcross populations., 2003, 107: 89–101.
[38] Mei H W, Li Z K, Shu Q Y, Guo L B, Wang Y P, Yu X Q, Ying C S, Luo L J. Gene actions of QTLs affecting several agronomic traits resolved in a recombinant inbred rice population and two backcross populations., 2005, 110: 649–659.
[39] Abdirad S, Majd A, Irian S, Hadidi N, Salekdeh G. H Differential adaptation strategies to different levels of soil water deficit in two upland and lowland genotypes of rice: a physiological and metabolic approach., 2020, 100: 1458–1469.
[40] Weng Q M, Wu W R, Li W M, Liu H Q, Tang D Z, Zhou Y C, Zhang Q F. Construction of an RFLP linkage map of rice using DNA probes from two different sources., 2000, 29: 129–133.
Mapping of QTLs for heading date of rice with high-density bin genetic map
ZHAO Ling, LIANG Wen-Hua, ZHAO Chun-Fang, WEI Xiao-Dong, ZHOU Li-Hui, YAO Shu, WANG Cai-Lin, and ZHANG Ya-Dong*
Institute of Food Crops, Jiangsu Academy of Agricultural Sciences / Nanjing Branch of China National Center for Rice Improvement / East China Branch of National Center of Technology Innovation for Saline-Alkali Tolerant Rice / Jiangsu High Quality Rice Research & Development Center, Nanjing 210014, Jiangsu, China
Identification of new loci and genes related to heading date is very important for the genetic mechanism research and molecular improvement in rice.A recombinant inbred lines (RILs) was developed by crossing therice TD70 and therice Kasalath with obvious difference in heading date. A high-density genetic linkage map with 12,328 recombination Bin markers was constructed based on the re-sequencing data of parents and RILs. The RILs and two parents were planted at the Jiangsu Academy of Agricultural Sciences, in Nanjing in 2018 and 2021. QTLs that controlled the heading date were analyzed by IciMappingv3.4 software with inclusive compound interval mapping method. 15 QTLs related to heading date of rice were detected, distributed on chromosome 3, 6, 7, 8, 10, and 12 in two years. The phenotype variation explained (PVE) and LOD value of single QTL ranged from 3.29%–14.73% and 2.58–10.68, respectively. Among them, seven QTLs were found to locate in the same interval or adjacent to previously QTLs, and four QTLswere detectedin twoyears indicating their genetic stabilityAccording to the annotation and sequences analysis of genes located in the region of repeatable QTLswe found thatseven annotated genes had non-synonymous mutations in the coding regions between TD70 and Kasalath. Based on the mutations in the coding regions, the haplotypes of seven genes were identified in RIL population. The heading date of RILs had significant difference between the RILs with different haplotype of four genes, indicating that they might be the candidate genes for heading date. These results could be useful for subsequent functional studies and molecular marker assisted breeding of heading date.
rice (L.); recombinant inbred lines; high-density bin map; heading date; QTLs
10.3724/SP.J.1006.2023.12089
本研究由江苏省重点研发计划项目(BE2019375)资助。
This study was supported by the Jiangsu Science and Technology Development Program (BE2019375).
通信作者(Corresponding author):张亚东, E-mail: zhangyd@jaas.ac.cn
E-mail: zhaoling@jaas.ac.cn
2021-12-28;
2022-05-05;
2022-05-13.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220512.1444.004.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).
