SREBP1c缺失对小鼠肠道菌群的影响
2022-12-19刘冰瑶郑宏庭廖晓玉
刘冰瑶,杨 航,郑宏庭,廖晓玉
400037 重庆,陆军军医大学(第三军医大学)第二附属医院内分泌科
成人肠道中有超过500种微生物,参与了机体营养物质的消化与吸收,与肠黏膜一起构成的肠道生物屏障能阻止病原体的入侵[1];还参与调节免疫、炎症、代谢等[2-3],与人体的健康密切相关。诸多研究表明肠道菌群的异常变化与各种疾病的发生发展有关,如免疫性疾病[4]、炎症性肠道疾病[5]、代谢性疾病[6-8]等。
在代谢性疾病中,肠道菌群参与脂代谢、糖代谢、胰岛素信号通路等的调控,肠道菌群自身也受到多种因素的调节,包括膳食、环境、遗传因素等[9-11]。固醇调节元件结合蛋白(sterol regulatory element binding proteins, SREBPs)是调控脂质代谢相关基因表达的关键性核转录因子,包括三种SREBP亚型,在脂质合成中扮演不同的角色。固醇调节元件结合蛋白1c(sterol regulatory element binding protein 1c, SREBP1c)是肝脏脂合成代谢的关键调控者,转录调控脂肪酸和甘油三酯合成过程的关键酶,在机体肥胖、胰岛素抵抗中发挥着重要作用[12-14]。有研究发现,无菌小鼠在接受常规喂养小鼠的粪菌移植后,体脂含量和胰岛素抵抗会增加60 %,菌群促进了肠道对单糖的吸收,上调肝内脂合成调控基因SREBP1c的表达,增加甘油三酯合成[15]。然而,SREBP1c对小鼠肠道菌群结构的影响并不清楚。本研究通过采集SREBP1c基因缺失小鼠和野生型小鼠的粪便,以及16S rDNA测序,分析SREBP1c基因缺失对小鼠肠道菌群结构的影响。
1 材料与方法
1.1 实验小鼠与鉴定
SREBP1c杂合缺失型小鼠(SREBP1c+/-)受赠于重庆医科大学第二附属医院杨刚毅教授,包括1只雄鼠,20周龄,体重31.4 g;1只雌鼠,20周龄,体重29.7 g。C57BL/6小鼠购买于江苏集萃药康生物科技有限公司,共2雄,2雌,5~6周龄,体重16.0~18.4 g。小鼠饲养于SPF级动物房,动物房温度25 ℃,湿度40%~60%,12 h昼夜更替。SREBP1c+/-小鼠通过杂交后获得SREBP1c纯合敲除型(SREBP1c-/-, SREBP1c-KO)和野生型(SREBP1c+/+, SREBP1c-WT)。
小鼠基因鉴定:采用鼠尾直接PCR试剂盒(Bimake,Cat:B40015)快速提取小鼠基因组DNA、PCR扩增,再通过凝胶电泳鉴定小鼠基因型。鉴定引物:SR1: 5′-ATCGGCGCGGAAGCTGGGGTAGCGTCT-3′; SR2: 5′-TCTCCAGATTTATGCAGGTCATAAATAGTAC-3′; SR3: 5′-GCTCAGTTCGAGGTGCTGTTTCTG-3′。PCR反应体系:DNA 1 μL,引物共1 μL,2X M-PCR混合液10 μL,H2O 8 μL,共20 μL;PCR反应程序:95 ℃预变性5 min,95 ℃变性30 s,60 ℃退火30 s,72 ℃延伸30 s,35个循环数。
1.2 小鼠粪便与组织样本收集
实验小鼠分为2组:野生型组(WT)和敲除组(KO),每组各5只雄鼠,均为6周龄。两组小鼠给予常规饮食,无特殊处理,记录小鼠体重变化与饮食情况。小鼠12周龄时,在超净工作台上,使用1.5 mL无菌EP管收集WT组和KO组小鼠粪便,收集后立即于液氮中速冻,再冻存于-80 ℃冰箱。小鼠粪便收集完成后,禁食过夜,安乐死后收集小鼠肝脏及肠组织,于液氮中速冻后再冻存于-80 ℃冰箱,用于后续实验。本研究经陆军军医大学实验动物福利伦理审理委员会审核,符合动物伦理和动物福利要求(批件号:AMUWEC2020194)。
1.3 荧光定量PCR
组织RNA提取与基因表达检测:采用RNAiso Plus试剂(Takara, Cat: 9109)提取收集的小鼠肝脏组织RNA,通过NanoDrop2000(Thermo Fisher Scientific,USA)检测RNA浓度,取1 μg RNA 逆转录为cDNA(PrimeScriptTMRT reagent Kit,Cat: RR047A),采用荧光定量PCR(TB GreenR Premix Ex Taq, Cat: RR820A)检测基因表达量。PCR反应程序:95 ℃预变性3 min,95 ℃变性10 s,57 ℃退火30 s,72 ℃延伸15 s,40个循环数。基因检测的引物序列见表1。

表1 引物序列
1.4 肝脏内甘油三酯(triglyceride, TG)、总胆固醇(total cholesterol, TC)含量检测
甘油三酯和总胆固醇检测采用南京建成生物工程研究所试剂盒(Cat: A110-1-1, A111-1-1)。首先采用RIPA裂解液(上海碧云天生物技术有限公司,Cat: P0013B)裂解收集的小鼠肝脏组织,于12 000 r/min离心20 min;取2.5 μL上清加到250 μL检测液中,混匀,37 ℃孵育10 min,用酶标仪(510 nm)检测各孔吸光值。蛋白浓度测定采用BCA法试剂盒(上海碧云天生物技术有限公司,Cat: P0012)。
1.5 16S rDNA测序
小鼠粪便样本的16S rDNA测序由上海美吉生物医药科技有限公司完成。具体操作步骤如下:
基因组DNA提取、扩增:DNA粪便菌群基因组DNA提取采用土壤DNA提取试剂盒 (Omega Bio-tek, USA)。DNA浓度通过NanoDrop 2000测定,并利用1%琼脂糖凝胶电泳检测样本基因组DNA质量。针对细菌16S rDNA基因的V3-V4高变区设计合成 引物:341F(5′-ACTCCTACGGGRSGCAGCAG-3′),806R(5′-GGACTACVVGGGTATCTAATC-3′),进行PCR(GeneAmp 9700, ABI, USA)扩增。PCR反应体系:4 μL 5×FastPfu缓冲液、2 μL 2.5 mmol/L dNTPs、上下游引物(5 μmol/L)各0.8 μL、0.4 μL FastPfu DNA聚合酶(TransGen, China),和10 ng基因组DNA,共20 μL反应体系。PCR反应使用以下程序:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,27个循环;最后再72 ℃延伸10 min。每个样本按照以上程序进行3个重复,同一样本的PCR产物混合后通过2%琼脂糖凝胶电泳检测,并使用AxyPrep DNA凝胶回收试剂盒(Axygen, USA)回收、纯化PCR产物;最后采用QuantiFluorTM-ST蓝色荧光定量系统(Promega, USA)对纯化的PCR产物进行检测定量,之后将每份扩增产物等量混合。
文库构建、测序:通过PCR将Illumina接头序列添加至目标区域外端(TruSeqTM DNA Sample Prep Kit, Illumina, USA);使用凝胶回收试剂盒切胶回收PCR产物,2 %琼脂糖电泳检测;采用氢氧化钠处理纯化的PCR产物,变性产生单链DNA片段,最后测序工作在Illumina MiSeq平台完成。
1.6 测序数据分析
Miseq测序得到的原始数据首先根据序列的重叠关系进行拼接,同时对序列质量进行质控和过滤。根据标签区分样本后,基于分类操作单元(operational taxonomic unit, OTU)对样本进行物种分类学分析、多样性指数分析、聚类分析、主成分分析(principal component analysis, PCA)等;基于分类学信息,在各个分类水平上,如门水平、科水平、属水平等,进行小鼠粪便样本中细菌群落结构的统计分析;最后在上述分析的基础上,对SREBP1c野生型和纯合敲除型两组小鼠粪便样本的群落组成进行差异显著性检验(Student’st检验)。
1.7 统计学分析
2 结果
2.1 SREBP1c敲除对小鼠体重的影响
如图1A、B所示,SREBP1c+/-小鼠通过配对繁殖获得SREBP1c纯合敲除型和野生型,即SREBP1c-/-(SREBP1c-KO)和SREBP1c+/+(SREBP1c-WT)。在常规饮食喂养下,SREBP1c-KO小鼠的体重与野生型小鼠无明显差异(图1C)。SREBP1c是通过调节脂肪代谢相关酶的基因表达影响体内脂肪合成,检测也发现SREBP1c敲除后,小鼠肝内脂肪合成关键酶的编码基因脂肪酸合成酶(Fatty acid synthase, FASN),硬脂酰辅酶A去饱和酶(Stearoyl-CoA desaturase, SCD1)表达显著降低(P<0.05),相应的肝脏中TG含量明显减少(P<0.05),TC含量无明显变化(图1D、E)。
2.2 SREBP1c基因敲除对小鼠肠道菌群的影响
两组小鼠共测序10个样本,平均测序序列数58 432.5,平均长度441 bp。以抽取的序列数及其对应的多样性指数(Shannon指数)构建Shannon-Wiener曲线。从曲线图可以看出,随着测序数据量的增加,曲线趋于平坦;当数据量大于5 000序列数时,Shannon指数达到峰值,说明样本测序数据量足够大,可以反映样本中绝大多数的微生物信息(图2A)。

A: 小鼠鉴定电泳图 M:DNA marker;1~2:SREBP1c-WT小鼠基因组DNA扩增产物电泳条带;3~6:SREBP1c-KO小鼠基因组DNA扩增产物电泳条带;B: SREBP1c-WT和SREBP1c-KO小鼠肝组织中SREBP1c mRNA相对表达水平;C: SREBP1c-WT和SREBP1c-KO小鼠体重比较;D: 两组小鼠肝组织中脂合成代谢酶编码基因的表达水平检测;E: 两组小鼠肝脏TG, TC含量检测 WT:野生型组;KO:敲除组;a:P<0.05,与WT组比较,Student’s t检验

A: 样本Shannon-Wiener曲线;B: 两组小鼠肠道细菌在OUT水平的维恩图分析 WT:野生型组;KO:敲除组
基于OUT水平,采用维恩图分析两组小鼠肠道菌群OUT数量及种类的相似性,如图2B所示,SREBP1c-WT和SREBP1c-KO小鼠总OUT分别为293和290个,特异性OTU分别为33和30个,共有OUT为260个,占总OUT数的80.5 %。α多样性分析发现,SREBP1c-KO组Shannon指数有所降低,Sobs指数显著降低(P<0.01),说明SREBP1c-KO小鼠的肠道菌群多样性显著低于野生型小鼠(图3)。

WT:野生型组;KO:敲除组;a:P<0.01,与WT组比较, Student’s t检验
进一步地,基于OUT水平的主成分分析也显示,两组小鼠肠道菌群组成有所不同(图4)。因此,我们在不同分类水平上对两组小鼠的物种组成差异进行了进一步的分析。在门水平,SREBP1c-KO小鼠肠道中厚壁菌门增加5.23 %,拟杆菌门减少5.34 % (图5A),厚壁菌门与拟杆菌门比值在SREBP1c-KO小鼠肠道中有上升趋势,但没有统计学差异(图5B)。
在科水平,Helicobacteraceae,Family_XIII,Clostridiaceae_1的相对丰度在SREBP1c-KO小鼠肠道中均显著降低(图5C);其中Family_XIII和Clostridiaceae_1均属于拟杆菌门,其差异与两组小鼠在门水平的组成差异相符。在属水平,相较于野生型小鼠,SREBP1c-KO小鼠肠道的Rikenella, Ruminiclostridium_9, Helicobacter, (Eubacterium)_ xylanophilum_group, Candidatus_ Arthromitus和(Eubacterium)_ brachy_group相对丰度均显著减少(图5D,P<0.05)。其中Rikenella(理研菌属)属于拟杆菌门理研菌科(Rikenellaceae),有研究报道发现Rikenellaceae与炎症反应[16-17]、高脂饮食[18]、内脏脂肪沉积相关[19]。
2.3 SREBP1c敲除小鼠的肠道屏障减弱可能与肠道菌群变化有关
有研究发现,岩藻多糖可增加小鼠肠道中理研菌属、粪球菌属等短链脂肪酸产生菌,降低炎症因子表达,增加紧密连接蛋白Occludin表达,改善小鼠肠道黏膜损伤[20]。那么,SREBP1c-KO小鼠肠道内理研菌属的降低,是否会影响小鼠肠道屏障。我们采用荧光定量PCR检测了两组小鼠肠道中抗菌肽Reg3β和Reg3γ,与紧密连接蛋白Occludin和ZO-1的表达水平。如图6所示,与野生型小鼠相比,SREBP1c-KO小鼠肠道内Reg3γ和Occludin表达水平均显著降低(图6,P<0.05),Reg3β和ZO-1表达也有所下降。结果表明,SREBP1c基因敲除引致的小鼠肠道屏障减弱,可能与小鼠肠道中理研菌属的丰度减少有关。

A: 在OUT水平,采用PCA法对两组小鼠肠道菌组成进行聚类分析;B: 两组小鼠肠道菌群PCA分析中的主成分PC1 WT:野生型组;KO:敲除组

WT:野生型组;KO:敲除组;a:P<0.05,与WT组比较,Student’s t检验
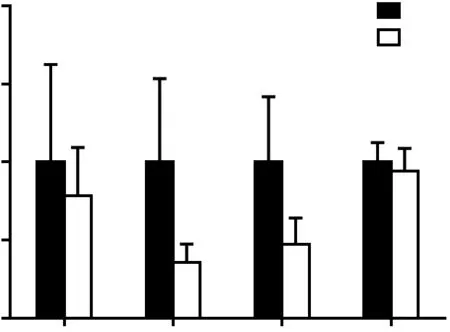
WT:野生型组;KO:敲除组;a:P<0.05,与WT组比较,Student’s t检验
3 讨论
人体肠道内寄生的细菌数目有40万亿,其基因总数约为人自身基因数目的150倍,因此肠道菌群也被称为人体的“第二基因组”。肠道菌群可以消化食物成分、合成必需维生素、刺激和调节免疫系统、排除病原体、支持肠道功能等。在代谢方面,越来越多的证据表明,肠道菌群的异常与糖代谢、脂代谢、肠肽分泌、胰岛素敏感性的紊乱密切相关[21-23]。
目前研究认为,肠道菌群的微生态平衡受饮食、药物、遗传因素等的影响。饮食是影响肠道菌群组成、多样性的关键因子之一,在肠道菌群的定植、成熟及肠微生态系统稳定的维持中都起到非常关键的作用。高脂-饮食的小鼠肠道中细菌α多样性降低,厚壁菌门与拟杆菌门比值增加,双歧杆菌数量减少[24];WU等[25]研究发现长期食用高动物蛋白高脂肪和低碳水化合物的受试者,肠道内拟杆菌属较高,普雷沃菌属水平较低,而低动物蛋白低脂肪高碳水化合物饮食的受试者肠道内细菌正好与之相反。饮食、药物等可能通过影响肠道菌群结构及肠道代谢物的产生,参与调控宿主糖脂代谢。在我们课题组以往的研究中,发现降糖药DPP-4i干预显著改变了高脂喂养小鼠和2型糖尿病患者的肠道菌群结构及肠道代谢物水平;DPP-4i改变的肠道菌群通过粪菌移植给无菌鼠,可显著改善高脂饮食诱导的葡萄糖不耐受[26],提示肠道菌群参与了降糖药DPP-4i改善糖代谢的作用。
除饮食、药物等外在因素,宿主自身遗传因素也对肠道菌群结构组成具有一定影响。近期研究发现髓样分化因子MYD88敲除小鼠肠道及多种组织内约氏乳杆菌的相对丰度显著增加;在体外,约氏乳杆菌主要持续存在于树突细胞中[27],可能与免疫调控有关。本研究首次探索了SREBP1c基因缺失对小鼠肠道菌群组成的影响。通过与野生型小鼠对比,我们发现SREBP1c敲除小鼠肠道菌群的α多样性降低,厚壁菌门增加5.23 %,拟杆菌门减少5.34 %,表明SREBP1c基因缺失在一定程度上影响了小鼠的肠道菌群结构组成。SREBP1c是促进肝脏脂肪生成的关键调控者,其调控的FASN、ACC1、SCD1等在脂肪生成过程中发挥关键作用[28-29]。在该研究中我们也发现,SREBP1c敲除显著下调了肝组织中FASN及SCD1的表达,相应的肝内甘油三酯含量也显著降低,然而SREBP1c表达水平是否通过影响脂肪酸含量从而影响肠道菌群组成值得进一步探究。
随着研究的深入, 肠道菌群和宿主之间的相互作用机理也越来越多被发现。宿主基因表达水平对肠道微生物组成结构可产生一定影响,而肠道菌群及其代谢产物也可调节宿主的基因表达水平、基因表观修饰等,从而参与代谢调控。葛根岑连汤是一种常用于2型糖尿病的中药配方,其关键组分小檗碱可调节肠道菌群组成,富集粪杆菌及丁酸盐产生菌,增加肠道短链脂肪酸水平,同时还下调免疫应答相关基因,上调脂代谢相关基因,缓解系统性及局部炎症[30]。新近研究还发现,肠道菌群还可调控宿主DNA甲基化修饰、组蛋白修饰和RNA甲基化修饰[31-32]。
除肠道菌群整体结构失衡对宿主的影响外,某些重要的单一菌属变化也会影响机体代谢水平和免疫反应。近期研究发现主要分布在肠道黏液层的艾克曼氏菌(Akkermansia muciniphila),可通过分泌黏蛋白保护肠道黏膜屏障,并被发现参与了一线降糖药二甲双胍的降糖机制[24]。本研究在不同分类水平上对两组小鼠肠道菌组成进行了差异分析,发现SREBP1c-KO小鼠肠道中理研菌属丰度显著降低,同时肠道屏障也减弱。理研菌可发酵肠道内未消化的膳食纤维,生成短链脂肪酸,为肠上皮细胞提供能量,使肠腔黏膜层增厚,维护正常的屏障功能,促进肠道健康。那么SREBP1c的缺失导致小鼠肠道菌群结构的变化,是否与肠道屏障的改变直接相关,也值得我们进一步探索。
综上所述,脂代谢关键调控基因SREBP1c缺失降低小鼠肠道菌群多样性,影响小鼠肠道菌群结构组成,可能与肠道屏障减弱有关,该研究结果为深入探讨宿主基因与肠道菌群互作关系提供了实验与理论依据。
