转座子编辑技术与CRISPR编辑技术在乳酸菌定向改良中应用的研究进展
2022-12-15艾连中熊智强杨昳津
杨 勇, 艾连中, 熊智强, 杨昳津, 宋 馨
(上海理工大学 健康科学与工程学院/上海食品微生物工程技术研究中心, 上海 200093)
乳酸菌(lactic acid bacteria,LAB)是一类可发酵碳水化合物产生大量乳酸的细菌统称,是食品工业重要的微生物。除了可用于生产酸奶、奶酪、酪乳和开菲尔等乳制品,发酵蔬菜、谷物和肉类,制作酒精性饮料外,乳酸菌还具备降胆固醇、维持菌群平衡、抑制致病菌、增强免疫等多种益生作用。乳酸菌主要包括乳杆菌属(Lactobacillus,Lb)、双歧杆菌属(Bifidobacterium)、乳球菌属(Lactococcus,Lc)、链球菌属(Streptococcus)和肠球菌属(Enterococcus)等。由于野生型乳酸菌菌株可能具有某些特性,如乳杆菌的益生特性、肠球菌的致病性等,为了充分发挥有益菌的功能潜力或抑制致病菌毒性,通常需要对其进行改良。
目前,乳酸菌改良策略包括传统改造(如随机突变、适应性进化和原生质体融合等)以及基因编辑技术。当下基因编辑工具(基于质粒的同源重组、重组酶介导的单双链DNA重组和CRISPR/Cas9系统等)存在的操作烦琐、脱靶率高、效率低等缺点限制了其在乳酸菌中的广泛使用[1],因此开发应用简便高效的遗传操作工具对于乳酸菌基因功能的探究和某些有害菌致病机制的阐释至关重要。转座子(transposon,Tn)是一类可以在生物基因组内移动和复制的遗传因子, 已作为重要的插入突变剂或分子标签广泛应用于细菌、真菌和其他微生物功能基因组研究中,成为有益微生物功能基因分析和致病微生物毒力基因挖掘的有力工具,如伤寒沙门氏菌(Salmonellatyphi)、霍乱弧菌(Vibriocholerae)、大肠杆菌(Escherichiacoli)等常见致病菌。基于CRISPR转座子编辑系统更可快速实现大片段DNA在细菌基因组中多个位点的高效、定向、无标记编辑整合,为千碱基规模的基因组工程提供了一种多功能的利器。本文以乳酸菌改良为出发点,简要概述了乳酸菌改良技术和应用前景,并着重介绍了基于CRISPR转座子编辑系统构建,以期为后续开发新型乳酸菌和丰富益生乳酸菌功能提供更多理论指导。
1 传统乳酸菌改良技术
野生型乳酸菌菌株可能具有某些遗传特性,比如良好的发酵性和较强的耐受性,但由于外界各种不良环境的胁迫及不利因素的影响,严重限制了乳酸菌的生长及代谢能力,阻碍了遗传功能的发挥,抑制了高效活性的利用。因此,为了充分发挥乳酸菌的潜力,通常需要对其进行特定改造。传统的乳酸菌菌种改良技术包括适应性进化、随机诱变和原生质体融合技术,见表1。

表1 乳酸菌传统改良技术的优缺点
1.1 适应性进化
微生物在长期受到新环境影响后,会逐渐改变自身遗传信息以适应外界环境变化,最终形成新的变异株。在新环境下能快速生长的菌体则成为优势株,而野生型和负突变型由于无法适应新环境最终被淘汰。适应性进化模拟微生物在自然进化过程中发生的随机变异及自然选择作用,通过设定特殊人工环境富集正向突变细胞,最后从变异株中筛选得到期望菌株。酸胁迫、冷冻胁迫、氧胁迫和渗透压胁迫都会影响乳酸菌的生存能力,尤其是双歧杆菌属,它们对外界环境压力的耐受性较差。Zhang等[2]将B.bifidumCCFM16置于实验室高渗透压环境下连续传代1 000代,所得突变体传代时间较原始菌株减少了2/3。Jiang等[3]通过酸胁迫提高了B.longumBBMN68的耐酸性,突变株在pH=2.5条件下,2 h内的耐酸能力有了显著提高。适应性进化在微生物育种进程中发挥了重要作用,存在靶向性强、效果明显、性状稳定的优点,但菌株低频率的自发突变以及进化周期较长等缺点限制了该技术的大范围推广使用。为了提高菌株的突变频率、加快微生物的进化速度,科研人员常在适应性进化过程中加入人工诱变策略。
1.2 随机诱变
随机诱变是一种常用的微生物菌种改良手段,通过诱变剂诱发基因突变,这样可以显著提高变异的频率,筛选到各种类型用于生产或研究的突变株。根据诱变剂的不同可以将随机诱变分为物理诱变和化学诱变。贺晓洁等[4]对筛选到的Lb.plantarumL-14进行二轮紫外诱变,突变株在产酸及耐酸、耐胆盐和耐盐等方面均优于原始菌株,这为高盐发酵剂的工业化生产提供了新途径。高钰淇等[5]分别使用紫外线、60Co-γ射线、硫酸二乙酯对Lb.acidophilusNX2-6进行诱变, 突变株的细菌素合成量最高达6 230.79 U/mL,较原始株提高了2.21倍。Zhao等[6]将紫外线照射与亚硝基胍联合作用于Lb.plantarumC88,使菌株的黏附能力得到显著增强。近年来,我国在常压室温等离子体(atmospheric and room temperature plasma,ARTP)、重离子束等新型随机诱变技术及装备上取得了重要进展,这些新一代随机诱变技术具有相对生物效应高、损伤的修复效应小、突变快、突变体稳定、遗传周期短等优点,被广泛应用于重要微生物菌种的选育。殷娜等[7]采用ARTP对从酸马奶中筛选的Lb.plantarumYL15进行诱变,选育出产酸能力高并且发酵性能稳定的乳酸菌菌株。
随机诱变虽然能够得到突变菌株,但后期筛选的工作量较大、获得的正突变较少,且突变株的稳定性不易控制,极易出现菌株衰退现象。因此,既能减少工作量,又能保证较高正突变率的诱变技术成为今后诱变筛选的研究方向之一。
1.3 原生质体融合
原生质体融合属于杂交育种方法,是人为地使遗传性状不同的两个亲株细胞的原生质体发生融合,并进而发生遗传重组以获得兼具两个亲本优良性状的遗传性稳定的融合子的过程。由于该技术具有重组频率高、重组类型多、能克服随机诱变出现的菌株衰退问题等优点,在乳酸菌菌种改良中得到广泛的应用。姜雄韬等[8]通过原生质体融合技术获得Lb.plantarumZJQ与Lb.plantarumZJ316的原生质体融合子, 成功提高了细菌素合成量。王慕华等[9]将Lb.bulgaricus和具有抗噬菌体能力的嗜热链球菌(S.thermophilus)融合得到的融合子Rh6具有抗噬菌体功能,并且具备优良的酸奶发酵特性。胡钰洁[10]通过将Lb.acidophilus同纳豆芽孢杆菌(Bacillusnatto)融合,得到的融合子发酵纳豆拉丝效果佳且无不良风味。原生质体融合技术获得的产品没有安全隐患,但该技术还存在着如遗传标记的选择、杂种的鉴定、融合菌株性状优化、杂种细胞间的异源基因相互排斥等问题,这些需要进一步的深入研究。
2 转座子编辑技术在乳酸菌改良中的应用
2.1 转座子分类及结构
根据物理结构和转座机制差异,可以将转座子分为两大类:RNA类转座子和DNA类转座子。RNA类转座子又称反转录转座子或逆转录转座子,通过“复制- 粘贴”机制以类似于逆转录病毒生命周期的方式移动:RNA中间体被逆转录成cDNA分子,然后通过逆转座酶将其插入到基因组中[11]。其结构复杂,根据功能划分为不同的结构域,如长末端重复序列、重复区和种属特异性抗原等。DNA类转座子无需RNA中间体介导,结构相对比较简单,通常包括编码转座酶基因、靶位点重复序列和两侧的末端反向重复序列,通过“DNA-DNA”的“剪切- 粘贴”机制移动,DNA转座子从一个位置切除并通过编码的转座酶靶向识别末端重复序列,将DNA转座子重新整合到靶位点[11]。
Tn916家族(Tn925、Tn919和TnF01等)、Tn917、Mu噬菌体、部分插入序列(insertion sequences,IS,如IS946、IS3家族和ISS1等)以及Mariner家族转座子是目前乳酸菌中应用较为广泛的几类转座子,而且都已被改造并成功应用于乳酸菌突变体库的构建及基因功能的研究中[12]。插入序列IS是最先被鉴定出的简单转座子,大多数IS包含单一转座酶编码基因,能够独立完成转座功能。Tn916和Tn917转座子同属一类转座子,这类转座子包含所编码的抗性基因、转座酶基因、解离酶基因、末端重复序列以及解离位点,其转座过程由所携带的转座基因和抗性单元完成。Mu噬菌体的DNA为线性分子, 不含末端重复序列,能随机地插入宿主染色体中,引发变异。上述几类均来源于原核生物,而Mariner转座元件是一种最简单的真核转座子,与其他转座子相比,Mariner转座元件具有插入位点随机性高,以及与品种特定的寄主因素无关、可以在不同品种间水平转移的特点。Mariner转座元件两端为30 bp的重复序列,元件内部具有单一阅读框(open reading frame,ORF),用于编码转座酶。转座酶的氮端为具有螺旋- 转角- 螺旋空间结构的DNA结合区域,碳端为3个天冬氨酸残基构成的催化结构域,结合区域与催化区域间存在高度保守的连接区域。上述转座子各自具有不同特点,在乳酸菌突变体的构建与功能研究方面表现出良好的应用前景。
2.2 转座子编辑技术的应用
转座子编辑技术即利用转座子特有的转座功能,对生物基因组进行基因突变并研究基因功能,如常利用转座子构建菌株突变体文库,其技术特征是所有转座子都携带转座所必需的基因,因此它并不依赖于转座子与靶点间的序列同源性。因转座子插入阻断了转录或翻译过程,导致靶点基因失活,而且插入往往表现出极性效应,即转座子在操纵子上游基因的插入影响到下游基因的表达,原因是转座子序列中含有终止子或终止密码子,造成转录或翻译的终止,因此影响后续基因的表达,进而依靠表型鉴定出突变体[13]。
Tn916家族是一类应用较多的转座子,已被证明能够在多种乳酸菌中进行转移[14](见表2)。Devirgiliis等[15]研究表明来自于Lb.paracasei的转座子Tn916转移至粪肠球菌(E.faecalis)JH2-2的频率极低(1×10-8),并且插入位点并不是文献报道的高频特征序列。由此可见,Tn916作为诱变转座子仍存在两个问题:转移和整合到各种乳酸菌的频率相对较低,无法构建庞大的突变体文库以供筛选;此插入位点存在偏好性也影响了突变文库的构建。Tn917在粪肠球菌中普遍存在,经常被用于乳球菌属、链球菌属、乳杆菌属和肠球菌属的随机插入突变,以挖掘表型相对应的未知基因。此外,Tn917的各种衍生物都具有无启动子的lacZ基因,从而能够与插入位点两侧的基因发生转录融合[16]。Israelsen等[17]构建的Tn917-lacZ衍生物在Lc.lactisMG1363中形成了一个随机插入的lacZ融合文库(包含2 500个整合子),并从该文库中鉴定出一系列受调控的启动子。Jones等[18]基于转座子标签诱变技术鉴定了新生鼠败血症感染模型中无乳链球菌(S.agalactiae)生长和存活所需的基因,通过对Tn7诱变筛选出大约1 600个S.agalactiae突变体,其中鉴定的120个突变体能在模型体内存活。同源插入序列(IS)也被用于乳酸菌突变体库构建。李晨等[19]利用载体pGhost9: ISS1,经复制型转座构建了Lb.delbrueckiisubsp.bulgaricusLb-MH的突变体,得到3 株具有抗后酸化能力的突变株LA-1、LA-2、LA-3。其他转座子如Mariner家族中的Himar1转座元件也已被成功地应用于肠球菌属和链球菌属[20]。由此可见,转座子诱变技术需要DNA链断裂,安全性低,且并不适用于广泛的宿主特性。

表2 转座子编辑技术在乳酸菌中的应用
3 CRISPR编辑技术在乳酸菌改良中的应用
在过去的30年里,各种基因编辑组件的开发,如基于质粒的同源重组、外源重组酶介导的单双链DNA重组等系统,使得乳酸菌的遗传操作工具得到了极大改进。但上述技术存在关键的问题需要解决,包括周期长、效率低、筛选标记不足、容易造成回复突变、无法实现基因组的无痕敲除等(表3)。成簇规律间隔短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)系统开辟了基因组编辑研究的新篇章。目前已在多种微生物中得到广泛应用,如大肠杆菌、黏细菌(Myxobacteria)、金黄色葡萄球菌(Staphylococcusaureus)、芽孢杆菌(Bacillus)、乳杆菌、嗜热链球菌和乳酸乳球菌等。CRISPR/Cas9系统由Cas蛋白、反式激活CRISPR RNA(trans-activating CRISPR RNA, tracrRNA)和CRISPR RNA阵列(crRNA,包含2个重复区和1个间隔区)组成,为简化用于目的基因的编辑,将crRNA和tracrRNA融合,形成单一的sgRNA(synthetic guide RNA)。Cas蛋白与sgRNA结合并识别基因组的原型间隔序列毗邻基序(protospacer adjacent motif,PAM)位点(-NGG),产生双链断裂,双链断裂后会激发非同源末端连接或同源定向修复机制进行修复。

表3 可用于乳酸菌基因组编辑的工具
3.1 在乳酸菌基因敲除中的应用
鉴于Cas蛋白可与具有不同位点的sgRNA共同作用,因此该系统可用于多个位点的同时编辑。Oh和van Pijkeren将CRISPR/Cas系统和单链DNA(ssDNA)重组技术相结合,成功对Lb.reuteriATCC6475进行基因编辑[21]。Huang等[22]通过RecE/T辅助修复的Cas9工具,在双质粒系统中成功实现植物乳杆菌和短乳杆菌单基因敲除,且敲除效率达到50%~100%,高效修复双链断裂。Song等[23]使用CRISPR/Cas9D10A在干酪乳杆菌中建立了一种快速且准确的基因编辑方法。利用该系统成功敲除了4个非必需基因,插入了1个绿色荧光蛋白报告基因,基因敲除和插入的效率为25%~62%。
3.2 在乳酸菌基因转录抑制中的应用
除了敲除或插入基因外,CRISPR编辑技术还能在转录水平对目的基因进行调控。基于Cas蛋白的修饰突变,衍生出了CRISPR干扰(CRISPR interference, CRISPRi)系统。将Cas9结构域同时失活构成的dCas9系统中的Cas9与目标基因结合后,失去靶向裂解基因的能力,成为实现基因沉默和转录调控的强大工具。目前已经有研究利用CRISPRi系统对乳酸菌基因转录进行调控的报道。Berlec等[24]通过CRISPRi系统靶向和沉默Lc.lactisNZ9000中的upp基因,使其mRNA的相对转录水平降低了50倍。Xiong等[25]在乳酸乳球菌中也对CRISPRi系统进行了研究,成功地将转录抑制从单基因扩展到多基因,抑制效率高达99%,并同时鉴定了一系列功能基因。
4 基于CRISPR的转座子编辑系统在乳酸菌改良中的应用展望
过去十年,转座子诱变仍然是构建突变体库和筛选关键基因的主要方法,但因其低效而未广泛使用。CRISPR系统虽设计简单、操作方便、效率高、特异性强,但同样依赖DNA断裂和同源重组,且存在严重的脱靶效应。最近,研究人员在对CRISPR系统生物多样性的探索中,发现了CRISPR相关转座酶的存在,并开始将转座酶的强大整合能力与RNA引导的Cas核酸酶的精确靶向结合起来,以实现靶向转座子整合[26-28]。目前的分析研究确定了含有crRNA阵列元件、Cas基因和转座酶特异性基因的CRISPR位点[28]。典型的例子就是CRISPR-Cas系统与Tn7样转座子结合进行基于RNA的靶向转座[29-30]。
4.1 基于CRISPR的转座子编辑系统的构建
在CRISPR系统中,Cas效应蛋白(如Cas9和Cas12)通过引导RNA定位于基因组中特定的DNA序列,诱导DNA双链断裂,然后通过同源重组或非同源末端连接进行修复,但这种类型的编辑效率往往不尽如人意[31]。基于转座子载体的插入效率在一定程度上高于宿主介导的同源重组,但局限于转座元件靶序列特异性较差的缺点。在2019年,Strecker等[27]在蓝细菌Scytonemahofmanni和Anabaenacylindrica中开发了用于DNA插入的CRISPR相关转座酶(CRISPR-associated transposase,CAST)系统(ShCAST和AcCAST)(图1)。该系统包含2个质粒,由单蛋白效应物Cas12k、转座酶复合物和crRNA阵列组成的辅助质粒(pHelper)以及由目的基因和转座子左右臂组成的供体质粒(pDonor)。首先Cas12k特异性识别具有5’-GTN的PAM序列,同时在tracrRNA协助下,crRNA间隔区与靶DNA链互补配对形成DNA-RNA杂交链,随后转座酶复合物识别引导目的基因整合至靶DNA中[32]。ShCAST和AcCAST系统的DNA整合分别位于PAM下游的60~66 bp和49~56 bp处的5 bp重复插入序列。虽然整合原理相同,但来自不同来源的酶的CAST引导的DNA定向整合具有不一致的位置偏好和插入效率,AcCAST系统的DNA插入效率仅为ShCAST系统的30%,且转座效率受插入序列大小和目标位点的影响,脱靶率较高(约50%)。CAST系统规避了对基因组DNA的切割和对DNA的损伤修复系统的利用,直接通过RNA引导的转座酶进行目的DNA的插入,相较于现阶段的遗传编辑技术存在很多优势。
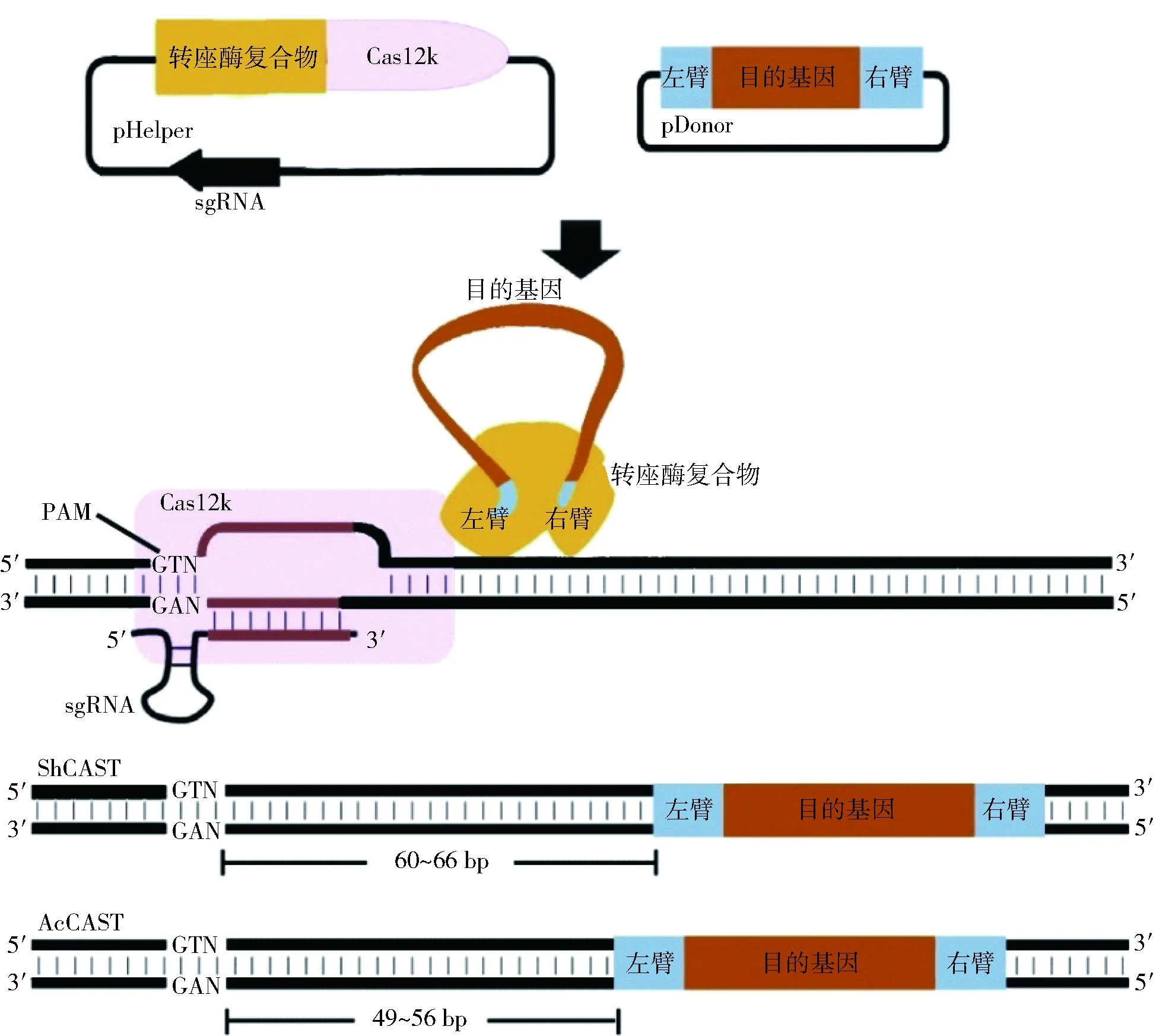
图1 CAST系统介导的外源DNA整合
美国哥伦比亚大学Klompe等[26]将来自于V.cholerae的天然转座子与CRISPR系统相结合,开发出一种强大的DNA插入系统(insert transposable elements by guide RNA-assisted targeting,INTEGRATE)(图2)。该系统包含3个质粒,由Cas678效应复合物、TniQ和crRNA阵列组成的级联复合质粒(pQCascade),由转座酶复合物(TnsA-TnsB-TnsC)组成的pTnsABC以及pDonor。首先Cas8通过靶DNA双链小沟序列特异性识别5’-CC的PAM序列,并稳定被打开的DNA双链,便于靶DNA链与crRNA的间隔区互补配对形成DNA-RNA杂交链,并由Cas7稳定,TniQ形成同源二聚体结合在Cascade复合物头部,分别与Cas7和Cas6相互作用,并负责招募非序列特异性DNA结合蛋白TnsC,转座子本身被TnsA和TnsB结合,形成一个配对末端复合物,被TnsC招募整合至目标DNA上[33]。整合位点位于PAM序列下游80~82 bp处,偏向于转座子右臂靠近靶位点。与CAST相比,它不需要tracrRNA,更适合直接插入多个基因组目标。此外,Cas678效应复合物的存在可能是该系统脱靶率低于5%的关键,但该系统的DNA插入没有方向性,大片段(3~10 kbp)转座效率低于CAST系统。随后,为了简化策略,消除抗生素承载和对多种转化过程的需求,提高整合效率,该团队在原来基础上通过将关键元件有效组合,构建出INTEGRATE单质粒系统pSPIN(图3),使得10 kb片段的整合效率达到100%,并在其他革兰氏阴性菌如产酸克雷伯氏杆菌(Klebsiellaoxytoca)及恶臭假单胞菌(Pseudomonasputida)中证实了pSPIN系统的有效性;相比之下,具有三质粒的INTEGRATE系统仅对低于1 kbp的片段具有较强的整合效率。此外,通过多阵列组合,并结合Cre-loxp系统,作者成功敲除了20 kbp的大片段,为微生物研究中实现更复杂的基因编辑操作提供了可能性。

图2 INTEGRATE系统介导的外源DNA整合
4.2 基于CRISPR的转座子编辑系统的应用
许多学者利用合成生物学研究表明:Cas核酸酶可以作为RNA引导的DNA结合蛋白域被重新用于操纵DNA序列和基因表达,如CRISPRi[34]、CRISPR激活(CRISPR activation,CRISPRa)[35]、FokI-dCas9二聚核酸酶[36]、碱基编辑器[37]和靶向组蛋白修饰[38]等。同样,转座酶也可以融合到dCas9上进行靶向转座。研究人员开发了一种新型合成转座子,Cas-转座子(CasTn),它可以将Himar1转座酶的DNA整合能力和dCas9的可编程基因组靶向能力结合起来,以实现在基因位点的定点转座子插入[39]。Vo等[40]将V.cholerae来源的CRISPR转座酶系统(VchINT)和S.hofmanni来源的CRISPR转座酶系统(ShoINT)联用,有效避免了已整合DNA片段的重新转座和整合,大大提高了多位点不同片段整合的特异性。转座子相关CRISPR-Cas系统的应用并不仅限于插入或敲除靶基因,与质粒依赖的表达策略相比,使用转座子相关CRISPR-Cas系统的染色体工程在控制拷贝数和保持异源基因的稳定性方面具有优势,而且该系统允许在细胞生长和繁殖时进行不间断的DNA整合,这对于构建多拷贝文库具有独特的吸引力。如Zhang等[41]通过多阵列组合的CRISPR相关转座酶策略建立的E.coli文库能在5 d内连续增加拷贝数至10个[图3(a)];而且,他还使用CAST系统实现了多拷贝染色体整合,并将其应用于N-乙酰葡萄糖胺(GlcNAc)的生产,得到的高产GlcNAc(11.59 g/L)突变体是含有pET-GNAc质粒菌株的6.2倍[42][图3(c)]。此外,转座子相关CRISPR-Cas系统也有可能应用于跨物种,这对混合细菌群落的基因操作特别有用。一项通过基因间结合将pSPIN系统从供体大肠杆菌递送到小鼠肠道菌群中的研究,成功实现了目标和物种的特异性整合[40][图3(b)]。

图3 基于CRISPR的转座子系统的相关应用
4.3 基于CRISPR的转座子编辑系统在乳酸菌定向改良中的应用展望
传统的乳酸菌改良技术虽能影响其生理代谢,致使部分基因发生突变,但却收效不佳;基于同源重组的基因编辑技术也存在一定的缺陷性。转座子是基因组重要组成部分,是微生物遗传操作的宝贵生物工具,对于基因功能分析至关重要。转座子诱变技术可将外源转座子转化至宿主细胞,并随机插入至宿主基因组中,使基因表达发生变化,从而产生新的性状。该技术虽可快速、准确、规模化寻找鉴定与某一表型相关的基因,但需要DNA链断裂,安全性低,且并不适用于广泛的宿主特性。基于CRISPR转座子编辑系统是一种新型的、强大的遗传编辑系统,它允许供体DNA的多重位点插入,并且不必依赖低效的同源重组和宿主细胞修复机制,因此可以大大提高插入效率和供体DNA的大小,安全性更高;特别是pSPIN系统,不仅消除了抗生素和多启动子的承载,简化了遗传转化过程,而且该系统还可以同其他操作系统联合使用,并具有广泛的宿主适应特性,是CRISPR相关转座子编辑系统的亮点。虽然目前还未有关于该系统在乳酸菌研究中的报道,但新型乳酸菌遗传功能分析、功能基因的精准靶向和高效整合技术一直是生物技术研究的焦点,基于CRISPR转座子基因编辑系统有望突破该瓶颈,为乳酸菌功能基因研究提供可靠的技术指导。当然,关于CRISPR转座子编辑系统的优化与安全性应用还需要更进一步地研究,比如精确的基因调节或无痕插入、详细的生化结构机制等。
5 总 结
乳酸菌是食品工业微生物中一类非常重要的细菌, 也是众所周知的维持人及动物健康的重要益生菌群。随着对乳酸菌功能研究的逐渐深入,人们对乳酸菌制品的要求也越来越高,菌种改良也就显得十分必要。利用基因编辑技术来改良乳酸菌,增强与修饰乳酸菌的生物学功能,筛选出安全稳定、生态效应强、更利于生产的优良菌种。而转座子技术的出现改变了以往人们认为遗传物质是固定排列在染色体上的观念,使得基因组编辑更为高效、简便。传统的基因编辑技术已很难满足乳酸菌复杂的功能研究,精准高效的基因编辑技术凭借其优势将会使乳酸菌在医疗、农业及食品工业中获得更为广泛的应用。
