腐生性钩端螺旋体Potoc 菌株全基因组学分析
2022-12-09张影石刚李彬杜宗利辛晓芳徐颖华
张影,石刚,李彬,杜宗利,辛晓芳,徐颖华
钩端螺旋体(简称钩体)属按照致病性不同可分为问号钩体和腐生性钩体,前者主要分离于病人和动物,具有寄生性;而腐生性钩体通常对人或动物不致病,常存在于环境土壤和水中[1]。人感染钩体病主要通过直接或间接接触了受问号致病性钩体污染的水、土壤或带菌动物的尿液。据统计报道每年全球仍有约 200 万钩体感染病例,其中大多数来自热带及亚热带地区的发展中国家和不发达国家,给当地人民健康和畜牧业生产造成了严重的影响[2-3]。为了更好预防和控制该传染病,国内外学者一直致力于研究钩体病的快速和早期诊断,例如建立了以腐生性钩体 Potoc 菌株为抗原的玻片凝集试验,并被证实该方法对钩体病的诊断具有一定帮助[4-5],但其方法内在机制尚未完全清楚。为了进一步了解其内在分子机制,本研究将腐生性钩体Potoc 菌株灭活菌液与国内 15 种不同血清群致病性钩体代表菌株血清进行免疫印迹分析,同时对Potoc 菌株进行全基因组测序,分析菌株的全基因组特征,并与已公布不同基因种的致病性钩体进行比较基因组学分析,构建系统发育树,从而为解析钩体属不同种基因组多样性和进化等研究提供新的线索。
1 材料与方法
1.1 材料
1.1.1 菌株 腐生性钩体 Potoc 型菌株(65-15)来源于中国食品药品检定研究院中国医学细菌保藏管理中心。
1.1.2 主要仪器和试剂 细菌培养箱为北京中仪国科科技有限公司产品;Vitek 全自动微生物鉴定仪为梅里埃诊断产品(上海)有限公司产品;EMJH培养基为美国 BD 公司产品;DNA 提取试剂盒为德国 Qiagen 公司产品;国内 15 种不同血清群致病性钩体代表菌株的兔血清来源于中国食品药品检定研究院;其他试剂均为国产分析纯。
1.2 方法
1.2.1 细菌培养 将腐生性钩体 Potoc 菌株接种至含 10% EMJH 培养基中,28 ℃ 培养 7~10 d,收集菌体;同时按照 DNA 试剂盒推荐操作步骤,提取菌株全基因组 DNA,DNA 样品放置于 -70 ℃保存备用。
1.2.2 全基因组序列测定与生物信息学分析 应用 Solexa 测序技术进行全基因组测序,并分别使用生物信息分析软件 velvet 1.2.03 和 glimmer 3.02 对获得下机数据进行拼装和基因预测,参考SEED 蛋白数据库进行测序基因组所包含的基因功能注释与直系同源簇(COG)分类。应用 CARD耐药抗性数据库(https://card.mcmaster.ca/)和VFDB 毒力基因分析数据库(http://www.mgc.ac.cn/cgi-bin/VFs/v5/main.cgi?func=VFanalyzer)在线工具对测序基因组进行耐药性和毒力基因预测分析。将测序获得全基因组序列与已发表的腐生性 Ames菌株(NCBI 登录号:NC_010842.1、NC_010845.1和 NC_010846.1)进行比较基因组学分析,并与已发表致病性钩体问号基因种 56601 菌株(NCBI 登录号:NC_004342.2 和 NC_004343.2)、波氏基因种 56142 菌株(NCBI 登录号:SRX673783)和中间型L.licerasiaeVAR010 菌株(NCBI 登录号:NZ_AHOO00000000.2)基因组进行共有与特有的基因比较分析。同时将上述 4 株钩体基因组与其他钩体菌株包括致病性钩体卫氏基因种 56679 菌株(NCBI 登录号:SRX673810)、L.kirschneri56611菌株(NCBI 登录号:SRX673784)、L.santarosai56180 菌株(NCBI 登录号:SRX673799)、L.noguchii56189 菌株(NCBI 登录号:SSRX673792)、L.alexanderi56159 菌株(NCBI 登录号:SRX672003)、L.alstonii56661 菌株(NCBI 登录号:SRX673771)、L.kmetyiBejo-Iso9T 菌株(NCBI 登录号:AHMP000000002)和中间型Leptospira johnsoniiE8T菌株(NCBI 登录号:BFAY00000000)进行核心基因的系统进化树分析。1.2.3 免疫印迹分析 按参考文献[6]方法进行Potoc 菌株灭活菌液与 1∶20 稀释的国内 15 种不同血清群致病性钩体代表菌株的兔血清进行免疫印迹分析。
2 结果
2.1 全基因组的特征
应用生物信息学软件,将测序菌株数据拼接,分析发现腐生性钩体 65-15 菌株染色体全基因组序列全长为 3 942 789 bp,预测基因组中所含编码基因为 3685 个,基因的平均长度为 997 bp,预测编码基因占整个基因组序列的 93.2%,其平均 GC含量为 39.0%。所含非编码区域大小为 267 456 bp,平均 GC 含量为 37.4%。此外还含有两个插入元件(ISLbi1 和 ISLbi2)。
以已发表的腐生性 Ames 菌株为参考进行比较分析,发现两株之间平均核苷酸一致性(ANI)为 99.98%,仅存在 179 个单核苷酸多态性位点(SNP)和 46 个插入或缺失(Indel)。SNP 包括103 个非同义 SNP 和 34 个同义 SNP,其中绝大多数非同义 SNP 影响转录、细胞运动相关编码蛋白。其余 42 个 SNP 均位于非编码区。
2.2 全基因组序列基因功能分析
应用 COG 数据库注释上述已获得的全基因组序列,发现腐生性钩体 65-15 菌株中共有2052 个基因具有明确的生物学功能,其中与信号转导功能相关基因数量高达 261 个,占总注释基因的12.7%,其次为细胞膜合成、氨基酸转运代谢、翻译后修饰、蛋白质周转、伴侣相关的基因(表 1)。此外,仍有 306 个(14.9%)的基因无法获得功能注释,有待后续进一步的分析。
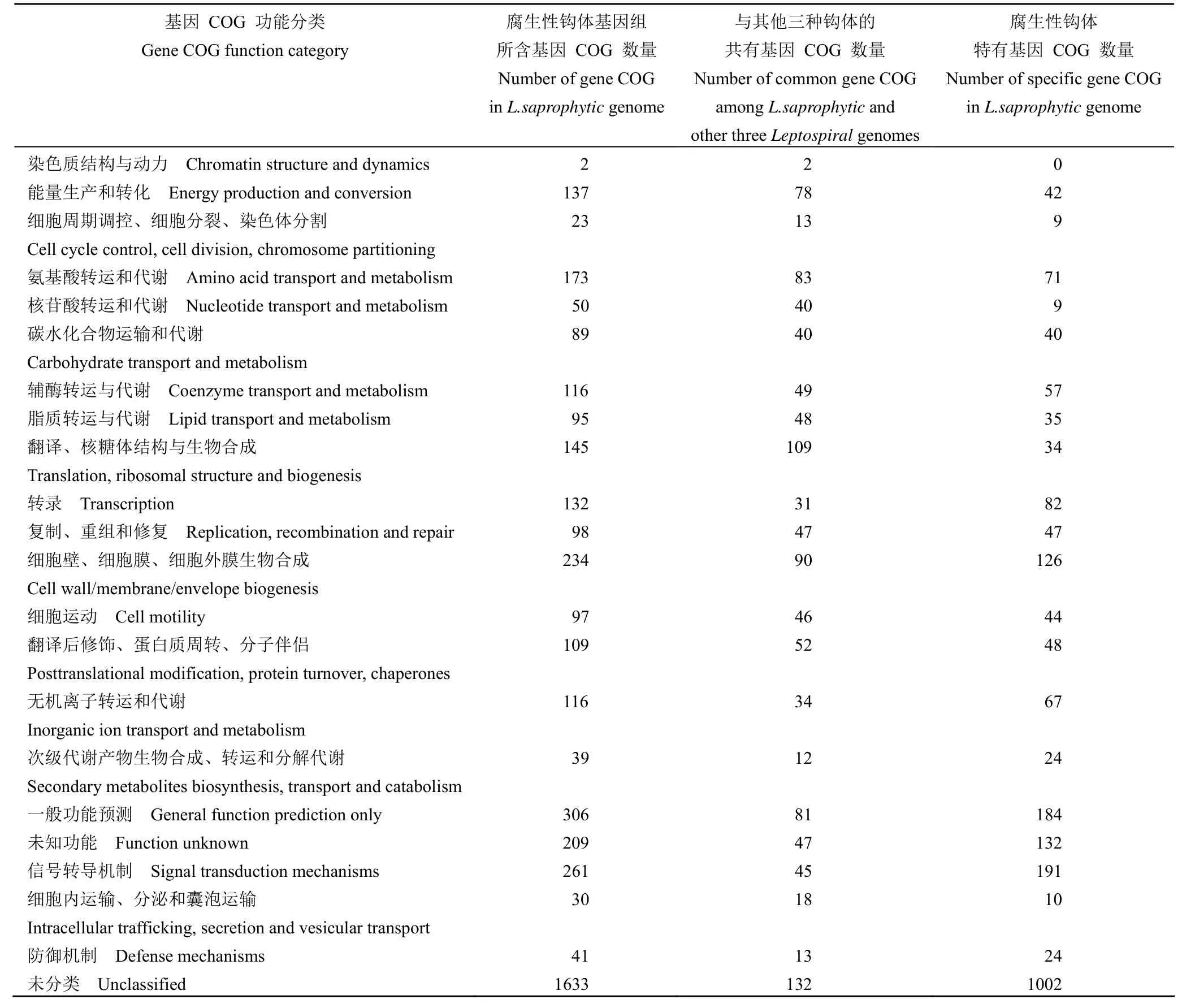
表1 腐生性钩体 Potoc 菌株基因组的 COG 比较分析Table 1 Comparative analysis COG of the genomes of Leptospira saprophytic Potoc strains
2.3 比较基因组学分析
应用比较基因组学分析与当前中国主要流行群菌株黄疸出血群 L45 菌株(NCBI 登录号:U61226.2)和波摩那群 L170 菌株(NCBI 登录号:AF316500)中负责 O 抗原生物合成基因簇(rfb)序列进行相似度比对分析,在测序的腐生性钩体65-15 菌株基因组中并没有发现类似rfb基因序列或相似基因。
同时,维恩图分析结果显示,与已发表致病性钩体问号基因种 56601 菌株、波氏基因种56142 菌株和中间型L.licerasiaeVAR010 菌株基因组存在共有基因 1110 个(图 1)。这些基因COG 功能分类主要集中在氨基酸代谢、生物合成等。而腐生性钩体 65-15 菌株含有 2278 个特有基因,COG 功能注释显示其中大部分基因与细菌适应不同环境相关。
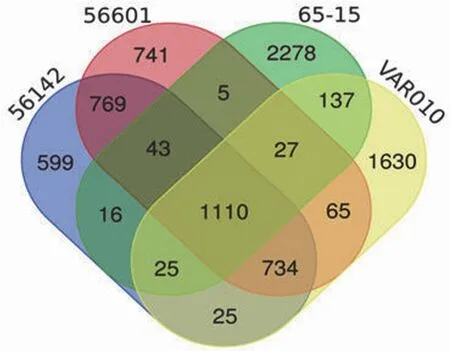
图1 腐生性钩体 Potoc 菌株与 3 株不同基因种钩体代表株的共有和特有基因维恩图(两种颜色交接处的数字表示颜色所指菌株的共有基因数)Figure 1 Venn diagram of common and unique genes between Leptospira biflexa Potoc strain and three different representative strains of Leptospira species (The figures in the two junction colors represents the number of common genes of both strains)
2.4 免疫印迹分析结果
免疫印迹分析显示,Potoc 菌株灭活菌液抗原与 15 种不同血清群致病性钩体代表菌株的兔血清呈现不同条带图谱。例如与拜伦群兔血清在65 kD 和 45 kD 处出现特异性条带,而波摩那群在 75 kD 处存在特异性条带。但与 15 种代表性菌株的兔血清在 25~50 kD 处均有明显的相同特异性结合条带(图 2),提示 Potoc 菌株中存在种属特异性的蛋白抗原成分。

图2 腐生性钩体 Potoc 菌株灭活菌液与国内 15 种不同血清群致病性钩体代表菌株兔血清的免疫印迹分析Figure 2 Results of Western blot analysis of inactivated bacteria solution of Leptospira biflexa Potoc strain and rabbit sera from fifteen Chinese representative different serogroup pathogenic Leptospira
2.5 进化分析
系统发育进化分析显示,不同致病性钩体分成不同进化分支,其中腐生性钩体菌株位于进化最外端,两株中间型钩体菌株L.johnsoniiE8T和L.licerasiaeVAR010 属于同一个进化分支内。9 种不同基因种的致病性钩体位于同一大进化分支中,并在进化过程中又分成不同亚分支。L.noguchii56189、L.kirschneri56611 和问号基因种 56601 菌株进化关系近,属于同一亚分支内。其他两株L.alstonii56661 菌株和L.kmetyiBejo-Iso9T 菌株形成独立亚分支,较其他 7 种基因种致病性钩体进化距离较远(图 3)。
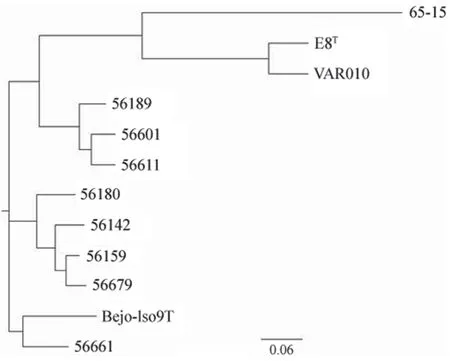
图3 腐生性钩体与致病型、中间型钩体全基因组系统发育进化分析Figure 3 Genome-wide phylogenetic analysis of Leptospira biflexa,pathogenic and intermediate Leptospira
3 讨论
随着生物技术的发展,快速、低成本的高通量测序技术已经广泛用于不同微生物的基因组学研究,在全基因组水平揭示其基因信息及生物学特征,推动了相关学科的深入研究[7-9]。本研究应用高通量测序技术首次对我国使用多年用于玻片凝集试验抗原制备的 Potoc 菌株进行分析,获得该菌种的全基因组特征,以此进行基因组 COG 聚类分析发现,参与信号转导(261 个基因)和细胞外膜合成(234 个基因)是高度富集的,推测可能与腐生性钩体在外界环境生存能力强,可在普通环境中长期存活的特性相关[10-12]。腐生性钩体 Potoc 株在我国被发现已经超过 40 年,其来源与历史不清楚[4-6]。将本研究获得的全基因测序数据与国外发表的腐生性 Ames 菌株的全基因序列比较,发现这两种菌株基因组之间的 ANI 高达 99.98%,仅存在179 个 SNP 和 46 个 Indel,提示国内这株腐生性钩体 Potoc 菌株与国外已发表的可能为同一来源,在各自实验室传代保存过程中,发生随机突变,从而产生一些碱基变化[13]。
致病性钩体 O 抗原是其重要的表面抗原,其结构多样性构成了不同血清群分类的基础[7]。不同血清型钩体之间,例如问号钩体哥本哈根型与哈焦型,两者rfb之间仅有 16 个基因相似,而相同血清型菌株整个rfb基因相似度可高达 90% 以上[7-8]。将本研究获得的 Potoc 菌株全基因组序列与国内最主要流行两个血清群黄疸出血群和波摩那群rfb基因序列比较分析,发现 Potoc 菌株基因组中并没有相类似rfb基因序列,提示 Potoc菌株中与致病性钩体发生特异性结合的并非 O 抗原。Western blot 分析显示的 25~50 kD 处的共同特异性结合条带进一步支持种属特异性可能是其他类型蛋白抗原成分[14]。事实上,我国研究人员发现三种不同毒力钩体外膜蛋白能与 Potoc 菌株特异性抗血清在 31 kD 出现特异性反应的阳性条带,建议这种 31 kD 外膜蛋白为潜在的种属特异性抗原之一[6]。这些种属特异性抗原不仅可作为钩体病实验室诊断方法的潜在靶标,同时也为未来钩体通用型疫苗的开发提供新的线索[15-16]。
比较基因组学分析结果发现腐生性钩体与其他中间型和致病性钩体的共有基因仅有 1100 余种,主要是维持微生物生存基本所需的,例如,氨基酸代谢、生物合成等[17]。而腐生性钩体特有的2278 个基因中,大部分与细菌适应不同环境相关。系统发育进化树进一步证实腐生性钩体位于系统进化树最外围,为致病性钩体种属的近祖先,而致病性和中间型钩体的遗传发育关系较近,在进化过程获得不同基因,形成不同的寄生能力和致病性[9-10]。
综上所述,本研究获得了我国的腐生性钩体Potoc 菌株的基本特征,探究了钩体属特异性抗原,揭示了不同基因种钩体基因组多样性,为后续致病性钩体的诊断和进化等研究奠定一定基础。
