薏苡bZIP基因家族全基因组鉴定与非生物胁迫表达分析
2022-11-11陈传敏周明强杨小雨班秀文刘凡值杨成龙
周 宇,陈传敏,王 健,周明强,杨小雨,班秀文,刘凡值,杨成龙*
薏苡bZIP基因家族全基因组鉴定与非生物胁迫表达分析
周 宇1,2,陈传敏3,王 健2,周明强2,杨小雨2,班秀文2,刘凡值2,杨成龙2*
1. 贵州大学农学院,贵州贵阳 550025;2. 贵州省亚热带作物研究所,贵州兴义 562400;3. 贵阳市第五中学,贵州贵阳 550003
薏苡是传统药食兼用经济作物,具有极高的营养价值和重要的药用价值,越来越受人们重视。转录因子是一类能够与调控基因的顺式作用元件或者功能基因特异性结合的重要调控蛋白质分子,在植物的许多生物学活动过程中起着重要的调控作用。碱性亮氨酸拉链(bZIP)转录因子家族在调控植物的生长发育及响应生物与非生物胁迫中起着重要作用。本研究利用北京薏苡(2n=20)基因组信息鉴定出83个bZIP基因家族成员,命名为~,2个bZIP基因(和)不能最终定位到任何连锁群通过生物信息学方法,分析了基因结构、理化性质、染色体分布,研究了其与其他物种的系统进化关系,并利用转录组技术研究了家族成员在栽培种兴仁小白壳苗期响应非生物胁迫的基因表达规律。结果表明:薏苡全基因组中bZIP成员分别编码氨基酸97~1008个,最小的是5(97个氨基酸),而最大的是28(1008个氨基酸);蛋白质分子量在11.33~110.03 kDa之间,pI在4.15(44)到12.33(50)之间;将其划分为A~I、K、S等11个亚组,不均匀地分布于基因组的10条染色体上;除了广泛分布的bZIP结构域的motif 1或motif 2外,每一类都具有相似基因结构与基序;然而,不同成员间存在外显子数目差异,最多为15个外显子(),最少为0个;干旱、高温、干旱和高温胁迫条件下,83个ClbZIP家族基因在干旱和高温胁迫后表达量的发生明显的变化,这说明这些基因可能在干旱和高温中的调控作用存在着不同。上述结果为更深入研究薏苡bZIP家族基因的生物学功能奠定基础及其在调控薏苡生长发育与响应生物与非生物胁迫机制提供参考。
薏苡;bZIP转录因子;生物信息学;干旱胁迫;高温胁迫;基因表达
薏苡(L.)又名薏仁米、薏苡仁和六谷子等,为禾本科玉蜀黍族薏苡属,一年生或多年生草本植物,起源于亚洲[1],我国西南部也是薏苡属植物的起源、演化和迁移的初生中心之一[2]。贵州是薏苡的主要栽培区,种植面积和产量均居全国第一,生产量占全国2/3,全省薏苡总产量达到28.8万t,全省种植和粗加工产值达43亿元,已建成全国最大的薏苡加工集聚区和产品集散地[3]。薏苡的营养和药用价值非常高,不仅被誉为“世界禾本科植物之王”,也被东亚和东南亚许多国家广泛用作药材和保健品[4]。薏苡仁中富含蛋白质、脂肪、糖等营养物质,具有较高的食用价值。薏苡整株均可入药,在医学方面具有健脾利湿、除脾止泻、清热解毒等功效[5]。因此,薏苡作为食物与中草药越来越受人们重视。
转录因子是一类能够与调控基因的顺式作用元件或者功能基因特异性结合的重要调控蛋白质分子,在植物的许多生物活动过程中起着重要的调控作用[6]。MYB、WRKY、AP2/EREBP、bZIP等转录因子是目前人们在植物中了解较早且研究较多的几大类转录因子家族。碱性亮氨酸拉链(basic leucine zipper, bZIP)转录因子家族是目前在植物中发现的64个基因家族中数量最大、最多样化且高度保守的基因家族之一[7]。bZI转录因子不仅参与调控植物的生长发育,而且在植物应对高温、干旱、病原防御等生物与非生物胁迫中也起着重要调控作用。bZIP转录因子家族成员已在多种植物中被鉴定,广泛存在于拟南芥(75个)[8]、玉米(125个)[9]、水稻(89个)[10]、谷子(85个)[11]等高等植物基因组。JAKOBY等[8]研究发现拟南芥基因通过与ABRE元件相结合,参与ABA信号传导通路,调控拟南芥植株的生长发育。KANG等[12]发现拟南芥的ABF3和ABF4蛋白通过调控ABA信号,可以增强拟南芥的耐旱性。NIJHAWAN等[10]通过过量表达水稻基因,可以提高水稻抗干旱与耐热能力。此外,水稻与基因共同过量表达,可以降低转基因植株的ABA和氯化钠的敏感性,从而提高水稻的耐受性[13-16]。WEI等[17]研究表明,通过诱导玉米基因表达,可以增强玉米耐盐性。LIM等[18]研究辣椒被沉默后,通过ABA诱导基因表达、ABA含量改变和关闭气孔,使其表现出干旱敏感和耐旱性。卢平等[11]研究谷子、和时,发现谷子在干旱处理下的根中表达量较高,参与根干旱胁迫应答。综上所述,植物bZIP转录因子家族在调控植物生长发育和植物响应非生物胁迫并提高其耐受性起着重要的调控作用。
目前栽培薏苡与野生薏苡基因组测序已完成,为薏苡抗性相关基因的挖掘与利用提供了重要的基因组资源。本研究利用栽培薏苡基因组信息和前期研究获得‘兴仁小白壳’转录组数据,通过生物信息学方法对薏苡bZIP转录因子家族进行全基因组鉴定,并分析了bZIP基因家族在薏苡苗期叶片响应干旱、高温和干旱高温胁迫的表达规律,为研究薏苡bZIP转录因子基因家族功能奠定基础,旨在挖掘薏苡抗旱候选基因,为解析薏苡抗旱和耐高温分子机理提供理论基础。
1 材料与方法
1.1 ClbZIP基因家族的鉴定
在薏苡中鉴定bZIP蛋白,本研究从NCBI(https://www.ncbi.nlm.nih.gov/bioproject/PRJNA544872)网站下载薏苡基因组序列,以及‘兴仁小白壳’干旱、高温和干旱高温胁迫转录组数据中编码序列(coding sequences, CDS)。另外,利用Pfam(http://pfam.xfam.org/)蛋白质数据库下载bZIP转录因子的隐马尔科夫模型文件(PF00170),利用HMMER 3.0软件中的Hmmsearch程序对薏苡蛋白质序列进行搜索,将得到的结果去除重复,并通过Pfam、SMART和CDD三大数据库进一步鉴定和筛选,最后得到薏苡bZIP家族蛋白质序列。薏苡bZIP转录因子基因的染色体定位和基因组位置信息来源于Phytozome数据库。然后,对错误预测的基因进行人工整理,其中一些基因通过PCR扩增和测序进一步验证。冗余序列通过手动剔除,经过全面整理,最终在薏苡基因组中鉴定出83个bZIP基因模型(ClbZIP)。使用ExPasy(http://web.expasy.org/protparam/)在线软件对ClbZIP蛋白的序列长度、分子量、等电点等进行理化性质预测。
1.2 ClbZIP基因家族的结构分析和染色体定位
根据薏苡基因组数据库中的物理位置信息,利用Mapchart 2.3软件(https://www.wur.nl/en)将所有ClbZIP基因定位到薏苡染色体上。采用多重共线性扫描工具包(MCScanX)以默认参数分析基因复制事件。为了展示从薏苡和其他选定物种获得的同源bZIP基因的共线关系,使用Dual-Systeny Plotter软件构建了共线分析图。使用TBtools在线软件绘制基因结构图;利用BLASTP对转录组鉴定的序列与薏苡基因组中的序列比较分析。
1.3 系统进化分析
利用MEGA 7.0软件把ClbZIP转录因子的氨基酸序列与拟南芥、水稻的bZIP蛋白序列进行多序列比对,并用邻接法(NJ)构建系统进化树,参数Bootstrap值设为1000,其他参数使用系统默认值。ClbZIP基因结构通过Gene StructureDisplay Server 2.0(http://gsds.cbi.pku.edu.cn/)网站预测,蛋白结构域通过MEME在线工具查询[19-21]。
1.4 ClbZIP家族成员启动子顺式作用元件分析
从薏苡全基因组数据库中提取每个薏苡bZIP基因启动子区域(上游1500 bp),并利用Plant CARE(http://bioinformatics.psb.ugent.be/webtools/ plantcare/html/)网站分析薏苡bZIP基因启动子的顺式作用元件种类、数目及功能。
1.5 基因表达分析
为了研究基因在非生物胁迫下的表达变化规律,本研究利用‘兴仁小白壳’作为试验材料,约1周左右,待材料幼苗生长至3片叶时,选择大小一致的材料分别进行干旱(15% PEG6000模拟干旱)、高温(40℃)和干旱高温(15%PEG6000+40℃)等胁迫处理不同时间(0、3、6、12、24 h)。胁迫后取叶片,每一个样品分别取3个生物学重复,液氮中速冻后保存于–80℃超低温冰箱中备用。每一个样品分别提取RNA构建文库并利用Illumina测序,经过注释获得差异表达基因。借助于Pheatmap对转录组数据进行均一化并鉴定基因差异表达,绘制基因表达热图,分析响应不同胁迫下基因表达的变化规律。‘兴仁小白壳’干旱、高温和高温干旱的转录组数据已上传至NCBI数据库(https://www.ncbi. nlm.nih.gov/),编号为PRJNA812268。
2 结果与分析
2.1 ClbZIP基因家族的鉴定
从Pfam数据库中获得的bZIP转录因子的隐马尔科夫模型文件(PF00170),结合bZIP蛋白保守序列,多重比对分析薏苡基因组数据库,通过剔除不含保守结构域的蛋白质序列,手动去除重复和不完整的序列。最后,基于存在明显完整的bZIP结构域,选择83个基因模型并注释为薏苡bZIP基因。最后,根据其在染色体上的位置依次命名为~(表1),2个ClbZIP基因(和)不能最终定位到任何连锁群,分别命名为和。
在83个ClbZIP蛋白质中,ClbZIP5被鉴定为最小的蛋白质,含有97个氨基酸,而最大的是ClbZIP28(1008个氨基酸)。蛋白质分子量在11.33~110.03 kDa之间,pI在4.15(44)到12.33(50)之间(表1)。

表1 ClbZIP基因家族理化性质分析
续表1 ClbZIP基因家族理化性质分析

Tab. 1 Analysis of the physicochemical properties of ClbZIPs (continued)
由图1可知,除了和外,薏苡bZIP基因家族成员不均匀地分布在薏苡10条染色体上,其中,1号染色体含有薏苡bZIP基因最多,有14个基因,占总数的16.87%;9号染色体含有薏苡bZIP基因最少,只有2个。
2.2 系统进化分析
对鉴定得到的83个ClbZIP基因进行基因结构分析,将薏ClbZIP转录因子的氨基酸序列进行多序列比对,并利用MEGA7.0软件构建薏苡、拟南芥和水稻bZIP家族成员构建系统进化树(图2)。结果表明,83个薏苡bZIP家族成员物种之间存在差异,与拟南芥亲缘关系较远,与水稻亲缘关系较近,并被划分为A(17个)、B(1个)、C(9个)、D(14个)、E(2个)、F(9个)、G(8个)、H(0个)、I(4个)、K(14个)、S(5个)等11个亚组。除H亚组外,均有ClbZIP基因家族成员分布,A亚组中最多,17个,B、E亚组中分布最少(1个,2个),和水稻(和)聚到一起,其同源性较高,并聚在B亚组,和两个成员亲缘关系较近聚集在E亚组。
2.3 ClbZIP基因家族成员的基因结构和基序组成
对所有已鉴定的ClbZIP基因的外显子-内含子组成特征进行分析,以进一步了解薏苡中bZIP家族的进化(图2、图3A)。结果表明,可划分为3个大类群,类群Ⅰ有57个基因,类群Ⅱ有7个基因,类群Ⅲ有19个基因;类群Ⅰ的57个ClbZIP基因可进一步划分为3个亚组,Ⅰa组有34个基因,Ⅰb组有9个基因,Ⅰc组有14个基因。由此可见,多组ClbZIP有较近的亲缘关系,如等均为旁系同源基因。
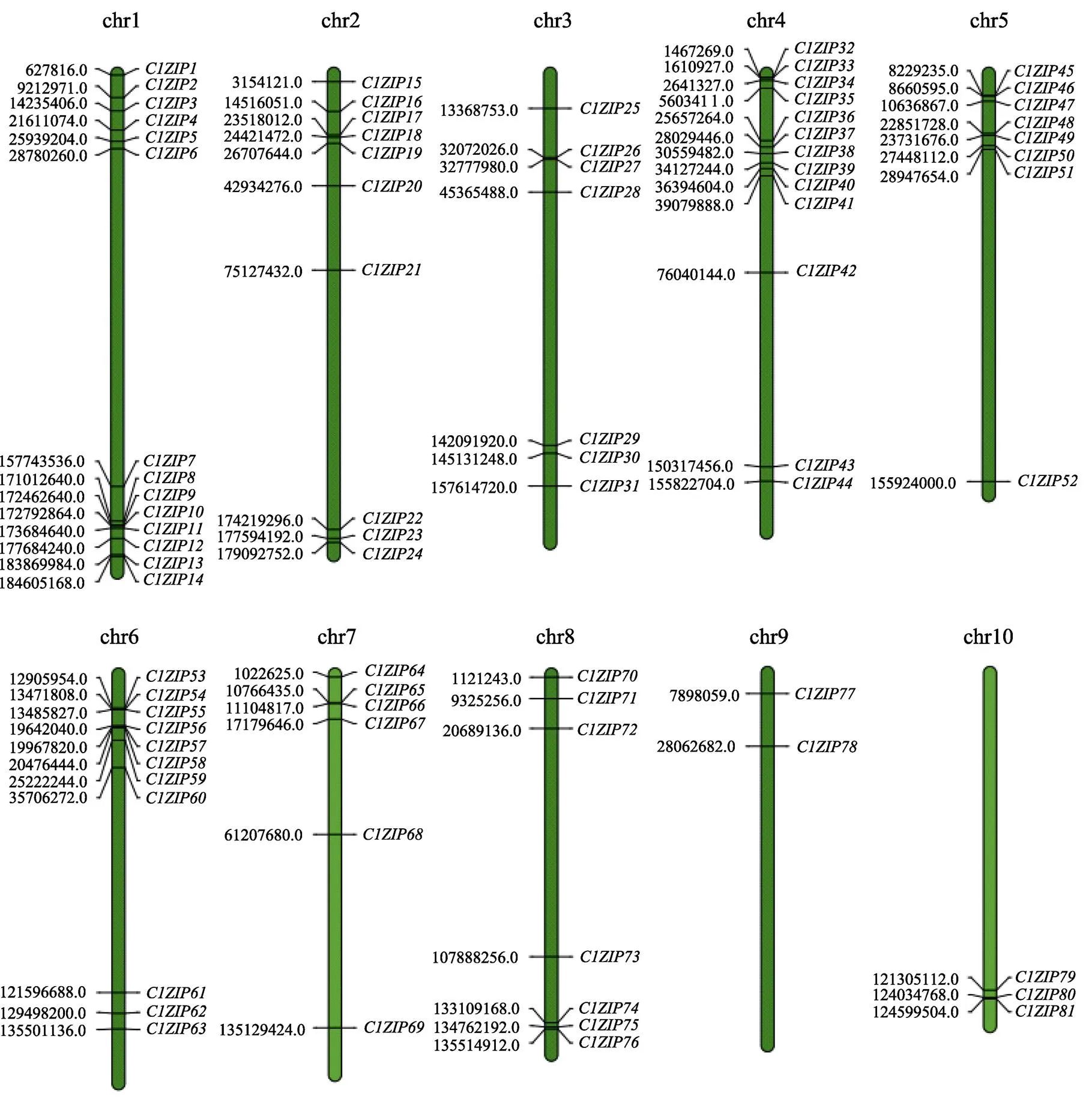
图1 ClbZIP基因家族的染色体分布
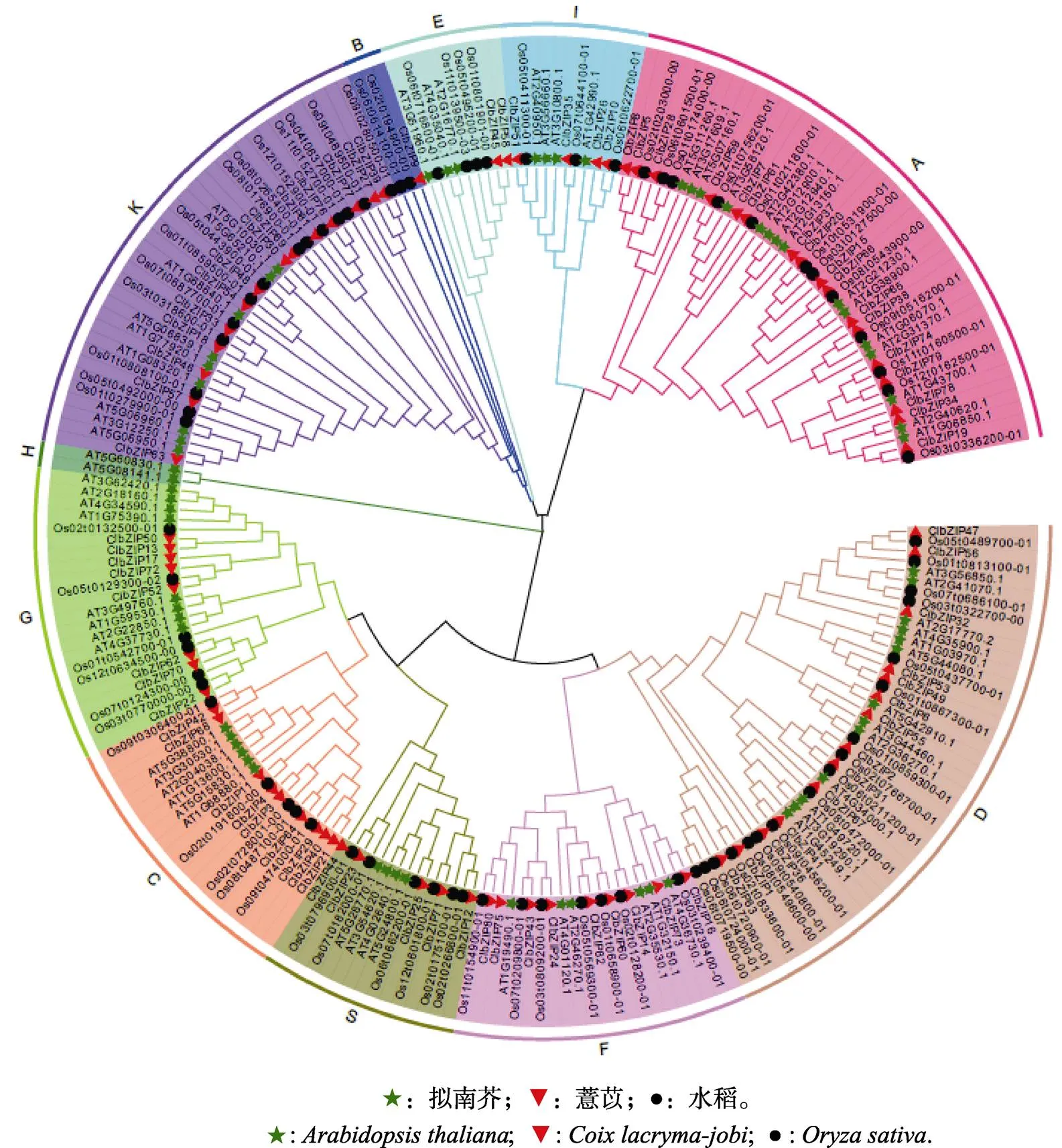
图2 薏苡与水稻、拟南芥的bZIP基因家族系统进化树
根据MEME motif分析结果构建了代表所有ClbZIP蛋白结构的示意图。如图3A所示,除了广泛分布的bZIP结构域的motif 1或motif 2外,同一组中的ClbZIP成员通常具有相似的基序组成,除Ⅰa类中有一部分包括motif 1和motif 2。例如,motif 9是I组一部分所特有的,而motif 3、4、5、6是Ⅲ组特有的且大部分含有多个motif。聚集的ClbZIP对,即,显示出高度相似的基序分布。亚组内ClbZIP蛋白之间相似的基序排列表明,该蛋白结构在亚家族中是相对保守。这些保守基序的大部分功能仍有待阐明。总体而言,同一类群中bZIP成员的保守基序组成和相似的基因结构,以及系统发育分析结果,可以有力地支持类群分类的可靠性。
如图3B所示,83个ClbZIP基因蛋白质保守基序均有所差异,含有0~7个不等。ClbZIP28中没有蛋白质保守基序,62个ClbZIP只含有1个保守基序,占薏苡ClbZIP全基因组的75%。Ⅰa组中,只有ClbZIP12含有Motif 2和Motif 7,ClbZIP21ClbZIP40ClbZIP42只含有Motif 2,其余基因均含有Motif 1。Ⅰb组中ClbZIP75ClbZIP8只含有Motif2,其余基因均只含有Motif 1。Ⅰc组中ClbZIP79只含有Motif 2,其余基因均含有Motif 2和Motif 7。类群Ⅱ中所有基因均只含一个保守基序,其中只有ClbZIP5含有Motif7,其余基因只含Motif 2。ClbZIP69ClbZIP77ClbZIP9ClbZIP27ClbZIP39ClbZIP48ClbZIP54ClbZIP76ClbZIP81ClbZIP30含有保守基序最多,有7个,均存在于类群Ⅲ中,并且ClbZIP71ClbZIP45ClbZIP17ClbZIP58均只含Motif 2。亲缘关系较近的基因蛋白质保守基序组成较相似,例如ClbZIP3/ ClbZIP4ClbZIP52/ClbZIP72均只含有Motif 2,且位置相近。
从图3C可以看出,ClbZIP基因家族成员的每个基因长度、内含子与外显子个数都有所不同,其内含子个数在0~15个之间,其中内含子数量最多的ClbZIP基因是有15个。均无内含子。

A:ClbZIP家族成员的系统进化树,不同颜色的枝代表不同的亚族;B:ClbZIP蛋白的基序组成,不同的基序用不同颜色的方框表示;C:ClbZIP基因的结构,框表示外显子,黑线代表内含子。
2.4 ClbZIP家族成员的染色体分布及共线性分析
图4所示,ClbZIP家族成员基因在10个薏苡连锁群(LG)上的分布不均匀。LG1、LG2、LG4含有最多的ClbZIP基因。一些连锁群(如LG1、LG2、LG4)有更多的基因,而其他连锁群则很少;有些LGs只有一个基因(例如LG9)。LG长度与ClbZIP基因数无显著正相关。根据200 kb内包含2个或更多基因的染色体区域被定义为串联复制事件,通过分析ClbZIP的基因复制事件,ClbZIP基因家族中不存在串联复制现象,有29对大片段复制基因。
为了进一步推断ClbZIP家族的系统发育机制,我们构建了2个代表物种相关的薏苡比较共线图,包括1个双子叶植物(拟南芥)和1个单子叶植物(水稻)(图5)。在薏苡与拟南芥的bZIP基因中仅有一对基因存在共线性关系,而薏苡与水稻的bZIP基因中存在52对基因存在共线性关系。在薏苡和拟南芥之间发现的共线基因对,在薏苡和水稻之间没有发现,这可能表明这些同源基因对是在双子叶植物和单子叶植物分化之后形成的。
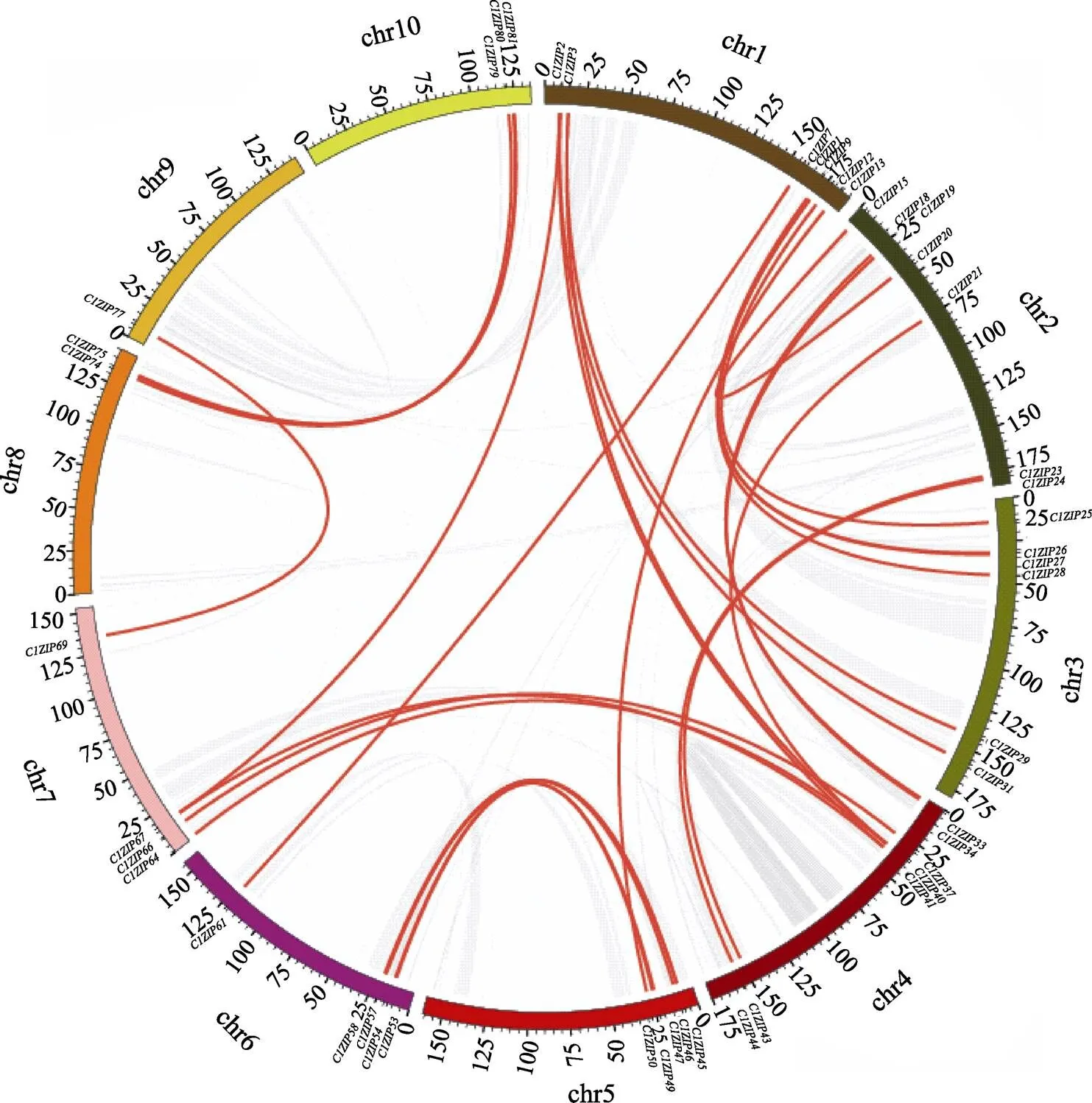
chr1~chr10表示薏苡的1~10号染色体;灰色部分表示薏苡全基因组的共线性基因;红色线条表示薏苡bZIP基因家族中的共线性。
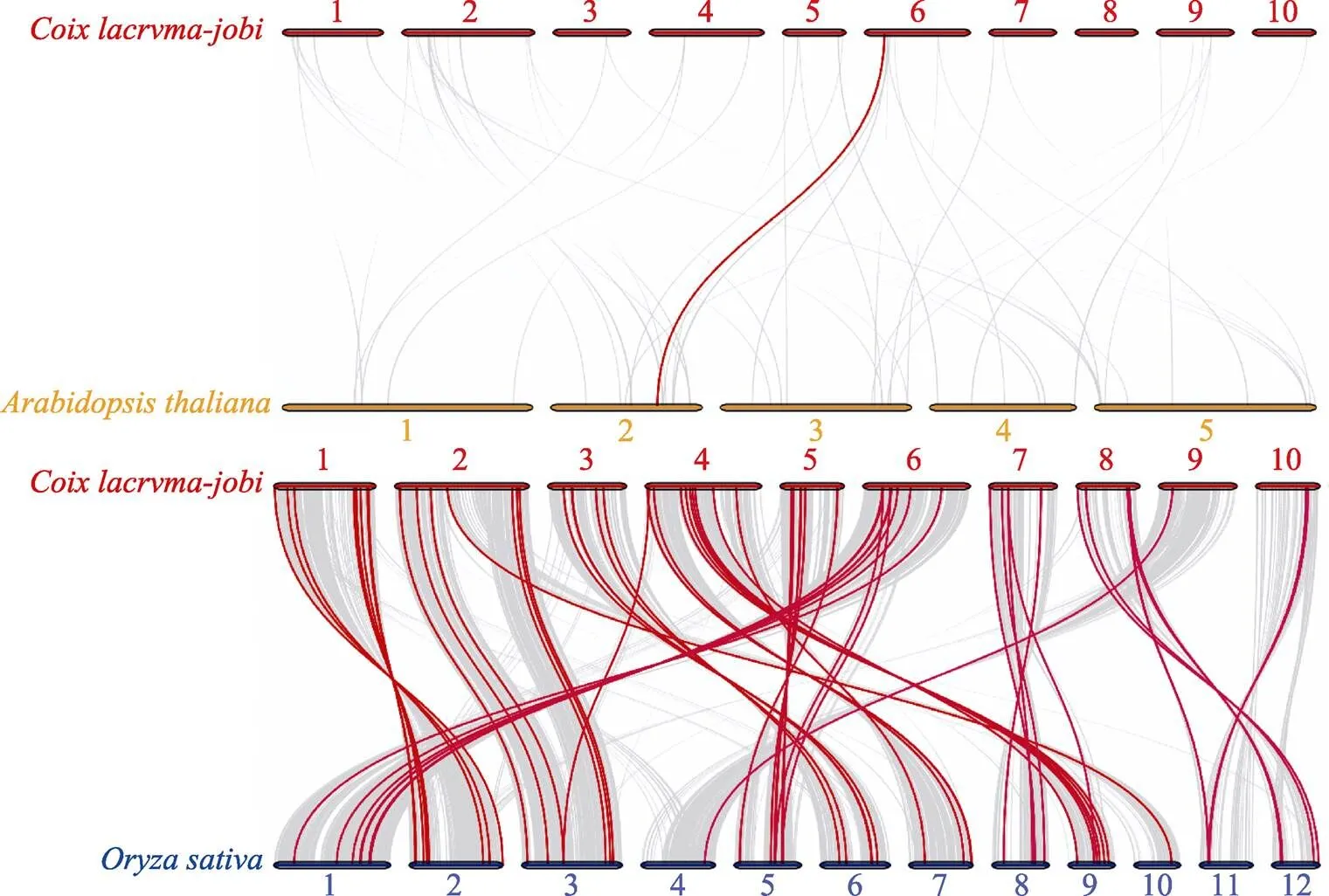
数字表示不同物种中的染色体;灰色部分表示2个物种间全基因组的共线性基因;红色线条表示2个物种间bZIP基因家族中的共线性。
2.5 ClbZIP基因家族启动子顺式作用元件分析
将ClbZIP基因起始密码子上游1500 bp序列用于顺式作用元件分析,如图6所示。ClbZIP基因启动子区域富含多个与ABA响应元件(ABRE)、植物光周期响应相关的顺式作用元件(AE-box、Box)。此外该基因家族还有与植物激素调节相关的顺式作用元件及与逆境响应相关的顺式作用元件。此结果表明ClbZIP基因在应对生物与非生物胁迫中起重要的调控作用。
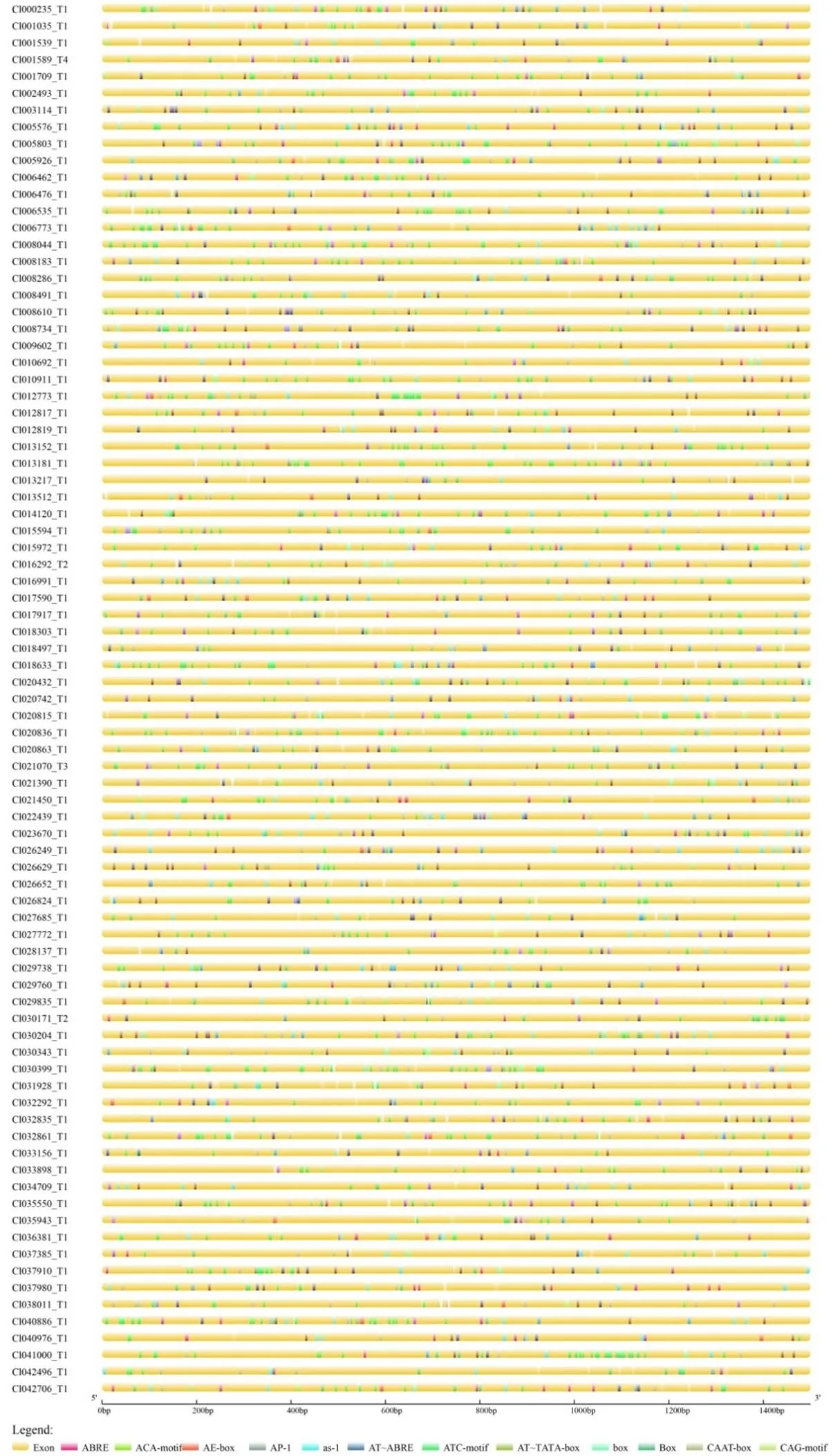
图6 ClbZIP基因家族顺式作用元件
2.6 ClbZIP基因家族表达分析
根据本研究‘兴仁小白壳’干旱、高温和干旱高温胁迫转录组中的FPKM数据,建立基因表达热图(图7、图8、图9),以研究ClbZIP家族基因在干旱和高温胁迫中所起的作用。从热图中发现,83个ClbZIP家族基因在干旱和高温胁迫后的表达量发生明显的变化,这说明这些基因可能在干旱和高温中的调控作用存在着不同。
如图7所示,在干旱胁迫3 h后,的表达量较高;在干旱6 h胁迫后,的表达量较高;在干旱胁迫12 h后,的表达量较高;在干旱胁迫24 h后,等基因具有较高的表达量。
在高温胁迫3 h后,的表达量较高;在高温胁迫6 h后,的表达量较高;在高温胁迫12 h后,的表达量较高;在高温胁迫24 h后,的表达量较高(图8)。
在干旱和高温共同胁迫3 h后,的表达量较高;在干旱和高温共同胁迫6 h和12 h下有较高的表达量;干旱和高温共同胁迫24 h后,的表达量较高(图9)。
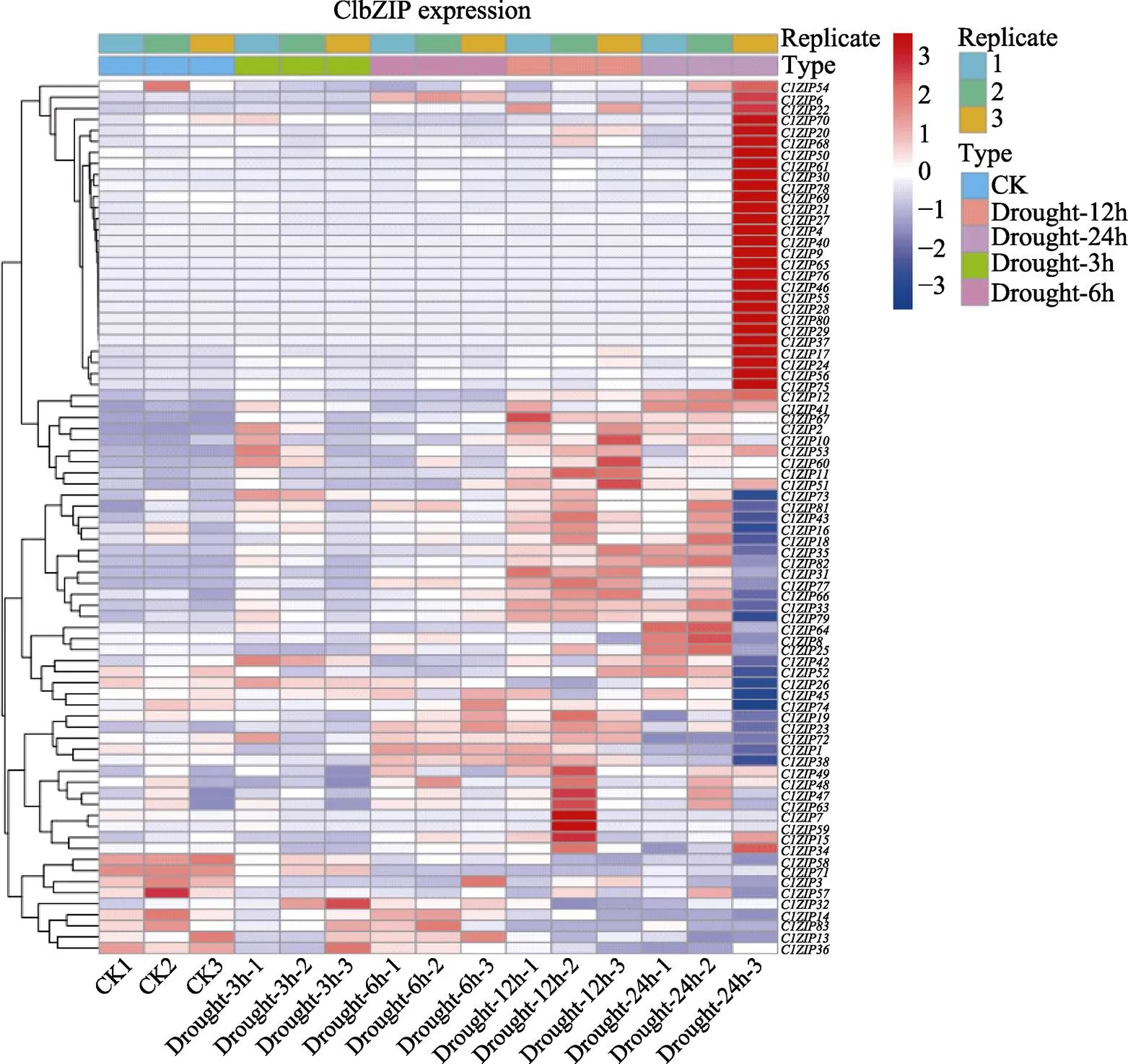
热图bar为log2;CK为未经干旱处理;Drought-3 h为干旱处理3 h;Drought-6 h为干旱处理6 h;Drought-12 h为干旱处理12 h;Drought-24 h为干旱处理24 h。

热图bar为log2;CK为未经高温处理,Heat-3 h为高温处理3 h;Heat-6 h为高温处理6 h;Heat-12 h为高温处理12 h;Heat-24 h为高温处理24 h。
3 讨论
bZIP基因家族普遍存在于植物中,其在植物生长、发育和抗逆等方面中起着重要作用,是最大的转录因子家族之一[22]。目前,bZIP基因家族已在多种植物中被鉴定出,但在薏苡的研究中尚未见报道。本研究通过对薏苡基因组数据中的蛋白质序列及兴仁小白壳的转录组数据进行分析,去除冗余后鉴定83个bZIP转录因子家族成员。本研究对鉴定的83个ClbZIP家族成员在染色体位置分析,发现ClbZIP基因不均匀地分布在1~10号染色体上。目前在与薏苡同为禾本科的谷子与水稻中分别鉴定出89和85个bZIP基因,其数量上相近。薏苡bZIP基因具有一定的保守性,亲缘关系相近的蛋白保守基序与基因结构也较为相似。薏苡bZIP转录因子家族的每个基因长度、内含子与外显子个数都有所差异,基因长度在97~ 1008个氨基酸之间,内含子个数最多的有15个,结构存在着复杂性,从而说明结构与功能存在关联性,复杂生物功能分化出不同类型基因结构。根据前人研究发现多种作物的bZIP家族基因中均普遍存在没有内含子的bZIP基因,如水稻、木薯、西洋梨,它们分别有15.3%、20.8%、28.8%的bZIP基因没有内含子[23-25]。基因复制事件在基因的进化过程中起着重要作用[26-27]。薏苡bZIP基因中存在29对大片段复制基因,基因片段复制是基因家族产生的重要因素,在大豆bZIP基因中有58%的基因存在片段复制现象[28]。薏苡与水稻的bZIP基因中存在52对基因存在共线性关系,而与拟南芥仅有1对基因存在共线性关系,说明薏苡与水稻亲缘关系较近。
前人研究发现,藜麦bZIP基因在不同组织部位均有表达[29]。谷子在暗处理后和的表达量较高[11]。水稻在干旱、PEG、ABA处理后具有较高的表达量[30-31]。苎麻在高盐胁迫下的表达量较高[32]。拟南芥正调控盐诱导基因,参与盐信号转导途径,并进行级联反应调控拟南芥的响应盐胁迫[33]。拟南芥和属于bZIP家族的TGA亚族,在抗逆过程中起着重要作用[34-35]。通常认为,聚类越近越相似,功能和结构上相似度越高。因此,推测拟南芥中的聚为一类的和基因可能参与薏苡抗旱的过程,参与调控激素GA和ABA的代谢过程,是否参与类似功能有待进一步验证。一般认为,基因表达量与其功能存在一定相关性,在转录组中发现薏苡bZIP家族多种基因在不同环境条件胁迫与不同胁迫时间条件下有不同程度的表达量,并且发现薏苡bZIP基因启动子含有多种调控生长发育及响应生物与非生物胁迫的顺式作用元件,但其是否在薏苡抗逆过程中起到重要的作用还需进一步验证。
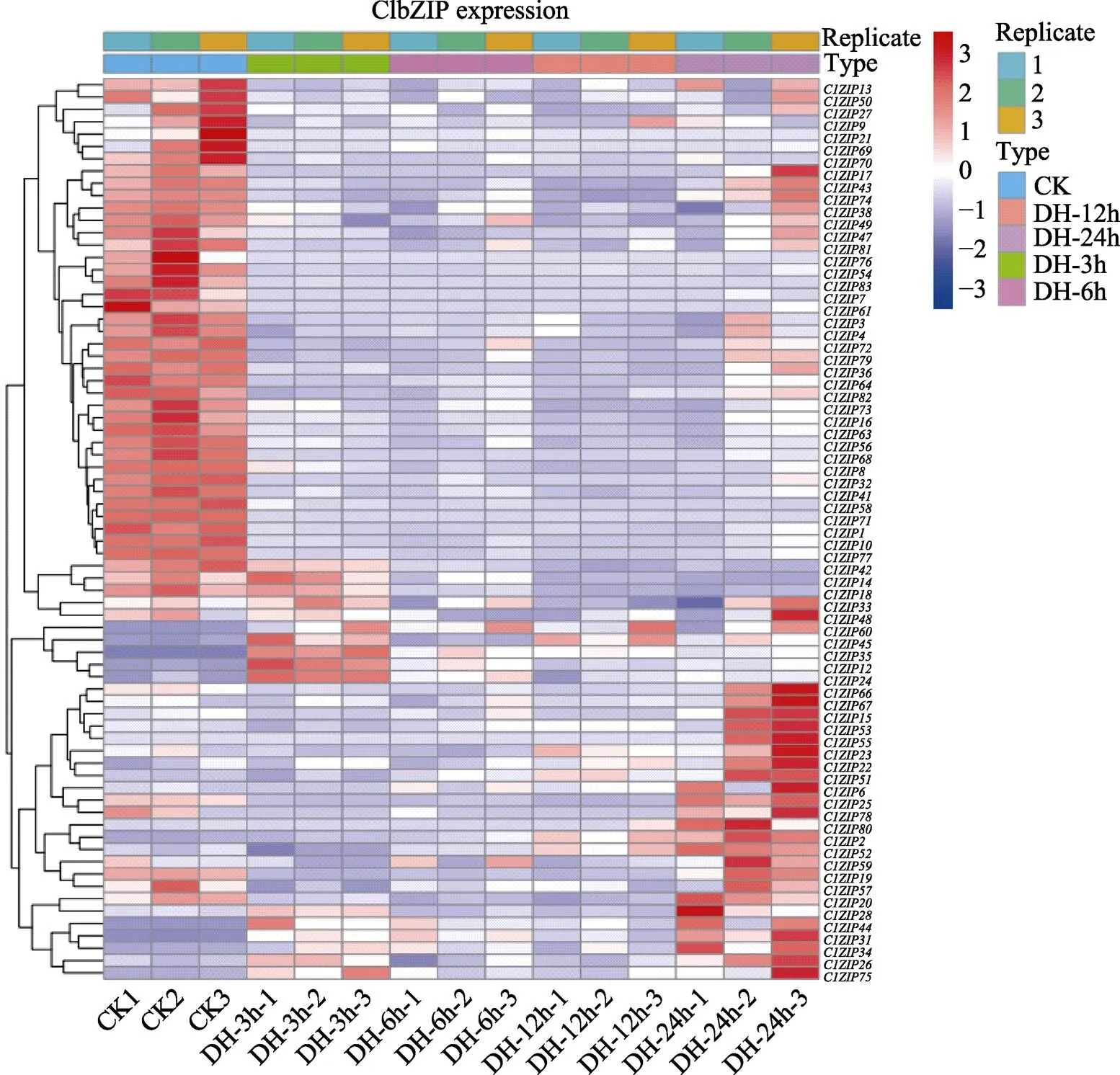
热图bar为log2;CK为未经处理;DH-3 h为干旱和高温处理3 h;DH-6 h为干旱和高温处理6 h;DH-12 h为干旱和高温处理12 h;DH-24 h为干旱和高温处理24 h。
薏苡作为一种粮药兼用作物,具有较高的营养与药用价值,但目前对其研究起步较晚并且尚未深入。bZIP转录因子是植物生长发育过程中重要的调控基因,并且在植物响应逆境环境胁迫中起重要的调控作用。尤其借助转录组与转录因子对薏苡抗性调控机制的研究,报道比较少。因此,对薏苡bZIP基因家族的研究是及有必要的,但在薏苡中研究抗逆性远没有拟南芥、水稻中的深入和清楚。本研究利用北京薏苡基因组和‘兴仁小白壳’干旱和高温胁迫的转录组对薏苡bZIP基因家族鉴定及干旱和高温胁迫后bZIP基因表达规律的研究,旨在为更深入研究薏苡bZIP基因家族的生物学功能奠定基础及其在调控薏苡生长发育与响应生物与非生物胁迫机制提供参考。
[1] ARORA R K. Job’s tears (L.) a minor food and fodder crop of Northeastern India[J]. Economic Botany, 1977, 31(3): 358-366.
[2] 陆 平, 左志明. 广西水生薏苡种的发现与鉴定[J]. 广西农业科学, 1996, 1: 18-20.
LU P, ZUO Z M. Discovery and identification ofRoxb. in Guangxi[J]. Guangxi Agricultural Science, 1996, 1: 18-20. (in Chinese)
[3] 李祥栋, 潘 虹, 陆秀娟, 魏心元, 陆 平, 石 明, 秦礼康. 薏苡种质的主要营养组分特征及综合评价[J]. 中国农业科学, 2018, 51(5): 835-850.
LI X D, PAN H, LU X J, WEI X Y, LU P, SHI M, QIN L K. Characteristics and comprehensive assessment of principal nutritional components in adlay landraces[J]. Scientia Agricultura Sinica, 2018, 51(5): 835-850. (in Chinese)
[4] 汪 灿, 周棱波, 张国兵, 张立异, 徐 燕, 高 旭, 姜 讷, 邵明波. 薏苡种质资源萌发期抗旱性鉴定及抗旱指标筛选[J]. 植物遗传资源学报, 2017, 18(5): 846-859.
WANG C, ZHOU L B, ZHANG G B, ZHANG L Y, XU Y, GAO X, JIANG N, SHAO M B. Identification and indices screening of drought resistance in job’s tears germplasm resources at germination stage[J]. Journal of Plant Genetic Resources, 2017, 18(5): 846-859. (in Chinese)
[5] 汪 灿, 周棱波, 张国兵, 张立异, 徐 燕, 高 旭, 姜 讷, 邵明波. 薏苡种质资源苗期抗旱性鉴定及抗旱指标筛选[J]. 中国农业科学, 2017, 50(15): 2872-2887.
WANG C, ZHOU L B, ZHANG G B, ZHANG L Y, XU Y, GAO X, JIANG N, SHAO M B. Drought resistance identification and drought resistance indices screening of job’s tears (L.) germplasm resources at seedling stage[J]. Scientia Agricultura Sinica, 2017, 50(15): 2872-2887. (in Chinese)
[6] 王金英, 丁 峰, 潘介春, 张树伟, 杨亚涵, 黄 幸, 范志毅, 李 琳, 王 颖. 植物转录因子家族的研究进展[J]. 热带农业科学, 2019(6): 39-45.
WANG J Y, DING F, PAN J C, ZHANG S W, YANG Y H, HUANG X, FAN Z Y, LI L, WANG Y. Research progress oflineage transcription factors in plant[J]. Chinese Journal of Tropical Agriculture,2019(6): 39-45. (in Chinese)
[7] PAULINO P R, MAURICIO R, GUEDES C, RENSING S A, BIRGIT K, BERND M R. PlnTFDB: updated content and new features of the plant transcription factor database[J]. Nucleic Acids Research, 2010, 38(suppl 1): 822-827.
[8] JAKOBY M, WEISSHAAR B, DRGE-LASER W, VICENTE-CARBAJOSA J, TIEDEMANN J, KROJ T, PARCY F, GROUP B.transcription factors in[J]. Trends in Plant Science, 2002, 7(3): 106-111.
[9] 于 滔, 王成波, 曹士亮, 孙培元. 玉米转录因子的生物信息学分析[J]. 黑龙江农业科学, 2016(4): 1-5.
YU T, WANG C B, CAO S L, SUN P Y. Bioinformatics analysis of thetranscription factor in maize[J]. Heilongjiang Agricultural Sciences, 2016(4): 1-5. (in Chinese)
[10] NIJHAWAN A, JAIN M, TYAGI A K, KHURANA J P. Genomic survey and gene expression analysis of the basic leucine zipper transcription factor family in rice[J]. Plant Physiology.2008, 146(2): 333-350
[11] 卢 平, 武懿茂, 武强强, 李雪垠. 谷子转录因子家族的全基因组鉴定与生物信息学分析[J]. 山西农业科学, 2020, 48(9): 1361-1370, 1430.
LU P, WU Y M, WU Q Q, LI X Y. Genome-wide identification and bioinformatics analysis oftranscription factor family[J]. Journal of Shanxi Agricultural Sciences, 2020, 48(9): 1361-1370, 1430.
[12] KANG J Y, CHOI H I, KIM S Y, IM M Y.basic leucine zipper proteins that mediate stress responsive abscisic acid signaling[J].Plant Cell, 2002, 14: 343-357.
[13] ZONG W, TANG N, YANG J, PENG L, MA S, XU Y, LI G, XIONG L. Feedback regulation of ABA signaling and biosynthesis by atranscription factor targets drought- resistance-related genes[J]. Plant Physiology, 2016, 171(4): 2810-2825.
[14] PANDEY A S, SHARMA E, JAIN N, SINGH B, BURMAN N, KHURANA J P. A ricetranscription factor, Os16, regulates abiotic stress tolerance when over-expressed in[J].Journal of Plant Biochemistry & Biotechnology, 2018, 27(4): 393-400.
[15] 李红婷. 拟南芥转录因子靶基因的筛选及鉴定[D]. 合肥: 安徽农业大学, 2020.
LI H T. Screening and identification of target genes ofin[D]. Hefei: Anhui Agricultural University, 2020. (in Chinese).
[16] 孙阳阳. 水稻转录因子Os18和OsBCAT家族基因的功能研究[D]. 武汉: 华中农业大学, 2020.
SUN Y Y. Function alanalysis of rice transcription factor Os18 and OsBCAT family genes[D]. Wuhan: Huazhong Agricultural University, 2020. (in Chinese).
[17] WEI K F, CHEN J , WANG Y M, CHEN Y H, CHEN S X, LIN Y N, PAN S, ZHONG X J, XIE D X. Genome-wide analysis of-Encoding genes in maize[J]. DNA Research, 2012, 19(6): 463-476.
[18] LIM C W, BAEK W, LEE S C. Roles of pepperprotein CaDILZ1 and its interacting partner RING-type E3 ligase CaDSR1 in modulation of drought tolerance[J].Plant Journal, 2018, 96(2): 452-467.
[19] JIN Z, WEI X, LIU A. Genomic surveys and expression analysis ofgene family in castor bean (L.)[J].Planta, 2014, 239(2).
[20] WANG M , YUAN F, HAO H , ZHANG Y, ZHAO H, GUO A, HU J, ZHOU X, XIE C G. BolOST1, an ortholog of open stomata 1 with alternative splicing products in, positively modulates drought responses in plants[J]. Biochemical & Biophysical Research Communications, 2013, 442(3/4): 214-220.
[21] YUAN F, WANG M, HAO H, ZHANG Y, ZHAO H, GUO A, HU J, ZHOU X, XIE C G. Negative regulation of abscisic acid signaling by theABI1 ortholog[J]. Biochemical & Biophysical Research Communications, 2013, 442(3/4): 202-208.
[22] DEPPMANN C D, TAPAROWSKY A E J. Cross-species annotation of basic leucine zipper factor interactions: insight into the evolution of closed interaction networks[J]. Molecular Biology and Evolution,2006, 23(8): 1480.
[23] HU W, YANG H, YAN Y, WEI Y, TIE W, DING Z, ZUO J, PENG M, LI K. Genome-wide characterization and analysis oftranscription factor gene family related to abiotic stress in cassava[J]. Scientific Reports, 2016, 6: 22783.
[24] 孙耀国, 蔡天润, 姬行舟, 张 军. 西洋梨全基因组基因家族生物信息学分析[J]. 林业与生态科学, 2021, 36(1): 24-34.
SUN Y G, CAI T R, JI X Z, ZHANG J. Genome-wide bioinformatics analysis ofgene family in[J]. Forestry and Ecological Sciences, 2021, 36(1): 24-34. (in Chinese)
[25] 金晓芬. 水稻类转录因子的功能分析[D]. 扬州: 扬州大学, 2008.
JIN X F. Functional analysis of, atranscription factor in rice (L)[D]. Yangzhou: Yangzhou University, 2008. (in Chinese).
[26] FLAGEL L E, WENDEL J F. Gene duplication and evolutionary novelty in plants[J]. New Phytologist, 2009, 183(3): 557-564.
[27] MOORE R C, MD P. The evolutionary dynamics of plant duplicate genes[J]. Current Opinion in Plant Biology, 2005, 8(2): 122-128.
[28] ZHANG M, LIU Y, SHI H, GUO M, CHAI M, HE Q, YAN M, CAO D, ZHAO L, CAI H. Evolutionary and expression analyses of soybean basic leucine zipper transcription factor family[J]. BMC Genomics, 2018, 19(1): 159
[29] LI F, LIU J, GUO X, YIN L, WEN R. Genome-wide survey, characterization, and expression analysis oftranscription factors in[J]. BMC Plant Biology, 2020, 20(1): 405.
[30] HAO C, WEI C, ZHOU J, HUANG H, CHEN L, CHEN H, XING W D. Basic leucine zipper transcription factor Os16 positively regulates drought resistance in rice[J]. Plant Science, 2012, 193/194: 8-17.
[31] HOSSAIN M A, CHO J I, HAN M, AHN C H, JEON J S, AN G, PARK P B. The ABRE-bindingtranscription factor OsABF2 is a positive regulator of abiotic stress and ABA signaling in rice[J]. Journal of Plant Physiology, 2010, 167(17): 1512-1520.
[32] HUANG C, ZHOU J, JIE Y, XING H, ZHONG Y, YU W, SHE W, MA Y, LIU Z, ZHANG Y. A ramietranscription factor Bn2 is involved in drought, salt, and heavy metal stress response[J]. DNA & Cell Biology, 2016, 35(12): 776-786.
[33] LIU J X, SRIVASTAVA R, CHE P, HOWELL S H. Salt stress responses inutilize a signal transduction pathway related to endoplasmic reticulum stress signaling[J]. The Plant Journal, 2007, 51(5): 897-909.
[34] MIAO Z H, LIU X J, LAM E. TGA3 is a distinct member of the TGA family oftranscription factors in[J]. Molecular Biology Reports, 1994, 25(1): 1-11.
[35] ZANDER M, LA CAMERA S, LAMOTTE O.class-II TGA transcription factors are essential activators of jasmonic acid/ethylene-induced defense responses[J]. The Plant Journal, 2010, 61(2): 200-210.
Genome-wide Identification and Expression Analysis of bZIP Gene Family under Abiotic Stress inL.
ZHOU Yu1,2, CHEN Chuanmin3, WANG Jian2, ZHOU Mingqiang2, YANG Xiaoyu2, BNA Xiuwen2, LIU Fanzhi2, YANG Chenglong2*
1. College of Agriculturre, Guizhou University, Guiyang, Guizhou 550025, China; 2. Guizhou Institute of Subtropical Crops, Xingyi, Guizhou 562400, China; 3. Guiyang Fifth Middle School, Guiyang, Guizhou 550003, China
Coix is a traditional economic crop that can be used both as food and medicine, and has high nutritional and medicinal value. As a kind of food and traditional Chinese medicine, coix is getting more and more attention. Transcription factors are important regulatory protein molecules that can specifically bind to cis-acting elements or functional genes of regulatory genes and play an important regulatory role in many biological processes of plants. The basic leucine zipper (bZIP) transcription factor family plays an important role in regulating plant growth and development and responding to biotic and abiotic stresses. In this study, 83 bZIP gene family members named1–83 were identified using the genomic information of Beijing Coix (2n=20). Twogenes (and) could not be located to any linkage group. Through bioinformatics methods, the gene structure, physical and chemical properties and chromosome distribution were analyzed, and its phylogenetic relationship with other species was studied. The gene expression rules of family members in response to abiotic stress at the seedling stage of Xingren small white shell were studied by the transcriptome technology. The results showed that the bZIP members in coix’s genome encoded 97–1008 amino acids, respectively. The smallest was(97 amino acids), and the largest was(1008 amino acids). The molecular weight of the protein was between 11.33–110.03 kDa, the pI was between 4.15 () and 12.33 (); They were divided into 11 subgroups such as A–I, K and s, which were unevenly distributed on 10 chromosomes of the genome. Except motif 1 or motif 2 of the widely distributed bZIP domain, each class had similar gene structures and motifs. However, there were differences in the number of exons among different members, with a maximum of 15 exons () and a minimum of 0. Under drought, high temperature, drought and high temperature stress, the expression of 83gene families changed significantly after drought and high temperature stress, indicating that the genes may have different regulatory roles in drought and high temperature. The above results would lay a foundation for further understanding the biological functions of the bZIP gene family inand provide reference for the regulation of growth and development and response to biological and abiotic stresses, and provide theoretical guidance for the study of the regulation of the bZIP gene family in drought-resistant and high-temperature environments.
L.; bZIP transcription factor; bioinformatics analysis; drought stress; high temperature stress; gene expression
S519
A
10.3969/j.issn.1000-2561.2022.10.006
2022-02-15;
2022-03-04
国家自然科学基金项目(No. 31701459);贵州省农业科学院院专项(黔农科院院专项(2016)019号);贵州省农业科学院青年基金项目(黔农科院青年科技基金[2022]22号)。
周 宇(1997—),男,硕士研究生,研究方向:作物遗传育种。*通信作者(Corresponding author):杨成龙(YANG Chenglong),E-mail:yangchenglong208@163.com。
