基于功能宏基因组学挖掘抗生素耐药基因研究进展
2022-11-05鲁兆祥王夕冉连新磊廖晓萍刘雅红孙坚
鲁兆祥 王夕冉 连新磊 廖晓萍 刘雅红 孙坚
(1. 华南农业大学兽医学院,广州 510000;2. 国家兽医微生物耐药性风险评估实验室,广州 510000;3. 岭南现代农业科学与技术广东省实验室,广州 510000)
抗生素被广泛的应用于临床治疗、疾病预防以及农业生产等诸多领域。抗生素的大量使用甚至一定程度上的滥用使得细菌耐药性问题逐渐成为威胁公共卫生与食品安全的一大难题。受制于微生物的培养,基于耐药细菌培养鉴定的传统耐药性研究方式无法获得综合全面的信息。而功能宏基因组学方法不依赖于对特定细菌的培养,并且将表型筛选与高通量测序有机结合,方便获得基于耐药表型的大量数据,因此在发掘新型耐药基因的应用上发挥了巨大的作用,展现了良好的发展潜力。但是功能基因组的发展依然存在大片段文库的构建方法和宿主外源表达的问题,随着以上问题的解决,功能宏基因组学将会在发掘新型耐药基因中发挥更大的作用。
1 抗生素耐药性问题
近年来随着抗生素的大量使用,不断发展的抗生素耐药性问题已经成为全球公共卫生面临的严峻威胁,现有的抗生素治疗方案已难以应对由耐药细菌引起的复杂感染。有文献报道,2014年全球有接近70万人死于由耐药细菌引起的感染,如果这一问题继续发展,30年后可能有超过1 000万人因此丧命[1]。目前新型抗生素的研发遇到瓶颈,新型药物研发耗时漫长,花费过高,致使在临床上治疗多重耐药细菌感染的有效药物不断减少[2-3]。
随着研究的深入,人们逐渐意识到细菌耐药性的产生是人类、动物和环境等多方面因素共同作用的结果,也同样影响着人类、动物和环境[4]。虽然抗生素耐药性最初被认为是人类健康问题,但由于当前的全球经济政策,人类、动物和环境越来越相互渗透,生态系统与人类活动互相影响,暗示着抗生素耐药性可能在生态系统中协同进化互相作用[5]。
已有多个研究表明,多种人类致病菌中的耐药基因,与环境中细菌的耐药基因具有高度相似的核苷酸序列[6],因此耐药基因可能通过水平转移,转入临床致病菌中,并进一步在新的宿主环境中发生转移与进化,造成了潜在的安全隐患。这一发现强调了对环境中大量未知抗生素耐药基因进行挖掘的必要性,不仅能够丰富人们对于更为复杂生态细菌中潜在耐药威胁的认知,深化理解细菌与环境的相互作用,而且还有助于人们提前防范未来超级细菌的产生。
1.1 各类抗生素的临床应用及耐药现状
近年来,各种新型耐药基因不断涌现,针对各类抗生素的耐药形势也越来越严峻。目前被广泛使用的抗生素在临床应用中均出现了明显的耐药情况。遏制细菌耐药性危机已经成为全人类生存发展亟待解决的问题。
β-内酰胺类抗生素是第一类被发现的抗生素,也是临床中最常用的抗生素之一[7]。β-内酰胺类抗生素是治疗革兰氏阴性菌感染的一线药物[8],由于其在畜牧养殖与临床诊疗中被广泛应用,使这类抗生素的耐药性持续增加[9]。之前的研究表明,从美国和欧洲的59个医疗中心的肺炎患者上分离得到的大肠杆菌对头孢曲松、头孢他啶、美罗培南等β-内酰胺类抗生素展现了较高的耐药率(表1)[10]。在中国,β-内酰胺类抗生素的耐药现状也十分严重,例如上海疗养院分离得到的大肠杆菌对β-内酰胺类抗生素广泛耐药,其中氨苄西林的耐药率高达97.5%(表 1)[11]。
四环素是广谱抗生素,广泛用于对抗革兰氏阴性和革兰氏阳性菌。由于四环素类药物具有广泛适用、成本低等诸多优点,近年来此类抗生素在动物和人类感染治疗中的应用不断增加[12-13]。这导致了耐四环素细菌的出现,现在开始逐渐限制此类抗生素的使用[14]。例如在中国四川省的肉鸡屠宰过程中,四环素类药物的耐药菌检出率就高达51.9%[15]。而对四川某医院的患者送样标本中的菌株分离鉴定发现,其对四环素类药物的耐药率大于70%[16]。
氨基糖苷类抗生素广泛用于医学[17-18]和农业领域[19-20],是最古老的抗菌药物类别之一。氨基糖苷类抗生素是杀菌抗菌剂,通过与 30S 核糖体亚基结合,损害细菌蛋白质合成而发挥作用。2013-2014年上海市的医院重症监护室临床病例标本分离的鲍曼不动杆菌对庆大霉素、头孢菌素、氨基糖苷类抗菌药物耐药率在96%以上。环境标本分离的鲍曼不动杆菌对氨基糖苷类抗菌药物阿米卡星耐药率最高,达62.39%[21]。其他类抗生素如大环内酯类、林可酰胺类等均表现出了显著的耐药情况(表1)。因此,进一步对临床细菌进行耐药性监测与发掘工作成为后续指导合理用药及遏制耐药性发生发展的关键任务[22]。
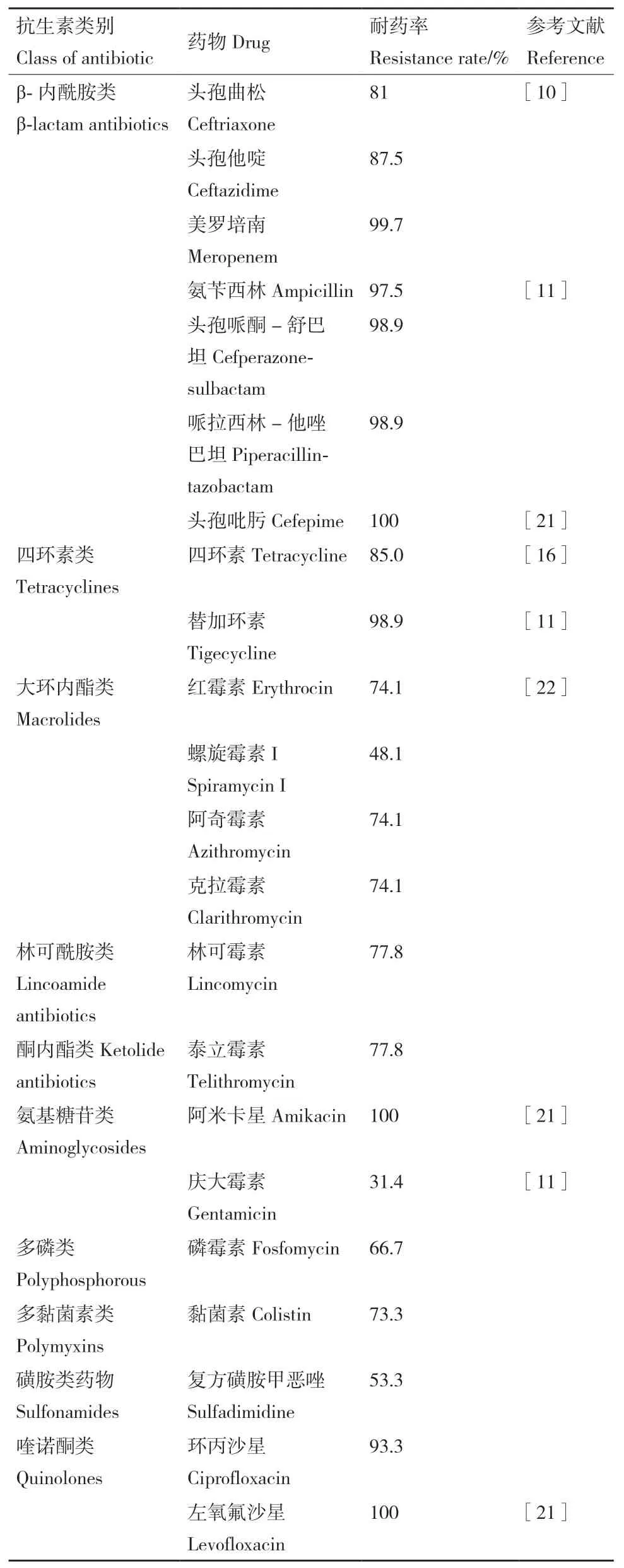
表1 临床抗生素耐药情况Table 1 Overview of antibiotic resistance in clinical isolates
1.2 耐药基因的检测及新型耐药基因的发掘
目前在实验室水平对细菌耐药基因检测主要依赖于两种传统的方法。一是基于可培养细菌中耐药基因的识别方法,作为鉴定抗生素耐药频率的常规方法,这是鉴定耐药细菌的“黄金标准”[23]。但是这类方法往往受制于所研究细菌的可培养性。例如土壤中也存在大量抗生素耐药基因,但土壤中可培养的细菌只占非常小的部分,因此基于可培养细菌的方法无法全面表征在不可培养的土壤微生物中的抗生素耐药基因[24]。二是使用有针对性的PCR的方法[25]识别和量化已知序列的抗生素耐药基因也因其准确性与可操作性被普遍用于耐药细菌的检测。但是这些技术要求我们已经获得耐药基因的相关信息,因此只能用于检测先前报道过的基因[26-27]。在过去的十年间,基于高通量测序技术的宏基因组学研究极大地拓展了我们的微生物学认知[28]。从单一菌株到微生物群落,从可培养微生物到不可培养微生物,使细菌耐药基因探索领域发生了革命性的变化。基于宏基因组学的方法,能够对所获得样本中的微生物群落的总DNA直接进行测序,无需培养,使宏基因组学技术手段被广泛地应用到探索环境中未知抗生素耐药性基因研究中[29-30]。使用这项技术,可以对种群中所有微生物的 DNA 进行整体分析,不仅可以做到揭示微生物物种信息,还可以深入了解微生物种群中可能存在的各种抗性因子。但是对微生物宏基因组进行测序,难以识别与已知耐药基因具有极低核苷酸相似性的耐药基因,同时低拷贝数的耐药基因也常常被忽略。此外,其最大的局限性在于无法确认假定的耐药基因的功能,因此需要进行额外的补充实验以阐明其功能。因此,在宏基因组学的基础上,结合表型筛选,功能宏基因组学被创立并发展成为鉴定新型耐药基因的有力工具,也是研究抗生素耐药机制及其相关基因的最有效和最强大的方法之一。
2 功能宏基因组学及其在耐药基因研究中的应用
2.1 功能宏基因组学的一般方法
如上所述,功能宏基因组学是近年来发展起来的一种用于鉴定抗生素耐药组(antibiotic resistome)的有效手段[31],此方法基于样品中的基因组,通过对样品宏基因组进行物理或生物手段剪切成功能性小片段。进一步的,将这些片段连接到表达载体中并转化到易感受体菌株中,从而得到功能宏基因组文库。针对获得的功能宏基因组文库,首先利用药理、生化和分析化学技术对文库进行功能或表型的筛选,以获得在特定环境下表达优势或功能优势的目的克隆,并进一步使用高通量技术建立起表型与基因型之间的联系,基本流程如图1所示。
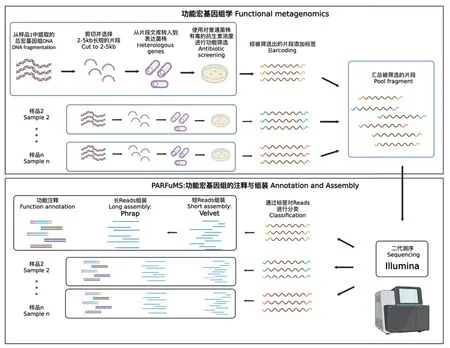
图1 功能宏基因组学方法的基本流程Fig. 1 Basic flow of functional metagenomics approach
由于抗性通常是可转移的,而水平基因转移是以前易感菌株获得抗性的最常见的方法。环境细菌拥有的抗性基因可能被人类病原体所获得,进而对人类健康产生威胁。针对上述问题,功能宏基因组学通过两个方面来开展研究:一是发现新型抗生素和抗感染药物;二是鉴定微生物种群中的抗性基因。相较于传统方法,功能宏基因组主要有以下优点:(1)不需要培养微生物;(2)不需要事先对耐药基因(antibiotic resistance gene,ARG)序列进行研究;(3)有耐药表型的克隆和测序得到的ARGs直接相关[32]。因此,功能宏基因组学技术成为揭示新型抗生素耐药基因并识别其功能的关键手段,为研究抗生素耐药基因的传播机制和作用方式拓宽了道路。
2.2 功能宏基因组学在微生物领域的应用
自创立以来,功能宏基因组方法已被运用于多种环境中识别许多新的基因,比如在发现编码碳水化合物酶基因过程中,利用功能宏基因组学将UDP-葡萄糖-4-差向异构酶鉴定为甲萘醌的抗性蛋白,加速了抗性功能新基因的发掘[33]。到目前为止,人体肠道微生物组中仅有约20% 的细菌物种被成功培养[24]。得益于基因组和宏基因组序列的大规模分析应用,大量与人类肠道微生物组中主要功能相关的蛋白质家族基因被挖掘出来[34-39]。例如在已有报道中,利用功能宏基因组方法从肠道细菌中成功鉴定出大量β-D-葡萄糖醛酸酶,扩大了已知的具有葡萄糖醛酸酶活性功能的酶的数量,同时也突出了肠道微生物组内葡萄糖醛酸酶多样性[40]。借助宏基因组学方法对复杂微生物群落的独特鉴别优势,Culligan等利用功能宏基因组学方法对人类肠道菌群中耐盐基因进行了筛选,研究结果有助于我们研究人类肠道菌群对渗透压的抵抗能力[41-44]。还有报道对工业甘蔗废渣进行采样并建库,发掘出新的编码糖基水解酶的基因[45],这证明了未培养微生物作为重要生物资源的巨大潜力。此外,依托于功能基因组技术的发展,编码氢化酶的基因[46]以及编码细菌磷酸酶的基因[47]也得到了发掘。由此可见,功能宏基因组在基于特定功能或表型上的新型基因发掘具有显著优势与应用价值,大量相关研究也为后续服务于耐药基因发掘与表征奠定了理论与实践基础。
2.3 功能宏基因组学在发掘耐药基因方面的应用
功能宏基因组学方法具有靶向特定表型进行特异性测序分析的优势,因此逐渐拓展到研究微生物耐药基因发掘领域。近年来,利用功能宏基因组学方法,大量新型耐药基因得以揭示[48-51]。更有报道指出,功能宏基因组能够对土壤中各类抗性蛋白进行高效筛选,这极大地丰富了在抗菌过程中各种抗性蛋白的发掘[52]。除了抗性基因本身,功能宏基因组还被用于发掘介导耐药性传导的可移动元件,利用其高通量的优势系统性地分析质粒的宏基因组,拓宽了宏基因组技术在耐药质粒上的应用[53]。
在一项最近的研究中,Martiny等[48]利用功能宏基因组技术研究海鸥栖息地的耐药基因多样性和遗传背景,并对野生动物中的β-内酰胺酶类耐药基因进行发掘。结果显示,共有31个未报道过的新型β-内酰胺酶类耐药基因被检出,暗示了多种耐药基因可能已经在野生动物体内驻留并发生潜在的进化。这项研究也表明了抗生素耐药性极有可能通过野生动物为媒介在人类主导的栖息地和周围环境之间进行传播。此外,还有通过功能宏基因组学阐明土壤微生物群中碳青霉烯酶编码基因的发生、多样性和功能的异质性。Gudeta等[49]利用功能宏基因组学方法对土壤样品中的微生物进行分析,发现了9种不同的金属-β-内酰胺酶(MBL),能够快速降解包括碳青霉烯在内的多种β-内酰胺类抗生素。有趣的是,功能宏基因组学产生的 MBL通常与基于土壤中可培养细菌中分离鉴定的MBL不同,表明这两种方法针对了土壤微生物群中的不同亚群。这意味着功能宏基因组学方法在分离抗生素耐药基因亚型方面有着独特的优势。
在针对四环素耐药基因发掘方面,功能宏基因组学方法同样被广泛应用。Gasparrini等[50]利用功能宏基因组技术首次报道了四环素类失活酶家族tet(X7),加深了学界对四环素降解酶类耐药基因的认知。此外,还有研究通过对暴露于抗菌剂合成的废水环境取样,并构建功能宏基因组文库,利用多种抗生素对文库进行筛选,成功鉴定了编码新型tetA型外排泵的基因以及新型1类整合子携带抗生素耐药基因,该tetA型外排泵被命名为tetA(62)[51]。这项研究通过功能宏基因组技术证明了工业废水的排放影响环境中存在的抗性基因的多样性和传播潜力,并为防控四环素耐药性的生成与传播提供了理论基础。
在针对土壤中抗性蛋白探究的过程中,通常存在由于编码抗性蛋白的基因在整个微生物基因组中的低丰度导致无法精确检测的问题。因此McGarvey等[52]利用功能宏基因组技术表征土壤中可能存在的抗性蛋白,通过表型筛选富集并揭示了几个抗生素抗性蛋白家族中天然存在的变异,具体包括ADP-核糖基转移酶、二氢叶酸还原酶、氨基糖苷类乙酰转移酶和转运蛋白。这些功能基因能够协助微生物获得针对特定抗菌药物的抗性。此研究的开展增加了学界对环境细菌中抗性决定因素的认识,展示了编码抗性决定因素基因序列的多样性,有助于对这些抗性蛋白进一步的探究。
除了细菌中可能存在的新型耐药基因,功能宏基因组还可以对固定生态位中的多种微生物群落进行耐药基因发掘。Moon等[54]对汉江水体中的病毒微生物组进行研究,通过功能宏基因组学手段建立文库对病毒组中的潜在耐药基因进行广泛筛查,发现了多种β-内酰胺酶、多药转运蛋白、多黏菌素抗性蛋白和万古霉素抗性蛋白基因,其中β-内酰胺酶甚至可以催化特定碳青霉烯类药物的降解。由于水体环境中存在较高的噬菌体丰度,这些病毒组中存在的耐药蛋白可能是由整合在噬菌体中的功能性基因编码。在淡水病毒组中发现这些新的抗性基因表明环境噬菌体可能是被长期忽视的耐药基因的重要储库。
如上文所述,功能宏基因组技术还可被用于针对菌群中新型质粒的鉴定,深入挖掘可介导耐药性传导的可移动元件。Zhang等[53]利用污水处理厂附近耐药基因高度富集这一特点,对所采的污泥样品进行质粒宏基因组分析并发现编码四环素(27.2%)、大环内酯(25.0%)和多药(24.9%)耐药性的ARGs在活性污泥中呈现高流行率。并且结合功能宏基因组研究发现这些ARGs可与毒力因子(VFs)一起被可移动遗传元件(包括质粒、整合子和转座子)携带,形成共转移的传播风险,并且这些携带耐药基因的可移动元件还呈现出较高的多样性和丰度。Marathe等[55]利用功能宏基因组技术,通过表型筛选结合下游的PacBio测序技术,不但筛选得到了几种新型碳青霉烯酶基因,还凭借PacBio测序深度优势完整描绘了耐药基因周围包括可以移动遗传元件在内的多种特殊基因环境与质粒类型,拓广了我们对于携带碳青霉烯酶耐药质粒的认知。
综上所述,功能宏基因组学因其结合表型筛选与高通量测序的特点,已在耐药性研究领域展现了巨大潜力并得到广泛应用,近年来利用功能宏基因组技术发掘或鉴定的耐药基因详见表2。
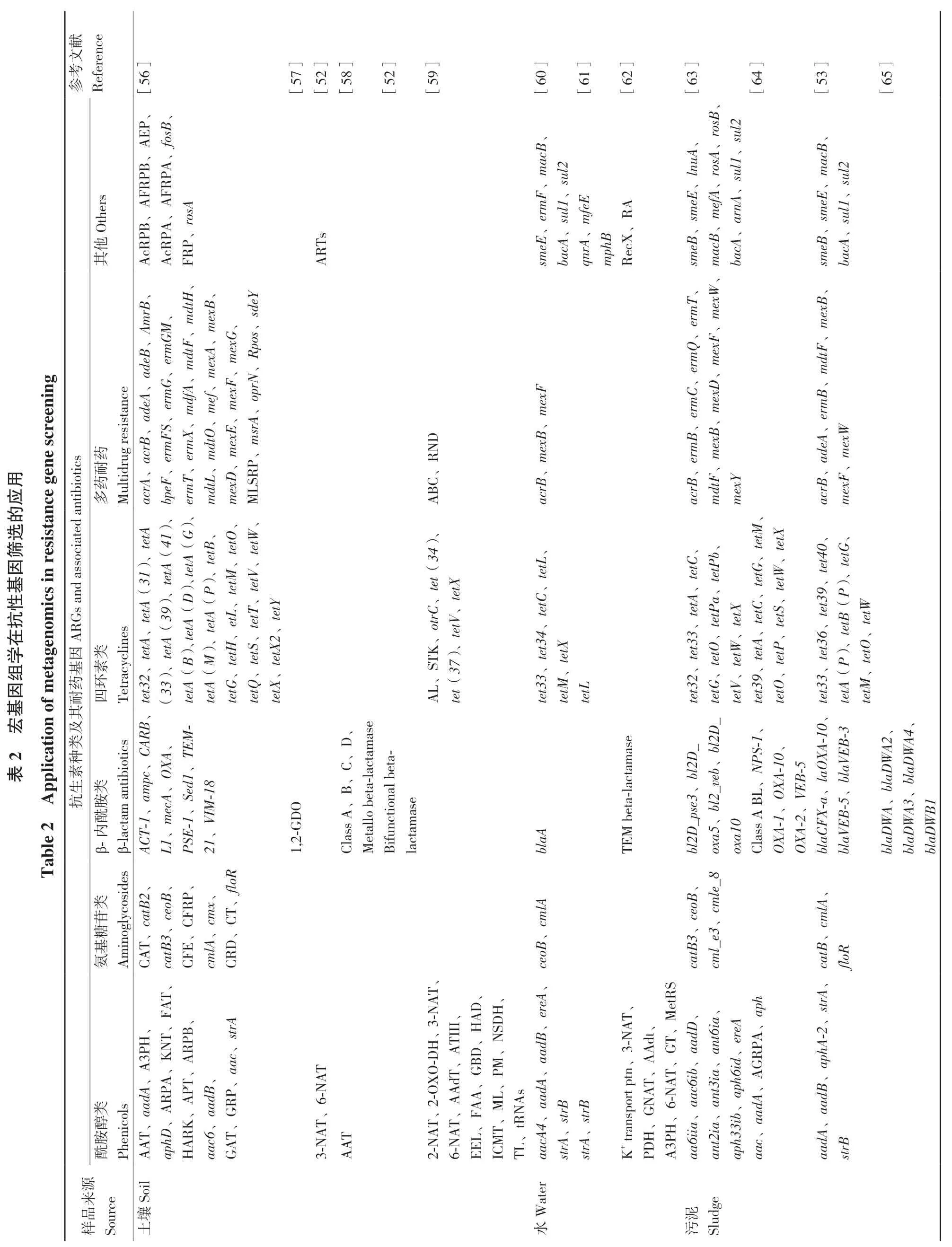
参Reference献文考[5 6]7][52][58][52][59][50][61][62][63][64][6[53][65]RP AE fosB、RP sul1、sul2 macB、B、l2ma A、A、AF Others AF B、P、rosB、A、rosA rn AcRP eE、erm、R P、FR feE m phB Ts AcRP A他eB、smeE、AR smeB、smeE、qnrA、m RecX sm其bacA、sul1、bacA、a cB、bacA、sul1、sul2 lnu A、macB、mefA、rosA、sm Am F、su tH、rB、M、mdexB、ermT、exG、Q、mexA、m mexB、dtF、Rpos、sdeY dtF、ermG e screening mexW、m ef、G、md mexF、m FS、ermfA、m mexF exE、msrA、oprN、ermC、erm adeA、adeB、m exD、mB、exF、ermB、m多Multidrug resistance药exW exB、crB、acrA、a药用耐ermT、ermdtO、D tL、m应nce gen m exD、mmd SR X、RN acrB、m bpeF、erm P、ML acrB、erm C、exB、AB tF、m mdacrB、adeA、mexY mexF、m性ics in resista的选筛tetA1)、tetW、tetX因tetA(G)、31)、34)、tetM、tetO、tetP tet40、Gs and associated antibiotics tetA、tetA(tetA(D)、tet(tetG、tetM、tetA(4 tetC、tetL、基tetA、tetC、b、tet33、tetP tetB、tetT、tetV、tet39、39)、在en、otrC、AR tetV、tetX tetXtetS、tetW、tetG、tet36、抗om tetW 37)、tetX、tetX2、tetY tet34、因类组etag D_ oxa 10 tet32、、STK a、药环因f m基表plication o tetA(P)、耐 四tet32、Tetracyclines基素tetA(B)、etL、tetA(M)、学tetG、tetH、tetQ、tetS、AL tet33、tet(tetL tetM、tetX tetG、tetO、tetV、tetW、tet39、tet33、tetA、tetC、tetB(P)、tetO、tetP、tetA(P)、tetM、tetO、类其2 宏 及antibioticsAR种B、L1、mecA、OXA、PSE-1、Sed1、TEM-、D M beta-lactam Ap pc、C、ase eta-lactam生类8 am nctional beta-、C素、N(33)、tetA(ase抗胺phClass A酰、B IM-1 β-lactam Table 2 ase T-1、Class A FX-a、laO 21、V Metallo b β-内WA、blaDW 1,2-GD Bifu O bl2D_oxa5、bl2_veb、bl2 lactam bl2D_pse3、ACblaA P S-1、OXA-1、OXA-10、OXA-2、VEB-5 TE BL X A-10、blaVEB-5、blaVEB-3blaC A 2、blaDWA3、blaDWA4、blaDWB1 blaD类苷CF糖CT catB 2、catB3、ceoB、、3、基氨Am inoglycosides RP T、CA E、CF lA、cmx、、floR cmCR D、ceoB、cmlA ceoB、cm l_e3、cm le_8catB tB、cm lA、floRca T、 、S D、FA AT HA H、SD-N TIII、ereA、、MetR、3-N strA T、c、aadA、aadB、3P、AD、PA RP tn、A、G PM aadA、A T、A Adt、aphA-2、strA、PT AT、3 6-NA 2-OXdT ATAT、NL、As GB aadD、a nt2 ia、an t3ia、an t6ia、aph33ib、aph6id、ereA、a aadA、A AAA、aa aa6iia、aac6ib、GR O-DH GR酰Phenicols类H、aphD、ARPA、KNT、GN、A醇3-NA P、2-NA T、T、FA RK、6-N T、T胺T、T、4、AA B、aac6、aadB、HA GA AA 6-NAL、、M EEMTICTL、tR NcAaa strA、strBstrA、strB K+ transport p H、PD A3PH aac、a adA、aa dB、strB源样Source来品Soil壤土W ater水污Slud泥ge
3 总结与展望
功能宏基因组学方法在新型基因检测的过程中展现出了巨大的价值和潜力,同时使人们能够接触到微生物世界的多样性,在不可培养的微生物研究方面做出了重大突破。但功能宏基因组学在技术上仍具有一定的局限性,其主要难点在于大片段文库的构建方法和宿主外源表达的问题[66]。作为功能宏基因组的理想宿主,其应具有支持多种基因外源表达的能力和转化率高的优点。虽然大多数研究均因易于培养而选择大肠杆菌,但大肠杆菌在外源基因表达时显示出了较大的局限性[26],只有极少数功能宏基因组学研究使用了大肠杆菌以外的宿主[67]。随着科研工作者的努力,多种用于鉴定具备天然优势的外源宿主及大型功能宏基因组文库构建方法被报道,以此来改进功能宏基因组文库的筛选方法[68]。随着这些材料和方法的优化也导致了越来越多有意义的化合物被发现。同时,蛋白质组学与功能宏基因组学相结合展现了良好的应用前景,使得每个DNA片段的潜在功能的筛选过程得以简化[69]。此外,测序技术的进步也极大地帮助了筛选出功能复杂的基因。随着测序深度的增加,或者长读数据技术发明(如Nano Pore Minion和PacBio SMRT),都将有助于解决功能宏基因组短读数据的组装所带来的相关问题,同时可以提供基因编码功能的更多信息,如多药耐药基因簇[69]。epicPCR(emulsion, paired isolation and concatenation PCR)也具有良好的应用前景[70],它与功能基因组学方法相结合后,可以将来自同一个宿主的两个基因连接成一个扩增子,再对扩增子进行测序。假如其中一个基因是16S rRNA基因,那么就可以对任意一个微生物群落中携带功能基因的宿主进行定位。高通量单细胞基因组测序技术可以同时分析多个细胞[71],这可能有利于功能基因组文库筛选得到的功能基因进行高通量测序。使 用 MALDI-TOFMS(matrix-assisted laser desorption/ionization time of flight mass spectrometry,基质辅助激光解吸电离飞行时间质谱)和MBT-ASTRATM的方法可快速检测单个细菌中的抗生素耐药水平[72],这可能将功能宏基因组筛选方法进一步优化。
综上所述,在细菌耐药形势逐渐严峻的今天,不断进化和广泛流行的耐药细菌对人类健康和社会经济构成重大威胁。因此越来越多的研究将焦点逐渐由来源于人类[73-74]和与人类密切相关环境中的耐药基因[75-76]转向环境中的土壤和水,甚至是野生动物这些耐药基因流行传播的重要环节。功能宏基因组学凭借其高通量、微生物无需培养等优点,在新型耐药基因的鉴别上展现了巨大的优势。随着文库构建技术的不断发展优化以及宿主外源表达问题的进一步解决,功能宏基因组学手段将在发掘新型耐药基因的研究中发挥更大的作用。
