CDCA7调控CCNB1IP1表达影响食管鳞癌发生发展的作用机制
2022-10-25李泓漪翁泳佳郭定和王少杰成晓龙
李泓漪,翁泳佳,郭定和,王少杰,成晓龙
1.山西医科大学转化医学研究中心,山西 太原 030001;2.山西医科大学基础医学院,山西 太原 030001
食管癌(esophageal carcinoma,ESCA)是全球第八大常见癌症类型[1],根据最新发表的2018年中国癌症报告,ESCA的发病率位居第6位,死亡率位居第4位[2]。根据病理学类型,将ESCA分为食管鳞癌(esophageal squamous cell carcinoma,ESCC)和食管腺癌(esophageal adenocarcinoma,ECA),腺癌为欧美国家的主要类型,占整个ESCA病例的70%~80%[3],而鳞癌为亚洲、非洲国家的主要发病类型[4]。我国90%左右的ESCA为鳞癌[5]。尽管随着诊疗技术的进步,ESCC的5年生存率有了一定提升,但全球仍维持在10%~30%的较低水平。我国癌症中心最新数据显示,ESCC 5年生存率约30%[6-7]。ESCC的发生严重威胁人类健康,且常见癌生物标志物在ESCC中的敏感性低、特异性差[8]。因此,深入研究ESCC的发生发展机理,探讨其早期诊断标志物显得尤为重要。
CDCA7基因为本课题组从31对ESCC的癌和癌旁配对样本中,通过全基因组测序发现的拷贝数扩增的基因之一[9]。已有文献报道,CDCA7基因在多种肿瘤中高表达,提示其在肿瘤的发生、发展中可能起关键作用[10]。同时,有文献报道,CDCA7基因可作为转录调控因子,促进三阴性乳腺癌的转移、侵袭[11],在肺腺癌中具有促进肺腺癌细胞增殖的作用[12]。本课题组前期在对CDCA7基因的研究中发现[9],敲除CDCA7基因可显著抑制ESCC的增殖及克隆形成能力,但具体的作用机制尚不明确。本实验探讨了CDCA7基因敲除和过表达后对ESCC细胞增殖表型及细胞周期的影响,并寻找CDCA7调控的下游靶基因CCNB1IP1,通过研究CDCA7基因表达水平变化后的一系列细胞周期蛋白和周期蛋白依赖性蛋白激酶表达水平的变化,分析其相关性以及相互作用机制,从而揭示CDCA7基因引起ESCC发生发展的机理。
1 材料与方法
1.1 细胞系、质粒及DNA人ESCC细胞系KYSE-450、KYSE150、KYSE180购自中国科学院上海生命科学研究院细胞资源中心;质粒pcDNA3.1-V5-HIS购自美国Addgene公司;质粒pGL3-Basic、pRL-TK购自美国Promega公司;KYSE150 cDNA和gDNA由山西医科大学转化医学研究中心实验室保存。
1.2 主要试剂及仪器 胎牛血清、胰蛋白酶、RPMI-1640培养基、青链霉素双抗混合液购自美国Gibco公司;TRIzol试剂、反转录试剂盒、荧光定量PCR Mix试剂盒、DNA聚合酶、DNA marker购自日本TaKaRa公司;内切酶、微量紫外分光光度计购自美国ThermoFisher公司;LipofectamineTM2000转染试剂购自美国Invitrogen公司;小干扰siRNA、对照NC、siRNA转染试剂购自广州锐博生物技术有限公司;双荧光素试剂盒购自美国Promega公司;Cell Counting Kit-8(CCK-8)试剂盒购自美国MCE公司;BCA蛋白分析试剂盒购自上海碧云天生物技术公司;CDCA7抗体(SAB4503307)购自美国Sigma公司;GAPDH抗体(60004-1-Ig)购自美国Proteintech公司;CCNB1IP1抗体(ab71977)、V5抗体(ab15828)、IRDye 800CW荧光二抗购自美国Abcam公司;PVDF膜、染色质免疫共沉淀试剂盒(17-295)购自美国Millipore公司;实时荧光定量PCR仪、普通PCR仪购自美国Applied Biosystems公司;核酸电泳仪、蛋白电泳仪、蛋白转印系统、核酸凝胶成像仪购自美国伯乐公司;冷冻非接触式超声细胞破碎仪购自美国Covaris公司;多功能酶标仪购自美国BIO Tek公司;双荧光素酶检测仪机购自德国BERTHOLD公司。
1.3 细胞培养 用RPMI1640完全培养基(含10%胎牛血清和1%双抗),在37℃,5% CO295%饱和湿度条件下培养细胞,隔天换液,待细胞生长密度为80%~90%时进行传代。
1.4 质粒构建 以KYSE150 cDNA和gDNA为模板,分别利用引物对CDCA7的CDS区(1 116 bp)、CCNB1IP1的CDS区(834 bp)及CDCA7与CCNB1IP1的DNA结合区域进行PCR扩增。引物序列见表1,引物由生工生物工程(上海)股份有限公司合成。反应条件:98℃10 min;98℃10 s,60℃15 s,72℃2 min,共30个循环。扩增产物经1%琼脂糖凝胶电泳鉴定并进行切胶纯化回收后,37℃,DNA内切酶酶切2 h,与经同样酶酶切的质粒pcDNA3.1-V5-HIS、pGL3-Basic,经T4 DNA连接酶4℃连接过夜;连接产物涂布LB板,16 h后挑取单克隆,菌落PCR及质粒测序(北京诺禾致源科技股份有限公司)鉴定过表达载体pcDNA3.1-V5-HIS-CDCA7、pcDNA3.1-V5-HIS-CCNB1IP1及双荧光素报告载体pGL3-Basic-CCNB1IP1。

表1 PCR扩增所用引物Tab.1 Primers for PCR amplification
1.5 siRNA和重组质粒转染 将KYSE150和KYSE450细胞按2×106个/孔接种10 cm培养皿,加入完全培养基至终体积为10 mL,当细胞密度达40%~50%时转染CDCA7siRNA,将1×ribo FEC-TTMCP Buffer 240 μL、20 μmol/L siRNA储存液10 μL、ribo FECTTM CP Reagent转染试剂20 μL混匀,室温静置15 min,加入10 cm培养皿,48~72 h后收集细胞。敲低细胞系分别命名为CDCA7-sh1/2组,对照细胞系分别命名为KYSE150/KYSE450-NC组。siRNA序列为:CDCA7-siRNA1:5'-GCCCTCAGAGAATTCTGTGACTGAT-3',CDCA7-siRNA2:5'-CATCCGTGACCCTTCCGCATATAAT-3';CCNB1IP1-siRNA1:5'-CGAAAGTGTCGCATCAAAC-3',CCNB1IP1-siRNA 2:5'-GGAGCGCAATCGTCAGTA-3'。
将KYSE180细胞按2×106个/孔接种10 cm培养皿,加入培养液至终体积为10 mL,当细胞密度达40%~50%时转染CDCA7过表达质粒,将1 mL Opti-MEM、24 μg pcDNA3.1-V5-HisB-CDCA7、pcDNA3.1-V5-HisB-CCNB1IP1以及50 μL转染试剂混匀,室温静置15 min,加至10 cm培养皿,48~72 h后收集细胞。过表达的细胞系为CDCA7-EXP组,阴性对照为KYSE180-NC组,分别用qRTPCR和Western blot法检测基因的敲除和过表达效率。
将CDCA7-sh组及对照组KYSE150细胞按0.8×105个/孔接种24孔板,当转染密度达40%~50%时,将800 ng pGL3-Basic-CCNB1IP1和2.4 ng内参质粒pRL-TK共转染至细胞中。
1.6 qRT-PCR用TRIzol试剂提取细胞总mRNA,反转录成cDNA(反转录体系:20 μL。反应条件:37℃15 min;85℃5 s),以其为模板,利用SYBR Premix EX Taq试剂盒进行qRT-PCR扩增。qRT-PCR所用引物见表2,引物由生工生物工程(上海)股份有限公司合成。反应条件:95℃10 min;95℃15 s;60℃50 s;共40个循环。以GAPDH为内参,采用2-ΔΔCt法计算产物相对表达量。

表2 qRT-PCR所用引物Tab.2 Primers for qRT-PCR
1.7 Western blot用含PMSF和蛋白酶抑制剂的RIPA裂解液提取细胞总蛋白,冰上超声裂解,4℃,10 000×g离心30 min,收集上清,BCA蛋白分析试剂盒进行蛋白浓度定量。经10% SDS-PAGE分离蛋白,湿转法(100 V,2 h)转移至0.45 μm PVDF膜上,5%脱脂牛奶室温封闭1 h;加入CDCA7(1∶1 000稀释)、CCNB1IP1(1∶1 000稀释)、GAPDH(1∶2 000稀释)抗体,4℃孵育过夜;TBST洗膜3次,加入IRDye 800CW荧光二抗(1∶10 000稀释),室温孵育2~3 h;TBST洗膜3次,显影。
1.8 CCK8试验 将对数生长期细胞按5×103个/孔接种96孔板,每组设5个复孔,分别于24、48、72、96、120 h时加入10% CCK8试剂,37℃孵育0.5 h;采用多功能酶标仪测定各孔在450 nm波长处的吸光度值,计算平均值和标准差。
1.9 硬克隆试验 将对数生长期细胞按1×103个/孔接种6孔板,用2 mL含10% FBS的RMPI1640培养基于37℃,5% CO2培养箱培养7~14 d;镜下观察到克隆形成后,弃培养基,4%多聚甲醛固定20 min;三蒸水清洗,结晶紫染色30 min;三蒸水清洗,观察形成的克隆并计数。
1.10 染色质免疫共沉淀测序(chromatin immunoprecipitation sequencing,ChIP-sequence) 按照染 色 质免疫共沉淀试剂盒说明书进行操作。将转染过表达载体pcDNA3.1-V5-HisB-CDCA748 h的KYSE150细胞,加入终浓度1%的甲醛交联,37℃孵育10 min;收集细胞,加入裂解液超声破碎(rc*750,功率25%,4.5冲击,9 s间隙,共14次),4℃,13 800×g离心10 min;收集上清,取100 μL,加入4 μL 5 mol/L NaCl至终浓度0.2 mol/L,65℃金属浴处理3 h,1.5%琼脂糖凝胶电泳鉴定解交联产物。取100 μL超声裂解液2管,分别加入900 μL ChIP Dilution Buffer和20 μL 50×PIC,再各加入60 μL ProteinA Agarose/Salmon Sperm DNA,4℃翻转混匀1 h;4℃,900×g离心1 min;取上清,1管加入1 μL V5抗体,1管不加,4℃翻转过夜;加入60 μL ProteinA Agarose/Salmon Sperm DNA,4℃翻转2 h;4℃,900×g离心心1 min;弃上清,缓冲液洗涤沉淀复合物,洗脱解交联,回收获得DNA片段,送北京诺禾致源科技股份有限公司测序。
1.11 双荧光素酶报告试验 将ChIP-sequence中分析得到的CDCA7与CCNB1IP1结合峰序列,构建至pGL3-Basic报告基因质粒中。将带有海肾荧光素报告酶基因(Rinilla luciferase)的pRL-TK质粒作为内参质粒,与重组质粒pGL3-Basic共转染至CDCA7-sh1和KYSE150-NC组细胞中,48 h后裂解细胞,取裂解液,分别检测萤火虫荧光素酶和海肾荧光素酶活性,计算萤火虫荧光素/海肾荧光素值,以此来表示双荧光素报告酶重组质粒的相对活性。
1.12 数据分析 通过美国TCGA数据库查阅CDCA7在各种肿瘤中的表达情况,并下载ESCC患者RNASeq数据,包括96例ESCC样本。同时获得CDCA7和CCNB1IP1在ESCC中的转录组数据,用GraphPad Prism 8.0软件进行相关性分析。用GEPIA数据库分析CDCA7在ESCA中的表达情况。
用LOGpc数据库对TCGA和GEO数据集的数据整合[13],将CDCA7按不同表达水平分组后,用Kaplan-Meier生存分析法分析患者的生存预后情况。
1.13 统计学分析 采用SPSS 19.0软件进行统计学分析。实验结果用均数±标准差(mean±SD)表示。多组计量资料的比较采用单因素方差分析(One way ANOVA),计量资料两组比较采用Student-t检验。以P<0.05为差异有统计学意义。
2 结果
2.1CDCA7在ESCA中的表达及临床预后情况GEPIA数据库中ESCA样本的表达数据显示,CDCA7在ESCA组织中相对于正常组织表达量升高,见图1。表明CDCA7可能作为一个癌基因在癌症中发挥作用。LOGpc数据库中的数据生存分析显示,CDCA7高表达的患者预后差,见图2。

图1 CDCA7在ESCA组织及正常组织中的表达情况Fig.1 Expression of CDCA7 in ESCA and normal tissues

图2 CDCA7表达水平的不同分组下ESCC患者的生存分析Fig.2 Kaplan-Meier survival analysis of patients with ESCC with various CDCA7 expression levels
2.2 ESCC细胞系中CDCA7敲低和过表达效率qRT-PCR和Western blot结果显示,转染siRNA序列的KYSE150和KYSE450细胞,即CDCA7sh1/2组,与KYSE150/KYSE450-NC组相比,CDCA7的mRNA和蛋白表达水平均明显降低(F分别为971 173、541.5、8 979、873.2,P均<0.001),见图3和图4;而转染过表达载体的KYSE180细胞,即CDCA7-EXP组,与KYSE180-NC组相比,CDCA7mRNA和蛋白表达水平均明显升高(t分别为9.503、38.61,P均<0.001),见图5和图6。

图3 KYSE150和KYSE450细胞中CDCA7敲低效率Fig.3 Knockdown efficiencies of CDCA7 in KYSE150 and KYSE450 cell lines

图4 KYSE150和KYSE450敲低细胞系中CDCA7蛋白表达水平的Western blot分析Fig.4 Western blotting of expression levels of CDCA7 protein in KYSE150 and KYSE450 knockdown cell lines

图5 KYSE180细胞系中CDCA7过表达效率Fig.5 Overexpression efficiency of CDCA7 in KYSE180 cell line

图6 KYSE180过表达细胞系中CDCA7蛋白表达水平的Western blot分析Fig.6 Western blotting of expression level of CDCA7 protein in KYSE180 overexpression cell line
2.3CDCA7对ESCC细胞增殖和细胞周期的影响CCK8和硬克隆试验结果显示,低表达CDCA7的KYSE150、KYSE450细胞,即CDCA7-sh1/2组,与KYSE150/KYSE450-NC组相比,增殖能力和克隆形成能力均明显减弱(F分别为9.386、219.7、55.89、707.4,P均<0.05),见图7和图8;高表达CDCA7的KYSE180细胞,即CDCA7-EXP组,与KYSE180-NC组相比,增殖能力和克隆形成能力均明显增强(t分别为10.53、93.34,P均<0.05),见图9。

图7 CCK8(A)、硬克隆试验(B)检测CDCA7对KYSE150细胞系增殖能力的影响Fig.7 Determination of effect of CDCA7 on proliferation of KYSE150 cell line by CCK8(A)and hard clone(B)assays

图8 CCK8(A)、硬克隆试验(B)检测CDCA7对KYSE-450细胞系增殖能力的影响Fig.8 Determination of effect of CDCA7 on proliferation of KYSE450 cell line by CCK8(A)and hard clone(B)assays
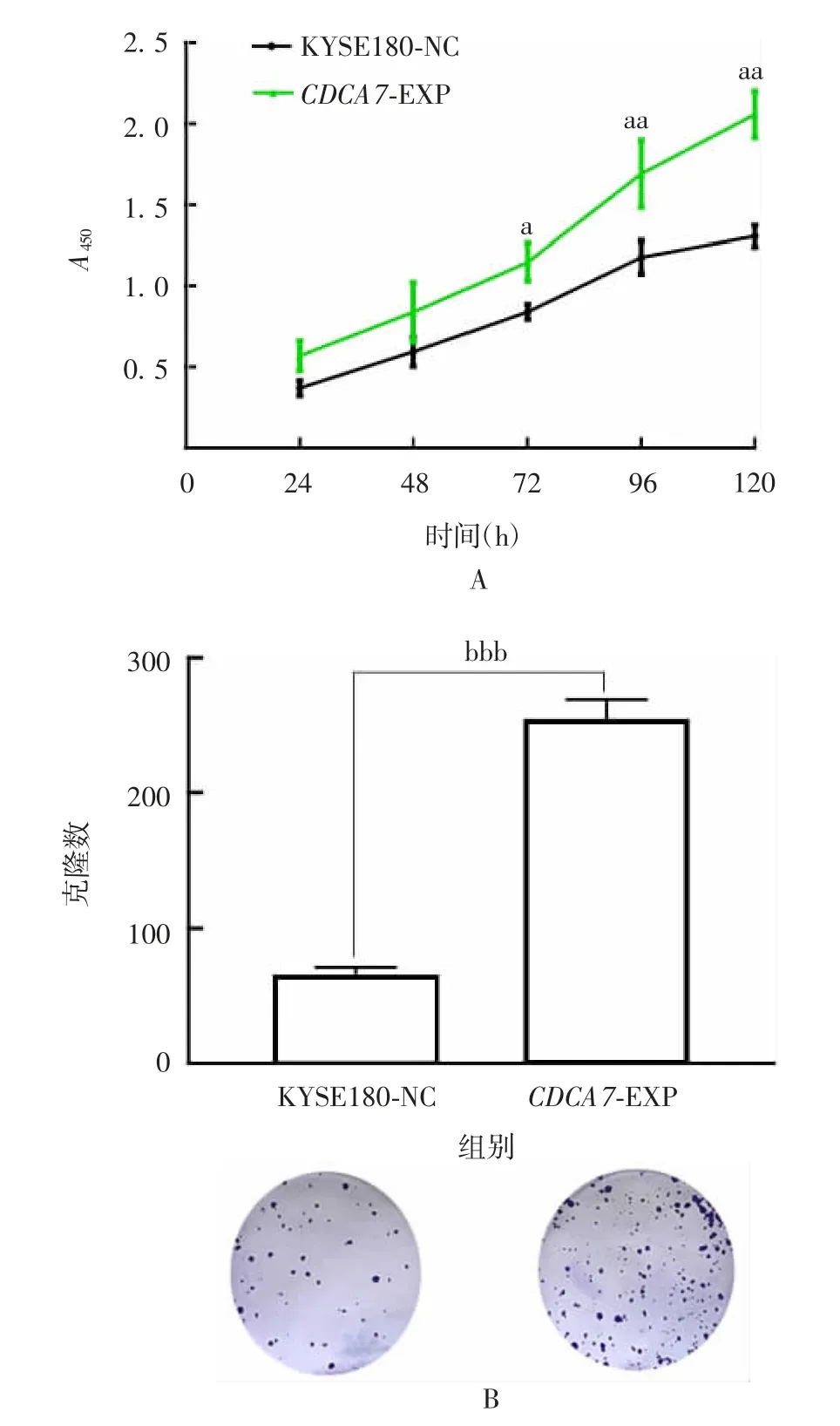
图9 CCK8(A)、硬克隆试验(B)检测CDCA7对KYSE180细胞系增殖能力的影响Fig.9 Determination of effect of CDCA7 on proliferation of KYSE180 cell line by CCK8(A)and hard clone(B)assays
2.4 不同水平CDCA7对CCNB1IP1的转录表达的影响ChIP-sequence结果显示,CCNB1IP1是能够与CDCA7结 合 的 下 游 靶 基 因,CDCA7与CCNB1IP1的-9 777~-10 182 bp区域结合,见图10。

图10 CDCA7与CCNB1IP1结合区域示意图Fig.10 Diagram of binding region of CDCA7 and CCNB1IP1
qRT-PCR和Western blot结果显示,CDCA7敲低的CDCA7-sh1/2组,与KYSE150/KYSE450-NC相比,CCNB1IP1的mRNA和蛋白表达水平均下调(F分别为808.1、14.99、396.1、5636,P均<0.05),见图11和图12;相反,CDCA7过表达的CDCA7-EXP组细胞,与KYSE180-NC组相比,CCNB1IP1的mRNA和蛋白表达水平均上调(t分别为5.556、43.79,P<0.01),见图13和图14。

图11 CDCA7敲低细胞系中CCNB1IP1表达水平Fig.11 Expression level of CCNB1IP1 in CDCA7 knockdown cell lines

图12 KYSE150和KYSE450敲低细胞系中CCNB1IP1蛋白表达水平的Western blot分析Fig.12 Western blotting of expression levels of CCNB1IP1 protein in KYSE150 and KYSE450 knockdown cell lines

图13 CDCA7过表达细胞系中CCNB1IP1表达水平Fig.13 Expression level of CCNB1IP1 in CDCA7 overexpression cell line

图14 KYSE180过表达细胞系中CCNB1IP1蛋白表达水平的Western blot分析Fig.14 Western blotting of expression level of CCNB1IP1 protein in KYSE180 overexpression cell line
皮尔森相关性分析结果显示,CDCA7与CCNB1IP1mRNA表达水平呈正相关(r=0.221 4,P<0.05),见图15。

图15 ESCC样本中CDCA7与CCNB1IP1 mRNA表达水平的相关性Fig.15 Correlation between mRNA expression levels of CDCA7 and CCNB1IP1 in ESCC samples
双荧光素酶报告试验结果显示,与KYSE150-NC组(82.42)相比,CDCA7-sh1组(4.0)细胞荧光素酶活性明显降低(t=9.656,P<0.001)。
3 讨论
本课题组前期ESCC组织及配对正常组织的组学测序发现,CDCA7基因所在区域存在显著且重复的拷贝数扩增;免疫组化和功能试验结果显示,ESCC组织中CDCA7表达量普遍高于其配对的正常组织,敲低CDCA7能够显著抑制ESCC细胞的增长速度。以上结果提示,CDCA7可能与ESCC的发生发展密切相关[9]。CDCA7又名JOP1,为细胞周期分裂相关蛋白家族成员之一,定位于2q31染色体。2005年就有文献报道,在人类多种肿瘤组织中,发现CDCA7高表达[10]。有研究显示,CDCA7可能通过影响细胞周期从而影响肺腺癌细胞的增殖生长[12];CDCA7可通过发挥转录调节因子的作用,正向调控EZH2基因的表达以促进三阴性乳腺癌细胞的侵袭和迁移能力[11]。但目前尚无ESCC中有关CDCA7的具体致病分子机制的研究报道。
细胞周期反应了细胞的增殖速度。哺乳动物的细胞周期分为G0/G1期、S期、G2期和M期,受不同细胞周期蛋白依赖性激酶(cyclin-dependent kinase,CDK)及其细胞周期蛋白(cyclin)控制[14],是一个有序而复杂的调控过程。cyclin具有调节CDK的功能,cyclin和CDK相互配合可精准调控细胞周期全过程。G1/S期和G2/M期恰好是细胞周期中2个重要检查点(check-point)。cyclin是调控细胞周期过程中最核心的分子,cyclin的表达调控,不仅保证了细胞周期的精准性,同时也介导了check-point功能。cyclin B在G1晚期开始合成,S期表达升高,于G2晚期和M期表达达到峰值并入核与CDK1结合形成复合物,该复合物称为成熟促进因子(maturation promoting factor,MPF),在G2末期引导细胞进入有丝分裂的M期[15-18]。在M末期,cyclin B经泛素途径分解,进入下一轮细胞周期或G0期。CCNB1IP1为E3泛素连接酶家族的成员之一,已有文献报道,CCNB1-IP1的缺失会抑制细胞增殖[19],其可通过与cyclin B相互作用并促进其降解来调节细胞周期进程。
肿瘤细胞中的细胞周期进程是高度紊乱的过程[20-23]。由于抑制基因的失活或突变以及癌基因的过度表达或扩增,可导致下游靶基因表达紊乱,影响cyclin的表达,从而引起不受控制的细胞周期进程和有丝分裂[24-25]。CDCA7在ESCC中存在高拷贝数和高表达现象。由于已有文献报道支撑CDCA7可发挥转录调控因子的作用[12],本研究通过ChIPsequence筛选出CDCA7转录调控的下游靶基因CCNB1IP1。当CDCA7高表达后,其可在转录水平上促进CCNB1IP1基因表达水平升高,而CDCA7敲低后则相反。双荧光素酶报告试验进一步明确了CDCA7与CCNB1IP1存在正向调控关系。当CCNB1IP1表达水平敲低后,ESCC细胞的增殖和克隆能力明显受到抑制;而当CCNB1IP1过表达后,则明显促进了ESCC细胞的增殖和克隆形成能力,从而促进细胞增殖速度,进一步加速ESCC的发生发展。但本研究有关ESCC中CDCA7下游靶基因CCNB1IP1对CCNB1产生的具体影响,以及是否通过影响CCNB1从而影响细胞周期,均仍需进一步深入研究。
综上所述,CDCA7基因为本课题组组学测序研究中发现的ESCC样本中拷贝数扩增的基因之一,其在ESCC肿瘤细胞中表达水平明显增高。CDCA7可通过转录调控CCNB1IP1基因的表达加速ESCC细胞的增殖及克隆形成能力,从而影响ESCC的发生发展。
