多重耐药胸膜肺炎放线杆菌GD2107株全基因组测序及生物信息学分析
2022-10-20徐民生柯海意杨冬霞施科达孙泽仪牛佳伟翟少伦臧莹安李春玲
徐民生,柯海意,杨冬霞,施科达,孙泽仪,牛佳伟,常 鑫,翟少伦,臧莹安,李春玲
(1.仲恺农业工程学院动物科技学院,广州 510305;2.广东省农业科学院动物卫生研究所,广东省畜禽疫病 防治研究重点实验室,农业农村部兽用药物与诊断技术广东科学观测实验站,广州 510640)
胸膜肺炎放线杆菌(Actinobacilluspleuropneumoniae,APP)是一种引起猪传染性胸膜肺炎(porcine contagious pleuropneumia,PCP)的革兰氏阴性杆状细菌,其定植于猪上呼吸道且具有高度传染性,可感染各年龄段猪并引发急性或慢性纤维出血性坏死性肺炎症状,严重情况下会导致死亡[1]。根据对烟酰胺腺嘌呤二核苷酸(nicotinamide adenine dinucleotide,NAD)的生长依赖性可将APP分为生物Ⅰ型和生物Ⅱ型[2]。Apx毒素在APP致病过程中起着重要作用,其中ApxⅣ在所有血清型的APP中均可分泌产生,可用于鉴别APP[3]。目前,基于衣壳多糖(CPS)的组成有19个公认的血清型,各血清型流行率具有一定的地理差异,治疗和防控此病仍依赖于抗生素和疫苗[4]。APP引起的急性或慢性病症导致较高的死亡率或治疗费用,给全球养殖业造成了重大损失[5]。
自1995年第一个完整的细菌基因组序列公布以来,全基因组测序技术(whole genome sequencing,WGS)得到了蓬勃发展[6]。从传统的Sanger测序到基于Illumina平台的第二代测序和PacBio平台的第三代测序,不仅提高了完整基因组的组装速度和质量,所需的时间和费用也逐步缩短和降低。全基因组测序技术作为一种有力测序工具在病原体的表征和流行病学调查中的运用也越来越多[7]。Li等[8]研究表明,全基因组测序技术可用来预测抗生素的耐药性,识别新的血清型以便用于研究药物和疫苗的作用靶点。全基因组序列正迅速成为预测APP生物信息的有利工具,尤其是对于了解相关菌株基因特征和进化关系具有重要意义[9]。
本研究以先前从广东省某发病猪场分离到的1株胸膜肺炎放线杆菌GD2107株为研究对象,药敏结果显示其具有多重耐药性。为进一步探索其致病和耐药机理,使用Illumina NovaSeq和PacBio SequeI测序平台进行全基因组测序和组装,通过生物信息学分析软件进行基因功能注释,了解其携带的耐药基因、毒力基因情况,分析菌株间亲缘关系,揭示菌株耐药与进化关系的相关性,以期为猪传染性胸膜肺炎的基础研究提供一个新的视角与借鉴。
1 材料与方法
1.1 材料
1.1.1 菌株来源 APP GD2107于2021年7月分离自广东省某规模化发病猪场感染传染性胸膜肺炎病死猪的肺部,由广东省农业科学院动物卫生研究所猪病研究室分离、鉴定和保存。
1.1.2 主要试剂与仪器 胰蛋白大豆肉汤培养基(TSB培养基)、烟酰胺腺嘌呤二核苷酸(NAD)均购自北京索莱宝科技有限公司;小牛血清购自广州蕊特生物科技有限公司;药敏纸片购自杭州微生物试剂有限公司;细菌基因组DNA提取试剂盒、2×FastTaqPremix酶、DL2000 DNA Marker均购自TaKaRa公司。电热恒温培养箱购自上海精宏实验设备有限公司;PCR仪购自杭州晶格科学仪器有限公司;电泳仪购自北京市六一仪器厂;显微镜购自Olympus公司。
1.2 方法
1.2.1 细菌培养及ApxⅣ基因扩增 将菌株GD2107冻存液以1∶100的比例加入含0.1% NAD和5%牛血清的TSB培养液中,37 ℃、220 r/min培养12 h。使用细菌DNA提取试剂盒提取基因组DNA。参考Zhang等[10]合成ApxⅣ基因特异性引物,引物序列为:F:5′-TTATCCGAA-CTTTGGTTTAGCC-3′;R:5′-CATATTTGATA-AAACCATCCGTC-3′,预期扩增片段长度为418 bp。引物由生工生物工程(上海)股份有限公司合成。以提取的基因组DNA为模板对菌株进行鉴定,PCR反应体系25 μL:2×rTaqMix 酶12.5 μL,上、下游引物(10 pmol/μL)各0.5 μL,DNA 模板1.0 μL,ddH2O 10.5 μL。PCR反应条件:94 ℃ 预变性5 min;94 ℃变性30 s,58 ℃退火30 s,72 ℃延伸1 min,共35个循环;72 ℃延伸10 min。PCR产物经1.0%琼脂糖凝胶电泳后,将ApxⅣ基因阳性PCR产物送至生工生物工程(上海)股份有限公司测序,菌株GD2107基因组DNA送至上海派森诺生物科技股份有限公司进行全基因组测序。
1.2.2 药敏试验 采用K-B药敏纸片法对分离菌株进行药敏试验。将分离菌株接种至含有5%牛血清及0.1% NAD的TSB培养基中,制成含菌量约为0.5麦氏浊度的菌悬液,吸取100 μL并用无菌棉签均匀涂布于TSA平板(含0.1% NAD和5%牛血清)表面,待液体吸干后用无菌镊子夹取抗生素药敏纸片贴在平板表面,置于37 ℃温箱中培养30 min,后倒置培养12~18 h,记录抑菌圈直径并参照杭州微生物试剂有限公司提供的判断标准进行结果判定。
1.2.3 全基因组测序 采用全基因组鸟枪法(whole genome shotgun,WGS)策略构建PE和S10K2个不同片段的文库,分别利用Illumina NovaSeq和PacBio SequeI测序平台对GD2107株进行全基因组测序。采用FastQc(v 0.11.7)[11]软件和AdapterRemoval(v 2.1.7)[12]和SOAPec(v 2.0)软件[13]对测序数据进行质量控制和过滤。使用HGAP(v 4.0)[14]和CANU(v 1.7.1)[15]软件进行拼装,得到contig序列后用pilon(v 1.22)[16]软件对结果进行校正,最终拼接得到完整序列。
1.2.4 基因组圈图绘制 首先将基因组序列、基因组预测及非编码RNA的预测信息整合成标准的GBK(GenBank)格式文件;然后采用cgview[17](http:∥stothard.afns.ualberta.ca/cgview_server)绘制该基因组的圈图;最后采用Photoshop CS对该图进行编辑。
1.2.5 功能元件分析 采用不同生物信息学分析软件对组装后的基因组、非编码RNA(ncRNA,主要类型包括sRNA、rRNA、tRNA、snRNA、snoRNA及miRNA等)、CRISPRs功能元件、原噬菌体(prophage)、基因岛(genomics islands,GIs)进行预测。用GeneMarkS(v 4.32)[18]软件预测GD2107全基因组基因序列;用tRNAscan-SE(v 1.3.1)[19]预测全基因组中的tRNA基因;用Barrnap(v 0.9)预测rRNA基因,其余ncRNA主要通过与Rfam(v 14.1)[20]数据库比对进行预测;用CRISPR finder(v 20170509)[21]预测全基因组中的正向重复序列和间隔区;用PhiSpy[22](http:∥phaster.ca)预测基因组中存在的原噬菌体;用IslandViewer4[23](http:∥www.pathogenomics.sfu.ca/islandviewer)预测基因组中存在的GIs。
1.2.6 蛋白编码基因功能注释 将所有预测得到的蛋白编码基因与数据库中包含的蛋白编码基因进行比对,若两者具有显著的序列相似性,则可初步确定其编码合成的蛋白具有相似甚至是相同的功能;采用diamond(v 0.9.10.111)软件完成序列比对[24]。序列比对的临界值选取为1e-6。序列的功能判别规则为:E-value<1e-6。
1.2.7 亚系统分析 对组装后的GD2107基因组进行碳水化合物活性酶(CAZy)、毒力因子(VFDB)和耐药因子(CARD)相关亚系统的信息学分析:采用hmmscan[25]软件与CAZy数据库比对来预测全基因组中存在的CAZy酶类基因;通过与VFDB[26]和CARD数据库[27]进行BLAST比对,预测存在的毒力基因和耐药基因。
1.2.8 系统进化树构建 将测序结果在GenBank数据库中进行BLAST比对。选取NCBI数据库中与GD2107株相似性相近的14条序列(表1),利用Mega 7.0软件构建系统进化树。
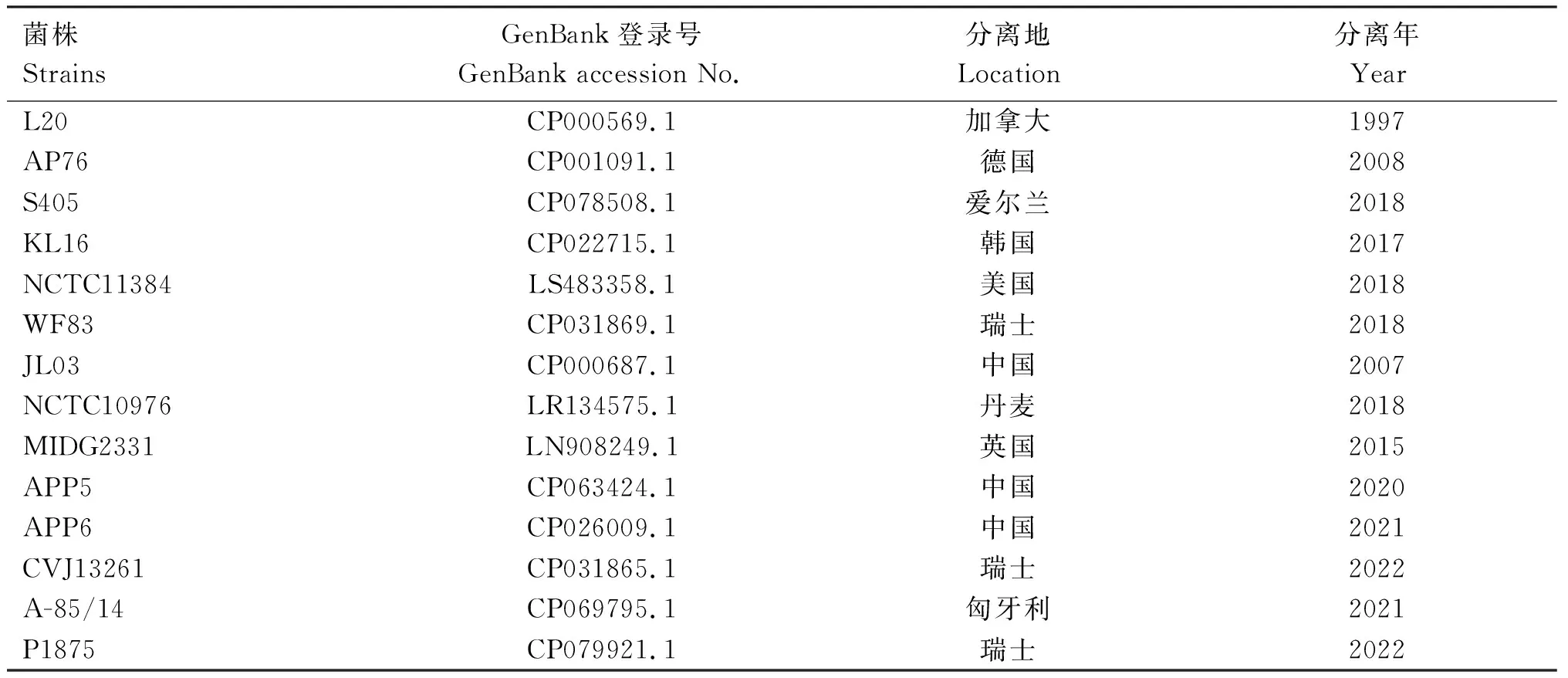
表1 参考菌株信息Table 1 Reference strain information
2 结 果
2.1 ApxⅣ基因扩增结果
采用针对APP菌种的特异性引物ApxⅣ基因扩增,结果获得大小为418 bp的特异性条带(图1),与预期结果相符。
2.2 药敏试验结果
药敏试验结果显示,菌株GD2107只对氨苄西林、阿莫西林/克拉维酸、头孢唑啉(先锋Ⅴ)敏感,对青霉素、头孢拉定(先锋Ⅵ)、卡那霉素、链霉素等14种抗菌药耐药(表2)。
2.3 GD2107株的基因组结构与分析
由图2可知,GD2107株的全基因组包括1条大小为2 271 987 bp大小的环状染色体,GC含量为41.21%,另存在2条大小为5 027和3 497 bp的环状质粒,GC含量分别为44.06%和36.06%。最后将基因组序列提交至NCBI数据库中,得到对应的染色体GenBank的登录号为CP097377,2条质粒的GenBank登录号分别为CP097378和CP097379。

M,DL2000 DNA Marker;-,阴性对照;+,阳性对照;1,分离株 M,DL2000 DNA Marker; -,Negative control; +,Positive control; 1,The isolate图1 分离株ApxⅣ基因PCR鉴定结果Fig.1 PCR identification results of ApxⅣ gene in the isolates

表2 药敏试验结果Table 2 Drug sensitivity test result mm

从内到外,第1圈代表刻度;第2圈代表GC Skew;第3圈代表GC含量;第4、7圈代表每一个CDS所属的COG;第5、6圈代表CDS、tRNA及rRNA在基因组上的位置 From inside to outside,the first circle represents the scale;The second circle represents GC Skew;The third circle represents GC content;The fourth and seventh circles represent the COG of each CDS;The positions of CDS,tRNA and rRNA in the genome in the fifth and sixth cycles图2 胸膜肺炎放线杆菌GD2107全基因组染色体圈图Fig.2 Circular map of Actinobacillus pleuropneumoniae GD2107 chromosome
测序得到的开放阅读框(ORF)数量有2 290个,总长度为1 972 470 bp,占全基因组的86.82%;在非编码RNA(ncRNA)中含有tRNA基因21个,rRNA基因19个(其中5S rRNA基因7个,16S rRNA基因6个,23S rRNA基因6个),其他ncRNA基因20个;使用PhiSpy预测基因组中存在的4个原噬菌体;经CRISPR finder预测发现菌株含有2组CRISPR相关序列;通过IslandViewer4预测到菌株GD2107基因组中有23个基因岛;基因组结构预测结果见表3。
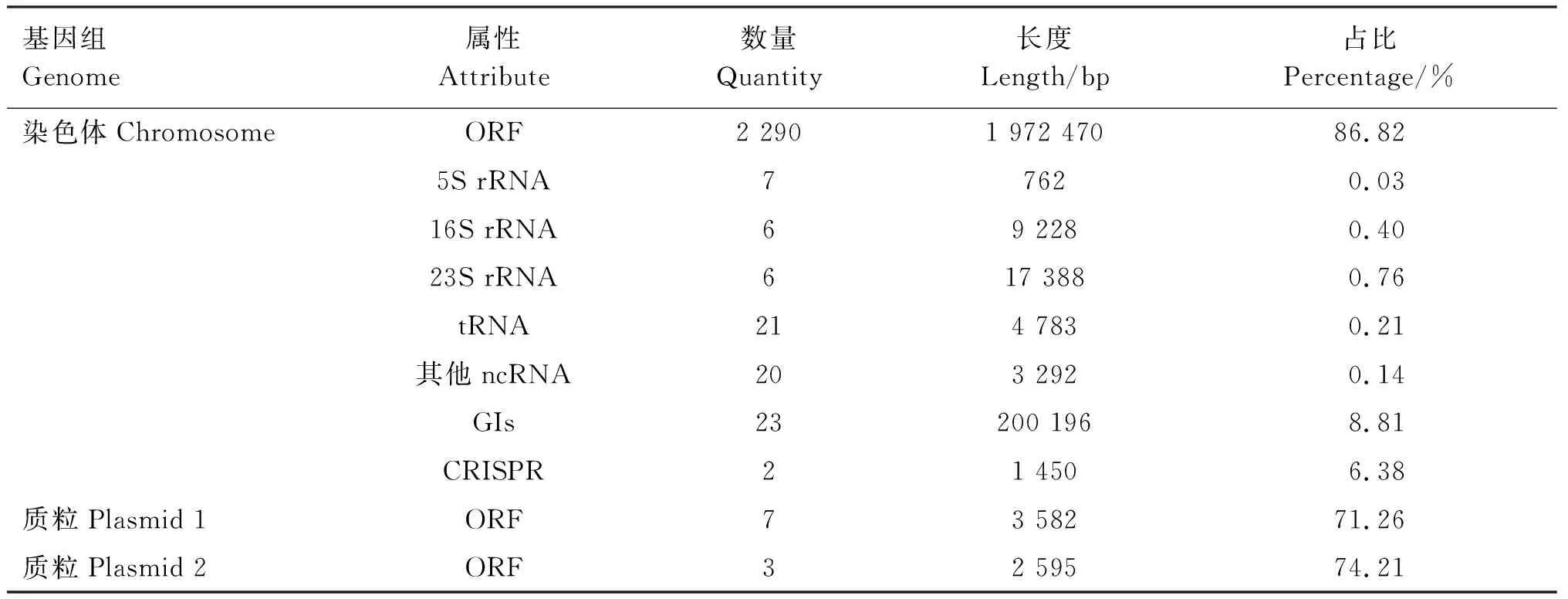
表3 基因组序列基本信息Table 3 Basic information of genome sequence
2.4 蛋白编码基因功能注释
将GD2107株基因的蛋白序列与表4中的功能数据库进行比对分析,对于每一条序列选取得分最高的比对结果进行注释;GD2107株的全基因组共有2 290个蛋白质编码基因,其中,分别有2 263、1 979、2 112、1 549、1 866、1 735、514、557个基因在NR、Swiss-Prot、COG、KEGG、GO、Pfam、TCDB、PHI数据库中得到注释(表4)。

表4 蛋白编码基因功能注释汇总Table 4 Summary of protein coding gene function annotations
2.4.1 COG数据库注释结果 结果显示,共注释到2 112个蛋白,占全部基因的92.23%,分布于25个COG的条目中(图3),其中,参与复制、重组和修复的基因有166个,占7.25%;参与翻译、核糖体结构和生物合成的基因有179个,占7.82%;与细胞壁、细胞膜、胞外被膜生物合成相关的基因有200个,占8.73%;参与氨基酸转运和代谢的基因有173个,占7.55%;与转录相关的基因有84个,占3.67%。

图3 COG功能分类图Fig.3 COG function classification diagram
2.4.2 KEGG和GO注释结果 GD2107菌株主要富集在碳水化合物代谢、膜运输、辅助因子、维生素代谢和氨基酸这些代谢途径,也是维持细菌新陈代谢所必需的。经KEGG注释可分为8类45个功能条目(图4),其分别注释为31.9%新陈代谢、7.1%遗传信息处理、6.5%环境信息处理、3.1%细胞过程、2.9%人类疾病及1.6%生物体系统,其中每一类型又包含有各自的亚型。
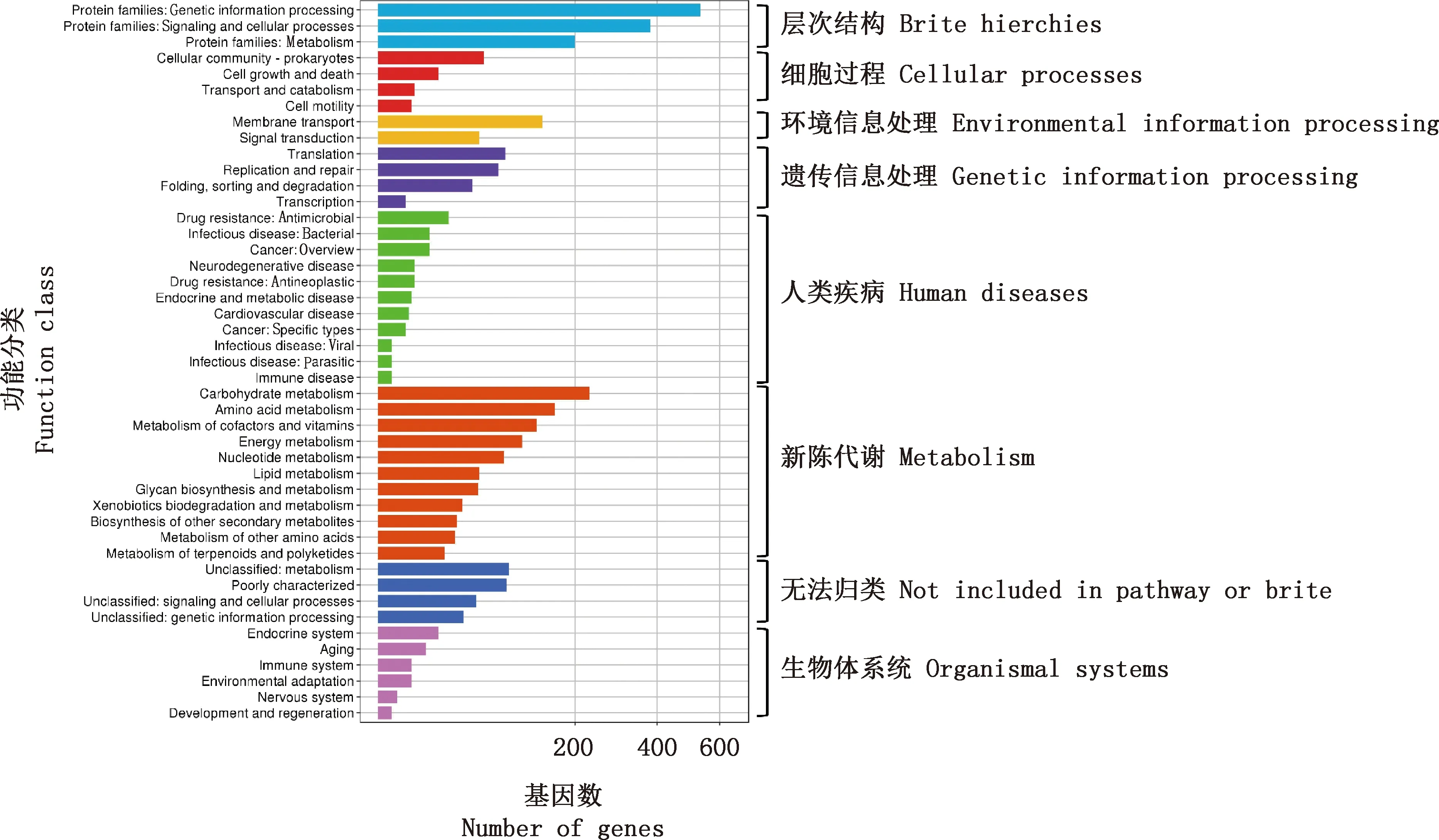
图4 GD2107基因组KEGG功能注释结果Fig.4 Results of KEGG functional annotation of GD2107 genome
通过GO注释,共有1 866个基因,其基因产物共分为三大类:生物过程(biological process,BP)占45.4%;分子功能(molecular function,MF)占23.1%;细胞组分(cellular component,CC)占31.5%(图5)。表明该菌株的基因产物主要集中在生物过程方面,其中大量基因与生物过程、细胞氮化合物代谢、生物合成和小分子代谢有关;在细胞组分方面,GD2107主要与细胞、细胞位置、胞外区、细胞质和浆膜等相关;在分子功能方面,主要与分子功能、离子结合等相关。

图5 GD2107基因组GO功能注释结果Fig.5 Results of GO functional annotation of GD2107 genome
2.4.3 TCDB和PHI注释结果 TCDB第一级统计结果见图6。由图6可知,共有514个基因注释到TCDB数据库的9个功能分类中,分别为:通道/孔隙(channels/pores)80个基因;电化学势驱动转运体(electrochemical potential-driven transporters)116个基因;主要活性转运蛋白(primary active transporters)221个基因;转录组(group translocators)21个基因;跨膜电子载体(transmembrane electron carriers)17个基因;运输中涉及的附属因素(accessory factors involved in transport)6个基因;不完全表征的运输系统(incompletely characterized transport systems)53个基因。
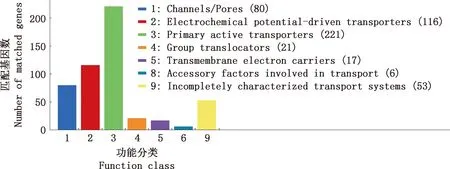
图6 TCDB功能分类图Fig.6 TCDB functional classification diagram
病原体PHI表型突变类型基因数目的统计情况见图7。由图7可知,这557个基因中有3个突变后致病菌株具有化学耐受性、2个具有化学敏感性、3个突变成效应子、42个突变后毒力增加、39个丧失致病性、338个突变后毒力降低、122个突变后不受影响。

图7 病原体PHI表型突变类型分布图Fig.7 Distribution of PHI phenotype mutation types of pathogens
2.5 亚系统分析结果
2.5.1 CAZy数据库注释结果 由图8可知,GD2107株中有72个基因注释到CAZy数据库中,其中包含与糖基转移酶(GTs)相关基因35个、多糖裂解酶基因2个、糖类脂解酶基因10个、氧化还原酶基因6个、碳水化合物基因5个、糖苷水解酶基因14个。

图8 CAZy功能分类图Fig.8 CAZy function classification diagram
2.5.2 VFDB基因预测分析结果 将GD2107株全基因序列与致病菌毒力因子综合数据库VFDB进行比对,共注释到48个毒力基因,其中毒力因子脂寡糖(lipooligosaccharide,LOS)和荚膜(capsule)的基因占比最大(70.8%)。质粒中并不存在毒力基因。
2.5.3 CARD分析 将测序序列与CARD进行比对分析,共获得了22个耐药基因,分别为aph(3″)-Ⅰb、macA、macB、sul3、tet34、tet35、tetO、parC、parE、gyrA、gyrB、floR、rpoB、rpoC、dfrA3、hns、tuf2、golS、folP、crp和cpxR,且分别对应表5中的抗菌药,其中氟苯尼考药物的耐药基因floR位于质粒上。

表5 GD2107株耐药基因及相对应抗菌药Table 5 Resistance genes and corresponding antibiotics of GD2107 strain
2.6 系统进化树分析
将PCR扩增得到的APP菌株经ApxⅣ基因扩增后进行测序,并将测序结果与14株参考菌株进行序列相似性比对分析,系统进化树结果显示,GD2107株与中国APP5株(CP063424.1)进化关系最近,处在同一分支;其次是中国分离株JL03株(CP000687.1);与韩国KL16、瑞士CVJ13216、瑞士WF83、爱尔兰S405等地的分离株进化关系均较近;而与美国NCTC11384、英国MDG2331、加拿大L20、匈牙利A-85/14、德国AP76、中国APP6、瑞士P1875、丹麦NCTC10976等APP分离株进化关系较远(图9)。

图9 GD2107分离株基于ApxⅣ基因序列构建的系统进化树Fig.9 Phylogenetic tree of GD2107 isolate based on ApxⅣ gene sequence
3 讨 论
APP是一种临床上常见的呼吸道病原菌,已成为目前影响猪场经济效益的重要细菌性疾病之一[28]。近年来随着饲料“禁抗”政策的推行和养殖场抗生素的滥用导致耐药性APP的滋生和扩散,这提示应长期对APP耐药性进行监测,尽量减少由APP感染引起的潜在危险[29]。
全基因组测序技术的快速发展给研究微生物多样性、进化及物种间相互作用带来了新的手段,利用全基因组测序技术进行DNA水平的测序分析已成为近年来的发展趋势[30],尽管细菌基因组信息的获取变得越来越容易,然而对APP完整基因序列的报道和研究相对较少。本研究所用GD2107株是从临床上1头发病猪的肺脏中分离得到的多重耐药菌株。通过全基因组测序和生物信息学分析了解GD2107株的基因组结构和功能,丰富了APP的基因组数据库,为进一步探明其致病机制和耐药机制提供重要的生物信息学依据。
通过PacBio及Illumina测序平台的相互结合,高效并准确地完成了对GD2107株全基因组的测序。测序到的蛋白质编码基因经GO、KEGG和COG数据库注释分析发现,3个数据库的注释结果高度一致,基因富集程度最高的都在代谢过程,这说明了代谢过程对于维持细菌的生命至关重要[31]。碳水化合物结合结构域能通过结合CAZy的底物提高CAZy催化底物的活性[32]。从注释结果可知,糖基转移酶和糖苷水解酶在GD2107株的碳水化合物活性酶中占比较大(68.1%)。特定的糖苷水解酶可使结构多糖酶解,这是碳水化合物跨环境转运的基本过程,而糖基转移酶是参与合成天然糖基结构的CAZy,高效的糖基转移酶对天然产物的糖基化至关重要,会影响其药理特性,从而对宿主的临床治疗起指导作用,也为生物技术和制药开辟了广阔的可能性[33]。
毒力因子是衡量微生物毒性的重要指标,是引起宿主发病的重要因素[34]。GD2107株有48个毒力因子得到注释,其中毒力因子中脂寡糖和荚膜的基因最多(70.8%)。由黏性表层组成的荚膜是APP的基本结构成分和致病因子,在感染过程中为保护细菌免受宿主防御提供了一种免疫学机制[35]。脂寡糖是革兰氏阴性菌细胞壁上的一种结构成分,同脂多糖结构相似,生物功能也相近。其他被注释的毒力因子还包括溶血素和黏菌素,与APP产生相互依赖的代谢生态系统相关,与蛋白酶相关的毒力因子具有防止吞噬作用和诱导脓肿形成的胶囊的作用,水解酶和脂多糖等毒力因子与降解免疫球蛋白和补体成分有关[36]。在毒力基因中,由某些基因共同调控形成的生物膜构成了抗生素耐药的第一道防线。
APP的多重耐药表现通常与其同时携带多种耐药基因有关。Bossé等[33]和Liu等[37]研究显示,除了大环内酯类药物外,全基因组测序技术可准确地预测APP对所测试的抗菌药的耐药性。经全基因组测序技术分析发现,在GD2107株中存在22种耐药基因。四环素是最早用于临床治疗PCP的主要药物,APP对四环素的耐药现象也已被广泛发现[38]。本试验药敏检测结果和测序到的耐药基因(tetO基因等)同样显示,GD2107株对四环素类药物耐药。Chang等[39]报道位于质粒上的耐药基因blaROB-1可介导APP菌株间阿莫西林的耐药性。GD2107株携的耐药基因(floR基因)也位于质粒上,推测可能与APP自身的耐药性和不同菌株间的传播有关,这增加了临床防控APP等细菌感染的难度。Kwong等[40]对肺炎克雷伯菌研究显示,抗生素的耐药基因在人类携带的分离株中也很常见,这些感染通常缺乏与侵袭性疾病相关的基因;COG注释结果中GD2107株存在大量与基因重组、基因转移相关的基因。这提示毒力与抗性基因的整合可能会导致变异强毒株的出现。因此,未来在疫苗研发时,除了需要充分了解菌种的生物学信息外,也需敲除其自身所携带的耐药基因和毒力基因等,以防止或减少细菌水平基因转移和转化,保证疫苗产品的安全高效[41]。
本研究通过构建GD2107株的基因ApxⅣ系统进化树发现,GD2107株与来自中国的APP分离株(CP063424.1)处在同一分支,进化关系最近;同国外的APP分离株进化关系较远,较符合疾病的流行规律,这与Donà等[42]研究APP的流行具有一定的地域流行性这一结论一致,为该病的流行病学调查提供了一定参考。
4 结 论
本研究完成了对菌株GD2107的全基因组测序与生物信息学分析,全面认识了该菌株基因组的结构和功能并探究了与耐药和致病机制中的相关基因,结果表明菌株GD2107呈多重耐药性且携带多种毒力和耐药基因,进化关系分析发现该菌株与国内分离株亲缘关系最近,具有一定的地域流行性。本研究为预防PCP的疾病流行和探索APP的致病机制提供了参考。
