3种单细胞全基因组扩增方法对1~4Mb拷贝数变异检测性能的研究
2022-10-09于婷王云云费嘉黄杰胡泽斌
于婷 王云云 费嘉 黄杰★ 胡泽斌★
拷贝数变异(Copy number variations,CNV)是胚胎植入前非整倍体遗传学检测(Preimplantation Genetic Testing for Aneuploidies,PGT-A)的重要检测对象。据Wang 等[1]对3 398 例羊水样本检测发现,3%(102/3 398)的样本存在致病性拷贝数变异,其中0.97%(33/3 398)CNVs 片段小于5 Mb。近年的研究发现,这些1~4 Mb 范围的CNV 可能也会带来严重的后果,比如被统称为微缺失/微重复综合征(microdeletion and microduplication syndromes,MMS)[2]的DiGeorge 综合征(22q11.2)、Wolf-Hirschhorn 综合征(4p16.3)、Prader-Willi/Angelman综合征(15q11-q12)、Smith-Magenis 综合征(17p11.2)等。然而目前对这些可能致病性的小CNV 多在流产物、患者血样、羊水提取物中进行分析,在预防遗传疾病最前端的PGT-A 检测中,却鲜有涉及。
PGT-A 检测方法有效应用的前提是单细胞全基因组扩增技术(whole genome amplification,WGA),其原理是对分离的单个细胞的微量全基因组DNA 进行扩增,获得高覆盖率的完整的基因组后进行高通量测序。因此,获得高覆盖度高保真性的全基因组扩增产物是准确全面的测序结果的保障。全基因组扩增技术从1992年出现,至今已开发了多种方法,主要有三类:一类为基于聚合酶链式反应(polymerase chain reaction,PCR)技术的WGA 方法,如简并寡核苷酸引物PCR(degenerate oligonucleotide primed PCR,DOP-PCR)[3]、SurePlex等;第二类为多次退火环状循环扩增(multiple annealing and looping-based amplification cycles,MALBAC)[4];第三类为恒温全基因组扩增反应,如多重置换扩增(multiple displacement amplification,MDA)[5];目前在临床使用的WGA 主要有3种:SurePlex、MALBAC 及MDA,且针对的是4 Mb以上分辨率PGT-A 分析。对于1~4 Mb 小CNV的准确检测,哪种方法性能更优,尚无太多文献报道。本文拟采用含有已知1~4 Mb 的CNV 培养细胞系样本,对上述3 种WGA 方法的检测性能进行综合评估,为PGT-A 选择适宜的方法提供数据支持。
1 材料与方法
1.1 细胞系
3 种含有1~4 Mb 的CNV 细胞系分别为:GM24312,XX,del(17)(p11.2-p11.2)(3.59 Mb);GM13325,XX,del(22)(q11.2-q11.2)(2.5 Mb);GM25372,XY,del(17)(p11.2-p11.2)(1.3 Mb),购自美国Coriell Institute for Medical Research 公司(https://catalog.coriell.org)。
1.2 主要试剂
WGA 试剂:SurePlex DNA Amplifiction System(美国Illumina 公司,货号:PR-40-415101-00,批号:002005028)、MALBAC 单细胞全基因组扩增试剂盒(亿康公司,货号:YK001B,批号:H210426)、Discover-sc single cell WGA kit(MDA)(诺唯赞公司,货号:N603-02,批号:7E551B1);文库构建试剂:TG DNA Library Prep Kit(Veriseq)(美国Illumina 公司,货号:20029274);定量试剂:Qubit 1X dsDNA HS Assay Kit(美国ThermoFisher 公司,货号:Q33231,批号:2342798);测序通用试剂(美国Illumina 公司,货号:FC-404-2005)。
1.3 主要检测仪器
QubitTM3 Fluorometer,货号为Q33216,美国ThermoFisher 公司;Life ECO 基因扩增仪,型号为TC-96/G/H(b)C,杭州博日公司;高通量测序仪,型号为NextSeq550,美国Illumina 公司生产。
1.4 方法
分别培养3 种细胞系,并以3~5 个细胞/管进行分装,每管样本体积不超过2 μL。严格按照3种WGA 试剂盒说明书操作,进行WGA 扩增,每种方法每个细胞系样本进行9 次重复,3 种细胞系共计27 个测试样本。使用Qubit 试剂对扩增产物进行定量,扩增产物按照Veriseq 试剂盒说明书操作,构建文库,并对建库产物浓度定量,最后使用Nextseq550 测序平台测序,采用SE75 试剂盒测序,以期每个样本获得10 M 以上有效reads。对下机数据(包括基因组比对率、已覆盖基因组比例、冗余序列比例、SD 值的数据资料),采用嘉宝仁和PGXCloud 生信分析平台结合散点图人工核对,进行PGT-A 分析。
1.5 统计学方法
采用SPSS 26.0 软件进行统计学分析;计量资料以(±s)表示,两组间比较采用t检验,三组间比较采用单因素方差分析;以P<0.05 为差异有统计学意义。
2 结果
2.1 3 种WGA 方法基本性能比较
SurePlex、MALBAC、MDA3 种方法在WGA 及以其产物进行文库构建中,均可获得足够的WGA产物,并成功构建了测序文库,满足后续PGT-A 测序分析要求。见表1。扩增产物浓度中,MDA>SurePlex>MALBAC,差异有统计学意义(P<0.05);建库产物浓度中,MDA>MALBAC>SurePlex,差异有统计学意义(P<0.05)。
表1 3 种WGA 方法基本情况比较(±s)Table 1 Comparison of three WGA methods'basic information(±s)
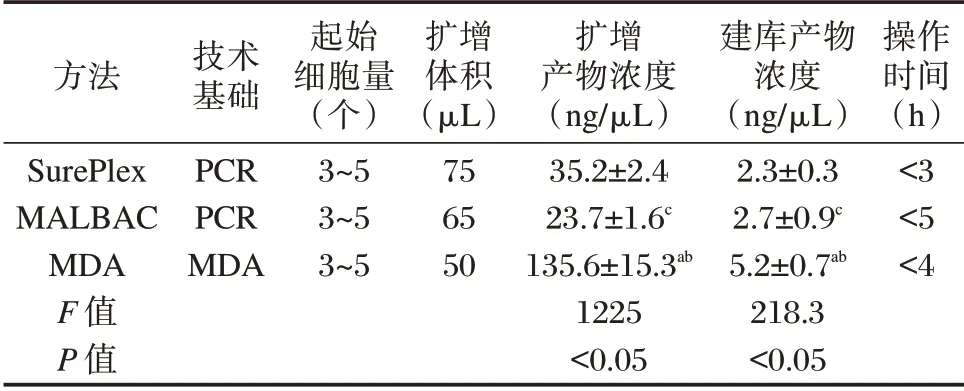
表1 3 种WGA 方法基本情况比较(±s)Table 1 Comparison of three WGA methods'basic information(±s)
注:MDA 组与SurePlex 组比较,aP<0.05;MDA 组与MALBAC 组比较,bP<0.05;MALBAC 组与SurePlex 组比较,cP<0.05。
方法SurePlex MALBAC MDA F 值P 值技术基础PCR PCR MDA起始细胞量(个)3~5 3~5 3~5扩增体积(μL)75 65 50扩增产物浓度(ng/μL)35.2±2.4 23.7±1.6c 135.6±15.3ab 1225<0.05建库产物浓度(ng/μL)2.3±0.3 2.7±0.9c 5.2±0.7ab 218.3<0.05操作时间(h)<3<5<4
2.2 3 种WGA 方法PGT-A 分析基本参数比较
3 种方法获得数据经PGXcloud 生信平台分析,相关的分析参数统计结果见表2。3 种方法测序获得有效reads 平均值在10 M 以上。原始数据与基因组的比对率均超过97%,达到预期目标。基因组比对率、已覆盖基因组比例、SD值比较,MDA>MALBAC>SurePlex,差异有统计学意义(P<0.05)。冗余序列比例的比较,SurePlex>MALBAC>MDA,差异有统计学意义(P<0.05)。
表2 测序数据结果比较(±s)Table 2 Comparison of sequencing data results(±s)
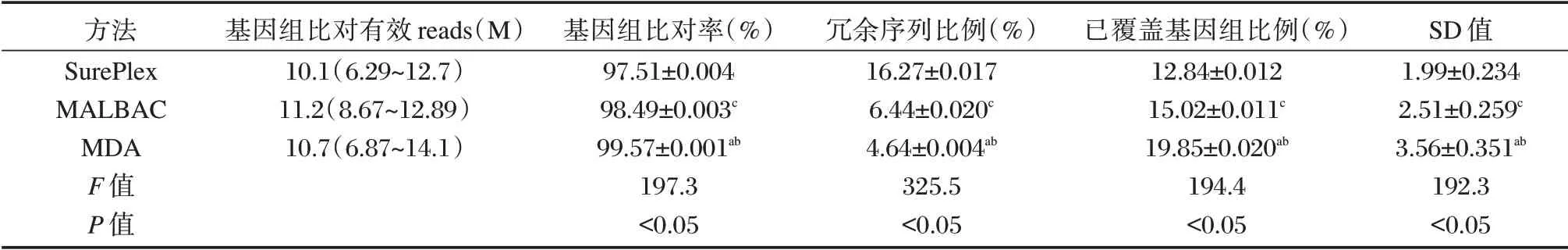
表2 测序数据结果比较(±s)Table 2 Comparison of sequencing data results(±s)
注:MDA 组与SurePlex 组比较,aP<0.05;MDA 组与MALBAC 组比较,bP<0.05;MALBAC 组与SurePlex 组比较,cP<0.05。
SD 值1.99±0.234 2.51±0.259c 3.56±0.351ab 192.3<0.05方法SurePlex MALBAC MDA F 值P 值基因组比对有效reads(M)10.1(6.29~12.7)11.2(8.67~12.89)10.7(6.87~14.1)基因组比对率(%)97.51±0.004 98.49±0.003c 99.57±0.001ab 197.3<0.05冗余序列比例(%)16.27±0.017 6.44±0.020c 4.64±0.004ab 325.5<0.05已覆盖基因组比例(%)12.84±0.012 15.02±0.011c 19.85±0.020ab 194.4<0.05
2.3 3 种WGA 方法PGT-A 对检测1~4 Mb CNV性能比较
3 种WGA 方法对3 种细胞系的27 个样本进行PGT-A 分析,散点图结果见图1~3。MDA 法可辨识出3.59 Mb(GM24312)左右的CNV。见图1C。而对于2.5 Mb(GM13325)及1.3 Mb(GM25372)则很难辨识。见图2C、3C。3 种WGA 方法PGT-A 检测1~4 Mb CNV 时,PGT-A 分析成功率、预期CNV检出率和非预期CNV 检出率三个指标方面的比较结果见表3。
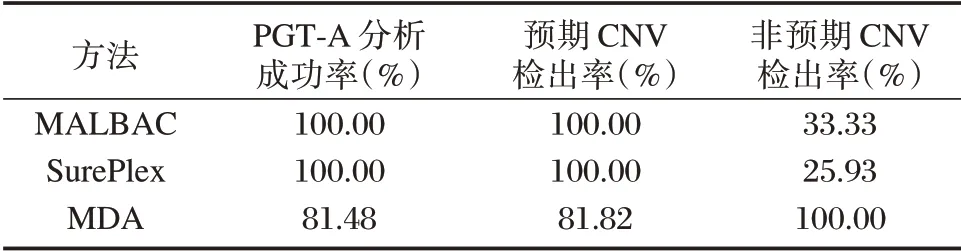
表3 3 种WGA 方法性能评估Table 3 Evaluation of three WGA methods'performance

图1 SurePlex、MALBAC 和MDA 对GM24312 细胞系CNV 检测结果Figure 1 Detection of CNV in GM24312 cell line by SurePlex,MALBAC and MDA
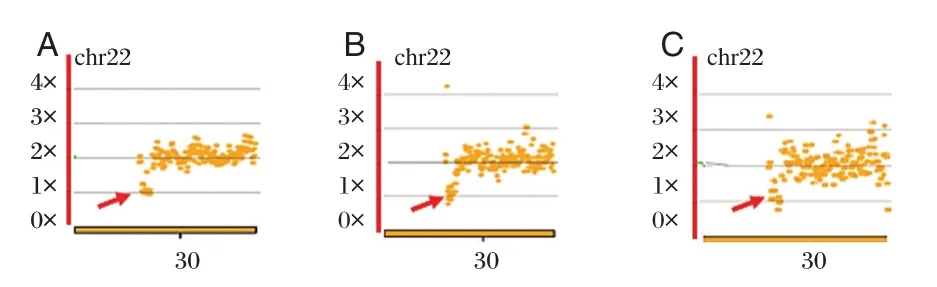
图2 SurePlex、MALBAC 和MDA WGA 对GM13325 细胞系CNV 检测结果Figure 2 Detection of CNV in GM 13325 cell line by SurePlex,MALBAC and MDA
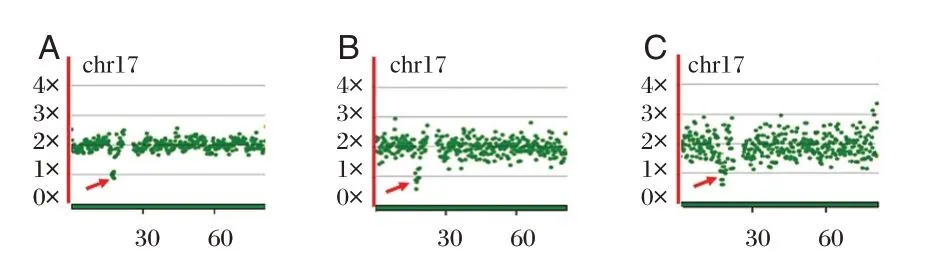
图3 SurePlex、MALBAC 和Discover-sc WGA 对GM25372细胞系CNV 检测结果Figure 3 Detection of CNV in GM 25372 cell line by SurePlex,MALBAC and MDA
3 讨论
CNVs 具有致病性、良性以及临床意义不明,随着NGS 技术的发展和CNVs 临床病例的积累,以及很多对CNVs 解读的分析标准完善,对CNV的认识也会越来越充分。以往认为是良性或者未知临床意义的CNVs 可能会对某些复杂性疾病的致病性具有潜在作用,因此提高CNV 解读的准确性,更有利于发现新的致病基因[6-7],提高我国的出生缺陷防控水平。Marcou 等[8]对DNA 微重复研究发现,片段长度<1 Mb 的CNV,并且临床意义未明(variants of uncertain significance,VUS)CNV 的重复DNA 片段,对提高致病性CNV(Pathogenic CNV,pCNV)诊断率的作用很有限。蔡艾杞等[9]推断除了导致单倍剂量不足的基因产生功能丧失突变的CNV,以及覆盖已知致病基因的CNV 外,片段长度<1MbCNV 很可能为不具有临床诊断意义的CNV,而对于长度≥1Mb 且多为新发突变的CNV 中,则意味着可鉴定诊断出更多的pCNV。PGT-A 检测核心技术是将pg 级别的微量DNA 进行均一保真有效地全基因组扩增,以获得足够的DNA 进行检测分析。因此,WGA 技术的基本要求是要扩增均一、基因组覆盖度高、保真度高。本研究所使用的3 种WGA 方法(SurePlex、MALBAC 和MDA),在临床上主要是对4 Mb 以上分辨率的PGT-A[10-13],每种方法各有所长,需要根据应用目的去选择合适的方法。然而,针对1~4 Mb CNV 在PGT-A 检测中的研究文献较少,因此本研究中开展了3 种WGA 方法对1~4 Mb CNV 的检测性能评估。
根据本研究的结果,MDA 法对单细胞全基因组的扩增能力最强,对相同起始量的细胞样本,其扩增产物是SurePlex 法及MALBAC 法得到的扩增产物的3~5 倍,建库产物是后两者的2倍左右,且操作时间居中(<4 h)。MDA 法获得基因组比对有效数据量在相近情况下(10M),冗余率最低(4.64%<6.44%<16.27%),对基因组的覆盖率最高(19.85%>15.02%>12.84%)。然而进行PGT-A 分析后,MDA 法的SD 值(3.56)远高于SurePlex 法(1.99)与MALBAC 法(2.51),这就表明MDA 法对基因组扩增的均一性远低于这两种方法,结果的可信度降低。因为SD 值代表了待测样本与正常基因组标准样本间的离散程度,SD 越小则越接近标准基因组,离散度越小,则可信度越高,反之则结果可信度越低。在对1~4 Mb CNV检测的评估中,MDA 法得到的结果显示其仅可准确分辨3.59 Mb CNV,而难以辨识2.5 Mb CNV 和1.3 Mb CNV。并且因SD 值过高,散点图离散程度较高,均出现一些非预期的CNV。有研究亦指出以PCR 技术为基础的MALBAC 和PicoPLEX single-cell WGA kit(NEB-WGA)与MDA 相比,更适合CNV 检测,MDA 更适合单核苷酸多态性分析[14]。
SurePlex 与MALBAC 两种技术的基本原理相同。本研究结果表明,无论是PGT-A 分析成功率、预期1~4 Mb CNV 检出率均为100%,与Li 等[15]在3~15 Mb CNV 测试对比的结果类似。不过,在数据量接近的情况下,SurePlex 法的冗余度远高于MALBAC,与基因组的匹配率也稍低。这就导致在进行PGT-A 分析中,唯一比对reads 数不如MALBAC 法得到的reads 数,对基因组的覆盖度稍低,这对PGT-A 分析不利。但同时SurePlex 获得的SD 值显著低于MALBAC,其散点图比MALBAC 的散点图更集中,目标CNV 的异常也更明确,从而检测结果可信度更高。这与Zhang[10]等的研究结果类似,他们在对比了多种WGA 方法后,认为这两种方法在扩增效率、准确性等方面得到同样结果。Deleye 等[16]研究结果显示SurePlex与MALBAC 相比,SurePlex 对基因组扩增有更好的均匀性,在CNV 检测中具有低的假阳性,因此SurePlex 在检测拷贝数变异方面更具优势。本研究结果也显示,MALBAC 法检出非预期CNV 的比例比SurePlex 法高出7.4%。所以,SurePlex 法获得数据的有效性上虽不如MALBAC 与MDA,但其SD 值最小,预期CNV 检出率为100%,而非预期CNV 检出率最低。因此3 种方法在检测1~4 Mb的CNV 时,在平均有效reads 为10 Mb 时,SurePlex法结果最可信,其次为MALBAC,MDA 表现最差。综合比较,SurePlex 法更优,可作为首选方法,MALBAC 可作为备选方法,而MDA 法不适合1~4 Mb 小CNV 的检测。
本研究中仅针对3 种已知大小CNV 的培养细胞系进行了分析,然而细胞系样本与囊胚滋养外胚层样本相比还是有一定差异,后者更具复杂多样。下一步将继续开展对临床已知由1~4 Mb CNV 引起疾病的废胚活检样本的研究,以期对胚胎染色体异常进行精准检测。
