基于WGCNA 方法识别影响猪肉质性状的关键模块和基因*
2022-09-22荣丽云王孝义李明丽鲁绍雄
荣丽云,黄 宁,王孝义,李明丽,陈 强 ,鲁绍雄
(云南农业大学 动物科学技术学院,云南 昆明 650201)
肉质性状是养猪生产中最重要的经济性状之一,对养猪生产效益有着重要影响。提高猪肉品质一直是猪育种工作的重要目标,但由于肉质性状活体难以准确测定,且包括肌内脂肪含量、大理石纹、嫩度、pH 值和滴水损失等诸多相互关联的子性状,常规育种方法改良难度大,难以获得理想的遗传进展。随着分子生物学及基因组学相关技术的发展,人们发掘了大量与猪肉质性状相关的基因[1-4]。尽管如此,关于猪肉质性状的分子机制迄今仍不甚清楚。
基因共表达网络分析(gene co-expression network analysis,GCNA)是基于对同一性状的基因在表达模式上具有共表达特征这一设定,从构建的表达调控网络中发现并识别目标性状的相关基因。该方法对目标性状关键新基因的发掘及其功能预测具有重要作用。加权基因共表达网络分析(weighted gene co-expression network analysis,WGCNA)是目前开展GCNA 最主要的方法[5],人们利用该方法鉴定了大量与复杂性状相关的基因共表达模块和关键基因[6-9]。
近年来,利用GCNA 方法探索畜禽重要经济性状分子机制的研究逐渐增多,有关猪肉质性状的基因共表达模块研究也有陆续报道[10-13]。如ZAPPATERRA 等[2]对意大利大白猪半膜肌进行研究,识别了4 个与肌内脂肪含量相关的基因共表达模块;ZHAO 等[14]利用WGCNA 法识别了与猪肉滴水损失相关的AASS、BCKDHB、ALDH6A1、MUT和MCCC1等5 个基因。这些研究为进一步揭示猪肉质性状形成的基因表达机制提供了有益探索,但目前关于肉质性状相关的基因共表达研究及可利用的基因共表达信息仍较少,尚需进一步深入研究。
相较于国外瘦肉型猪种,中国地方猪种肉质优良的遗传特性突出。由于两类猪种在肉质性状等方面存在较大差异,基于两类猪种的比较分析是揭示肉质性状分子机制的一种有效途径。本研究基于公共GEO 数据库中大蒲莲猪(中国地方品种)和长白猪(国外瘦肉型品种)背最长肌基因表达数据集,通过WGCNA、蛋白质与蛋白质互作(protein-protein interaction,PPI)网络及中心性分析等方法,挖掘与肉质性状相关的关键基因共表达模块和关键基因,并对识别的关键基因表达进行qPCR 验证,以探索猪肉质性状形成的分子机制,为联合运用经典育种和分子育种手段改良猪肉质性状奠定基础并提供科学依据。
1 材料与方法
1.1 数据来源
数据来源于美国生物技术信息中心(NCBI)基因表达数据库(GEO,https://www.ncbi.nlm.nih.gov/geoprofiles/)中的数据集GSE119368,包括8 头大蒲莲猪(中国地方品种)和8 头长白猪(国外瘦肉型品种) 6 月龄背最长肌基因表达数据。猪群饲料营养相同、饲养管理条件相近。基因表达数据由Illumina HiSeq 2500 测序平台产生。
1.2 差异表达基因分析
差异表达基因分析利用基于R 软件的limma包[15](version 3.44.3)进行,以矫正的P<0.05 和|log2fold change|>1 作为基因差异表达的显著性阈值。差异表达基因(differentially expressed genes,DEGs)分布的火山图和表达热图采用ggplot2 软件包(https://cran.r-project.org/web/packages/ggplot2/,version 3.3.3)绘制。
1.3 基因共表达模块分析
基因共表达模块分析采用WGCNA 方法[5],使用基于R 环境的WGCNA 软件包(https://cran.r-project.org/web/packages/WGCNA/,version 1.70-3)进行。以大蒲莲猪为处理组、长白猪为对照组,质控后获得的12 494 个基因按如下流程进行分析:
(1)计算两两基因间的皮尔逊相关系数(r),并据此构建基因表达相似矩阵;(2)将基因表达相似矩阵转换成邻接矩阵,并通过软阈值β (β=10,无尺度拓扑标准R2>0.85)产生增强邻接矩阵;(3)使用皮尔逊相关分析构建增强邻接矩阵的无监督基因共表达关系矩阵,并基于基因间的关联程度指标拓扑重叠尺度(topological overlap measure,TOM)将其转化为拓扑矩阵;(4)基于基因间相异度(1-TOM),使用平均连锁层次聚类法将具有相似基因表达模式的基因归为同一个基因模块,采用动态剪切算法从系统聚类树中识别基因共模块,并将相似度高于75%的基因模块进行合并。
1.4 基因功能分析
差异表达基因及关键模块中基因的功能采用基因本体论(gene ontology,GO)富集及京都基因和基因组百科全书(Kyoto encyclopedia of genes and gnomes,KEGG)通路富集方法,利用在线的DAVID 软件[16](https://david.ncifcrf.gov/,version 6.8)进行分析,通路显著性富集阈值为P<0.05。
1.5 PPI 网络构建
关键模块中编码蛋白基因间的互作关系采用PPI 网络进行分析,互作关系信息来源于在线数据库STRING[17](http://string-db.org,version 11.5),筛选标准为互作得分大于0.7。PPI 网络采用Cytoscape 软件[18](https://cytoscape.org/,version 3.8.0)构建。
1.6 高度关联模块及关键基因识别
PPI 网络中与肉质性状高度关联模块的分析采用分子复合检测(molecular complex detection,MCODE)算法,并利用Cytoscape 软件中的MCODE 插件[19](version 1.5.1)进行,相应参数设置为Degree Cutoff=2、Max Depth=100、K-Core=2 以及Node Score Cutoff=0.2。关键基因的识别采用Subgraph、Eigenvector、Information、Closeness、Betweenness、LAC 和Network 等7 种中心性方法,并利用Cytoscape 软件中的CytoNCA 插件[20](version 2.1.6)进行,其中,7 种方法得分最高的前10 个基因即为关键基因。
1.7 关键基因验证
选择彼此无亲缘关系的地方品种撒坝猪和引进瘦肉型品种大白猪各3 头,在相同饲养管理条件下饲养至体质量为90~100 kg 时屠宰,采集背最长肌组织用于肉质关键基因验证。从每个识别的肉质性状高度关联模块中各选择3 个关键基因,采用qPCR 方法检测关键基因在撒坝猪和大白猪背最长肌组织中的表达水平,并采用非配对试验的t检验进行差异分析,差异显著和极显著阈值分别为P<0.05 和P<0.01。
2 结果与分析
2.1 DEGs 识别及功能分析
差异基因表达分析结果如图1 所示。在大蒲莲猪和长白猪的背最长肌组织中共识别出390 个DEGs,其中,大蒲莲猪中显著高表达基因189 个、显著低表达基因201 个。

图1 大蒲莲和长白猪差异表达基因分布及表达水平Fig.1 Distribution and expression level of differentially expressed genes in Dapulian and Landrace pigs
GO 分析结果(表1)显示:189 个高表达基因极显著富集在3 个GO 生物学过程、3 个GO 细胞组分和2 个GO 分子功能上,201 个低表达基因极显著富集在12 个GO 生物学过程、5 个GO细胞组分和2 个GO 分子功能上。高表达基因主要与胶原纤维组织、成肌细胞融合和PI3K-Akt信号传导等生物学过程相关,低表达基因主要与骨骼肌细胞分化、蛋白质折叠及神经元凋亡过程调控等生物学过程相关。

表1 差异表达基因极显著富集的GO 条目Tab.1 GO terms with extremely significant enrichment of differentially expressed genes
KEGG 通路分析结果(表2)显示:189 个高表达基因和201 个低表达基因分别极显著富集在5 个和3 个KEGG 通路上。其中,高表达基因主要与蛋白质消化吸收、醛固酮调节的钠重吸收以及心肌细胞的肾上腺素能信号等通路相关,低表达基因主要与MAPK 信号通路、内质网蛋白质加工以及雌激素信号通路等相关。

表2 差异表达基因极显著富集的KEGG 通路Tab.2 KEGG pathways with extremely significant enrichment of differentially expressed genes
2.2 基因共表达模块分析
聚类结果显示:大蒲莲猪和长白猪样本均各自聚成一类(图2a)。根据计算的基因间相异度,识别了6 个基因共表达模块(图2b);合并相似性高于75%的模块后,获得红色、蓝色和绿色3 个基因共表达模块(图2c)。其中,红色模块(r=0.91,P=7E-7)和蓝色模块(r=0.89,P=5E-6)与肉质性状的关联度最高(图2d),两模块中基因表达与肉质均呈极显著相关(r=0.93,P<1E-200;r=0.90,P<1E-200)(图2e 和2f)。
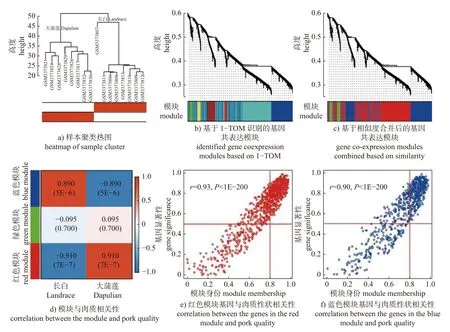
图2 WGCNA 分析结果Fig.2 The results of WGCNA analysis
2.3 与肉质性状最显著关联的模块基因功能分析
与肉质性状最显著关联的红色模块660 个基因极显著(P<0.01)富集到17 个GO 生物学过程和12 个KEGG 通路上(显著性最高的前5 个生物学过程和KEGG 通路见表3),主要与蛋白质折叠、骨骼肌细胞分化、糖原代谢过程和雌激素信号、蛋白质加工以及蛋白激酶等信号通路相关;蓝色模块527 个基因极显著(P<0.01)富集到12个GO 生物学过程和10 个KEGG 通路上(显著性最高的前5 个生物学过程和KEGG 通路见表4),主要与肌肉组织形态形成、心脏肌肉收缩、快慢纤维转换过程和蛋白质消化吸收、ECM-受体相互作用以及黏着斑等信号通路相关。

表3 红色模块基因极显著富集的前5 个GO 条目和KEGG 通路Tab.3 Top 5 GO terms and KEGG pathways extremely significantly enriched by the genes in the red module

表4 蓝色模块基因极显著富集的前5 个GO 条目和KEGG 通路Tab.4 Top 5 GO terms and KEGG pathways extremely significantly enriched by the genes in the blue module
2.4 PPI 网络构建及关键基因识别
所构建的红色模块基因PPI 网络包括163 个基因(节点)和371 条边(图3a),最高得分的子网络包含11 个基因(FBXL4、RNF115、FBXO40、ASB4、ASB15、UBE2L6、RNF41、ASB5、HERC5、SOCS1、ASB11)和55 条边(图3b),11 个基因的中心性综合得分相同(表5),为肉质性状相关的关键基因。

表5 红色模块基因PPI 子网络基因中心性分析Tab.5 Centrality analysis of genes in the PPI sub-network of the red module

图3 基因互作网络与高关联度模块识别Fig.3 Gene interaction network and highly related module identification
所构建的蓝色模块基因PPI 网络包含128 个基因和341 条边(图3c),最高得分的子网络包含22 个基因和112 条边(图3d),中心性综合得分最高的前10 个基因为COL3A1、COL5A1、COL1A2、COL14A1、CRTAP、COL5A3、LEPREL1、DEPTOR、COL4A2和COL6A2(表6),为肉质性状相关的关键基因。

表6 蓝色模块基因PPI 子网络基因中心性分析Tab.6 Centrality analysis of genes in the PPI sub-network of the blue module
2.5 关键基因表达分析及qPCR 验证
由图4 可知:2 个模块各挑选的3 个肉质性状关键基因在大蒲莲猪和长白猪背最长肌中的表达水平均呈极显著差异,除RNF115(P=0.001 84)外,COL3A1(P=0.000 16)、COL5A1(P=0.001 65)、COL1A2(P=0.000 073)、FBXL4(P=0.000 97)和FBXO40(P=0.000 54) 5 个基因的表达水平均呈现为长白猪高于大蒲莲猪。

图4 大蒲莲和长白猪背最长肌中6 个关键基因的表达水平Fig.4 The expression levels of six key genes in longissimus dorsi muscle of Dapulian and Landrace pigs
qPCR 结果(图5)显示:蓝色模块中的COL-3A1(P<0.000 1)、COL5A1(P=0.000 6)和COL1A2(P<0.000 1)在撒坝猪和大白猪背最长肌中的表达水平呈极显著差异,而红色模块中3 个基因(RNF115、FBXL4和FBXO40)的表达水平在2 个品种间则无显著差异(P>0.05)。

图5 6 个关键基因在撒坝和大白猪背最长肌中的表达水平Fig.5 The expression of six genes in longissimus dorsi muscle of Saba and Large White pigs
3 讨论
本研究基于WGCNA 方法识别了2 个与猪肉质性状显著关联的基因共表达模块,并通过构建的模块基因间互作关系网络分别识别出10 个和11 个关键基因。
在蓝色模块识别的10 个猪肉质性状关键基因中,有7 个基因(COL3A1、COL5A1、COL1A2、COL14A1、COL5A3、COL4A2和COL6A2)为胶原蛋白家族相关基因,该家族基因编码一类胶原蛋白,主要存在于动物结缔组织中。有研究表明:胶原蛋白对动物肌内脂肪形成、肌肉嫩度、系水力及滴水损失等具有重要影响[21-24]。NAKAJIMA等[25]研究了牛胸最长肌的肌内脂肪,发现胶原蛋白V 和VI 型对肌内脂肪的形成具有正向调控作用;曾勇庆等[26]对莱芜猪背最长肌的胶原蛋白含量及性质进行分析后发现:随着胶原蛋白含量的逐渐增加,猪肉的大理石纹和持水性能逐渐增加。
已有研究[27-29]表明:COL3A1、COL5A1、COL1A2和COL14A1等4 个胶原蛋白家族基因与猪肉质性状相关。LIM 等[27]对巴克夏猪背最长肌转录组的研究显示:COL1A2、COL5A1和COL14-A1与猪肌肉肌内脂肪含量显著相关;BAO 等[28]对大白猪杂交群体的研究发现:COL3A1基因的表达上调有利于猪肌纤维的生成;FERNÁNDEZ-BARROSO 等[29]对伊比利亚猪的研究发现:COL14A1基因对猪肉嫩度有显著影响。可见,胶原蛋白家族基因可能主要与猪肉的肌内脂肪含量和嫩度等相关。本研究的qPCR 验证结果表明:COL3A1、COL5A1和COL1A2基因在肉质差异较大的地方品种撒坝猪和引进品种大白猪背最长肌中的表达水平差异极显著,进一步提示上述基因对肉质性状有着潜在重要影响。而有关COL-5A3、COL4A2和COL6A2基因对猪肉质性状的影响迄今未见相关研究报道,其确切功能尚需进一步研究。此外,本研究识别的另外3 个关键基因(CRTAP、LEPREL1和DEPTOR)中,CRTAP是软骨关联蛋白编码基因,主要与骨质发育相关,有研究表明该基因参与胶原形成和胞外基质降解[30],而LEPREL1和DEPTOR基因对肉质性状的影响目前仍未见报道,2 个基因与肉质的确切相关性仍需进一步通过功能试验确认。
在红色模块识别的11 个关键基因中,已有研究[31-37]显示其中的一些基因与肉质性状相关,如FBXL4、FBXO40、ASB4和ASB15等。FBXO40为F-box 蛋白家族成员之一,该基因的敲除可显著促进猪骨骼肌纤维增粗[31-32]。FBXL4是一个与线粒体功能控制有关的核基因,与FBXO40同属于F-box 蛋白家族,当FBXL4基因敲除时小鼠出现线粒体功能障碍及体质量减轻等现象[33]。ASB4、ASB15和ASB5同属ASB 基因家族成员,在骨骼肌和心肌中呈高表达,被认为是猪肉质性状遗传改良的潜在候选基因[34-35]。UBE2L6在动物脂肪细胞中可作为介导脂肪分解的负调节因子促进高脂饮食诱导产生肥胖,敲除该基因能够抑制脂肪生成和减少脂肪细胞数量[36-37]。目前,关于RNF41、RNF115、HERC5和SOCS1基因与肉质性状的相关性尚未见报道。
尽管撒坝猪和大白猪背最长肌中FBXL4和FBXO40基因的表达差异与大蒲莲和长白猪的趋势一致,但RNF115基因的表达差异则呈相反趋势,且这3 个关键基因在撒坝猪和大白猪中的表达水平差异均不显著。究其原因,可能在于撒坝猪和大蒲莲猪虽同为肉质优良的中国地方猪种,长白猪和大白猪均为国外瘦肉型猪种,但同类猪种在部分肉质指标上仍存较大差异,如体质量为100 kg 的撒坝猪肌内脂肪含量和肉色评分分别为(6.53±1.65)%和(3.19±0.31)分[38],而大蒲莲猪则分别为(4.63±0.88)%和(4.25±0.14)分[39],可见其肉质形成的分子机制可能存在一定差异,这可能也是造成验证结果不一致的原因之一。如能在不同猪种中进一步开展表达水平验证,重点比较不同地方品种(如撒坝猪和大蒲莲猪)的基因表达差异情况,深入揭示这些基因影响的具体性状及其效应,将有助于猪肉质性状的基因发掘并为精准育种提供更有价值的分子标记。
4 结论
本研究基于WGCNA 方法识别了与猪肉质性状相关的2 个关键基因共表达模块和21 个关键基因,其中7 个胶原蛋白基因家族相关基因COL3A1、COL5A1、COL1A2、COL14A1、COL5A3、COL4A2和COL6A2在猪肉质性状形成中可能有重要作用。大蒲莲猪和撒坝猪虽同为地方猪种,但其肉质性状形成的分子机制可能存在较大差异。
