MALDI-TOF MS自建库对不同来源大肠埃希菌的鉴定能力验证
2022-09-16张浩科赵智龙李紫薇宫海燕
张浩科,赵智龙,李紫薇,宫海燕*
(新疆医科大学1第一附属医院药学部,乌鲁木齐830054;2第五附属医院检验科,乌鲁木齐830011)
基质辅助激光解析电离飞行时间质谱(Matrix assisted laser desorption/ionization flight mass spectrometer,MALDI-TOF MS)是近年来新发展起来的一种软电离生物质谱,只需1~2个纯菌落,2~3 min即可获得鉴定结果。具有简单易学、快速准确、成本低廉和高通量等优点[1]。传统的细菌鉴定时间较长,至少需要8~18 h或更长的时间(如苛氧菌、厌氧菌、真菌),而如聚合酶链反应(Polymerase chain reaction,PCR)、DNA芯片、微阵列技术等分子生物学方法虽敏感性较高,时间较短,但费用昂贵,对操作人员、设备和环境要求严格,不适合临床常规应用[2-3]。MALDI-TOF MS技术将激光作为能量来源辐射结晶体,以离子质荷比为横坐标,离子峰为纵坐标形成特异性的病原菌蛋白质组指纹图谱,并与图谱库进行对比,获得鉴定结果,最终确定细菌种类[4]。即MALDI-TOF MS技术的核心是其数据库[5-7]。由于不同地区的优势菌存在差异,且受当地环境条件及生活习惯的影响[8],数据库中微生物的参考质谱图并不能客观代表其他微生物实验室分离的同种典型菌株。因此将分离的本地典型菌株作为参考填充至自定义数据库中,可有效提高微生物的鉴定率。本研究旨在建立具有特征的大肠埃希菌MALDITOF MS自建库,并分析MALDI-TOF MS自建库对不同来源的大肠埃希菌的鉴定能力,充实MALDI-TOF MS技术在本地临床微生物鉴定中的应用。
1 材料与试剂
1.1 菌株收集采集2019年3月—2020年8月新疆医科大学第五附属医院检验科临床标本分离的人源性大肠埃希菌142株,采集自北京市怀柔区和延庆区养殖场分离出的40株动物源性大肠埃希菌菌株。
1.2 材料与试剂MALDI-TOF MS所用色谱纯甲酸(LOT:151817)、乙腈(LOT:167900)、α-氰-4-羟基肉桂酸(LOT:SHBD0586V)、三氟乙酸(LOT:153080)、无水乙醇(LOT:159116)均购自赛默飞世尔;5%羊血营养琼脂培养基(LOT:190124)、营养琼脂平板(CM107,批号:190902)购自北京陆桥技术有限公司;标准菌株为大肠埃希菌ATCC 8739(CICC,编号CICC10302)。Gel Extraction Kit试剂盒(OMEGA,LOT:410800);Easy-Pure Genomic DNA Kit(北京全式金,LOT:202005);Applied Biosystems Sanger测序试剂盒包括ExoSAP-IT Express试剂、BigDye Terminator v3.1循环测序试剂盒和BigDye XTerminator纯化试剂盒(ABI公司);Taq酶使用TaqHotStart DNA Polymerase(Takara公司)。
1.3 仪器与设备质谱仪Clin-TOF II型基质辅助激光解析/飞行时间质谱(北京毅新博创生物科技有限公司),Vitek 2 Compact全自动微生物分析系统(法国生物梅里埃公司)。数据采集软件为Shimadzo Biotech MALDI-MS 2.9.3(北京毅新博创生物科技有限公司)。微生物鉴定软件及聚类分析软件均为MicroID version 4.0(北京鑫汇普瑞公司),Verity PCR扩增仪(ABI公司),测序使用3730XL测序仪(ABI公司)。数据库采用EXS3000 V1.0.0.0(重庆中元汇吉生物)。
1.4 菌株的处理
1.4.1 菌株的培养 将菌株接种于5%羊血营养琼脂培养基,37℃培养24 h,重复划线培养2~3次,获得纯菌落。其培养方法和条件均参照临床实验室标准化协会(Clinical and laboratory standards institute,CLSI)标准规范进行。
1.4.2 菌株的冻存 挑取单菌落转移至营养琼脂平板划线培养,37℃培养24 h;用无菌棉签刮取菌落,置于含等量营养肉汤和30%灭菌甘油的冻存管中,混匀,分别于-20℃、-80℃逐级降温,-80℃保存备用。
1.4.3 菌株的复苏 将菌液于-80℃、-20℃、室温逐级复融。用接种环将菌液置于5%羊血营养琼脂培养基,37℃划线培养24 h。重复培养一次,获得复苏的纯菌落。
1.5 菌株的初步鉴定临床样本分离培养的菌株通过Vitek 2 Compact全自动微生物分析仪进行菌株种的初步鉴定和药敏检测。
1.6 菌株的16S rRNA测序鉴定
1.6.1 目的基因的提取 根据Easy Pure Genomic DNA Kit试剂盒使用说明书,将复苏的纯菌落溶于2 mL无菌盐水,1 000 r/min离心30 s,弃上清沉淀;加入1 mL 2%十六烷基三甲基溴化铵(CTAB)裂解液混匀,加入20 μL蛋白酶K,涡旋振荡混匀;56℃温浴1~2 h;每隔20 min颠倒混匀;加入350μL酚、350μL三氯甲烷,充分混匀;静置3 min;室温12 000 r/min离心12 min;将上清转移至2 mL离心管中,加入1.2 mL无水乙醇,颠倒混匀20次;-20℃放置30 min;室温10 000 r/min离心10 min;弃上清,加入700μL,70%无水乙醇,颠倒混合,室温静置3 min;10 000 r/min离心5 min;弃上清,10 000 r/min离心1 min;吸净管底残余液体,开盖晾干5~10 min;加入60~100μL,Tris-Cl(pH8.0-8.3)10 mmol/L,65℃溶解1 h。5倍稀释8μL Tris缓冲液与2μL原液。电泳0.8 %琼脂糖凝胶,在点样板加入2μL进行跑胶。
1.6.2 目的片段扩增 用Primer5软件设计并合成通用引物,引物序列如下:上游:5'-AGAGTTTgATCCTGGCTCAg-3';下游:5'-GGTTACCTTGTTACGACTT-3',产物长度1 500 bp。采用Applied Biosystems Sanger测序试剂盒的反应体系,反应条件:95℃预变性5 min,循环1次;95℃变性0.5 min,58℃变性0.5 min,72℃延伸1 min,共循环35次;72℃保温10 min,循环1次。PCR反应体系(25μL):5μL10×Buffer,1.5μL引物(10 μmol/L),2μL dNTP,0.25μL Taq(5 U/μL),2μL DNA,加14.25μL双蒸水(ddH2O)。
1.6.3 16S rRNA测序 按照Applied Biosystems Sanger测序试剂盒说明书进行测序。用Binding Buffer 56℃,间断振荡混合7 min,回收含有目的条带的琼脂糖凝胶,将其转移至核酸纯化柱(Spin Column)中,并插入1.5 mL离心管中,8 000 r/min离心1 min,弃滤液;向Spin Column加入Washing Buffer700μL,室温,8 000 r/min,离心1 min,弃滤液,重复上述步骤1次;将Spin Column置于新的1.5 mL离心管上,在Spin Column膜的中央加入25μL Elution Buffer,室温静置1 min,8 000 r/min离心1 min,洗脱DNA;回收产物用正、反向引物做测序,并将测序结果进行拼接。
1.7 菌株的MALDI-TOF MS鉴定
1.7.1 样品前处理 用接种环取约5 mg菌落,置于盛有300μL纯水的1.5 mL离心管中,震荡均匀,加入900μL无水乙醇,震荡,漩涡仪混匀3~5 s,12 000 r/min离心2 min,弃上清,12 000 r/min离心2 min,吸弃剩余液体,置于室温干燥5 min。加入40μL 70%甲酸,充分震荡,漩涡仪混匀3~5 s,加入40μL乙腈,充分震荡,漩涡仪混匀3~5 s,12 000 r/min离心2 min,移液枪吸取1μL上清,点至靶板上,室温干燥;将1μL基质溶液点至已干燥的样品点,室温自然晾干。
1.7.2 数据采集与处理 为保证建库质量,降低系统及人工误差。由3位实验员分别对同一菌株提取蛋白质,鉴定72个位点(72张图谱)。采用寻峰算法对各菌株数据进行预处理,消除原始数据中的基线漂移、明显噪声等,确定明显的特征峰(信号表现点),排除特征不清晰的图谱(特征峰扎堆或特征峰不明显)。
1.7.3 图谱特征峰聚类分析 对样品特征峰通过聚类分析后进行数据评价,质谱相似性在0%~100%,百分比越低质谱差异越大。通过相似性验证筛选出最终可用于聚类分析的谱图,由于相似性低于70%的谱图聚类效果较差,因此对相似性高于70%的谱图进行聚类分析,创建肽质量参考谱。
1.7.4 细菌肽质量谱参数 激光能量64%,每个靶点谱图数100张,每个谱图轰击5次,线性正离子模式下检测质荷比(m/z)。
1.8 自建库的构建
1.8.1 蛋白质指纹图谱数据库的构建 釆用MicroID 4.0的自建库功能进行构建。参数设置如下:质量区间3 000~15 000 PPM,最大质量误差600 PPM,最小匹配率60%,最大峰数量70。每张入库参考谱经归一化、平滑、基线校正、峰挑选后形成谱图。通过聚类分析得出显著共性峰系列。将自建库中大肠埃希菌的特征峰值与重庆中元肽质谱菌库(EXS3000 V1.0.0.0)中的特征峰值进行比对,分析筛选本地特有的特征峰值。
1.8.2 自建库的验证与评价 采用交叉验证,按MicroID 4.0系统(鑫汇普瑞公司研发)的判读标准确定识别程度。分值介于0~10分,<6分为不可识别,6~8分为可参考,8~9.2分为可信(属水平的鉴定),>9.2分为高度可信(种水平的鉴定)。本研究中,抽取40株合格的大肠埃希菌菌株用于自建库的鉴别能力验证。
1.9 统计学处理采用SPSS 22.0统计软件进行分析,计数资料以例(%)表示,采用χ2检验,检验水准α=0.05。通过美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)网 站 数 据 对16S rRNA测序结果进行比对鉴定。MicroID 4.0进行菌株鉴定及蛋白质指纹图谱的聚类分析。
2 结果
2.1 大肠埃希菌的标本类型与科室分布142株人源性大肠埃希菌通过初步鉴定与药敏分析,其中44株为敏感菌株,98株为非敏感菌株(至少对一种抗生素耐药或中介)。临床分离的人源性大肠埃希菌的标本类型主要有痰、尿液、血液、分泌物等,其中检出率最高的是尿液标本(图1),分布科室主要有泌尿外科、胃肠外科、肾内科等(图2)。
2.2 16S rRNA测序鉴定结果142株大肠埃希菌16S rRNA测序鉴定结果显示,5株未扩增出,8株被鉴定为非大肠埃希菌,其余129株均为大肠埃希菌,部分PCR产物电泳结果见图3。
2.3 MALDI-TOF MS鉴定及自建库的建立剔除16S rRNA测序鉴定的非大肠埃希菌菌株,最终筛选出40株合格的大肠埃希菌用于自建库的建立,对其897张合格的蛋白质指纹图谱进行归一化处理(图4)。MicroID 4.0自建库功能将收集到的蛋白质指纹图谱进行数据库检录。其结果显示,在m/z3 131、m/z4 166、m/z4 364、m/z4 535、m/z4 775、m/z5 096、m/z5 382、m/z5 751、m/z6 257、m/z6 317、m/z9 550处具有特征峰值。将特征峰值与重庆中元肽质谱菌库(EXS3000 V1.0.0.0)中的特征峰进行匹配,其中m/z4 166(10 %)、m/z4 364(39 %)、m/z5 751(42 %)、m/z9 550(59 %)四个特征峰值与EXS3000 V1.0.0.0标准菌库特征峰值的匹配度存在差异性(图5)。

图1 大肠埃希菌的标本类型分布
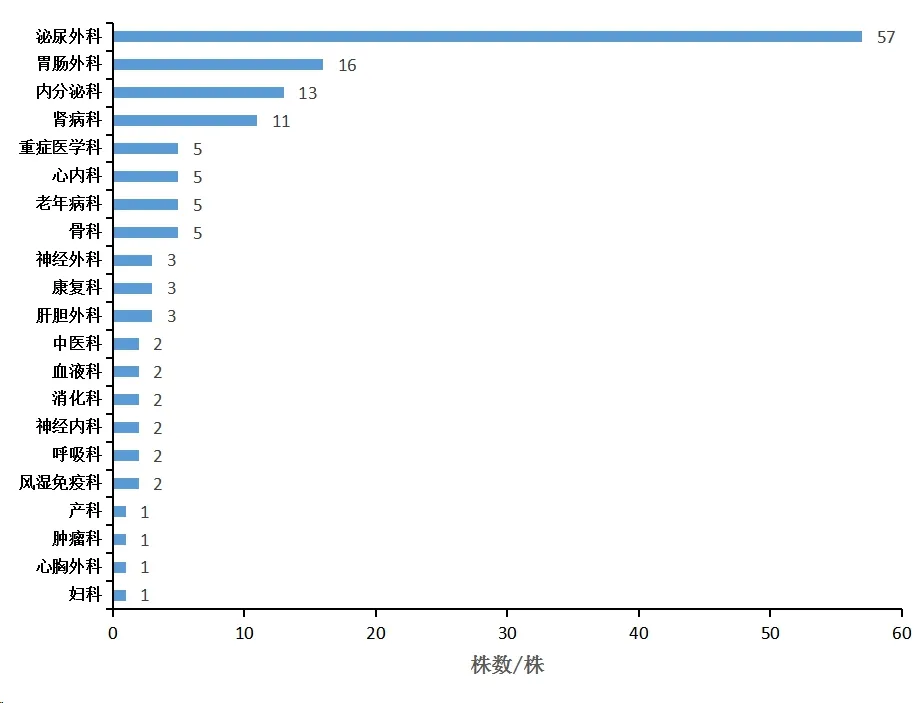
图2 大肠埃希菌标本科室分布

图3 部分菌株PCR产物电泳结果

图4 大肠埃希菌的蛋白质指纹图谱
2.4自建库的鉴定能力验证本次采集的142株人源性大肠埃希菌经16S rRNA鉴定,129株为合格菌株,其中40株用于自建库的鉴别能力验证。用于验证的40株人源性大肠埃希菌菌株的鉴定结果显示,96%为高度可信,4%为可信。动物源性大肠埃希菌对自建库鉴定结果显示,83%为高度可信,17%为可信。人源性和动物源性大肠埃希菌鉴定数据的可信程度差异有统计学意义(P<0.05)。

图5 大肠埃希菌的MALDI-TOFMS特征峰值比对结果
3 讨论
菌株蛋白质指纹图谱信息的丰富是准确鉴定菌株的基础,不同来源的菌株受多种因素的影响,蛋白质表达存在差异化特征,在选择扩充蛋白质特征图谱数据库的菌株要有代表性,要囊括尽量多的型别和不同地区的分离株,尤其要包括本地区种群的菌株,以便获得更准确的匹配结果。本研究探索MALDI-TOF MS技术对人源性和动物源性大肠埃希菌的差异鉴定能力,旨在提高细菌鉴定的准确率,分析蛋白质指纹图谱的特征峰值,为进一步探索大肠埃希菌特征蛋白质差异提供研究基础。目前,基于MALDI-TOF MS技术的自建库多用于临床病原菌的快速诊断,其培养条件的改变、不同地区、不同生长阶段、不同温度和不同培养基等因素都会导致微生物内生物大分子发生改变[9]。因此为实现最佳匹配,提高MALDI-TOF MS的鉴定能力,需对特征图谱数据库进行补充。随着对生物特征的广泛研究和数据库的不断完善,MALDITOF MS将得到广泛应用[10]。
本研究共收集142株本地人源性大肠埃希菌菌株,经16S rRNA鉴定129株是合格的大肠埃希菌菌株。从中筛选出40株符合建库条件的菌株进行建库,其中在m/z3131、m/z4166、m/z4364、m/z4 535、m/z4775、m/z5096、m/z5382、m/z5751、m/z6257、m/z6317、m/z9550处具有共同的特征峰值。将此次采集的特征峰与EXS3000 V1.0.0.0数据库中的特征峰进行匹配发现,m/z4166(10%)、m/z4364(39%)、m/z5751(42%)、m/z9550(59%)四个特征峰值在库中匹配度低,说明本自建库具有大肠埃希菌的地域特征峰,即具有本地代表性。
本研究建立的自建库验证结果显示,人源性和动物源性的大肠埃希菌菌株的可信程度差异具有统计学意义,说明此次建立的自建库可用于区分鉴定人源性与动物源性大肠埃希菌菌株,可用作不同地区菌株的鉴别与参考图谱数据库的补充。
