玉米11个ZmbZIP基因的鉴定及表达特征分析
2022-09-15贾利强刘洋赵秋芳陈曙
贾利强,刘洋,赵秋芳,陈曙
玉米11个基因的鉴定及表达特征分析
贾利强1,刘洋2,赵秋芳3,陈曙3
(1.贵州师范学院生物科学学院,贵州 贵阳 550018;2.嘉兴职业技术学院现代农业学院,浙江 嘉兴 314036;3.中国热带农业科学院南亚热带作物研究所,广东 湛江 524091)
以玉米自交系郑58为试验材料,利用生物信息学方法和qPCR技术,设置200 mmol/L NaCl、20%PEG6000、4 ℃低温和硝态氮或铵态氮缺乏等胁迫试验,对11个基因进行生物信息学分析,探测这些基因应答低温、干旱、盐害、缺氮等胁迫时的响应表达模式。系统进化树分析结果表明,11个基因可以细分为2个亚组;qPCR分析结果表明,不同亚组的基因在玉米不同组织中呈现不同的表达特征,同一亚组的基因呈现类似的表达模式,反映了基因在玉米生长发育进程中生物学功能的保守性和分化性。人为模拟盐、干旱、低温和氮缺乏等胁迫的结果表明,基因受到各种逆境因子的广泛调控,亚组I中的、和的表达量同时受NaCl溶液胁迫诱导而上调(相对表达量上调2倍),受PEG6000胁迫抑制下调;亚组II中的、、和同时受盐、PEG6000和低温胁迫诱导上调,硝态氮缺乏胁迫抑制、和基因表达,而铵态氮缺乏胁迫下,这3个基因表达明显上调,推测这些基因广泛参与应答逆境胁迫响应途径。
玉米;碱性亮氨酸拉链;基因家族;逆境胁迫;基因鉴定;基因表达;进化树
功能保守的转录因子家族在维持植物正常的生长发育以及抵御外界生物或非生物逆境胁迫进程中发挥着广泛而又重要的作用。当植物在生长过程中遇到低温、干旱、盐害或者缺氮等胁迫时,转录因子就会受到激发,结合靶基因的顺式作用原件,进而调控逆境途径相关基因的表达,使植物发生生理生化、形态等方面的变化,以抵御逆境胁迫等不利环境[1]。植物bZIP家族是分布较广泛、数量较多的转录因子家族之一。bZIP家族起源于真核生物历史进程的早期阶段,并在植物生长发育[2–4]、细胞生长与分化、逆境响应以及代谢平衡[5–10]等方面发挥着重要作用。
bZIP转录因子家族都含有典型的高度保守的bZIP结构域,一般由60~80 aa组成。N端含有高度保守的N–X7–R/K–X9蛋白基序,起着蛋白核定位和结合特定核苷酸序列的作用;C端为1个或多个由7个氨基酸组成的亮氨酸拉链蛋白基序构成的α螺旋二级结构。 bZIP转录因子通过亮氨酸区域构成同源或异源二聚体结构,调控bZIP转录因子与特异核酸序列的结合。近些年来,随着植物基因组技术的发展,拟南芥[3]、水稻[11]、葡萄[12]、玉米[13]、油菜[14]、大豆[15]、辣椒[10]、橄榄[16],红花[17]、荠麦[18]和马铃薯[19]等植物的bZIP家族成员相继被报道。
培育高产、稳产、综合抗性好的玉米新品种对保障粮食安全至关重要。玉米在生长过程中经常遭受各种逆境胁迫,严重影响玉米产量[20];因此,挖掘玉米抗逆因子,阐明玉米抗逆的分子生理生化机制对选育玉米新品种具有重要意义。本研究中,模拟盐、干旱、低温和不同氮形态缺乏胁迫,研究玉米自交系郑58中11个基因的响应表达模式,以期为阐明这些基因的生物学功能提供依据。
1 材料与方法
1.1 材料
玉米核心自交系郑58保存于中国热带农业科学院玉米研究中心。待郑58自交系生长至授粉扬花期,采集根、茎、叶、雄花、授粉2周的幼穗和苞叶等组织器官,液氮速冻,–80 ℃保存,备用。
1.2 方法
1.2.1玉米bZIP亚家族的生物信息学分析
11个玉米ZmbZIP和78个拟南芥AtbZIP蛋白序列、基因序列和CDS序列来源于Phytozome v9.1数据库(http://www.phytozome.net/)和MaizeGDB (Maize Genetics and Genomics Database)数据库(http://www.maizedb.org/)。运用在线工具ExPASy (http://expasy.org/tools/protparam.html)对ZmbZIP蛋白的相对分子质量、理论等电点等理化性质进行预测。运用在线软件MAFFT进行多重序列联配比对分析,去除不保守蛋白序列;运用MEGA 10,采用邻接法(NJ)构建拟南芥–玉米bZIP蛋白系统发育树,并使用Pairwise Deletion处理缺失数据,选择P–distance模型,校验参数Bootstrap重复次数设置为1000次。根据基因在玉米染色体上的位置,运用Mapinspect绘制其在染色体上的分布(http://www.plantbreeding.wur.nl/uk/software–mapinspect.html)。运用在线工具ExPASy(http://expasy. org/tools/protparam.html)预测ZmbZIP蛋白的相对分子质量大小和理论等电点;采用在线工具GSDS(http://gsds.cbi.pku.edu.cn/index.php)绘制玉米基因结构图。
1.2.2逆境胁迫试验
玉米自交系郑58种子经5%次氯酸钠溶液消毒,28 ℃黑暗催芽后置于网纱上,于Holland营养液中28 ℃光照16 h、25 ℃黑暗8 h,生长至3叶期时进行逆境胁迫处理。干旱或盐胁迫处理,选取生长一致的玉米幼苗转移至20% PEG 6000或200 mmol/L的NaCl溶液;缺氮胁迫处理,选取生长一致的玉米幼苗转移至硝态氮或氨态氮缺乏的营养液中;低温处理,选取生长基本一致的玉米幼苗转移至4 ℃条件下。分别在处理的0、1、6、24 h时取叶片样品,进行液氮速冻,保存于–80 ℃冰箱,备用。
1.2.3RNA提取及荧光定量PCR分析
取约0.1 g根系或叶片样品组织,液氮快速研磨粉粹,采用华越洋植物RNA快速提取试剂盒提取样品总RNA,进行RNA浓度与纯度检测(NanoDrop2000,ThermoScientific,Massachusetts, USA),用DNase I (TaKaRa,Tokyo,Japan)除去残留的核酸,反转录cDNA(Prime–ScriptTMRT reagent Kit with gDNA Eraser,TaKaRa),稀释至100 μg/μL,作为扩增模板。利用Oligo 7,根据基因的保守区域设计特异性引物,以为内参基因,引物序列见表1。实时定量荧光PCR反应在Roche Light Cycler 480 System(Roche, Basel, Switzerland)上进行,试剂盒 Thermo Fisher Scientific,反应体系10 μL,包括2×SYBR Green Master MIX 5 μL、上下游引物(10 μmol/L)各1 μL、cDNA 1 μL和ddH2O 2 μL。采用2–△△Ct算法进行数据计算。每个处理3次生物学重复。

表1 11个ZmbZIP基因的实时荧光定量引物
1.3 数据分析
采用Excel 2003进行数据统计分析和绘图。
2 结果与分析
2.1 11个玉米ZmbZIP基因的生物信息学分析
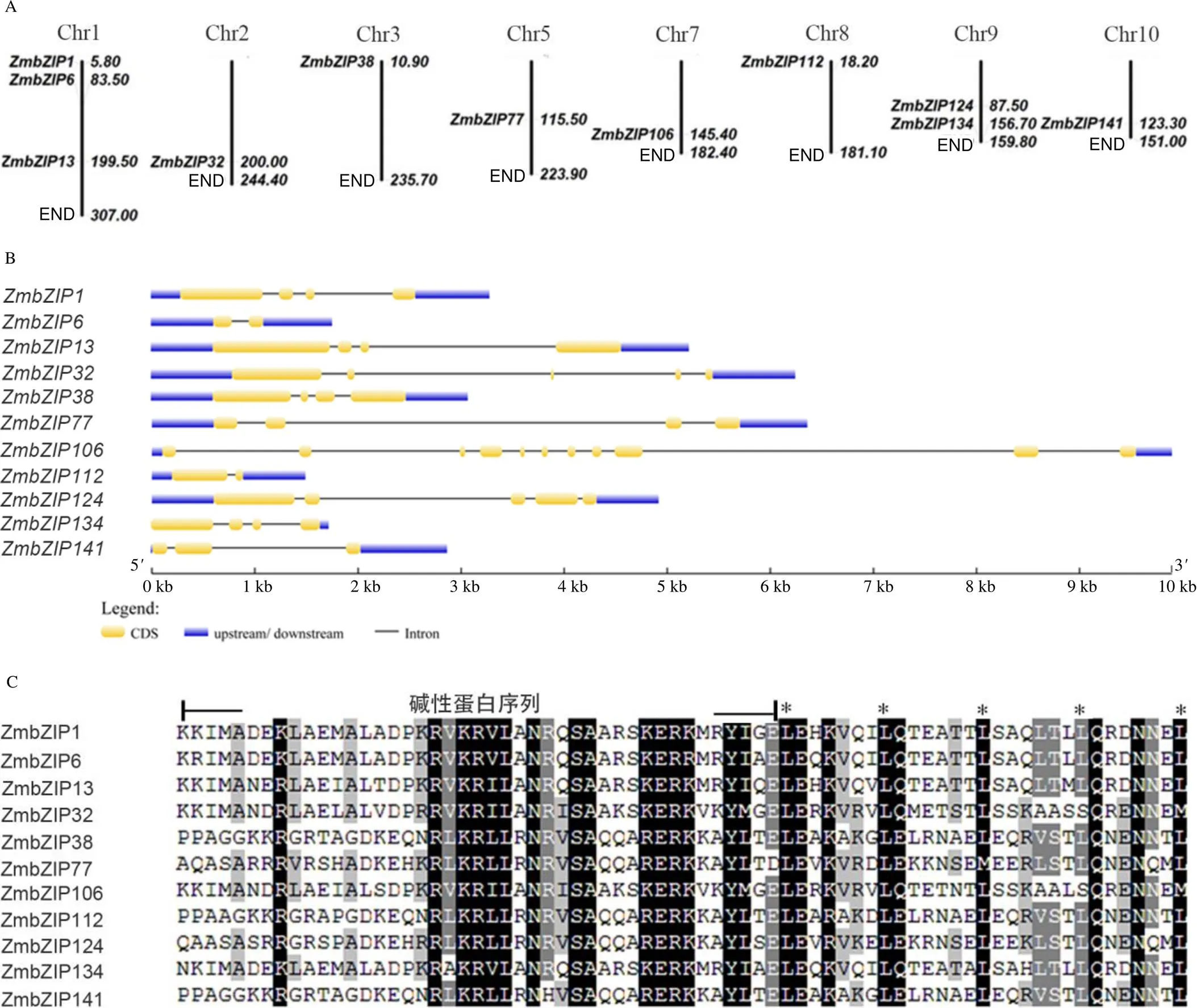
从表2可以看出,11个ZmbZIP蛋白的长度为138~479 aa,蛋白质相对分子质量为15 190~51 650。11个基因散布于8条染色体上,其中Chr1有3个基因,Chr2和Chr9各有2个基因,Chr3、Chr5、Chr7、Chr8和Chr10上各有1个基因(图1–A)。11个基因的内含子大多为3~4个,和基因各含有1个,含有10个内含子,基因含2个内含子(图1–B)。11个ZmbZIP蛋白的N端含有保守的bZIP结构域,包含碱性蛋白序列和亮氨酸拉链序列(图1–C)。进化树分析结果显示,11个基因分成2个家族(图1–D)。亚家族I中含有6个基因,II中含有5个基因,表明这2个亚家族的进化路径不同。

表2 玉米ZmbZIP蛋白的理化性质
2.2 玉米ZmbZIP基因的组织表达分析
为探测基因在玉米不同组织器官中的表达水平,在玉米自交系郑58扬花授粉期,采集玉米根、茎、叶、雄花和授粉15 d的幼穗等组织,利用qRT–PCR技术,检测这些基因的组织表达模式。由图2可知,这些基因的组织特异性表达模式不同,同一亚组的基因大多呈现类似的表达模式,、和聚合成1个亚组。和在所检测的组织中都有明显的表达,其中在根系和幼穗中的表达量较高;Z和在根系和茎中的表达量较高,其表达量达其他组织器官的2倍以上。可见,亲缘关系较近的基因具有相似的表达模式。主要集中在幼穗中表达,表达量是在其他组织器官的5倍左右,预示该基因在幼穗发育中起重要作用。亚组II包含、和等5个基因,其中和在根系中的表达量较高,体现了同源基因表达模式的保守性;和主要在根系和幼穗中表达,在根系、叶和雄花中表达量较高,说明这3个基因在进化历程中表达模式发生了分化,预示着其生物学功能的分化。这些数据表明基因在玉米组织器官生长发育进程中功能的多样性。
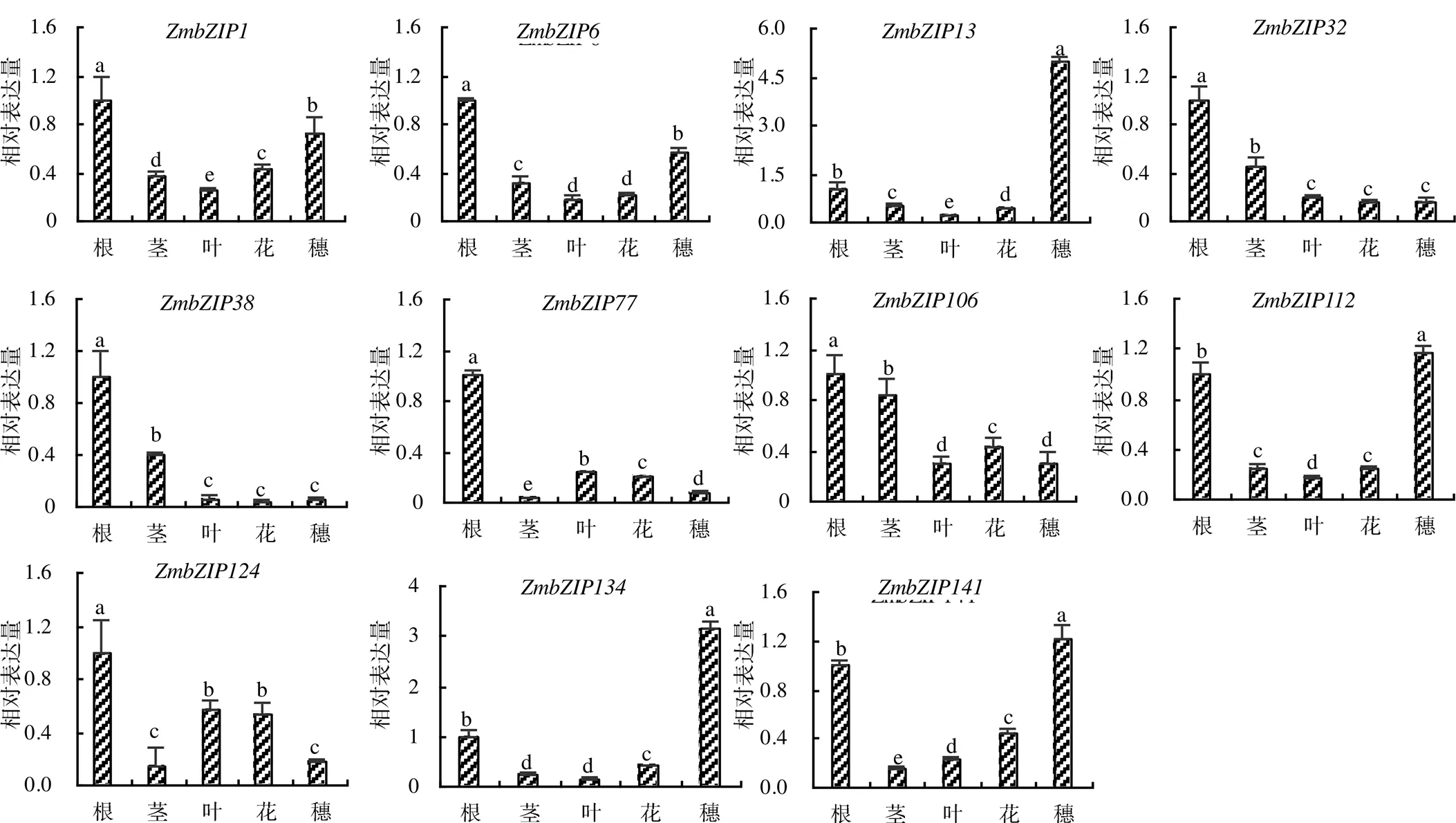
不同小写字母表示同一基因在不同组织中的表达量差异有统计学意义(P<0.05)。
2.3 ZmbZIP基因应答逆境胁迫的表达分析
为进一步了解基因应答非生物胁迫(盐、干旱和低温)响应表达模式,模拟200 mmol/L的NaCl溶液、20%PEG6000和4 ℃低温胁迫,运用qRT–PCR探测了玉米三叶期幼苗叶片中基因应答表达模式。由图3可知,同亚组的基因呈现相似的表达模式。亚组I中的和表达量同时受NaCl溶液胁迫诱导而上调(相对表达量上调2倍以上);受PEG6000胁迫抑制下调,胁迫处理24 h时,和的表达量比处理前分别下降了70%左右,表明这4个基因在应答盐和干旱胁迫响应途径中扮演着不同的重要角色。低温对、和的表达影响不同,早期受低温胁迫的诱导,在胁迫处理1 h时,其表达量比处理前增加2倍,说明该基因是低温胁迫的早期响应基因;在胁迫24 h时受到明显的抑制;的表达受低温胁迫的影响不明显。说明这3个同源基因在应答低温逆境胁迫时表达模式出现了分化,预示着生物学功能的分化。亚组I中的另2个基因,和受PEG6000胁迫抑制下调,胁迫处理24 h时,其表达量比处理前分别下降了70%左右;而盐和低温胁迫对这2个基因的表达水平影响不同,盐胁迫诱导基因的表达,抑制的表达,反映了这2个同源基因表达模式和功能的分化。亚组II中的、和血缘关系较近,和血缘关系较近,除外,其他4个基因的表达模式具有相似性。3种逆境胁迫6 h时,、和的表达量上升至最高值,其中的表达量上升超过5倍,之后出现回落,体现了表达模式的保守性,预示着这4个同源基因功能的冗余性;而的表达模式和其他4个基因差异明显,预示着该基因功能的分化。这些结果表明,基因应答各种不同逆境胁迫时具有复杂的表达模式,反映了这些基因在逆境胁迫响应途径中扮演着多样的生物学功能。
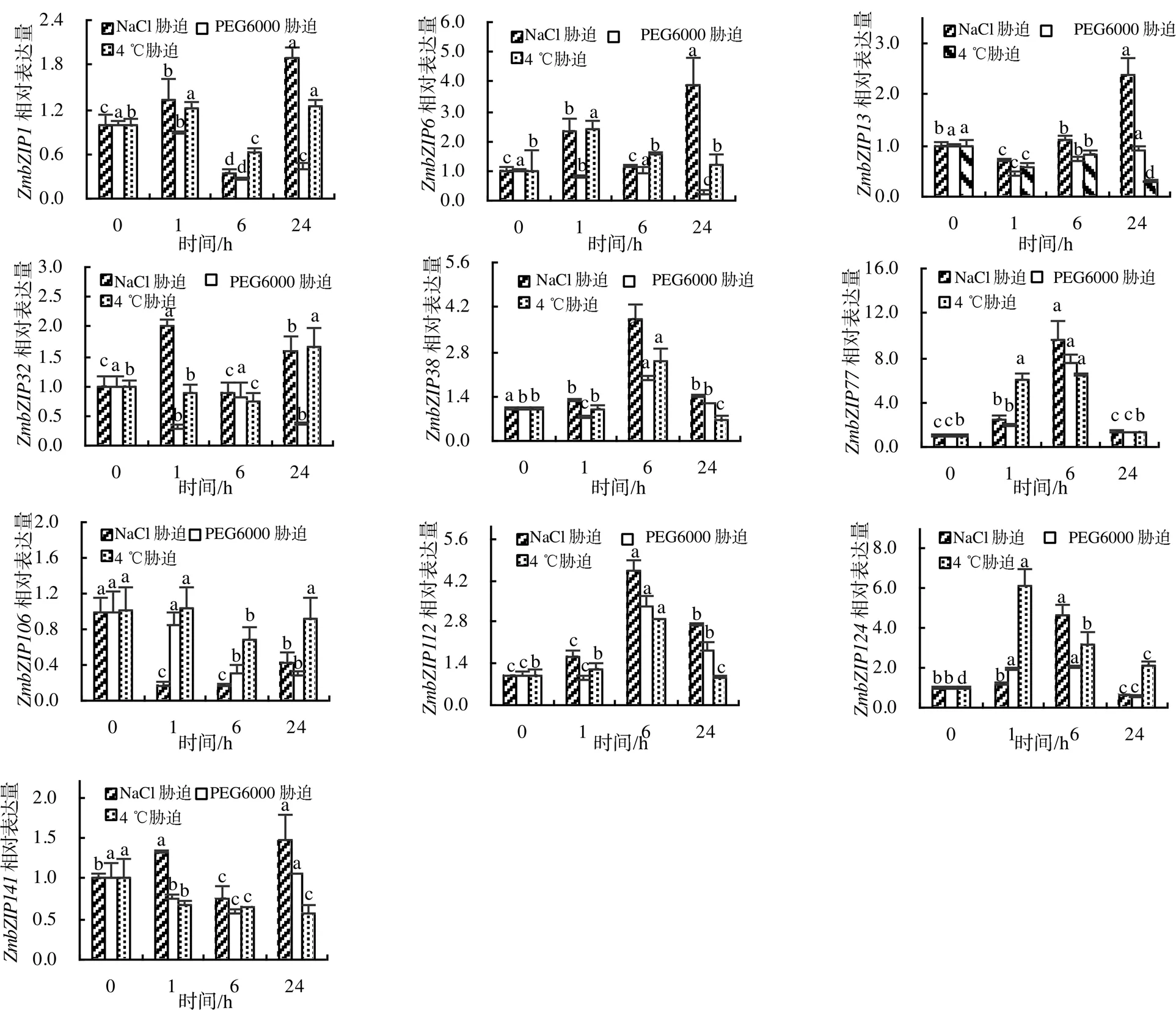
不同小写字母表示同一基因在不同时间同一胁迫处理中的表达量差异有统计学意义(P<0.05)。
2.4 ZmbZIP基因应答硝态氮和氨态氮胁迫的表达分析
由图4可知,叶片中基因的表达模式明显受硝态氮或铵态氮缺乏胁迫的影响。亚组I中,和应答硝态氮或铵态氮缺乏胁迫时的表达模式类似,硝态氮缺乏胁迫时抑制这3个基因的表达,在胁迫处理24 h时,3个基因的表达量下降了90%;而早期铵态氮缺乏胁迫诱导这3个基因的表达,在胁迫处理24 h时,其表达量出现回落。类似的表达模式预示着同源基因进化上的保守性。另1个同源基因在应答硝态氮或铵态氮缺乏胁迫时呈现完全不同的表达模式,早期硝态氮缺乏胁迫诱导其表达,之后出现回落,而铵态氮缺乏胁迫则抑制该基因的表达,说明该基因的表达模式及生物学功能出现了分化。亚组II中,在硝态氮或铵态氮缺乏胁迫条件下,和的表达量下降,表明这3个基因在硝态氮缺乏胁迫中可能发挥类似功能;在硝态氮缺乏胁迫条件下,和表达量下降,而对铵态氮缺乏胁迫呈现不同的表达模式,显示这2个基因与同组成员在进化早期出现分化,预示着其在应答氮缺乏胁迫时存在不同的生物学功能。

不同小写字母表示同一基因在不同时间、同一胁迫处理中的表达量差异有统计学意义(P<0.05)。
由图5可知,玉米根系中受硝态氮或铵态氮缺乏胁迫的调控。亚组I中,在2种不同氮形态缺乏胁迫24 h时,和表达量下降,而和受2种氮缺乏胁迫的影响各异,和受铵态氮缺乏胁迫出现明显诱导而上调,其最高值比处理前分别上升了8.7和27.7倍。亚组II的基因表达具有明显的保守性,和受2种氮缺乏胁迫的抑制,其中受抑制程度最显著,胁迫处理24 h时,其表达量下降90%以上;呈现不同的表达模式,硝态氮缺乏胁迫抑制其表达,而铵态氮缺乏胁迫显著诱导其表达,胁迫处理24 h,其表达量上升了24倍,显示该基因在参与硝态氮或铵态氮缺乏胁迫响应途径时具有不同的生物学功能。根系基因应答氮缺乏胁迫时表现出类似的表达模式,显示了同源基因的表达和功能的保守性,也预示着这些基因在发挥生物学功能时可能存在冗余性,但少数基因的表达模式出现分化,预示这些基因可能具有新的生物学功能。

不同小写字母表示同一基因在不同时间同一胁迫处理中的表达量差异有统计学意义(P<0.05)。
3 结论与讨论
植物在长期的进化进程中拥有一系列抵御逆境胁迫的防御机制,包括激活转录因子、调控抗逆途径基因的表达、增强植物的抗逆性等。近些年来,发现有越来越多的转录因子如bZIP、WRKY、MYB、bHLH和NAC等[21]参与调控植物应答逆境胁迫响应,通过改变这些基因的表达量,能显著提高植物的抗逆性[22]。众多作物包括玉米[13]、水稻[11,23]、番薯[21]、辣椒[10]等的基因表达受各类逆境胁迫的调控。和是逆境胁迫响应基因,改变这2个基因能显著改变植物的抗逆性[9,24]。小麦和玉米也具有类似的功能[25–26]。本研究中,玉米大部分基因受各类逆境胁迫的调控,这和前人[1]的研究结果基本一致。
近些年来,玉米中众多受逆境胁迫诱导基因被鉴定出来,其中包括转录因子[27]。根系和雌穗中受逆境胁迫的诱导,超表达可以促使玉米根系伸长,提高抗逆性。HY5、bZIP转录因子也具有类似的功能[27]。本研究中,在根系中的表达模式和应答不同逆境胁迫时的表达模式比较复杂,和在根系中的表达量较高,同时受高盐、干旱和低温等胁迫的明显诱导,推测这些逆境胁迫诱导根系中和基因的表达,提高抗逆性,而在硝态氮和铵态氮缺乏胁迫时,根系中这2个基因都受到明显抑制,反映了和基因在调控不同逆境胁迫时的分子调控机制的差异。
氮素是影响作物产量的重要因素之一,可影响植物生长发育和水分利用效率[28]。研究[29–30]表明,转录因子可直接调控氮吸收相关基因的表达,也可以通过各种信号途径调控根系发育及氮素代谢平衡。本研究中,探讨了硝态氮或铵态氮缺乏胁迫对玉米根系和叶片中基因表达水平的影响。铵态氮缺乏胁迫下,叶片中的和表达水平呈下降趋势;在铵态氮缺乏胁迫1 h时,根系中的和出现明显上调,硝态氮缺乏胁迫24 h时,受明显诱导上调,可能这3个基因在玉米抵御铵态氮或硝态氮缺乏胁迫中起着重要作用。JAGADHESAN等[31]研究报道,低氮条件下,水稻叶片中转录因子表达量很低,而根系中在胁迫早期其表达量显著上调,超表达该基因显著提高了水稻的氮吸收效率和产量。、和同属于亚家族I,但在应答铵态氮缺乏或硝态氮缺乏胁迫时表现出不同的表达模式,也反应了该家族基因的表达模式和蛋白功能的分化。在今后的研究中,可以将该家族基因作为调控氮素代谢平衡的重要候选基因。
[1] CAO L R,LU X M,ZHANG P Y,et al.Systematic analysis of differentially expressed maizegenes between drought and rewatering transcriptome reveals bZIP family members involved in abiotic stress responses[J].International Journal of Molecular Sciences,2019,20(17):4103.
[2] HUANG X,OUYANG X H,YANG P Y,et al.FHY3 and HY5 positively mediate induction of COP1 transcription in response to photomorphogenic UV-B light[J].The Plant Cell,2012,24(11):4590–4606.
[3] JAKOBY M,WEISSHAAR B,DRÖGE-LASER W,et al.bZIP transcription factors in[J].Trends in Plant Science,2002,7(3):106–111.
[4] LINDEMOSE S,O'SHEA C,JENSEN M K,et al. Structure,function and networks of transcription factors involved in abiotic stress responses[J]. International Journal of Molecular Sciences,2013,14(3):5842–5878.
[5] SORNARAJ P,LUANG S,LOPATO S,et al.Basic leucine zipper(bZIP) transcription factors involved in abiotic stresses:a molecular model of a wheat bZIP factor and implications of its structure in function[J].Biochim Biophys Acta,2016,1860,46–56.
[6] ZANDER M,LA CAMERA S,LAMOTTE O,et al.class-II TGA transcription factors are essential activators of jasmonic acid/ethylene-induced defense responses[J].The Plant Journal:for Cell and Molecular Biology,2010,61(2):200–210.
[7] AGARWAL P,BARANWAL V K,KHURANA P. Genome-wide analysis of bZIP transcription factors in wheat and functional characterization of a TabZIP under abiotic stress[J].Scientific Reports,2019,9:4608.
[8] WANG W W,WANG Y F,ZHANG S M,et al. Genome-wide analysis of the abiotic stress-related bZIP family in switchgrass[J].Molecular Biology Reports,2020,47(6):4439–4454.
[9] YANG S Q,XU K,CHEN S J,et al.A stress-responsive bZIP transcription factorimproves drought and oxidative tolerance in rice[J].BMC Plant Biology,2019,19(1):260.
[10] GAI W X,MA X,QIAO Y M,et al.Characterization of the bZIP transcription factor family in pepper (L.):positively modulates the salt tolerance[J].Frontiers in Plant Science,2020,11:139.
[11] NIJHAWAN A,JAIN M,TYAGI A K,et al.Genomic survey and gene expression analysis of the basic leucine zipper transcription factor family in rice[J].Plant Physiology,2007,146(2):323–324.
[12] LIU J Y,CHEN N N,CHEN F,et al.Genome-wide analysis and expression profile of the bZIP transcription factor gene family in grapevine()[J].BMC Genomics,2014,15:281.
[13] WEI K,CHEN J,WANG Y,et al.Genome-wide analysis of bZIP-encoding genes in maize[J].DNA Research,2012,19(6):463–476.
[14] ZHOU Y,XU D X,JIA L D,et al.Genome-wide identification and structural analysis of bZIP transcription factor genes in[J].Genes,2017,8(10):288.
[15] YANG Y,YU T F,MA J,et al.The soybeantranscription factor geneGmbZIPconfers drought and salt resistances in transgenic plants[J].International Journal of Molecular Sciences,2020,21(2):670.
[16] RONG S Y,WU Z Y,ZHANG Z,et al.Genome-wide identification,evolutionary patterns,and expression analysis ofgene family in olive(L.)[J].Genes,2020,11(5),510–515.
[17] LI H Y,LI L X,SHANGGUAN G,et al.Genome-wide identification and expression analysis ofgene family inL[J].Scientific Reports,2020,10:15521.
[18] LIU M Y,WEN Y D,SUN W J,et al.Genome-wide identification,phylogeny,evolutionary expansion and expression analyses oftranscription factor family in tartaty buckwheat[J].BMC Genomics,2019,20(1):483.
[19] LI D Y,FU F Y,ZHANG H J,et al.Genome-wide systematic characterization of the bZIP transcriptional factor family in tomato (L.)[J]. BMC Genomics,2015,16:771.
[20] WANG W B,QIU X P,YANG Y X,et al.Sweetpotato bZIP transcription factorIbABFconfers tolerance to multiple abiotic stresses[J].Frontiers in Plant Science,2019,10:630.
[21] HOSSAIN M A,CHO J I,HAN M,et al.The ABRE- binding bZIP transcription factor OsABF2is a positive regulator of abiotic stress and ABA signaling in rice[J].Journal of Plant Physiology,2010,167(17):1512–1520.
[22] HERATH V,VERCHOT J.Insight into thegene family in:genome and transcriptome analysis to understand the roles of gene diversification in spatiotemporal gene expression and function[J]. International Journal of Molecular Sciences,2020,22(1):253.
[23] CHANG Y,NGUYEN B H,XIE Y J,et al. Co-overexpression of the constitutively active form of OsbZIP46 and ABA-activated protein kinase SAPK6improves drought and temperature stress resistance in rice[J].Frontiers in Plant Science,2017,8:1102.
[24] ZHANG L N,ZHANG L C,XIA C,et al.A novel wheat bZIP transcription factor,TabZIP60,confers multiple abiotic stress tolerances in transgenic[J]. Physiologia Plantarum,2015,153(4):538–554.
[25] YING S,ZHANG D F,FU J,et al.Cloning and characterization of a maize bZIP transcription factor,ZmbZIP72,confers drought and salt tolerance in transgenic[J].Planta,2012,235(2):253–266.
[26] MA H Z,LIU C,LI Z X,et al.ZmbZIP4contributes to stress resistance in maize by regulating ABA synthesis and root development[J].Plant Physiology,2018,178(2):753–770.
[27] 周文涛,龙文飞,戈家敏,等.增密减氮对双季稻光合特性及水分利用率的影响[J].湖南农业大学学报(自然科学版),2022,48(1):1–7.
[28] GAUDINIER A,RODRIGUEZ-MEDINA J,ZHANG L F,et al.Transcriptional regulation of nitrogen-associated metabolism and growth[J].Nature,2018,563:259–264.
[29] CHEN X B,YAO Q F,GAO X H,et al.Shoot-to-root mobile transcription factor HY5 coordinates plant carbon and nitrogen acquisition[J].Current Biology,2016,26(5):640–646.
[30] JAGADHESAN B, SATHEE L, MEENA H, et al. Genome wide analysis oftranscription factors reveals their role in nitrogen stress tolerance of rice[J]. Scientific Reports,2020,10:9368.
The identification of 11of maize and their expression pattern analysis
JIA Liqiang1,LIU Yang2,ZHAO Qiufang3,CHEN Shu3
(1.School of Life Sciences, Guizhou Education University, Guiyang, Guizhou 550018, China; 2.College of Modern Agriculture, Jiaxing Vocational and Technical College,Jiaxing, Zhejiang 314036, China; 3. South Subtropical Crop Research Institute, Chinese Academy of Tropical Agricultural Sciences, Zhanjiang, Guangdong 524091, China)
Using maize inbreed line Zheng58 for experimental material, bioinformatic and expression pattern analysis of 11genes were performed at the experimental conditions of 200 mmol/L NaCl, 20% PEG6000, 4 ℃ and nitrate or ammonium deficiency stresses. The phylogenetic tree analysis showed that 11could be subdivided into 2 subgroups. RT-PCR results revealed that different expression patten ofwere detected in different tissues, indicating different biological roles in plant growth and development. Under simulated salt, drought, low temperature and nitrogen stress,were widely regulated,andof subgroup I were obviously upregulated by salt by two fold, and inhibited by PEG6000, while,,andof subgroup II were upregulated by salt, PEG6000 and low temperature stresses. And,andwere obviously downregulated by nitrate deficient stress, while upregulated by ammonium deficient stress, indicating different biological function roles of these genes in responsive to various stresses.
maize; basic leucine zipper; gene family; adverse stress; gene identification; gene expression; phylogenetic tree
S513;Q786
A
1007-1032(2022)04-0407-09
贾利强,刘洋,赵秋芳,陈曙.玉米11个基因的鉴定及表达特征分析[J].湖南农业大学学报(自然科学版),2022,48(4):407–415.
JIA L Q,LIU Y,ZHAO Q F,CHEN S.The identification of 11of maize and their expression pattern analysis[J].Journal of Hunan Agricultural University(Natural Sciences),2022,48(4):407–415.
http://xb.hunau.edu.cn

2021–08–26
2022–06–06
贵州师范学院博士项目(2021BS016)
贾利强(1976—),男,河北石家庄人,博士,副教授,主要从事植物分子生物学研究,liqiangj@163.com
责任编辑:毛友纯
英文编辑:柳正
