Sigma54依赖性转录起始
2022-09-07梅子青
洪 婷, 梅子青, 王 丰*
(1)北京理工大学生命学院,分子医学与生物诊疗工业和信息化部重点实验室, 北京 100081;2)北京科技大学化学与生物工程学院,北京 100083)
基因转录是指RNA聚合酶以DNA双链中的一条链(无义链)为模板,以核苷三磷酸(ATP、GTP、CTP和UTP)为底物,通过碱基互补配对原则合成核糖核苷酸(RNA)过程[1-4]。基因转录对于延续及维持生命活动极为重要,始终受到机体的严格调控[5, 6]。基因转录主要包括起始、延伸和终止3个过程[4]。起始是指RNA聚合酶全酶(holoenzyme)结合在启动子区域,形成启动子-聚合酶复合物,打开DNA双链形成转录泡,并以无义链为模板,按照5′—3′方向启动合成RNA的过程。延伸是指RNA聚合酶全酶在转录起始合成10 nt左右RNA后进行的延伸反应。RNA聚合酶全酶在此过程中需要解开下游的DNA双链,并使后方的DNA恢复双链状态。终止是指转录完成前,RNA聚合酶全酶合成一段能形成特定结构的核苷酸序列,使转录停止、释放RNA的过程[4]。
RNA聚合酶全酶是基因转录的主要承担者,其亚基组成极为保守。原核细胞的RNA聚合酶全酶由RNA聚合酶核心酶(core RNA polymerase,RNAP)及可变的sigma因子组成,其中RNAP由5个固定亚基α1、α2、β、β′和ω组成[7, 8],sigma因子则多种多样。由于不同类型的sigma因子对应着不同基因的启动子,所以RNAP结合不同sigma因子后会选择性启动其对应基因的转录[9-11]。
Sigma因子根据功能不同主要分为2类:一类为sigma70,识别启动子-35 ~-10区域,主要辅助RNAP催化管家基因的自发转录[12, 13];另一类为sigma54,识别启动子-24 ~-12区域,主要辅助细菌中响应环境胁迫信号及代谢信号相关基因的转录[10, 14-17]。sigma54依赖的基因转录不能自发起始,需在感知到外界胁迫及代谢信号后才能启动[13, 18]。感受外界信号并将其传递给sigma因子的蛋白质为细菌增强子激活蛋白质(bacterial enhancer binding protein,bEBP),在接收到特定胁迫或者代谢信号后,结合在启动子上游UAS(upstream activating sequence)的bEBP会与RNA聚合酶全酶相互作用[19, 20],进而解除sigma54对RNA聚合酶的转录抑制,打开DNA双链,激活下游基因转录[21, 22]。
近年来,对原核细胞转录调控机制的研究大多都聚焦在起始阶段,尤其是bEBP和sigma54因子所参与的基因转录[7]。一些关键的结构生物学证据已经部分揭示出sigma54抑制RNAP、bEBP蛋白解除sigma54对RNAP的抑制,以及促进启动子DNA双链打开等不同转录起始阶段的分子机制。本文从结构生物学角度详述由bEBP和sigma54激活的基因转录起始调控机制的进展。
1 Sigma54因子与转录起始
Sigma54是细胞内响应胁迫信号和代谢信号的转录因子,由3个保守区域RⅠ、RⅡ和RⅢ组成(Fig.1)。其中,RⅠ由N端2个α螺旋构成,1~56位氨基酸,主要负责与DNA、bEBP及RⅢ的ELH结构域相互作用[23];RⅡ由57~120位氨基酸组成,序列保守性较差,在转录起始的部分阶段中占据了DNA及DNA/RNA通道[24];RⅢ由121~477位氨基酸组成,由CBD、ELH-HTH和RPoN3个结构域构成。其中CBD是sigma54与RNAP相互作用的主要区域[25];RPoN结构域负责与启动子DNA的-24位点相互作用[26, 27];这2个结构域中间有一段长螺旋结构(extra-long helix, ELH),其C端紧跟着螺旋-转角-螺旋(helix-turn-helix, HTH)结构域,HTH结构域结合在DNA的-12位点[1, 22, 28]。

Fig.1 Structural domain of sigma54 Structure of sigma54 derives from the structure of holoenzyme (PDB:5BYH)[1]
最近,一系列结构生物学研究揭示了sigma54依赖的基因转录起始过程中的部分复合物结构[1](Fig.2),包括:RNA聚合酶全酶、封闭式复合物(RNA polymerase closed complex,RPc)、中间复合物(RNA polymerase intermediate complex,RPi)、DNA部分装载的中间复合物(intermediate partially loaded DNA complex,RPip)和开放式启动子复合物(RNA polymerase open complex,RPo),这些结构解析了由bEBP激活的sigma54依赖性基因转录起始过程的关键步骤。
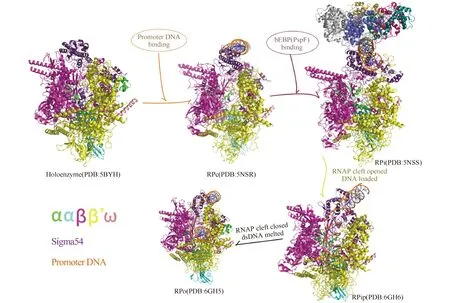
Fig.2 Process of transcription initiation[1] There are five structures of the transcription initiation process, which are holoenzyme (PDB:5BYH), RNA polymerase closed complex RPc (PDB:5NSR), RNA polymerase intermediate complex RPi (PDB:5NSS), intermediate partially loaded DNA complex RPip (PDB:6GH6) and RNA polymerase open complex RPo(PDB:6GH5)
1.1 结合sigma54因子的RNA聚合酶全酶
RNA聚合酶全酶由5个核心酶亚基α1、α2、β、β′和ω以及sigma54因子组成。通过组装来自肺炎克雷伯菌的sigma54因子与大肠杆菌RNAP的5个亚基(α1、α2、β、β′和ω)以及28bp的启动子DNA片段。研究者制备并解析了分辨率为3.76Å的RNA聚合酶全酶冷冻电镜结构[24]。结果正如Fig.3A所示,sigma54整体位于RNAP上方,钩子状的sigma54-RI与ELH位于RNAP裂隙正上方,RII.1的2个α螺旋占据了RNAP的DNA通道;由无规卷曲组成的sigma54-RII.2 和RII.3包埋在RNAP内部的RNA通道位置[29]。由2个α螺旋亚结构域组成的sigma54-CBD分别与RNAP的β翼(β flap 835~937位氨基酸)、β的C端(1 267~1 320位氨基酸)、β′拉链(β′ zipper 35~100位氨基酸)、β′对接结构域(β′ docking domain 370~420位氨基酸)以及α亚基的C端(282~308 位氨基酸)区域发生明显的相互作用[29]。RI的α1螺旋位于ELH结构域上方,并与其相互作用,挡在了RNAP裂隙上方[28, 29]。sigma54的C端的RPoN结构域与sigma54的其他结构域,以及RNAP并未发生相互作用[22, 29]。尽管在制样时添加了DNA,但在此结构中未发现任何DNA密度。
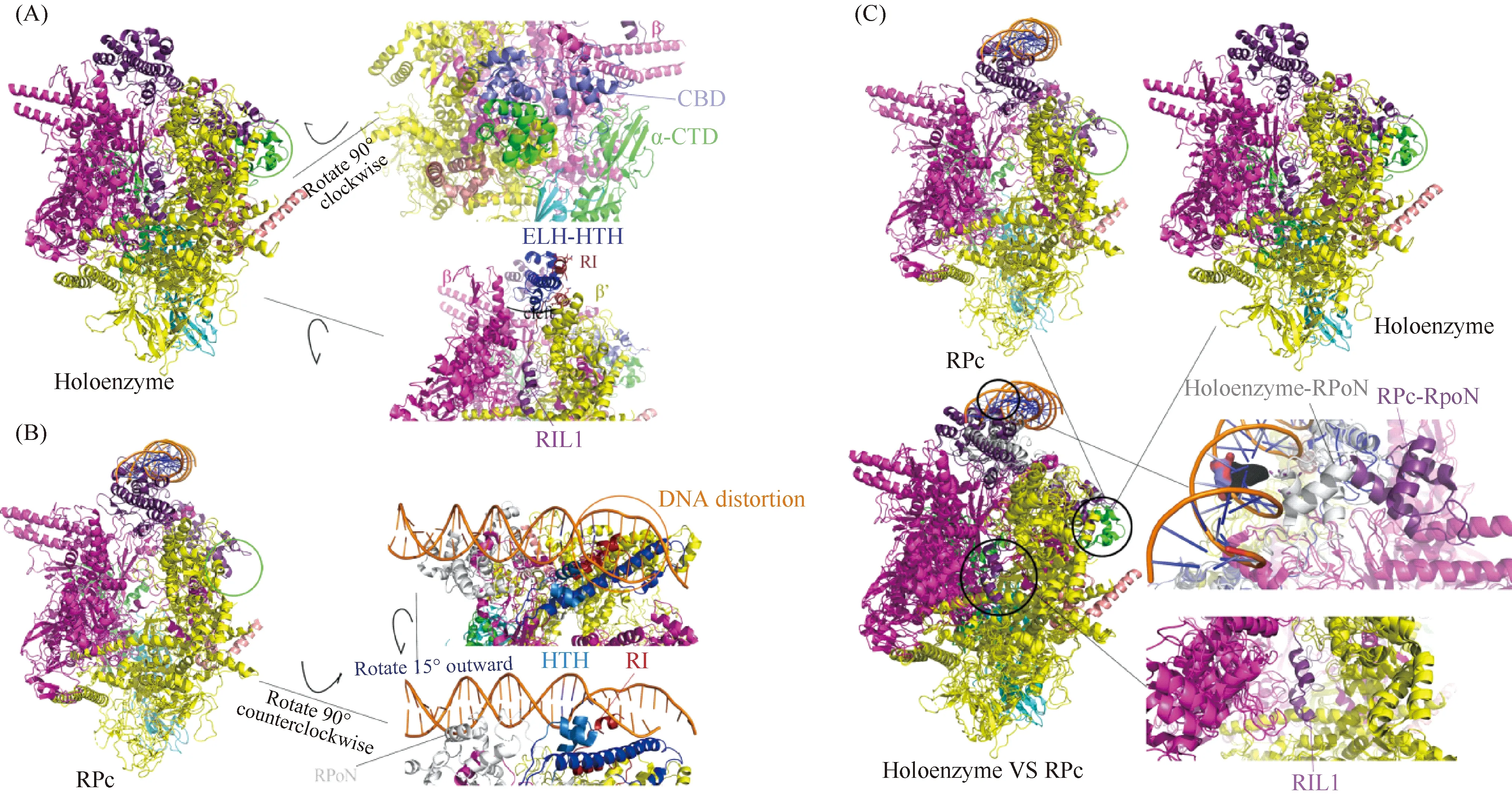
Fig.3 Structural comparison of RPc with holoenzyme (A) In holoenzyme, sigma54-CBD binds to RNAP, and RII.1 is in the crack. (B) The DNA in RPc is twisted and interacts with sigma54-RPoN, HTH and RI. (C) Comparison between RPc and holoenzyme: RPoN turns, C-terminal of α subunit of RNAP is lost and RII.1 is unseen in RPc
1.2 封闭式复合物(RPc)
RPc是RNA聚合酶全酶结合启动子DNA的转录起始早期状态。通过组装来自肺炎克雷伯菌的sigma54、大肠杆菌的RNAP及苜蓿中华根瘤菌的固氮酶启动子DNA(-35—+28、其中-12/-11错配)获得封闭式复合物,并解析了分辨率为3.0 Å的冷冻电镜结构[22]。将RPc与RNA聚合酶全酶进行结构对比(Fig.3)可得出如下信息:(1)sigma54-RI 是抑制RNAP转录起始的关键区域,位于DNA进入RNAP裂隙的入口处。在RPc中,sigma54-RI通过与sigma54-ELH发生相互作用,使得sigma54在RNAP裂隙上方形成空间位阻,阻碍DNA进入RNAP裂隙。已有的生化实验显示,将sigma54-RI删除或者将sigma54-ELH中336位Arg突变为Ala,在预先形成转录泡的情况下,不需要bEBP的参与,即能自发起始转录[26, 29]。在RPc中,sigma54被锁定在裂隙上方,由β和β′亚基的钳子部分组成的RNAP裂隙处最窄只有15 Å,无法容纳A型(直径25.5 Å)、B型(直径23.7 Å)和Z型(直径18.4 nm)等任何双链DNA的进入。与RNA聚合酶全酶不同,RPc中未观察到sigma54-RII.1占据在RNA聚合酶的裂隙中。(2)由于裂隙不能容纳DNA,在RPc中仅能看到sigma54因子与DNA相互作用;RPoN结构域相较于RNA聚合酶全酶发生了约60°的顺时针旋转,结合在DNA的-24处;sigma54-HTH与-12处DNA相互作用;sigma54-RI与-12下游处DNA也存在相互作用;位于HTH和RI之间的sigma54-ELH刚性结构会使DNA伸展,导致-12下游的DNA小沟明显变宽,产生不同于B型DNA的扭曲(Fig.3B,3C)。(3)sigma54中主要的RNAP结合结构域CBD位于RNA出口处。原本在RNA聚合酶全酶中与CBD相互作用的α亚基C端,在RPc中因密度低而不可见,可能是因为结构变化导致相互作用丧失。而CBD与RNAP的β、β′亚基相互作用,依然挡在RNA出口[22]。总之,该结构显示,在RPc结构中,sigma54对转录起始有明显的抑制作用,主要是因为RI通过与ELH相互作用挡在了RNAP裂隙上方,阻止了DNA进入;在HTH、RI及ELH的作用下,DNA已经开始发生扭曲[2]。
1.3 中间复合物(RPi)
RPi是指RPc向开放式启动子复合物过渡的中间状态,称为中间复合物。已有生化实验证明,在ADP·AlFx(ATP水解过渡态类似物)存在时,bEBP与sigma54相互作用较强[30]。据此,在有ADP·AlFx条件下,将PspF(1种bEBP)与RNA聚合酶全酶及启动子DNA组装成RPi复合物,并解析了分辨率为5.8 Å的冷冻电镜结构[22]。结果正如Fig.4A所示,在RPi结构中,不对称的6聚体 PspF位于RNA聚合酶全酶和DNA的上方,与DNA以及sigma54直接相互作用。其中,PspF的L1/L2 环分别与-24 — -12间的DNA骨架及-12下游的沟槽相互作用。sigma54-RⅠ是sigma54中与PspF相互作用的主要区域。在RPi中,sigma54-RⅠ的α1区域密度更清晰,长度较RPc明显增长,并向上旋转,与PspF的L1/L2环相互作用,形成楔子形结构插在DNA双链中,帮助稳定转录泡形成,进而撬动DNA链分离(Fig.4B)[22]。与RPc相比,RPi中sigma54-RⅠ与ELH相互作用的区域随着RⅠ的旋转发生了位置改变,导致两者不再完全阻挡在RNAP裂隙上方,同时DNA也随ELH发生了偏移;RNAP裂隙顶部比在RPc中拓宽15 Å左右,更加有利于双链DNA进入通道。此外,在RPi中,CBD结构域上移与RPoN相互作用,不再阻挡RNA出口。综上,在RPi中,sigma54对转录起始的抑制作用已经被bEBP基本解除[22]。但转录的最终启动还需要进一步打开DNA链,形成转录泡,并将DNA载入RNAP裂隙的活性位点处。PspF可与DNA和sigma54分别相互作用,暗示着PspF等bEBP蛋白极有可能利用其水解ATP释放的能量来改变与其相互作用的sigma54及启动子DNA区域的双链构象,以解除sigma54对RNAP的抑制,并解开DNA双链,最终激活转录起始。
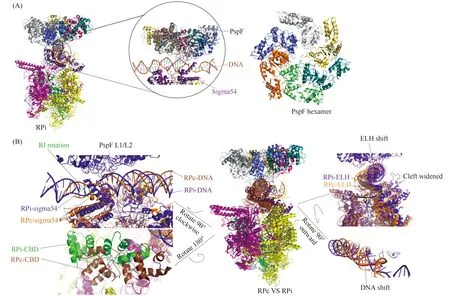
Fig.4 Structural comparison of RPi with RPc (A) PspF hexamer interacts with DNA and sigma54. (B) Comparison between RPi and RPc: The α1 of RI stretches, rotates and interacts with L1/L2 of PspF, as does DNA; ELH and DNA also shift with a certain degree, and the gap expands; CBD moves upward
1.4 DNA部分装载的中间复合物(RPip)
RPip是指部分DNA装载的起始复合物的中间状态。已有研究表明,在预先形成转录泡的情况下,将sigma54-ELH中336位Arg(R)突变为Ala(A),转录起始不再需要bEBP的参与即能自发进行[26, 29]。据此,将肺炎克雷伯菌的sigma54-ELHR336A突变体与非模板链上-10 — -1位完全错配的苜蓿中华根瘤菌启动子DNA及大肠杆菌RNAP进行组装,捕获到了DNA部分装载中间复合物即RPip,并解析出其4.1 Å分辨率的冷冻电镜结构[31]。在引入ELHR336A突变及DNA错配的RPip结构中,sigma54的RPoN与HTH的相对位置与RPc、RPi中类似,仍分别结合在DNA的-24位点和-12位点(Fig.5A)。但相较于RPi,RPip中的sigma54和DNA的整体结构发生顺时针旋转,向RNAP裂隙偏移(Fig.5C)。值得注意的是,RPip中的RNAP裂隙比RNA聚合酶全酶、RPc和RPi中更宽阔(约56 Å)(Fig.5A,B)。与RPi相比,RPip中的DNA在-10位置发生了约30°扭转,导致-10 — -1的DNA夹在sigma54-ELH与β叶(372~375)和β′卷曲之间。来自β叶(β lobe)(372~375)的Phe、Gly、Glu和Phe(PGEP)4个氨基酸同β′卷曲及其周围带有大量正电荷的结构域,通过相互作用来稳定-10 — -1 DNA,引导DNA进入RNAP裂隙的下游DNA通道中。此外,在RPip中,ELH变短,且密度缺失,暗示ELH可能因不再与RⅠ相互作用,在DNA装载过程中发生了构象变化(Fig.5C)。
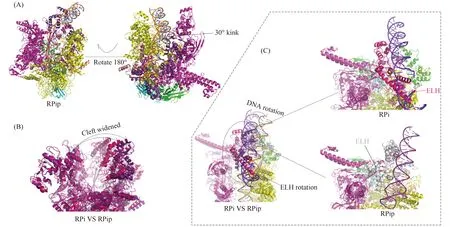
Fig.5 Structural comparison of RPip with Rpi (A) The DNA in RPip is partially loaded, and a 30° twist is generated. (B) The cleft in RPip is further enlarged compared to RPi. (C) Compared with RPi, RPip rotates clockwise with ELH-HTH, RPoN and DNA; and the ELH domain becomes shorter
1.5 开放式启动子复合物(RPo)
在RPo中,局部解链的启动子DNA结合在RNAP裂隙中,RNAP裂隙关闭。RPo与上述RPip在同一样品中获得,分辨率为3.4 Å(Fig.6)。在RPo中,RNAP裂隙再次关闭,恢复到与RPc同等宽度(Fig.6B)。sigma54-ELH相较于RPip中变得更长且发生弯折,插入到NT(非模板链)和T(模板链)之间,将DNA双链在-11处打开(Fig.6C),这与sigma70依赖的基因转录起始过程明显不同。在sigma70依赖的基因转录开放复合物中,RPo依靠sigma70中保守的Trp来稳定-11位的核苷酸[32, 33]。结果正如Fig.6A所示,RPo中形成的转录泡含有13个单链DNA核苷酸(-11 — +2)以及1个双链DNA核苷酸对(+3)。其中,非模板链NT位于sigma54-ELH上方,沿着由β叶和β′卷曲形成的含大量带正电氨基酸的通道放置,而模板链T则处于β亚基(538~543)、β′卷曲环(318~323) 和sigma54-RII.3 (107~112) 构成的“隧道”中。与sigma54-CBD相连的sigma54-RII.3(107~112)含有较多酸性氨基酸,可能参与引导RNA从RNA出口释放[29]。sigma54-CBD在RPo中的位置与RPc中相同,也挡在RNA出口处。此外,在转录泡下游双链解开的位置,+2NT插入到β亚基的疏水口袋中,+1NT与β亚基的183位Trp发生疏水相互作用,这与sigma70依赖的基因转录中的开放式复合物十分相似[33]。
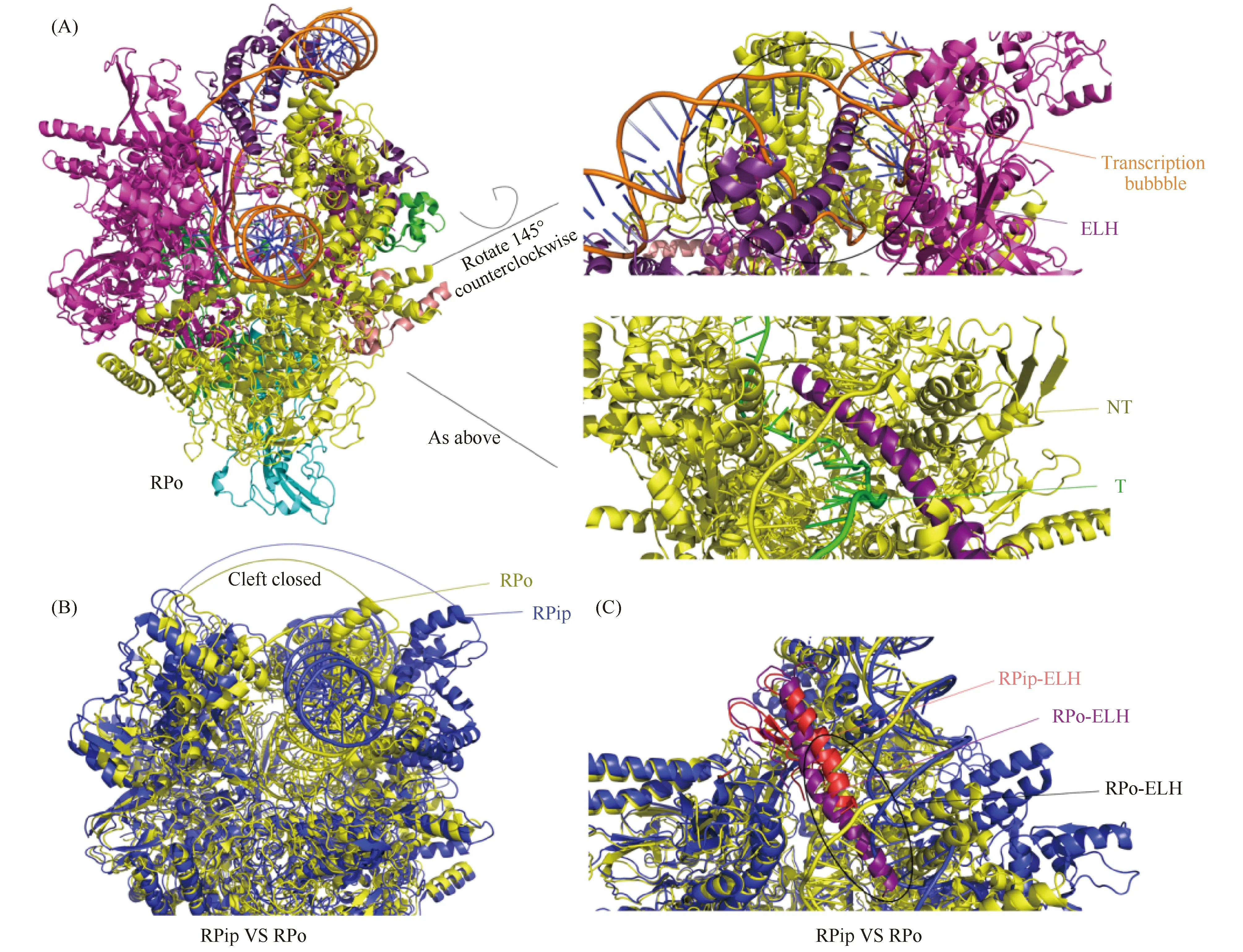
Fig.6 Structural comparison of RPo with Rpip (A)The transcription bubble is formed in RPo, and the ELH is inserted into the DNA double strand to stabilize the transcription bubble. (B) Compared to RPip, the cleft in RPo is closed again. (C) Compared with RPip, ELH in RPo bends and extends
1.6 Sigma54依赖性基因转录起始的装载-解链耦联模型
通过比较RPip和RPo两个状态的结构,英国帝国理工大学张晓东课题组提出了sigma54依赖性转录中DNA装载和转录泡打开过程的分子机制模型[1]。结构证据显示,RPip到RPo的转变是由β′钳绕夹钳底部旋转22°引发,这一旋转导致与DNA链相互作用的β′螺旋线圈向下推动,由此产生的挤压力迫使DNA双链解开,并将DNA加载到裂隙的活性部位中。简而言之,夹钳闭合会导致DNA同步解链及最终装载,即装载与解链耦合机制[31, 34, 35]。该模型与sigma70依赖的基因转录起始过程类似,但与sigma70依赖性转录的“先解链、后装载”模型以及“先装载、后解链”模型明显不同[36]。
2 bEBP的结构与功能
bEBP是sigma54依赖性基因转录起始的激活蛋白质,通常含有3个结构域:N-端调节结构域、中间的AAA+结构域和C端DNA结合结构域。其中调节结构域可通过结合配体(各种胁迫信号分子或代谢信号分子)、被磷酸化修饰或者与其他蛋白质相互作用等来调控bEBP的活性;AAA+结构域负责bEBP的寡聚化、水解ATP分子及通过对sigma54进行结构重塑来激活转录[37];DNA结合结构域主要负责结合启动子上游的增强子(enhancer/UAS-upstream activating sequence),帮助bEBP靠近结合在启动子区域的RNA聚合酶全酶[38]。并不是所有bEBP都同时包含这3个结构域,有的bEBP缺少调节结构域,有的bEBP缺少DNA结合结构域,有的bEBP例如FleT甚至会同时缺失调节结构域和DNA结合结构域,暗示bEBP生物功能在进化过程中分化出多样性[39-41]。根据生物功能和调控模式的不同,bEBP可分为5类(Table 1)[2, 42]。
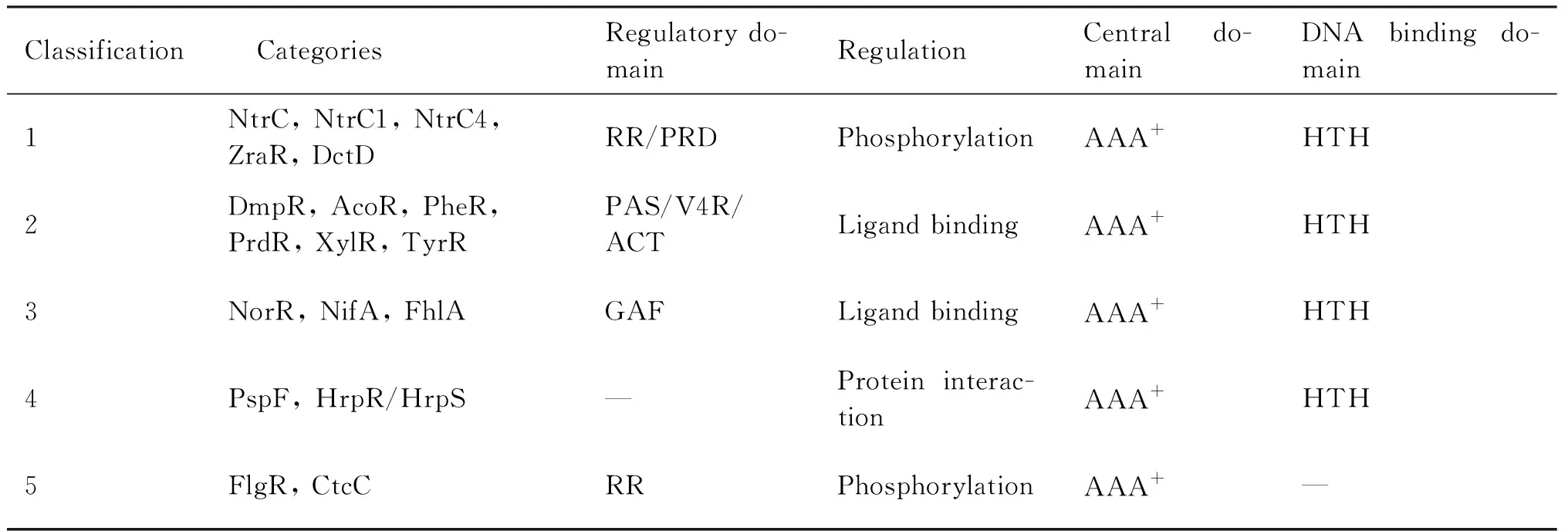
Table 1 bEBP protein classification and domain composition[2]
2.1 bEBP的AAA+结构与功能
位于bEBP序列中间的AAA+结构域是AAA超家族(ATPases associated with diverse cellular activities superfamily, AAA)成员的特征性结构域[43]。该家族成员大多借助于AAA+结构域形成寡聚体,并通过水解ATP参与细胞内蛋白质降解、膜融合、DNA复制、微管运输以及转录激活等多种生命事件[43, 44]。
经典的AAA+结构域含有7个保守区域:C1-C7[45]。PspF、NtrC及ZraR等bEBP蛋白的晶体结构表明,bEBP可通过AAA+结构域以寡聚环状形式组装在一起来执行功能[30, 46-48],常以六聚或七聚状态发挥活性[42]。寡聚化对于bEBP激活转录起始十分重要[49]。每1个AAA+结构域单体都含有1个α/β亚结构域和1个小的α螺旋亚结构域[49]。ATP结合位点位于寡聚物中相邻2个单体的裂隙[48]。AAA+结构域含有2个重要的基序:Walker A和Walker B。Walker A负责结合ATP[43],其保守序列GxxxxGK(T/S,AAA蛋白中Walker A 序列后保守的丝/苏氨酸在EBP蛋白中全都变成了天冬/谷氨酸)形成的P-loop可与ATP分子的磷酸基团相互作用[50],是bEBP蛋白结合ATP分子的必要条件[51, 52]。Walker B基序负责水解ATP,其保守序列为hhhhDE (h为疏水氨基酸)。Walker B是bEBP蛋白水解ATP分子所必须的基序,其保守的Asp在水解核苷酸时,负责螯合镁离子[51, 52]。除了Walker A和Walker B外,AAA+结构域还含有sensor I 和sensor II 两个重要保守区域,分别位于C6和C7[53]。sensor I上保守的Thr可通过1个水分子与Walker B中的Glu相互作用,推测与bEBP蛋白水解ATP分子引起L1、L2 loop的构象变化有关[54]。sensor II位于小α亚结构域的第3个α螺旋,推测与核苷酸结合有关,其附近的精氨酸手指(arginine finger)参与了ATP水解驱动的构象变化[43, 49, 53, 55-57]。在相邻亚基间隙形成的ATP水解空腔中,一个亚基的精氨酸手指会伸入相邻亚基的活性位点,与ATP的γ-磷酸基团相互作用[58]。综上,AAA+结构域的寡聚化可帮助形成水解ATP分子的空腔,而ATP水解导致的构象变化对于bEBP发挥作用十分重要。
除了含有AAA+结构域的常规特征外,bEBP蛋白的AAA+结构域还有2段额外的插入序列,在AAA+结构域上形成2个暴露在外的无规卷曲环,命名为L1和L2。其中L1插在C3的C端,是bEBP蛋白的AAA+结构域中一段保守的无规卷曲环,其保守序列为GAFTGA,位于α/β亚结构域[49, 53];L2插在C5和C6之间[42]。因此,在AAA+结构域因水解ATP发生构象变化时,L1和L2 loop很可能会发生位置变化,引发相应的生理事件。
2.2 bEBP的谷氨酸开关模型
bEBP蛋白能够在结合与水解ATP分子的过程中,与sigma54发生相互作用,引发sigma54和RNAP构象重塑,进而启动转录。研究表明,在结合ADP·AlFx时,bEBP的L1从朝向环中心转变为向寡聚化的平面外伸展,并通过L1 loop上的GAFTGA与sigma54维持稳定的相互作用[30, 49, 53]。这种构象变化可用bEBP蛋白的“谷氨酸开关”模型加以解释[2]。以PspF(1~275)为例,Walker A在结合不同核苷酸时,其Walker B的E108位点会采取不同的构象,这种构象变化能够被sensor I中的Thr148和Gln64所感应,随后传递到L1和L2 loop[30, 55, 59]。许多AAA+蛋白都利用“谷氨酸开关”模型来行使功能[60]。
因此,bEBP很可能是通过其调节结构域来感知胁迫信号或者代谢分子信号的变化,然后改变自身的寡聚化状态,利用水解ATP分子提供的能量,重塑sigma54甚至转录起始复合物的构象,最终调控基因转录[2](Fig.7)。
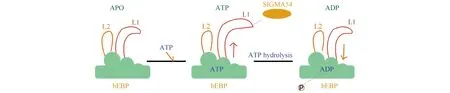
Fig.7 Schematic diagram of glutamate switch model[3] The conformation of bEBP protein changes greatly during the process of binding ATP and hydrolyzing ATP. Upon bEBP binding to ATP, L1 extends out of the oligomerization plane and can interact with sigma54. After hydrolyzing ATP to release the free phosphate group, the L1 of bEBP retracts toward the oligomerization plane. This dynamic process leads to the conformational remodeling of sigma54 and the transcription complex
2.3 bEBP在转录中的作用
当前已解析出结构的样品中,只有RPi是含有bEBP的转录中间状态复合物(Fig.4A)。该状态下的bEBP分辨率低,只能从电子密度中辨别出bEBP的L1/L2结构域与DNA以及sigma54-RⅠ有相互作用。当bEBP结合到转录复合物上,sigma54的 RⅠ/ELH以及CBD结构域的构象发生改变,并且RNAP裂隙的宽度较RPc扩大了约15 Å。此外,RⅠ的N端明显变长并向上偏转,导致其可以贯穿DNA与bEBP,形成楔子样结构,以撬动DNA解链。显而易见,bEBP能够帮助DNA双链解链。目前的数据显示,RNAP裂隙的扩大主要由β′钳子移动造成(Fig.4B),但β′钳子的移动是否由接近它的ELH N端以及RⅠ的α1引起目前仍不清楚。删除sigma54-RⅠ或者将sigma54-ELH 366位Arg突变为Ala,皆可去除sigma54对RNAP的抑制,在bEBP不存在的情况下也能启动转录。这些结果提示,bEBP可以解除sigma54-RⅠ与ELH之间的相互作用。甚至bEBP主要通过与RⅠ的相互作用来改变sigma54的构像。但bEBP如何解除sigma54-RⅠ与ELH的相互作用,进而帮助转录起始的机制尚缺乏报道。在RPi中,sigma54-RⅠ与ELH发生整体偏移,不再完全阻挡在裂隙上方,但二者的相互作用仍然存在,暗示着bEBP与转录复合物的结合并未破坏RⅠ/ELH的相互作用,只是使RⅠ的构象发生变化。若要真正地破坏两者之间相互作用,或许仍然需要bEBP通过水解ATP来将化学能转化为机械能才得以实现[2]。在RPip中,不仅RNAP裂隙显著拓宽,sigma54和DNA整体偏向裂隙,而且RⅠ不可见(Fig.5)。这些结构特征进一步提示:bEBP可以直接作用于DNA,并且会通过改变RⅠ构象来改变ELH和HTH等结构域,从而影响裂隙的开合和DNA的装载。因此,为激活转录起始,bEBP还需要进一步发生结构变化,来帮助DNA解链并装载入RNAP裂隙。
3 问题与展望
3.1 Sigma54与bEBP在转录起始中的生理角色
Sigma54的3个保守区域RⅠ、RⅡ和RⅢ在转录过程中均发挥着至关重要的作用。在全酶和RPc结构中,RⅠ与来自RⅢ的ELH结构域相互作用,在RNAP裂隙入口处,担负着DNA进入开关的角色。来自RⅢ的CBD结构域与RNAP的β、β′、α亚基相互作用,位于RNA出口处,作为RNA释放的开关[22]。而RⅡ结构域在转录起始的很多状态中均处于不可见状态,只有sigma54-RⅡ.1在RNA聚合酶全酶中占据DNA通道,阻止DNA结合[29]。
在RPi中,由bEBP引发的RⅠ/ELH整体偏移使其不再完全阻挡RNAP裂隙。此时,β′亚基钳子也随RⅠ/ELH发生偏移,从而扩大RNAP裂隙。β′亚基钳子的位置变化提示其顶端可能与ELH的N端发生相互作用,但目前因电镜结构分辨率较低,缺少可信的证据支持。裂隙打开的RPip与RPc在制样方法上的区别仅是将sigma54的336位Arg突变为Ala,这个事实进一步支持了维持裂隙闭合的力量是由RⅠ与ELH之间的相互作用所提供这一观点。RⅠ的N端1~15位氨基酸部分在RNA聚合酶全酶和RPc中均不可见,但是8~15位氨基酸部分在RPi中以明显变长的α螺旋呈现,且向上旋转约45°插入DNA中。因此,RⅠ很可能是bEBP蛋白质用以撬动转录复合物与推动DNA双链打开的一个作用支点。此外,bEBP的结合导致来自sigma54 RⅢ的CBD结构域整体向上移动,离开RNA出口。RPc、RPi、RPip和RPo等结构的对比分析结果显示,CBD结构域的位置仅在RPi中发生上移,离开RNA出口;而在其他结构中的位置均挡在RNA出口[22, 31]。这些结果表明,使bEBP解除sigma54抑制转录起始发生的关键因素很可能是sigma54-RⅠ/ELH的移动[22],而并不是CBD结构域的上移。
在RPip中,sigma54上1~114位氨基酸部分的密度不可见,提示RⅠ所参与的相互作用在此构象中很可能已被解除。此外,与RⅠ距离较近的ELH上293~335之间的区域在RPip中也变得柔性不可见,导致ELH变短。此时,sigma54与DNA一起发生旋转偏移,偏向RNAP裂隙。通过比对发现,在整个偏移过程中,sigma54的ELH-HTH、RPoN以及DNA的相对位置并未发生改变,表明它们是以固定相互作用模式进行整体移动[22, 31]。但sigma54整体偏移方向与RPi中RⅠ/ELH偏移的方向相反,且β′亚基钳子中距离ELH最近的2个α螺旋相对位置在RPi中发生扭曲,而在RPip中则为同步偏移[22, 31]。RPip中不仅裂隙拓宽,且DNA部分装载,之后自发形成RPo构象;RPi的变化与RPip矛盾,提示RPi不是RNAP裂隙打开的关键步骤,而仅是bEBP与RⅠ和DNA相互作用引起的构象改变。真正有效的裂隙打开应该发生在RPip之前,由RPi之后RⅠ与ELH之间的相互作用解除而引发,但直接导致裂隙打开的作用力来自哪一结构域仍不清楚。从RPi中可以发现RⅠ与bEBP有明显的相互作用,而且bEBP是该转录体系引入外力的一个重要作用点,所以目前认为,bEBP能够通过水解ATP改变转录复合物的构象,尤其是破坏sigma54中RⅠ与ELH的相互作用和直接改变DNA结构来起始转录。
在RPo中,ELH基本恢复其在RPc中的长度,弯折且插入到DNA双链中,此时RNAP的裂隙也再次关闭。此外,在RPo中,ELH由在RPip中较短且处于DNA外侧,变得更长且插入到DNA双链中,所以ELH更有可能是引发结构自RPip向RPo转变的关键结构域。RPip中ELH变短且N端与β、β′亚基相距较远,无明显相互作用;而RPo中的β、β′钳子相互靠近,ELH也延长,两钳子位于裂隙中的无规卷曲(β:356~385,β′:251~325)与ELH的距离非常近,认为ELH与这些卷曲有相互作用。由于RPip与RPo来自同一样品,不会有额外的物质造成这种结构变化。所以我们推测,RPi到RPo的结构变化来自于ELH N端的随机摆动,以及其N端与β、β′卷曲间的相互作用,当三者发生相互作用时,两钳子之间的距离被拉近,并导致DNA的解链和装载。但由于结构分辨率较低,与ELH相互作用的氨基酸目前仍无法确定。
3.2 展望
细菌中依赖sigma54进行转录的基因大多与胁迫和代谢信号相关,且无法自发起始转录,需要其激活蛋白质bEBP参与才能启动转录起始。细胞内的小分子代谢物质可结合在bEBP的调节结构域以增强其活性。例如:2-OG和NO可结合GAF结构域,黄素等分子可结合PAS结构域。调节蛋白质也可通过结合bEBP调控其活性。例如:NifL与NifA结合后,会抑制NifA的活性;HrpS与HrpR结合,可共同发挥活性。此外,磷酸化修饰也能改变bEBP的活性状态,例如:磷酸化可激活NtrC,启动转录起始[42]。未来,结合小分子代谢物、配体蛋白质或者磷酸化修饰的bEBP三维结构的解析,将极大推动我们对bEBP感受并传递信号分子机制的认识。
在sigma54依赖性转录起始方面,尽管科学家们利用冷冻电镜技术已经解析了一系列sigma54依赖的基因转录复合物的结构,在sigma54如何抑制转录起始、DNA如何进入RNAP裂隙、bEBP如何与sigma54相互作用,以及bEBP作用的转录进程等方面给出了更为清晰的答案[22, 31]。但关于sigma54依赖的转录起始机制仍然存在许多未解之谜。目前报道的sigma54依赖性转录起始相关的各种结构,都是通过组装异源的sigma54与RNAP、启动子DNA所得,这些结构是否能够完全代表来自于同一物种的真正的转录起始过程仍有待确认。此外,当前获得的转录复合物电镜结构的分辨率普遍较低,一些关键的相互作用信息尚无法判断,阻碍了对转录起始机制的深入理解。另一个重要问题是,目前已经解析出的在ADP·AlFx存在下,bEBP结合的转录复合物结构仅能提供有限信息。在未来,来自同一物种的转录起始复合物的结构解析将描绘出更为准确的转录起始过程。而结合不同状态核苷酸的bEBP-RNA聚合酶全酶复合物的结构解析,将有助于理解bEBP水解ATP过程中的构象变化,及其对转录复合物结构的重塑作用。
