高通量测序技术在细菌耐药中的应用
2022-09-07陈慧娟刘琪琦
陈慧娟, 刘琪琦
(1)北京工业大学环境与生命学部生命科学与化学学院生物医学工程系, 北京 100124;2)军事科学院军事医学研究院辐射医学研究所, 北京 100850)
在全球范围内,细菌耐药(antimicrobial resistance, AMR)已成为威胁公共健康的重要因素之一[1]。世界卫生组织2020年全球抗生素耐药性和使用监测系统(Global Antimicrobial Resistance and Use Surveillance System, GLASS)报告显示:细菌耐药已在多数国家出现,并且在中低收入国家更为严重(https://apps.who.int/iris/bitstream/handle/10665/332081/9789240005587-eng.pdf?ua=1)。抗生素的广泛使用、误用及滥用是导致各种耐药菌甚至“超级耐药菌”出现的主要因素。目前几乎所有类型的抗生素,包括一些应用于多重耐药菌治疗的广谱抗生素均已出现了耐药性[2, 3]。细菌耐药的检测、监控及预测对于抗生素的合理使用、耐药细菌感染的控制及耐药细菌的监控至关重要。药敏试验(antimicrobial susceptibility testing, AST)是目前实验室较常用的检测细菌耐药方法。药敏试验能够提供细菌耐药的表型信息,但不能直接检测临床样本,需要培养才能鉴定出耐药菌株,周期较长,且针对培养受限的菌株无法进行检测[4]。细菌耐药通常由耐药基因编码产生,随着各种耐药基因的发现,基因芯片、微流体芯片、飞行时间质谱、聚合酶链反应等各种基于耐药基因检测细菌耐药的检测技术迅速发展。此类技术只能检测已知耐药基因,检测基因数目非常有限,对未知耐药基因无法检测,无法发现新的细菌耐药基因、耐药机制,并且无法进行细菌耐药监测和追踪。高通量测序技术可一次性并行的对几千条甚至数十亿条核酸分子进行序列测定。不依赖于培养,高通量测序可以快速、准确无差别的对培养菌株,以及不可培养或培养受限细菌中的耐药基因进行快速鉴定,可作为药敏试验或其他检测方法进行细菌耐药检测的重要补充[5]。高通量测序还可用于细菌耐药基因组研究,可发现新的耐药基因和耐药机制,并可同时监测、追踪耐药基因及耐药菌全球分布、传播、流行的机制和特征。近年来,随着高通量测序技术的发展、测序速度的提升和测序成本的下降,高通量测序技术在细菌耐药领域的应用越来越广泛。本文就高通量测序技术、测序平台、数据分析方法等在细菌耐药领域的应用进展,以及细菌耐药数据库的开发更新进展进行综述。
1 高通量测序技术在细菌耐药领域的应用
高通量测序技术在细菌耐药领域应用范围包括全基因组测序(whole-genome sequencing, WGS),目标区域测序(target next generation sequencing, tNGS)和宏基因组测序(metagenomics next generation sequencing, mNGS)。NGS获得细菌基因序列信息的主要流程包括:(1)核酸提取和纯化:从纯培养物(WGS)或原始样本(tNGS、mNGS) 中提取微生物DNA。(2)片段化基因组DNA:通过机械或化学方法等(WGS、mNGS)将基因组DNA随机片段化,或通过特异性引物扩增、探针杂交捕获等方式(tNGS) 获得目的片段化DNA。(3)文库构建:在片段化的DNA加上接头和唯一性标签序列(测序后区分待测的DNA片段),随后进行克隆化扩增或直接进行片段筛选后得到均一化的可测序的文库。(4)测序:边合成延伸边测序,通过捕捉合成链末端的荧光信号或化学信号来获得DNA序列信息。(5) 生物信息学分析及注释:通过不同生物信息学分析流程得到耐药基因信息并进行注释。下面将简要介绍WGS、tNGS及mNGS三种检测流程在细菌耐药领域的应用情况。
1.1 全基因测序技术
全基因组测序即WGS,是指获得一个物种全基因组序列的过程。在细菌耐药领域,从纯培养物(单一菌落)开始,WGS可快速准确均一的得到1个菌株的所有耐药基因包括耐药质粒的信息。采用WGS检测耐药基因和耐药机制可预测细菌耐药表型情况。多项研究表明,基于WGS的细菌耐药检测结果与细菌表型耐药检测结果间存在较高的一致性。虽然检测性能因所检测的细菌不同略有差异,总体检测灵敏度(真阳性率,基因预测耐药结果与表型耐药结果百分比)>87%,总体检测特异性(基因预测敏感结果与表型敏感结果百分比)>98%[9-12]。结核病综合耐药性预测国际联合会(Comprehensive Resistance Prediction for Tuberculosis: an International Consortium, CRyPTIC)一项基于10209个结核培养菌株研究显示:WGS测序结果对异烟肼、利福平、乙胺丁醇及吡嗪酰胺等4个一线抗结核药物的检测灵敏度分别为97.1%、97.5%、94.6%和91.3%;检测特异性分别为99.0%、98.8%、93.6%和96.8%[13]。结合深度学习WGS还可预测抗生素的最小抑菌浓度,对肺炎克雷伯菌平均准确度为92%[17],而对非伤寒沙门菌平均准确度可达95%[18]。除以上用途外,WGS还可应用于耐药性细菌,例如产OXA-48和CTX-M-15的高毒力肺炎克雷伯菌[14]、耐万古霉素肠球菌[15]等爆发流行的确认和监控,及新的耐药基因或机制的发现,例如抗粘菌素新的耐药基因mcr-7.1[16]。WGS检测的样本类型主要为纯培养物[6],其检测周期较长。另外检测样本若存在其他细菌,尤其是检测因携带耐药质粒或其他移动因子所导致的耐药时,会严重影响结果准确。虽然目前已有一些采用WGS直接检测非培养样本中肺炎分支杆菌耐药基因的报道[7, 8],但针对难培养或无法培养的细菌,WGS依旧不太适用。
1.2 目标区域测序技术
目标区域测序即tNGS,是指仅对所感兴趣的目标区域或基因进行测序的技术。一般在测序前会对目标区域或基因进行选择性富集,目前常用的富集方法有:PCR扩增(扩增子测序)、探针杂交捕获和CRISPR-Cas9技术[19, 20]。tNGS可直接检测原始样本,检测灵敏度高,检测周期短。因仅检测目标区域或基因,所需测序数据量少,测序成本低。同时tNGS通过对目标区域或基因选择性富集,解决了mNGS所面临的从大量背景核酸序列(人类或其他物种基因组)中“大海捞针”般的寻找少量目的片段的困境[21]。近年来已有多篇关于tNGS在细菌耐药领域的应用报道。有研究报道,采用Thermo Ion Ampliseq扩增子富集技术检测结核分枝杆菌9个抗生素耐药基因,对利福平、异烟肼、乙胺丁醇及吡嗪酰胺检测特异性分别为96%、88%、97%、100%,检测灵敏度分别为100%、88%、60%和67%[23]。Urbaniak等采用基于Thermo Ion AmpliSeq技术的定制panel可同时检测多种微生物中518个耐药基因的情况[22]。而Lanza等基于SeqCapEZ技术所开发ResCap杂交捕获流程,通过采用设计的探针可同时检测88.13 Mb (Million bases)目的区域内47806个抗生素耐药基因和30794个杀菌剂和金属抗性基因的状况[24]。另有研究报道,采用37826个探针组合可同时检测2021个抗生素耐药基因,同时采用人类肠道菌群样本进行验证该panel具有较好的敏感性[25]。虽然tNGS还存在诸如均一性较差、只能检测已知耐药基因等诸多局限性,但快速、成本低和检测灵敏度高等特点,为其在临床细菌耐药检测领域的应用提供了较大的可能。
1.3 宏基因组测序技术
宏基因组测序技术即mNGS,可直接同步无偏差的检测原始样本中整个微生物群落(例如细菌、病毒、真菌等)、耐药基因、毒力因子甚至与特定疾病阶段相关的宿主生物标记物[26]。在临床检测领域,mNGS已成功应用于多个组织部位临床感染微生物的诊断和鉴定[27-29],同时也成功的鉴定了一些新发、少见和非典型的临床感染微生物[30]。在临床细菌耐药检测中,Yan等研究发现:基于CosmosID生物信息分析平台,mNGS对葡萄球菌中mecA-介导的耐甲氧西林的检测灵敏度为77.4%,检测特异性为100%[31]。除在临床细菌耐药检测中的应用外,mNGS还可应用于环境例如医院污水、自然水源和土壤等[32]或食源性动植物例如猪肉、鸡肉等中细菌耐药基因丰度的检测和监控,进而可尽早采取措施防止耐药基因在环境微生物中富集和传播。近期有研究采用mNGS检测潮白河(北京)河水发现:在潮白河水中共检测到22类442种耐药基因,平均丰度为1.1×10-1-8.1×10-1拷贝ARG/16S-rRNA基因,远远高于深海和极地等无人类活动/活动较少的区域[33]。同WGS,mNGS还可以发现新的耐药基因和其作用机制。Gloria等从土壤样本中发现了11个新的耐药基因(3个氨苄青霉素、2个大庆霉素、2个氯霉素和4个甲氧苄氨嘧啶抗性基因)[34]。Forsberg等采用mNGS发现9个与降低四环素降解酶活性相关的耐药基因,进而发现了四环素耐药的机制[35]。虽然近一、两年来mNGS的检测流程和数据分析流程在逐步优化,但依旧存在诸多例如检测灵敏度低、所需数据量大测序成本高、无标准化“湿实验”和数据分析流程等需要近一步完善和优化之处。
2 测序平台
目前在细菌耐药领域,主要使用的测序平台为基于短片段测序技术的二代测序平台。基于长片段测序技术的三代测序平台近两年也开始逐步被应用。二代测序平台主要有:Illumina系列、Ion Torrent系列和BGI系列。Illumina 测序平台是目前应用最为广泛的二代测序平台。该测序平台采用可逆性测序末端边合成边测序:在碱基延伸过程中,每个循环反应只延伸一个正确互补的带荧光碱基,通过收集四种不同的荧光信号确认碱基种类。Illumina测序平台的测序试剂均采用卡盒式结构,人工操作较少,自动化程度较高。该测序平台测序时间较长,MiSeq双端300bp测序一次测序需要超过60 h。另外该测序平台存在标签跳跃(index hopping)的问题,在其高通量测序机型如HiSeq3000、4000、NovaSeq等该问题更为严重[36]。对于细菌耐药检测而言,标签跳跃会导致样本间的交叉污染,尤其是丰度较高的样本对低丰度样本的污染,在实际应用中需特殊注意。
Ion Torrent基于半导体测序技术,不需要光学系统,通过测序过程中DNA合成时所释放H+导致的测序环境中pH值的变化进行碱基的识别。无卡盒试剂,文库在测序前需进行乳液PCR、纯化等,操作比较繁琐,对操作人员的依赖性较强。BGI采用DNA纳米球测序技术:利用滚环式复制让待测的DNA扩增形成DNA纳米球,通过组合探针锚定连接法进行边合成边测序。在每轮测序前先加入与接头匹配的锚序列,然后加入大量只有一个荧光标记碱基的探针,通过检测与待测序列结合的标记探针的荧光信号得到序列信息。滚环式复制中所有扩增模板均为最初的插入片段,不会产生PCR错误的累积。另在测序过程每一碱基的判读不依赖上一碱基,测序错误更加随机,相较其他二代测序平台BGI测序平台的碱基准确率更高。
Oxford Nanopore Technologies纳米孔(Nanopore sequencing)和Pacific Biosciences (PacBio)的SMRT (Single Molecule, Real-Time) 单分子测序技术通常被称为三代测序技术。三代测序技术的特点为单分子测序技术,测序过程中无需PCR扩增。目前Nanopore 和PacBio SMRT 已逐渐成为三代测序的代名词。Nanopore采用电泳技术,通过电泳使每个单分子逐一通过纳米孔,每个纳米孔仅允许单个核苷酸通过,通过ATGC带电性质差异的检测碱基的类别[37]。纳米孔可对穿过的任意长度的DNA片段进行测序,无长度限制,可以得到Mb级别长度的序列[38, 39]。SMRT技术边合成边测序,荧光标记的脱氧核苷酸与待测DNA链形成化学键时,荧光会因荧光集团被切除而消失,通过检测记录荧光强度的变化可获得相应的碱基序列。Pacbio对碱基判断会出现随机误差,误差率大概为12.5%;另Pacbio聚合酶可以1 s钟合成3个碱基,测序速度较快。光照可能造成待测DNA的缺口和聚合酶的变性,会限制测序读长。相较于二代测序,三代测序尚存在检测通量较低、测序错误率较高等诸多不完善之处。
三代测序所具备的长读长、测序时间短等优点为三代测序未来在细菌耐药领域的广泛应用提供了可能。长读长可大大降低测序序列的拼接难度,对基因组序列较小细菌甚至可以直接完成全基因组测序。相对于二代测序从样本到报告一般需花费5~10 d的漫长报告周期,三代测序可在数分或数小时内实时获得相应测序数据。有研究报道采用Nanopore测序可在4 h内完成肺炎链球菌的耐药基因从样本到报告的检测,检测灵敏度为75%,检测特异性为100%[40]。
3 基于高通量测序的细菌耐药检测数据分析方法
高通量测序所获得的原始reads数据(原始的碱基序列信息),需要通过生物信息学软件、流程进行分析以获得特定耐药基因的信息。首先,需采用诸如FastQC,SolexaQA等程序进行测序序列质量控制和过滤(去除低质量、重复序列去除);过滤后的质量合格的序列可进行后续分析。基于mNGS流程所得到的序列,需先将测序序列与人类基因组进行比对,去除人源基因组序列后再进行后续分析。目前耐药基因分析主要有序列组装和直接序列分析两种分析流程。序列组装是将检测出的微生物短reads拼接为完整的基因组序列,再与参考数据库比对得到耐药基因信息的过程。序列组装目前多采用基于De Bruijn graph (DBG) 策略的算法,例如SPAdes[61]、Velvet[62]等。该算法首先将reads分割成一系列连续的短序列(称为k-mers),然后利用k-mers间的重叠构建DBG图,从而得到全基因组序列。该算法对内存消耗少,但会受测序过程中产生的碱基错误的影响[63]。为改善碱基错误对序列组装正确性的影响,一些针对单一细菌基因组中重复序列或质粒序列拼接的算法,例如SAUTE[68]等和针对mNGS测序序列组装的算法,例如MetaVelvet[64]、 SPAdes[65]等,以及一些提高碱基判读准确度的方法[66]陆续被开发和广泛应用。序列组装完成后,通过采用BLAST、USEARCH等工具将组装的全基因组序列与细菌耐药基因参考数据库比对,可获得耐药基因的注释。
直接序列分析是将质量合格序列直接与参考数据库比对,并得到耐药基因信息的过程。主要有两种方式:一种通过双序列比对工具,例如Bowtie2 (如SRST[67]) 、BWA等将过滤后质量合格的序列直接与细菌耐药基因参考数据库比对。另一种为先将reads分割成k-mers (如KmerResistance[69]) 后与参考数据库比对。此类分析流程可在因测序序列中存在实验室或宿主污染所导致的背景噪音,或测序序列不足等所导致的序列组装流程无法进行的情况下进行耐药基因注释。该类分析流程无法对单核苷酸多态性位点(single nucleotide polymorphisms, SNPs)引起的耐药进行注释。无需组装后对比,占用内存较少且速度快,近年来此类分析流程在需要及时预测细菌耐药基因的场景中应用较为广泛。目前正在使用的多种数据分析方法各有千秋,关于如何选择合适的分析方法并无统一的规则。建议根据所检测的样本来源、微生物生物属性、文库构建方法、测序平台、测序深度等多种因素综合考量选择最适合的方法。
4 细菌耐药基因数据库
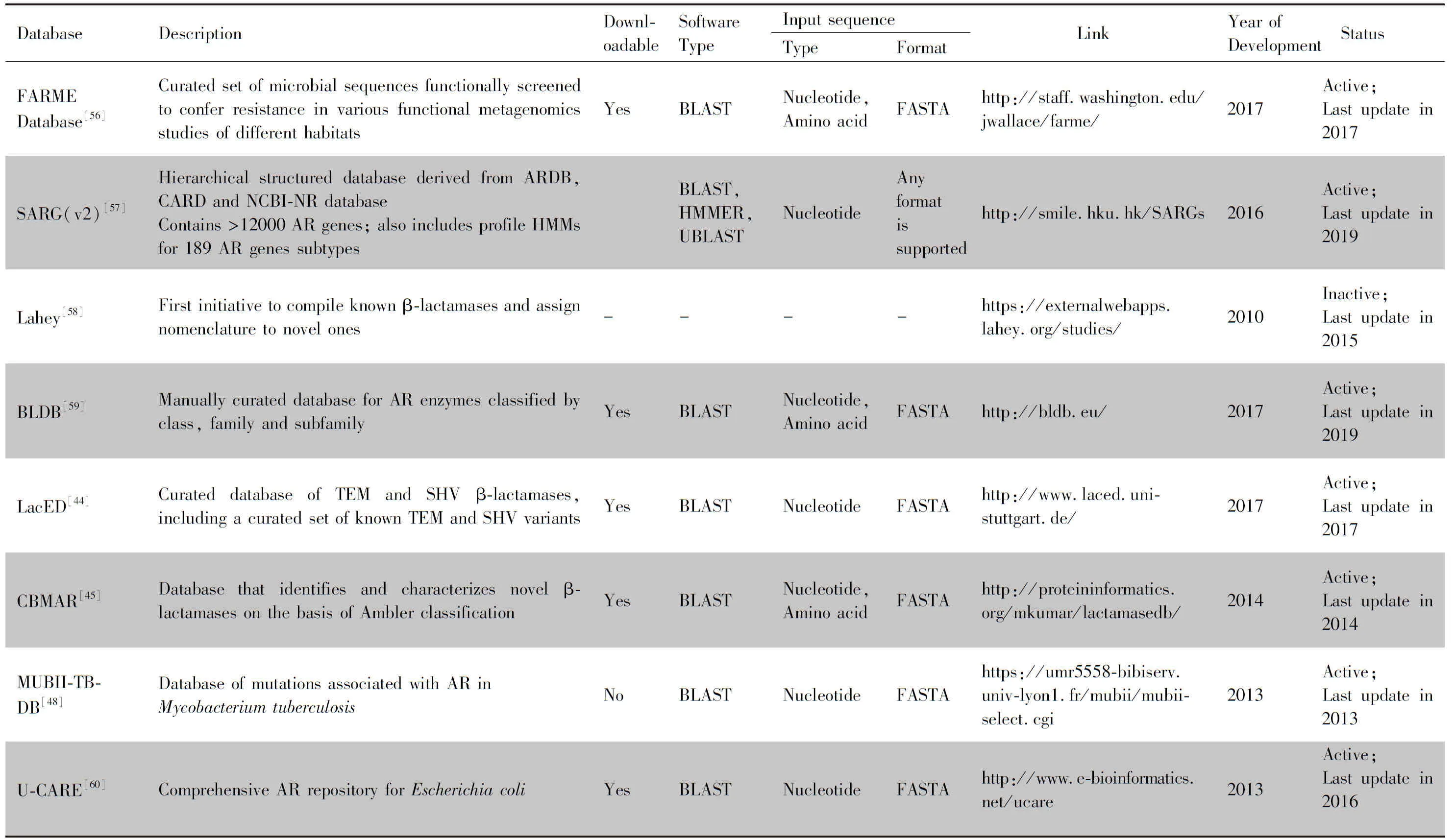
细菌耐药基因预测细菌耐药表型的准确性在很大程度上依赖于成熟的专业耐药基因数据库。目前细菌耐药基因数据库主要有通用型数据库和特定型数据库两种。通用型数据库包含多种抗生素耐药基因及耐药机制的信息。CARD数据库(Comprehensive Antibiotic Resistance Database)为目前使用范围最广的通用型数据库 (Table 3)。CARD数据库目前涵括4833个ARO (Antibiotic Resistance Ontology)、3339个参考序列和3385个AMR检测模型。CRAD数据支持网上耐药基因预测分析,有BLAST和RGI(Resistance Gene Identifier)两种模式:BLAST基于NCBI中BLAST软件,将所得序列(核酸或蛋白序列)与参考序列比对后进行相应注释;RGI是CARD数据库团队自主研发的基于蛋白质序列预测耐药基因序列的软件,目前仅能分析蛋白质序列[41]。特定型数据库包括针对特定耐药基因和针对特异性菌种两种(Table3)。菌种特异性耐药基因数据库对于了解特定菌种的耐药性机制,尤其是对某些抗生素的内源性耐药具有重要的作用[47]。另外,此类数据库能迅速、有效的收集新的耐药基因和耐药机制信息。

Table 1 Advantages and disadvantages of high throughput sequencing applications in antimicrobial resistance detection
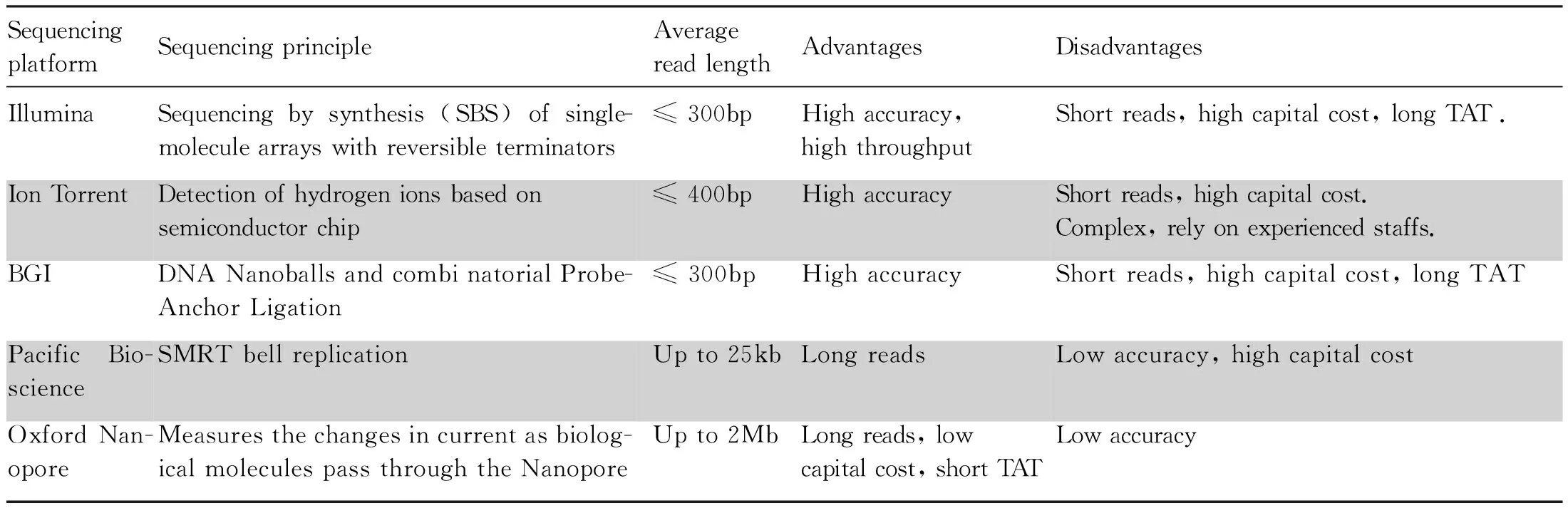
Table 2 Advantages and disadvantages of 5 major sequencing platforms
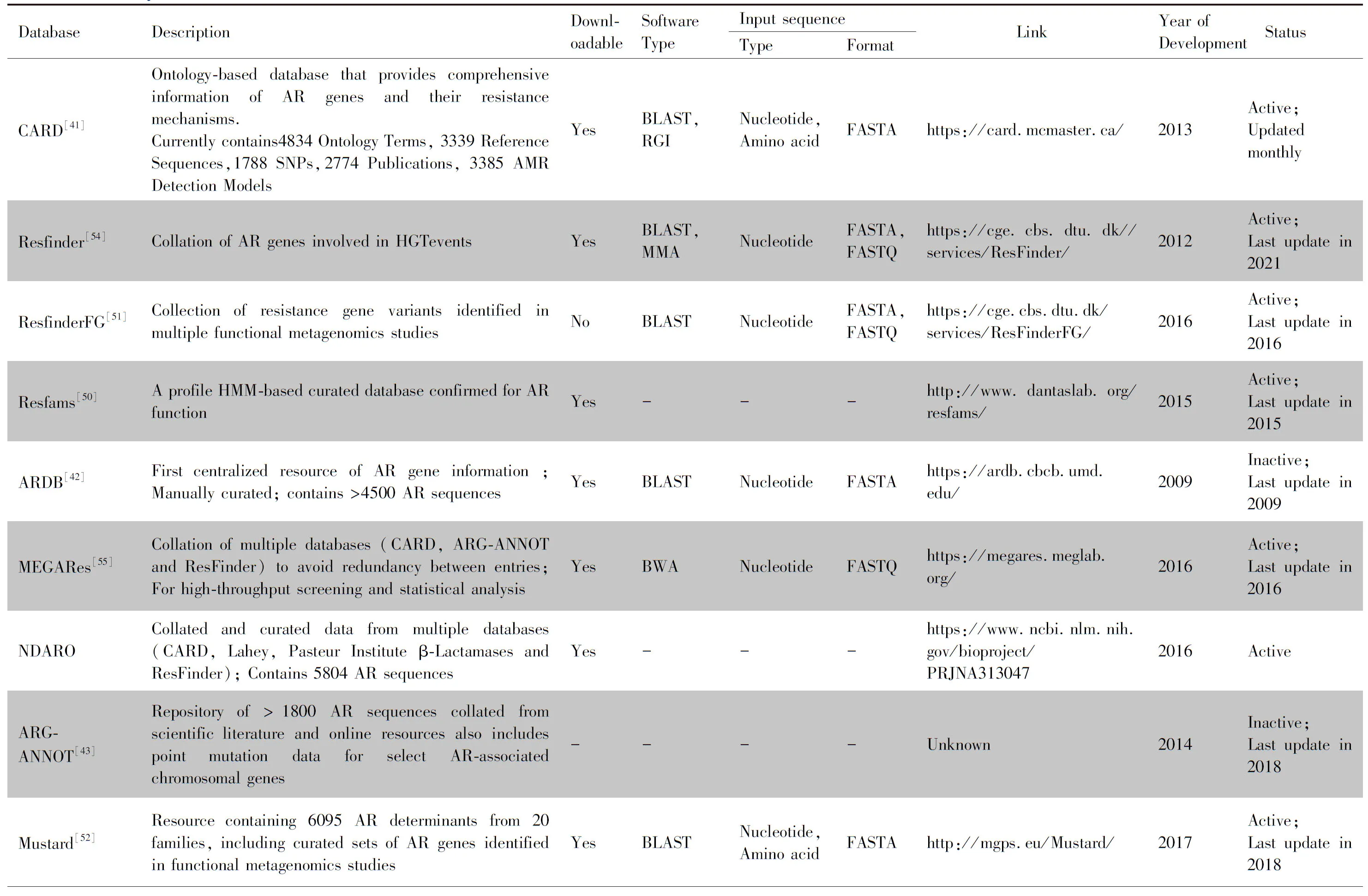
Table 3 Summary of antimicrobial resistance reference databases
Continued Table 3
通用型数据库和特定型数据库所包含的多为人类致病菌及易培养菌的耐药基因信息。而此类数据库无法对较难培养的或无法培养的菌种的耐药基因进行鉴定[49],而隐马尔可夫模型(hidden Markov model, HMM)数据库可对此类耐药基因进行鉴定。其流程为:将待鉴定序列与HMM数据库的参考序列进行多序列比对,能找到序列不一致但具有相似功能的参考序列,通过参考序列的耐药性可对待鉴定序列的耐药性进行预测。HMM类数据库包括Resfams (通过与CARD、LacED等数据库中耐药基因蛋白质参考序列进行多序列比对)[50]、ResfinderFG[51](与Resfinder数据库进行比对)及Mustard[52](结合蛋白质3D结构预测耐药基因)等。虽然近年来各种耐药基因数据库在逐步完善,但依旧仍存在诸多弊端:1) 除极少数数据库外,大多数数据库无持续有效的收集更新数据的机制,导致数据库更新不及时或停止更新;2)命名规则混乱,目前大多数耐药基因既可根据核苷酸亦可通过蛋白来命名,同一耐药基因在不同数据库命名不同,例如二氢叶酸还原酶在一些数据库是dhfr而在另一些数据库为dfrA[53],会引起使用者的困惑;3)目前几乎所有的数据库仅关注编码蛋白质的耐药基因的特性,而针对诸如rRNA 的原发突变、调控因子等其他可能导致的耐药机制关注较少。
5 问题与展望
随着测序技术的发展,各种检测、分析方法及耐药基因数据库的逐步开发和完善,高通量测序技术已广泛应用于细菌耐药领域。因细菌耐药的复杂性,该技术在此领域的应用尚存在诸如:检测成本相对其他基于核酸检测方法高、无标准化的检测和分析流程、自动化程度低、对实验操作人员和数据分析人员要求高、参考数据库的准确度及范围需要进一步提升等诸多需要改进之处。高通量测序技术在细菌耐药领域的应用能够使人类更快、更全面、更准确的检测细菌耐药基因,可指导临床合理用药,在实现感染患者治疗个体化、精准化的同时可减少抗生素的误用和滥用;另细菌耐药是一个系统性、高度复杂的体系,目前在人类、动物及环境中所发现的耐药基因、耐药机制仅仅为冰山一角。高通量测序技术的应用可加速新的耐药基因、耐药机制的发现,预测、监控耐药基因传播流行趋势,使人类能够更深入、更全面的了解细菌耐药的机制和流行趋势,不但有助于遏制新的耐药微生物出现和传播,而且可以使人类尽早的采取预防措施,对于全球性的耐药微生物防控具有重要的意义。
