学科交叉与知识扩散相关性研究
——以基因工程领域为例
2022-09-01朱世琴范丹丹苟文静
朱世琴 范丹丹 苟文静
(华东理工大学科技信息研究所,上海 200237)
科学技术的每一个学科都会或多或少、或远或近地与其他任何一个学科相关联[1]。学科间通过引文知识扩散,并不断借鉴其他学科有益的理论和方法,逐渐交叉渗透与融合,从而达到知识创新、科学发展的目的。知识扩散反映了知识的传递、继承、应用与创新[2]。探讨论文的知识扩散对科研评估、科学预测以及准确衡量学科交叉产生的科研成果影响力具有潜在价值。十四五规划明确提出:为推动科技自立自强,需要加强基础研究、注重原始创新,优化学科布局和研发布局,推进学科交叉融合。可见,学科交叉研究受到政府和学术界的高度关注。目前,国内外对学科交叉与知识产出的关系已开展了广泛的研究,主要关注学科交叉与论文学术影响力的关系,但对于论文中知识扩散的广度、速度及强度鲜有探究,没有关注学科交叉程度与知识扩散的关系。本研究旨在计算论文的学科交叉度,探索学科交叉的实际效果和影响,从而判断学科交叉程度与知识扩散的关系。
1 国内外研究现状
1.1 学科交叉研究现状
学科交叉反映了不同学科之间的知识流动和渗透,在科学研究中发挥着关键作用,并产生了许多开创性的科学发现。但学科交叉程度对论文价值的影响还有待思考。许多学者对学科交叉的意义进行了深入研究,尝试从不同角度探索学科交叉成果的实际效果和影响力。
1)学科交叉与被引频次的关系研究
Stopar K等[3]从主题和参考文献两个角度研究林业学科论文,研究发现论文学科交叉程度越高,被引频次越高,学科交叉研究对林业论文的学术影响力具有促进作用。Alfredo Y Y等[4]从参考文献多样性、均衡性、差异性角度出发,使用Tobit回归模型分析学科交叉程度与论文影响力的关系,研究发现学科多样性与影响力呈正相关关系,多样性处于中等程度的论文影响力最强。Levitt J M等[5]对图书情报领域前0.1%的高被引论文进行分析,发现多数高被引论文至少涉及两个学科,即学科交叉研究相比单一的学科研究能产生更高的学术影响,推广学科交叉研究有利于提高图书情报领域的论文质量。张琳等[6]引入PLOS官方平台的使用数据,结合引用数据对学科交叉研究成果进行综合评价,发现学科交叉水平与论文影响力有一定正相关关系,学科交叉程度对使用数据和引用数据之间的相关关系有显著影响。Chen S等[7]从不同维度探讨学科交叉程度与高被引论文影响力的关系,发现无论是否考虑控制变量,多样性和差异性明显促进论文的影响力,而平衡性不利于论文的影响力。
虽然学科交叉研究一直被各界广泛关注,产生了许多高影响力的学术成果,但学科交叉是否产生更高的实际效果和影响力仍存在分歧。Levitt J M等[8]将自然科学和医学类期刊作为研究对象,研究发现多学科期刊的引文数量远少于单一学科期刊。Zhang L等[9]对7种不同学科的TOP期刊研究发现,学科交叉程度和引文数量的关系因学科而异,数学学科交叉程度越高,引文数量越多,与之相反,生物信息学的引文数量随交叉程度提高而降低。
2)学科交叉与其他指标的关系研究
除了对学科交叉程度与影响力的关系进行探讨外,学者们还从不同层面对学科交叉成果的价值进行测度。Bromham L等[10]对学科交叉程度与基金资助的关系进行探究,发现学科交叉程度越高,基金申请成功率越低。王菲菲等[11]使用Tobit回归模型对学科交叉程度与论文价值成长老化指标进行分析,发现学科交叉度与论文成长老化指标呈倒U型关系。开滨等[12]采集了6个SCI学科中的睡美人文献,对其学科交叉特征的相关性、独特性和差异性进行分析,发现论文的睡美人特征越强,学科交叉程度越高。徐璐等[13]以图书情报领域18种核心期刊为研究样本,探讨了期刊的跨学科引用对知识输出的影响,发现期刊跨学科引用有助于扩大知识输出的学科范围,增强了该学科对其他学科的影响力,但降低了跨学科知识输出的速度。
1.2 知识扩散研究现状
知识扩散作为知识生产和传播的重要组成部分,在知识创新过程中发挥着重要作用[14]。关于知识扩散的相关研究,国内学者已经取得了一些研究成果。
现有学者多是将知识扩散作为评价指标,如赵蓉英等[15]通过作者知识扩散广度、深度等指标,评价作者知识扩散的贡献程度。Ding J等[14]使用知识扩散广度、强度、速度和主题探讨图书情报学的知识扩散发展趋势及其对其他学科的影响。魏绪秋等[2]以图书情报学领域的图书为研究对象,发现图书知识扩散深度、扩散速度、扩散指数和扩散强度4个指标与图书被引频次相关性极强。但缺乏对影响知识扩散的因素的研究,仅张雪等[16]以中国医药领域为研究对象,分析专利的知识吸收对知识扩散广度、强度、速度的影响,发现不同的知识吸收指标对知识扩散广度、强度、速度产生不同影响。
纵观上述研究可知:现有研究主要聚焦在学科、期刊及作者的知识扩散特征上,忽视了将学术论文这一学科交叉成果作为研究对象,论文知识扩散能力代表了知识传播与知识流动能力,知识在不同学科之间流动,有助于产生新的知识点,了解论文在不同学科的知识扩散可以更好地提高知识价值。目前对学科交叉成果的科学认定及价值评估的角度较为单一,可以尝试从论文知识扩散视角进行佐证,了解学科交叉与知识扩散的相关性可以为评估学科交叉成果提供参考。但是现有研究尚未提及学科交叉与知识扩散的关系研究。
据此,本文提出以下研究问题:学术论文的学科交叉程度与知识扩散是否有相关关系?学术论文的学科交叉对知识扩散会产生怎样的影响?本研究将尝试以基因工程领域论文为研究对象,通过参考文献从学科交叉综合指标和3个单一维度指标测度论文的学科交叉程度;通过引证文献从论文知识扩散广度、知识扩散强度、知识扩散速度视角衡量论文的知识扩散程度;并采用Tobit回归模型对论文学科交叉和知识扩散之间的关系进行探析。
2 测度指标及方法
2.1 知识扩散指标
为探析论文知识扩散特征,本研究结合Liu Y等[17]在2010年提出的学科知识扩散广度、强度以及速度,引入论文知识扩散广度、强度以及速度3个测度指标。
1)论文知识扩散广度(KDB)。引证学科数量从学科层面量化了学术论文影响力广度,对于一篇论文,引证该篇论文所属的JCR学科(去重)数量就是该篇论文知识扩散广度。学术论文的引证文献所属学科越多,学术论文影响广度越大。
2)知识扩散强度(KDD)。对于一篇论文,知识扩散强度等于该论文某一学科引证该论文最高的次数。引证某一学科论文数量越多,说明该论文的方法技术、理论知识对该学科影响力越强。
3)知识扩散速度(KDS)。一篇论文发表后,引用该论文的(去重)学科数量与统计起点到终点年数的比值,可以用来量化论文知识的整体扩散速度。
2.2 学科交叉指标
陈仕吉[18]指出,导致学科交叉研究与影响力关系不同的一个原因在于学者们在测度学科交叉程度时,并未使用统一的测度指标,只是涉及学科多样性的一到两个维度。为排除学科交叉测度指标不同所带来的影响,本文根据Stirling A[19]提出的学科多样性3个测度属性,即多样性、均衡性、差异性,选择V、B、D作为学科交叉单一测度指标,Rao-Stirling指标作为学科交叉综合测度指标,在文中简称为R。
1)V指标。指一篇论文参考文献所属JCR学科的类别,被用作测度学科交叉多样性。若参考文献所属学科类别越多,则学科交叉多样性越强。
V=n
(1)
2)B指标。使用香农熵表示一篇论文引文学科均衡性,如果论文每个学科参考文献的数量相同,说明该论文引文学科分布均衡,若引文学科分布越均衡,则学科交叉程度越高。
(2)
3)D指标。通过计算学科之间不同距离分析学科交叉程度,若一篇论文引文所属学科差异性较大,则该论文学科交叉程度更高。
(3)
4)R指标。从多样性、均衡性及差异性综合测度论文学科交叉程度。若一篇论文参考文献所属学科类别非常相似,则该论文学科交叉程度较低,反之,则越高。
R=∑i,j(1-Si,j)pipj
(4)
其中n为参考文献所属学科类别,pi表示学科i的被引频次占所有学科总被引频次的比例,i不等于j,Si,j表示学科i和学科j的相似性程度矩阵中学科i和学科j之间的相似性。
2.3 研究方法
由于本研究选取的因变量在正值上连续分布,存在大量0值,且呈现离散分布,而传统的最小二乘法回归模型忽略了0值的重要意义,故不适用于本研究。离散分布数据通常采用Tobit、泊松分布、负二项分布以及零膨胀分布等模型进行拟合。在选择具体模型时,本研究结合样本数据,对模型一一进行检验,最终得到适合本研究所有样本数据集的最佳模型为Tobit模型。Tobit模型适用于受限因变量的情况,采用最大似然法对参数进行估计,能够较好地规避参数估计有偏的问题[20]。因此,本研究选择Tobit模型对论文学科交叉和知识扩散指标进行回归分析。其公式具体如下:
(5)
Y*=β0+βiXi+ε
(6)
式中,Y为因变量,Y*为潜在变量,Xi为自变量,βi为对应的相关系数,i为第i个变量,ε为随机误差项。
3 基于基因工程领域的实证分析
3.1 样本采集
本研究选取Web of Science核心合集的SCIE数据库作为数据源,以基因工程领域文献为研究样本,限定在标题或者作者关键词字段检索,数据检索时间为2021年1月。检索式为TI=(“gene*engineering”or“DNA engineering”or“gene* manipulat*”or“DNA manipulat*”or“gene*recombinat*”or“transgen*”or“gene*clon*”or“molecular clon*”)or AK=(“gene*engineering”or“DNA engineering”or“gene*manipulat*”or“DNA manipulat*”or“gene*recombinat*”or“transgen*”or“gene*clon*”or“molecular clon*”)。文献类型限定为Article,由于论文存在“延迟引用”现象,绝大部分论文在发表3年后才被引用[21],为保证一定的引用时间窗口,所以时间跨度限定为2000—2018年,最终命中50 142条文献。选择“全记录与引用的参考文献”格式,导出论文的题录信息,同时利用Python爬取每篇论文的引证文献。
3.2 数据处理
1)构建期刊全称—缩写1—缩写2—学科类别对照表。WoS学科分类体系以期刊为分类对象,更容易获取参考文献的学科数据,并且WoS学科包含254个学科类别,划分粒度细致,涵盖学科最全,故研究选择WoS学科分类方法[22]。为计算单篇论文的学科交叉指标和知识扩散指标,研究需要根据论文所属期刊的学科类别将每篇参考文献和引证文献映射到特定的学科类目。首先从JCR下载期刊学科对应信息,对照ESI期刊列表构建期刊全称—缩写1—缩写2—学科对照表;其次发现部分期刊未被收录在JCR期刊中,故对丢失学科信息的期刊进行手动补足;最后成功将12 268本期刊匹配到学科。
2)数据清洗。由于题录信息中包含参考文献在内的多个字段,故需要对其进行清理。首先删除参考文献为0以及题录信息不全的论文,最终共得到49 997篇论文;其次对导出的论文提取学科类别、作者、机构、参考文献等字段,为了避免其他因素对研究结果产生影响,本研究设置4个控制变量,即合著人数(AU)、合著机构数量(N_inst)、国际合作(I_collab)和国内合作(N_collab)。如果合著机构来自不同的国家,则I_collab取1;如果至少有两个不同的机构来自同一个国家,则N_collab取1;否则I_collab和N_collab均取0。这些数据通过Excel进行手动提取得到。
3)学科映射。通过论文的参考文献可以有效获取各个学科的知识和信息的输入情况,了解论文知识的学科来源,测度论文的学科交叉程度[23]。利用Python提取每篇论文的参考文献的期刊刊名,通过构建的期刊—学科对照表,对每个刊名进行一一匹配。如果参考文献和引证文献涉及的期刊所属学科有两个以上,为保证数据准确性将其全部计入统计结果。根据上述步骤,获得参考文献共2 045 283篇,期刊未匹配到学科的参考文献共212 247篇,匹配率达89.6%。通过Python共爬取1 510 875条引证文献,期刊未匹配到学科的引证文献共62 735篇,匹配率达95.8%。
4)指标计算。利用Python计算每篇论文的学科交叉程度和知识扩散程度。由于自变量中学科交叉多样性指标(V)的取值范围在[1,50],为了让学科交叉指标更加统一,同时为消除奇异样本带来的不良影响,因此采用最大—最小值标准化方法,将其区间映射在[0,1]。而每篇论文的知识扩散广度、知识扩散强度、知识扩散速度服从偏斜分布,为了减弱这些变量的偏斜分布,使得数据更加平稳,本研究对这些因变量进行加1取对数处理[4]。为保证计算结果的科学性与准确性,本研究将引证文献和参考文献的期刊学科匹配率不足50%的论文删除,使用Vlookup函数对保留的论文进行匹配,最终共得到47 873篇论文。采用Stata 15.1统计软件对数据进行分析。
4 结果与分析
4.1 描述性统计
对观测样本变量进行描述性统计,结果如表1所示。可以看出,多数变量标准差较大,呈离散分布,且部分变量存在0值,适用于Tobit模型。
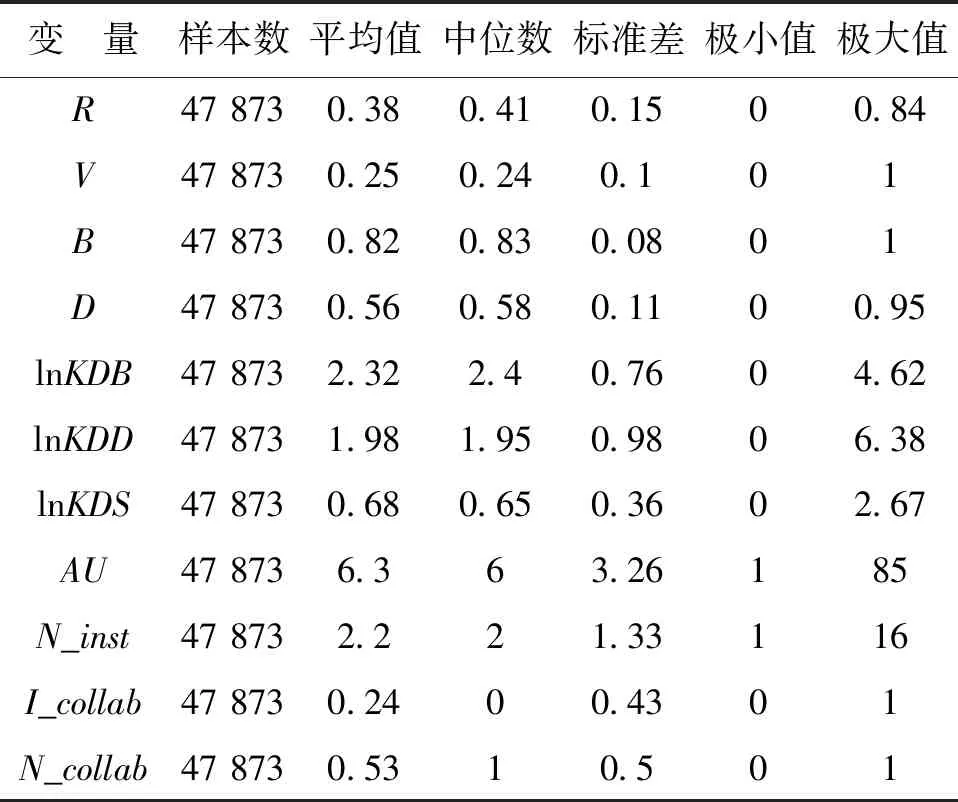
表1 各变量描述性统计分析
1)学科交叉论文分布。图1展示了4个学科交叉指标的论文频数分布,可以看出,基因工程领域学科交叉综合程度较低,交叉水平呈现较大差异,分布均衡;引文学科多样性集中在较低水平、而差异性呈中等水平,引文学科分布偏向均衡。说明基因工程领域的论文引文学科多样性不高,但偏向于引用学科差异性中等的论文,且引文学科分布均衡,故基因工程领域学科交叉整体水平偏低。但是与已有研究[11,25]相比,可以发现基因工程领域论文的学科交叉的综合程度、多样性、均衡性、差异性均高于医学信息学和纳米科技与纳米技术等领域,适合作为研究对象。
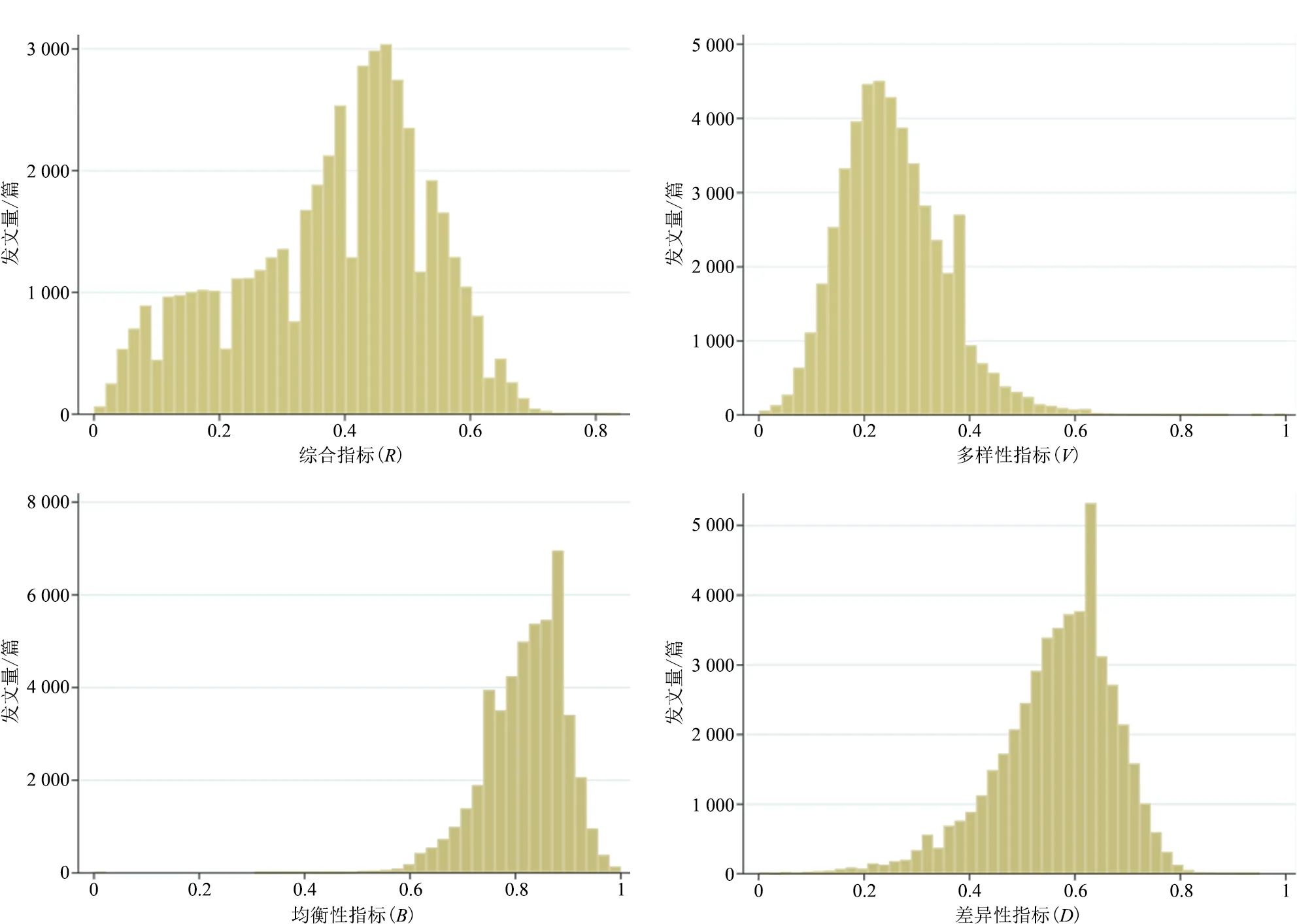
图1 学科交叉指标论文频数分布直方图
2)知识扩散论文分布。3个知识扩散指标的论文频数分布如图2所示,可以看出,论文知识扩散两极差异明显、分布不均衡,论文分布主要集中在中等扩散广度、低扩散强度以及低扩散速度区域。说明该领域论文虽然知识流向学科范围广,但影响力不强,传播速度慢。
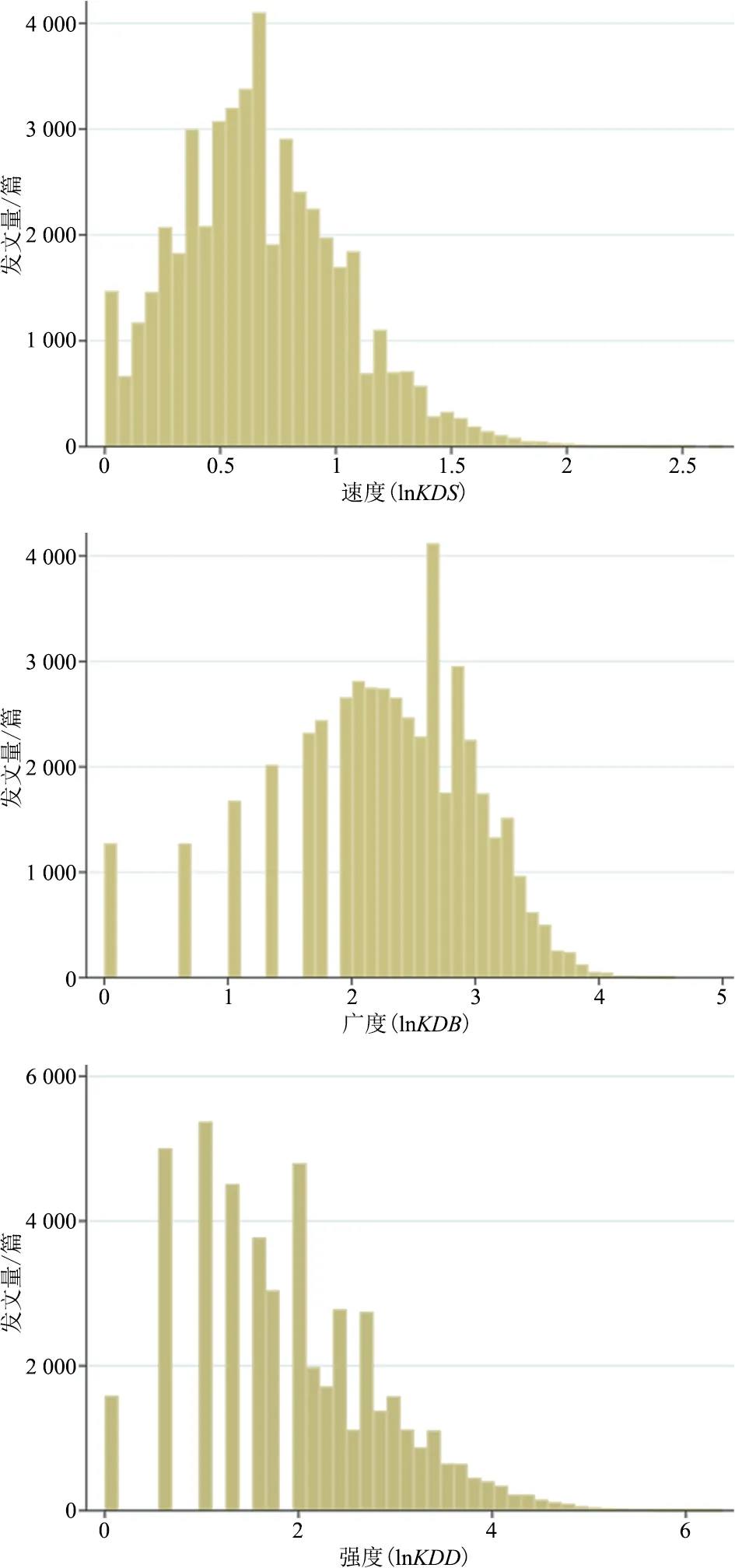
图2 知识扩散指标论文频数分布直方图
4.2 相关性分析
本研究采用Spearman秩相关分析,构建了学科交叉综合指标、学科交叉多样性指标、学科交叉均衡性指标、学科交叉差异性指标、知识扩散广度指标、知识扩散强度指标、知识扩散速度指标、合著作者数、合作机构数、国际合作和国内合作的相关关系矩阵,如表2所示。
从表2可以看出,学科交叉综合指标、多样性、均衡性、差异性指标与知识扩散广度、强度、速度的相关性不同,说明从不同维度研究学科交叉对知识扩散的影响具有必要性。变量全部通过了相关性系数显著性检验,所选取的变量具有一定的影响力,且相关性系数很低,进一步检验变量的方差膨胀因子,结果显示模型并不存在严格意义上的多重共线性,可以将全部变量纳入回归模型进行回归分析验证。

表2 学科交叉和知识扩散各指标变量的相关矩阵
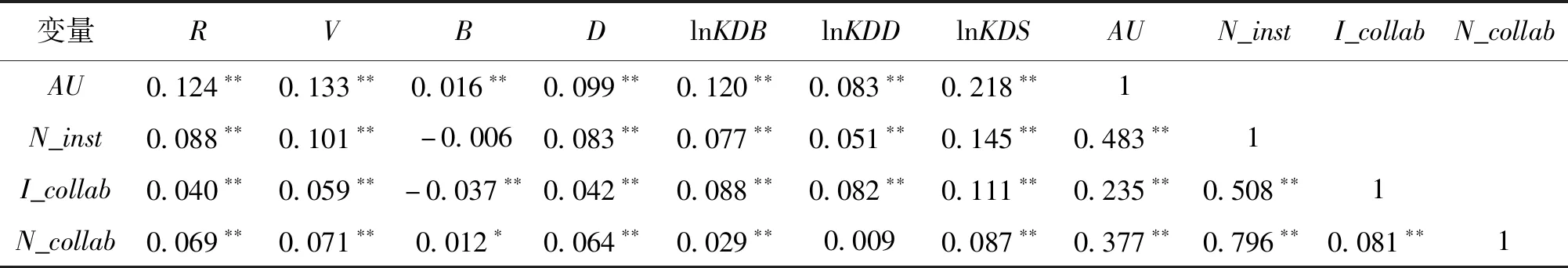
表2(续)
4.3 学科交叉与知识扩散回归分析
4.3.1 模型估计结果
为更加深入地研究变量之间的依赖关系,本文采用Tobit模型研究。将自变量(学科交叉多样性、差异性、均衡性、综合指标)、因变量(知识扩散广度)、控制变量(合作机构数、合著作者数、跨国合著机构、国内合著机构)代入回归方程,使用Tobit模型估计学科交叉研究对知识扩散广度的影响,其中Prob>chi2=0.0000,说明模型可信度较高。从表3可以看出,除机构数外各变量均在至少0.1的水平下通过了显著性检验。从模型1和3可以看出,学科交叉综合指数对知识扩散广度的影响在0.05水平上呈正向促进作用;学科交叉多样性和差异性对知识扩散广度的影响也在0.05水平上呈正向作用;而学科交叉均衡性对知识扩散广度的影响则在0.05水平上呈负向作用。为了验证学科交叉与知识扩散广度是否存在U型或倒U型关系,对知识扩散广度的所有一次回归函数均加入二次项,获得模型2、4、5和6,具体结果如表3所示。

表3 Tobit模型估计学科交叉研究对知识扩散广度的影响
采用Tobit模型估计学科交叉研究对知识扩散强度、知识扩散速度的影响,其研究过程与上述研究相同,研究结果和表3类似,不再列出。
4.3.2 学科交叉综合指标与知识扩散指标的回归分析
根据Tobit模型估计学科交叉研究对知识扩散指标的回归结果发现,学科交叉综合指标与知识扩散广度、强度、速度之间存在统计学上的显著关系,其关系式为:
ln(KDB)=-1.566*R2+1.512*R+1.829
(7)
ln(KDD)=0.941*R2-1.397*R+2.128
(8)
ln(KDS)=0.951*R2-0.322*R+0.482
(9)
一篇论文的学科交叉融合与多样性越高,吸收的学科知识差异越大,R值越大。在一定程度上,该论文的知识基础越全面与均衡,越有助于提高学科影响范围和扩散速度。学科交叉综合指标与知识扩散广度两者呈倒U型关系,表明论文学科交叉综合指标在达到某个最优值前,有利于提升知识扩散的广度;而达到最优值后,则会降低知识扩散的广度。对于知识扩散强度而言,学科交叉综合指标对其影响呈现U型左半边,最低值为0.74。说明当R小于0.74时,论文多样性越高,分布越均衡,学科差异越大,知识扩散强度越低,反之则越高。对于知识扩散速度而言,学科交叉程度对其影响则呈现U型右半边,当R大于0.17时,学科交叉综合指标越高,知识扩散速度越快,反之则越低。说明论文学科知识融合更全面、均衡,在一定程度上会被更加迅速地传播到更多学科,但同时由于论文知识专业度不强,会降低知识扩散强度。
从表1可知,学科交叉综合指标的平均值为0.38,如果想要提高论文知识扩散广度、速度使其达到最优值,则需融合种类更多、差异性更强的学科知识。学科交叉综合指标与知识扩散广度、强度、速度关系如图3所示。
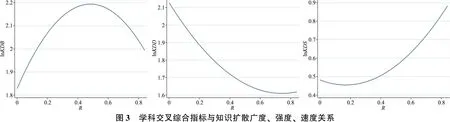
提取处于极值点的论文进行分析。研究发现共1 404篇论文处于知识扩散广度极大值点,篇均被引频次为36.2,发文量最多的学科为Neurosciences;2篇论文处于知识扩散强度极小值点,篇均被引频次为20,所属学科为Biology;共531篇论文处于知识扩散速度极小值点,篇均被引频次为31.1,发文量最多的学科为Plant Sciences。
学科交叉综合指标从学科多样性、均衡性及差异性3个维度综合测度论文的学科交叉程度。为了更加深入分析学科交叉各维度的影响,本研究分别探讨了学科交叉多样性、均衡性、差异性3个指标对知识扩散指标的影响。
4.3.3 学科交叉多样性指标与知识扩散指标的回归分析
根据Tobit模型回归结果发现,学科交叉多样性指标与知识扩散广度、强度、速度之间存在统计学上的显著关系,其关系式为:
ln(KDB)=-3.045*V2+2.758*V+2.198
(10)
ln(KDD)=-4.658*V2+3.511*V+3.199
(11)
ln(KDS)=-0.534*V2+1.521*V+0.510
(12)
当论文引用学科类别越丰富时,知识倾向于多元发展,其V值越高,说明该论文掌握了多个学科的理论和方法,而这些学科知识的交叉和融合使得论文的知识扩散学科范围更加宽泛,影响力更大,被学科吸收速度更快。学科交叉多样性与知识扩散广度和强度呈倒U型关系,说明若要提高论文的学科扩散的范围及影响力,需要融合更多学科的知识,但存在一个阈值,当学科种类超过一定数量时,可能会造成论文知识冗杂,主题定位不清晰,科学性受到质疑,从而降低论文的知识传播范围和影响力。结合表1可知,基因工程论文学科交叉多样性的平均值为0.25,低于对知识扩散广度和强度影响的最优值,表明论文可以适度地增加不同学科参考文献引用,提高知识综合能力。对学科交叉多样性与知识扩散速度关系进行倒U型检验,发现两者不存在倒U型关系,即学科交叉多样性达到知识扩散速度最优值的点大于V的最大值,论文引用学科类别越多,知识传播速度越快。学科交叉多样性与知识扩散强度、广度、速度的关系如图4所示。
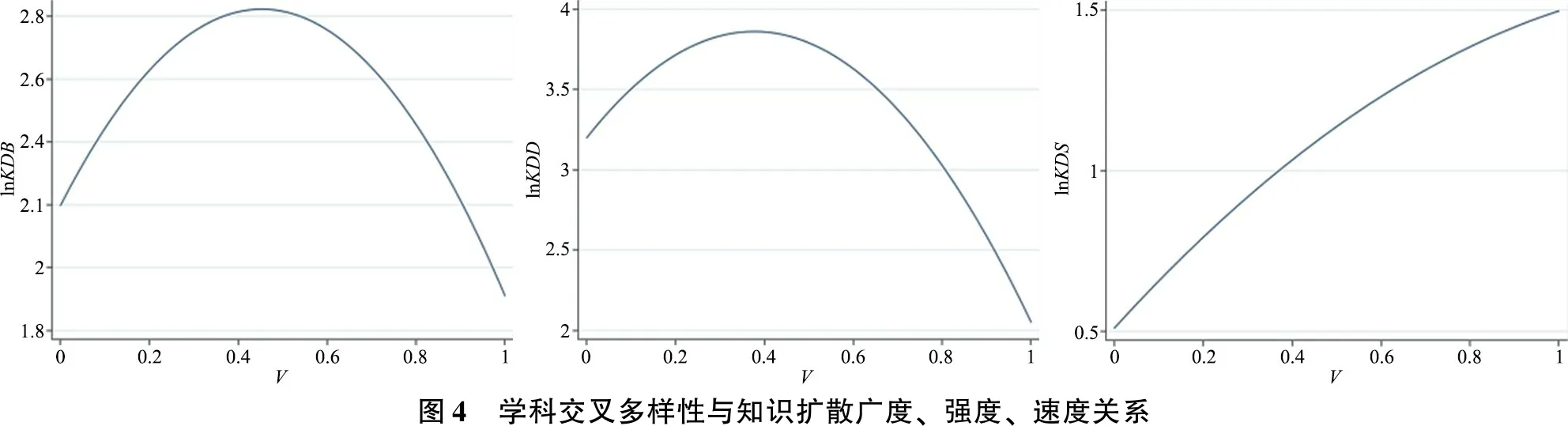
研究发现,共568篇论文处于知识扩散广度极大值点,篇均被引频次为24.9,发文量最多的学科是Biochemistry & Molecular Biology;共1 539篇论文处于知识扩散强度极大值点,篇均被引频次为30.2,发文量最多的学科也是Biochemistry & Molecular Biology。
4.3.4 学科交叉均衡性指标与知识扩散指标的回归分析
Tobit模型回归结果显示,学科交叉均衡性指标与知识扩散广度、强度、速度之间存在统计学上的显著关系,其关系式为:
ln(KDB)=-2.245*B2+2.849*B+1.050
(13)
ln(KDD)=-5.098*B2+6.220*B+0.464
(14)
ln(KDS)=-0.550*B2+0.491*B+0.217
(15)
论文所引用各学科的参考文献数量越均衡,理论基础和系统知识越坚实,B值越大,其科学性越高[11],吸引更多学科引用,领域知识在学科中传播速度越快,影响力越大。学科交叉均衡性与知识扩散广度、强度、速度均呈倒U型关系,如图5所示。表明论文引用学科分布越均衡,知识融合越全面,影响力更加广泛和强大,传播更加迅速。但当论文学科交叉均衡性超过阈值时,可能会表现出太过中庸,主题不清晰,论文缺乏新颖性和创造性,很难被扩散到更广的学科范围,产生更深的影响力;只有适当降低论文学科分布的均衡性,明确研究重点,论文的学术价值才会提升,知识扩散广度、强度和速度才会提升。
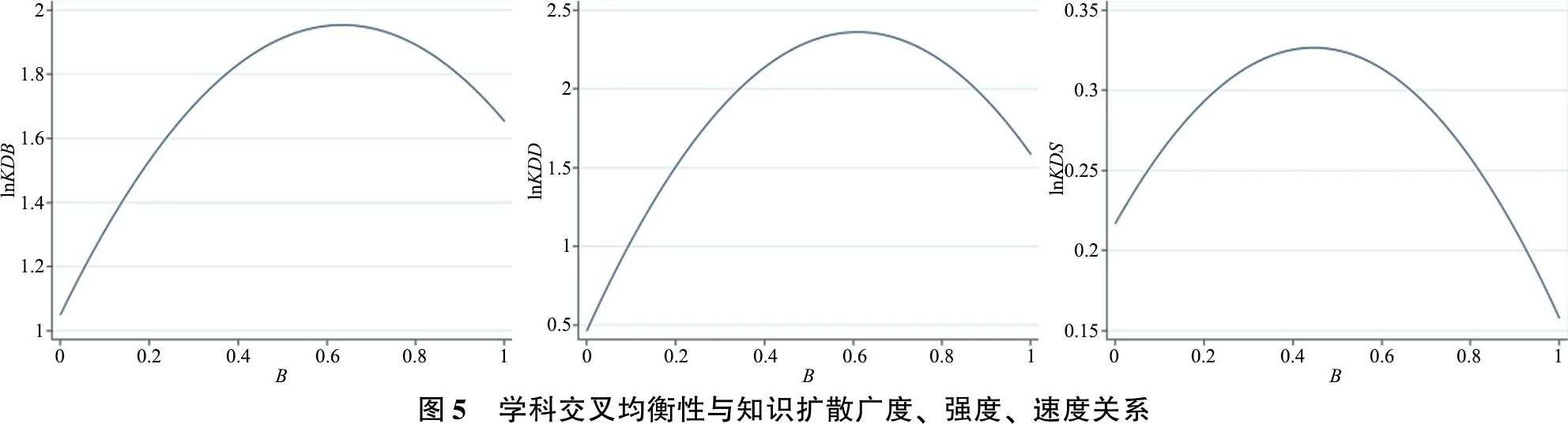
结合表1可知,基因工程领域论文均衡性的平均值为0.82,高于达到知识扩散广度、强度和速度的最优值,表明论文引用学科均衡性过高,知识扩散偏弱,该领域学者应当提高论文专业水平,明确重点,突出研究主题,才能更好地提高知识扩散广度、强度和速度。
研究发现,共190篇论文处于知识扩散广度极大值点,篇均被引频次为29.3,发文量最多的学科是Plant Sciences;共105篇论文处于知识扩散强度极大值点,篇均被引频次为36.8,发文量最多的学科也是Plant Sciences;仅1篇论文处于知识扩散速度极大值点,该论文被引频次为65,所属学科为Entomology。
4.3.5 学科交叉差异性指标与知识扩散指标的回归分析
Tobit模型回归结果显示,学科交叉差异性指标与知识扩散广度、强度、速度之间存在统计学上的显著关系,其关系式为:
ln(KDB)=-2.344*D2+2.478*D+1.813
(16)
ln(KDD)=-2.366*D2+1.536*D+2.903
(17)
ln(KDS)=-0.186*D2+0.170*D+0.496
(18)
论文所引用的参考文献归属学科的差异度越高,知识越不容易整合[24],学科间知识流动更稀少,从某种程度上来说这篇论文的创新性越强[11],因而在一定程度上使知识扩散广度、强度和速度越高。学科交叉差异性与知识扩散广度、强度、速度均呈倒U型关系,如图6所示,即知识属性上学科差异度高的论文会产生更强、更广泛、更迅速的影响力。但论文学科交叉差异性并非越高越好,当学科交叉差异性达到阈值后,论文融合的学科知识差异度过大,导致凝聚力不强,论文科学性受到质疑,知识扩散广度、强度和速度下降。基因工程领域差异性均值为0.56,低于论文达到知识扩散广度最优值,若要提高论文影响范围,需加强对差异性较大学科的引用,即论文探索、融合更多相差较大的学科知识,将此类学科中有价值的理论方法应用到该学科的研究进程,并与该学科的知识理论相结合。
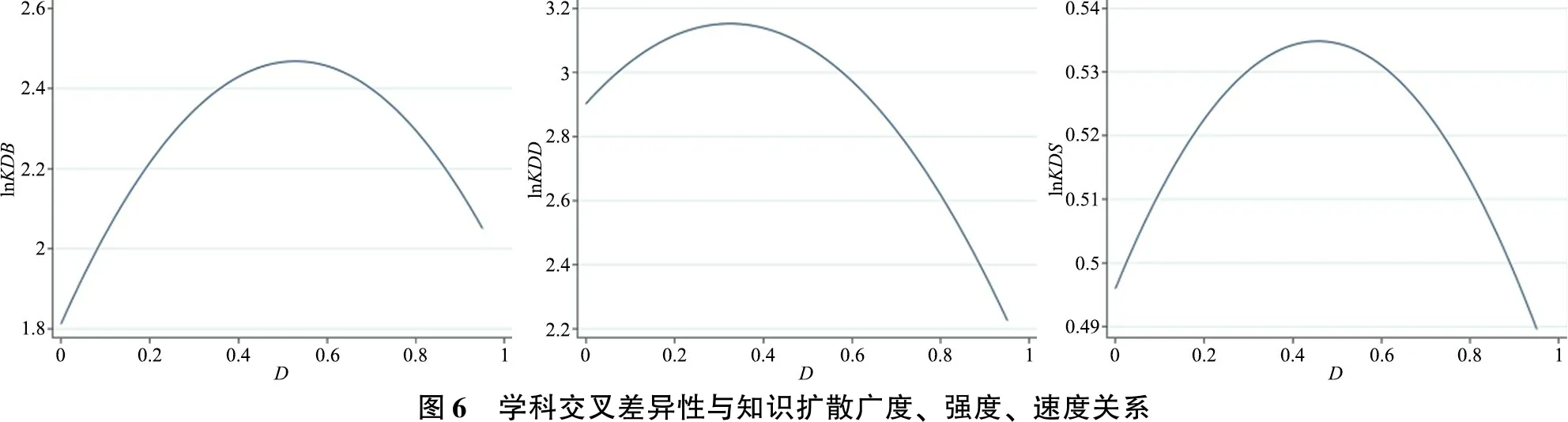
结合表1可知,论文达到知识扩散强度、速度最优值的差异性指标略低于均值,说明基因工程领域不能一味地追求打破学科壁垒、扩大基础知识的差距,而应当在保持论文专业性的同时,适当整合差异性较强的学科知识,才能最大限度地促进论文知识传播与扩散。
研究发现,共1 508篇论文处于知识扩散广度极大值点,篇均被引频次为36.4,发文量最多的学科是Biochemistry & Molecular Biology;共203篇论文处于知识扩散强度极大值点,篇均被引频次为54.9,发文量最多的学科是Plant Sciences;共872篇论文处于知识扩散速度极大值点,篇均被引频次为34.7,发文量最多的学科也是Plant Sciences。
5 结 论
本研究以基因工程领域2000—2018年发表论文为研究对象,以合著作者数、合著机构数、国内及国外合著情况为控制变量,采用Tobit模型分析基因工程领域论文的学科交叉综合指标、多样性、均衡性、差异性对知识扩散广度、强度、速度的影响。研究得出以下结论:
1)学科交叉和知识扩散基本呈现U型和倒U型关系。从多样性、均衡性、差异性3个维度更能细致地分析学科交叉对知识扩散的影响。学科交叉综合指标与3个单一维度指标和知识扩散广度全部呈倒U型关系;学科交叉综合指标与知识扩散强度呈U型关系,而3个单一维度指标则呈倒U型关系;学科交叉综合指标与知识扩散速度呈U型关系,而均衡性、差异性呈倒U型关系,多样性则不存在U型或倒U型关系。
2)学科交叉对知识扩散的影响存在最优值。在基因工程领域中主要学科发展到一定程度后都会出现难以逾越的知识屏障,研究成熟的学科在产生这些屏障时会向发展程度低一级的学科渗透。起初,发展较低的学科由于成熟学科的跨界会得到迅速的发展,论文的知识多样性增加,研究质量与知识扩散基本提高,但是过度的学科合作使得知识体系融合产生一定难度,并不能解决问题,论文研究容易停留在低层次的形式上,缺乏实质性的融合,反而对知识扩散产生不利影响。基因工程领域的研究主要以Plant Sciences、Neurosciences等学科提供的理论方法为基础,脱离这些学科,论文专业性难以被认同,可能流于肤浅。
3)基因工程领域论文应该适当增加引文的学科类别,降低引文均衡性,明确研究重点,突出研究主题,适当调整对差距较大学科的引用。目前基因工程领域大部分论文的引文学科多样性不高,且偏向于引用学科类别跨度相对均匀、距离中等的论文,学科交叉整体水平偏低。推动论文学科交叉指标达到实现知识扩散的最优值,有助于论文获得更加广泛、深远、迅速的影响力。
本文以基因工程领域的论文为研究对象,研究对象较为单一,虽然具有参考价值,但研究结论可能不适合其他领域;本文以JCR分类为基准进行学科期刊匹配时较为准确和高效,但存在少量论文所在期刊未被JCR期刊收录,可能会在一定程度上影响学科交叉度指标和知识扩散指标计算的精确度。今后研究可拓展到其他领域,并在提高精确度方面做进一步探讨。
