鹅产蛋性状全基因组选择信号分析
2022-08-23唐碧徽李海英赵晓钰吴盈萍丁雅文吴利楠梅志勇
唐碧徽,潘 璐,李海英,赵晓钰,吴盈萍,蒋 腾,丁雅文,吴利楠,曹 妍,梅志勇
(新疆农业大学动物科学学院,乌鲁木齐 830052)
随着人们对鹅产品需求的不断增长,低繁殖率严重阻碍了鹅产业的规模化发展[1],鹅的生产和繁殖能力取决于遗传因素和许多非遗传因素(饲养密度、公母比例、性成熟时体重、饲料组成和光照方案等),其中遗传因素更为重要,当非遗传因素得到充分解决,再加上适当的基因型选择与人工选择措施,便可以起到改善鹅的繁殖性能的作用[2]。全基因组重测序是指在已知某一物种基因组序列的基础上对该物种不同个体的整个基因组序列进行测序[3]。通过比对测序获得的序列和已知该物种的参考序列可以得到大量的基因组遗传信息,挖掘不同个体或群体基因组间的结构差异,探讨功能基因或区域定位[4]。全基因组重测序数据分析内容中的高级数据分析包含选择信号分析[5]。刘刁等[6]对深县猪和梅山猪进行全基因组重测序,采用滑动窗口方法进行群体选择信号分析,结果显示,在受选择区域筛选后得到受选择位点580个,定位于23个基因,通过GO功能和KEGG通路富集分析显示,筛选到3个与深县猪繁殖相关的候选基因。吕世杰等[7]对郏县红牛和中国荷斯坦奶牛进行选择信号检测,筛选种间差异的基因组区域,通过在动物数量性状座位(QTL)数据库中的比对,寻找到3个与牛繁殖性状相关的候选基因。Liu等[8]对狮头鹅、浙东白鹅、太湖鹅和籽鹅4个品种共35个个体运用全基因组重测序方法进行选择信号分析,在4个鹅品种基因组中筛选出许多候选基因,发现了可能与狮头鹅产肉性状和籽鹅产蛋性状的相关通路。伊犁鹅是新疆特有的优良地方禽种,耐粗放饲养、肉质优良,但产蛋性能较低,且就巢性强,成年母鹅年均产蛋仅为8~12枚[9]。霍尔多巴吉鹅是由欧洲引进的一种绒蛋兼用型优良品种,其繁殖能力优于伊犁鹅,成年母鹅平均年产蛋为40~50枚[10]。伊犁鹅与霍尔多巴吉鹅在产蛋数量上差异较大,适合作为研究鹅产蛋性状的素材。本研究利用全基因组重测序技术,通过选择信号分析,筛选与鹅产蛋相关的候选基因,以期为伊犁鹅和霍尔多巴吉鹅的群体遗传分析和产蛋性能的研究提供新的思路。
1 材料与方法
1.1 样品采集
随机选取健康状况良好、饲养管理水平一致的2岁伊犁鹅母鹅和霍尔多巴吉鹅母鹅各24只(伊犁鹅(Y1~Y24);霍尔多巴吉鹅(H1~H24)),翅下静脉采血5 mL于EDTA抗凝管中,置于-20 ℃保存,用于DNA提取。所用试验动物均由新疆额敏县恒鑫实业有限公司国家级伊犁鹅保种场提供。
1.2 DNA提取及重测序
采用磁珠法从全血样本中提取基因组DNA,通过1.0%琼脂糖凝胶电泳检测DNA完整性,并使用分光光度计(NanoDrop 2000)测定DNA浓度。检测合格的DNA样品通过Covaris超声波破碎仪随机打断,经末端修复、加A尾、加测序接头、纯化和PCR扩增等步骤完成整个文库制备。库检合格后,将不同文库按照有效浓度及目标下机数据量的需求进行Illumina Hiseq PE150测序,经过对测序数据的严格过滤,得到高质量的clean base。
1.3 SNP变异检测
使用BWA软件(参数:mem-t4-k32-M)将读取到的高质量数据比对到天府鹅的参考基因组(https:∥ftp.cngb.org/pub/gigadb/pub/10.5524/100001_101000/100789/),采用SAMTOOLS等软件对样本进行群体SNP检测。经过条件为dp 7(保留样本深度不低于7×的数据)、Miss 0.1(保留样本缺失率不高于0.1的数据)、maf 0.05(保留样本最小等位基因频率不小于0.05的数据)过滤后,获得高质量的SNP位点用于后续分析。
1.4 群体系统进化树构建
经过SNP检测后,得到的个体SNP可用于计算种群之间的距离。运用TreeBest(http:∥treesoft.sourceforge.net/treebest.shtml)软件计算距离矩阵,通过邻接法(Neighbor-Joining method)构建系统进化树。
1.5 群体遗传结构分析
采用frappe进行群体结构分析。首先创建PLINK的输入文件-Ped文件,然后利用frappe软件构建群体遗传结构和群体世系信息。
1.6 选择信号分析
1.6.1 群体分化指数 利用工具vcftools计算伊犁鹅和霍尔多巴吉鹅2个群体之间的群体分化指数(Fst)值,使用窗口大小为40 kb、滑动步长为20 kb的滑动窗口,计算每个窗口内所有标记的Fst平均值,将其作为此窗口的Fst值。
1.6.2Fst和核苷酸多样性的联合分析 基于Fst值再分别计算伊犁鹅和霍尔多巴吉鹅的核苷酸多样性(Pi)值,使用窗口大小为40 kb、滑动步长为20 kb的滑动窗口,计算每个滑动窗口SNP的Pi平均值作为此窗口的Pi值,进一步与Fst值联合进行窗口的筛选。
1.7 候选基因注释及基因功能富集分析
从参考基因组对应的基因注释文件中,将筛选出的受选择位点定位到基因。使用pfam和KOBAS 2.0数据库进一步对基因进行GO功能和KEGG通路富集分析。
2 结 果
2.1 全基因组重测序结果
伊犁鹅和霍尔多巴吉鹅2个群体的全基因组重测序结果见表1,测序共获得1 066.665 Gb的原始数据,各样本的原始数据在12 911 727 038~31 532 656 750 bp之间,经过滤处理后得到的有效数据在12 493 713 313~31 423 739 525 bp之间,平均有效数据率达96.76%,Q20平均达到96.47%,Q30平均达到91.80%,GC含量在44.32%~45.90%之间。表明本次测序数据质量较高,可满足后续试验要求。
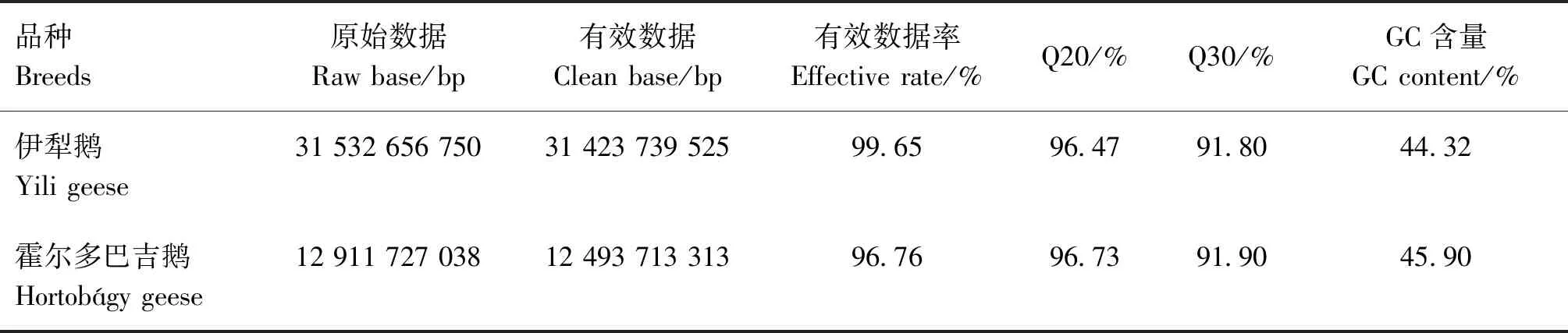
表1 伊犁鹅与霍尔多巴吉鹅测序数据平均统计表
2.2 测序数据与参考基因组的比对分析
质控过滤后的测序数据通过BWA软件比对到鹅参考基因组。由表2可知,本试验测序数据与参考基因组的比对率在97.31%~97.44%之间,测序深度在9.49×~21.06×之间,1×覆盖率在98.18%以上,4×覆盖率在83.90%以上。表明本试验的测序物种与参考基因组相似度高,测序数据准确,可进行后续分析。

表2 伊犁鹅与霍尔多巴吉鹅平均测序深度及覆盖度统计表
2.3 SNP变异检测结果
本试验对2种鹅群体SNPs进行变异检测,结果见表3。由表3可知,共检测到15 406 790个SNPs位点,对检测到的SNPs位点通过ANNOVAR软件进行注释发现,SNPs在基因间、内含子和外显子区域的比例分别为65.35%、29.49%和1.77%。其中外显子区域中,少量的SNPs获得终止密码子的变异(2 003个)或失去终止密码子的变异(282个),但大多数SNPs导致同义突变(154 517个)或非同义突变(117 659个),转换(Ts)为10 838 735个,颠换(Tv)为4 568 055个,Ts/Tv为2.372。

表3 SNPs分类结果
2.4 群体系统进化树分析
伊犁鹅群体和霍尔多巴吉鹅群体的系统进化树结果见图1。由图1可知,两种鹅群体明显分为两大支,伊犁鹅群体和霍尔多巴吉鹅群体各自聚为一支,说明两种鹅群体之间的亲缘关系存在一定的距离。
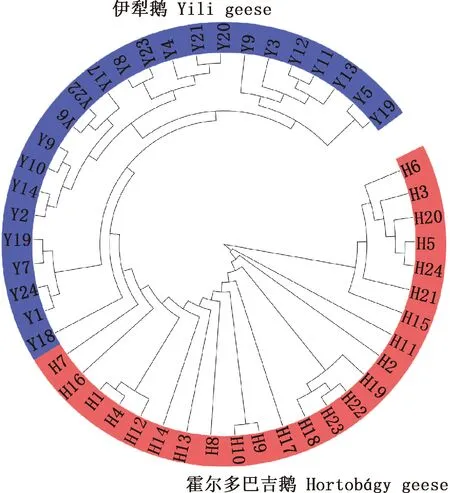
图1 伊犁鹅与霍尔多巴吉鹅群体系统进化树Fig.1 Phylogenetic tree of Yili and Hortobgy geese populations
2.5 群体遗传结构分析
由图2可知,K=2时,霍尔多巴吉鹅群体全部为一个颜色,说明霍尔多巴吉鹅群体全部在一个亚群群体中;而伊犁鹅群体出现了两种颜色,说明其可能来自于2个亚群群体。
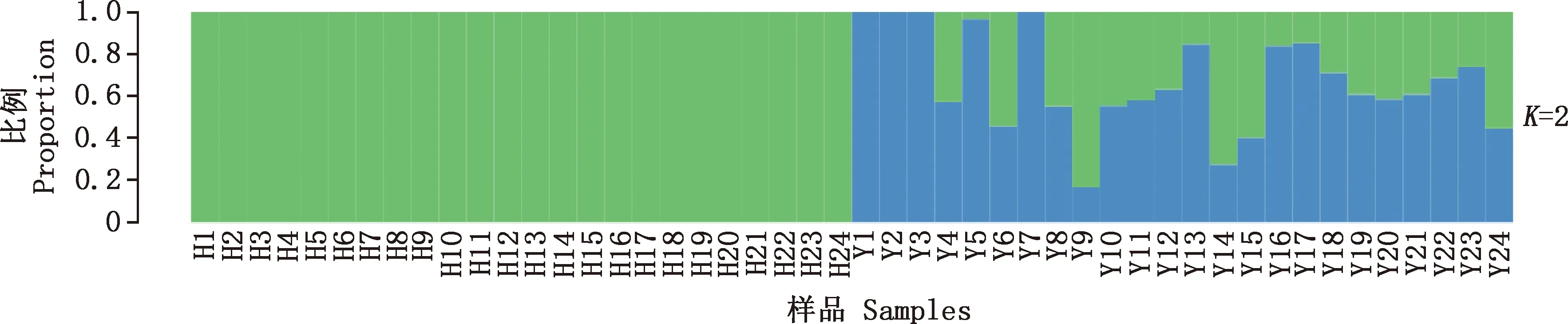
K=2代表有2个不同的亚群,不同颜色代表不同的亚群,如某个样品由多种颜色构成,则对应着纵坐标可看出每个亚群所占的比例K=2 represents the two of different subgroups,and different colors represent different subgroups,if a sample is composed of multiple colors,the proportion of each subgroup can be seen according to the ordinate图2 伊犁鹅与霍尔多巴吉鹅遗传结构图Fig.2 Genetic structure diagram of Yili and Hortobgy geese
2.6 群体分化结果
由图3可知,以top 5%为阈值作为筛选标准,即top 5%的Fst值为0.228的窗口被视为受选择的窗口,共筛选到1 231个受选择的候选区域,其中有5个选择信号窗口的Fst值>0.5,位于5和22号染色体,Fst值最高的选择信号窗口位于22号染色体。

虚线代表选择阈值(top 5%)The dotted line represents the selection threshold (top 5%)图3 伊犁鹅与霍尔多巴吉鹅Fst图Fig.3 Fst diagram of Yili and Hortobgy geese
2.7 Fst & Pi联合分析结果
本试验中Fst&Pi选取阈值为top 5%,关联Fst&Pi提取相应候选区域,并提取相应区域内的变异位点信息,采用选择性清除方法检测到相应的选择信号区域,伊犁鹅和霍尔多巴吉鹅基于Fst&Pi的选择消除分析结果见图4。由图4可知,伊犁鹅群体与霍尔多巴吉鹅群体之间的Fst值为0.198,Pi值分别为-0.203 93和2.050 78,在伊犁鹅群体中Fst和Pi的选择性消除分析中有10个受到选择的区域,其中得到注释的基因有5个;在霍尔多巴吉鹅群体中Fst和Pi选择消除分析中有353个受到选择的区域,其中得到注释的基因有263个。
2.8 基因富集分析
对伊犁鹅和霍尔多巴吉鹅2个群体中受到选择的基因分别进行GO功能和KEGG通路富集分析。在GO功能富集分析中,2个鹅群体中受到选择区域中的基因主要富集在生物过程,如DNA重组(DNA recombination)、内稳定过程(homeostatic process)、Wnt受体信号通路(Wnt receptor signaling pathway)和细胞的自动调节(cellular homeostasis)等过程。在KEGG通路富集分析中,伊犁鹅群体显著富集到细胞黏附分子(cell adhesion molecules)信号通路,霍尔多巴吉鹅群体主要富集到了Wnt信号通路(Wnt signaling pathway)、TGF-β信号通路(TGF-beta signaling pathway)、MAPK信号通路(MAPK signaling pathway)、胞质DNA感应途径(cytosolic DNA-sensing pathway)和细胞因子-细胞因子受体相互作用(cytokine-cytokine receptor interaction)等通路。本试验从这些通路中筛选到了6个与产蛋性状相关的候选基因:BMP2、BMP6、ENO1、MIS、LIF和EP300(表4),还筛选到了1个与免疫相关的基因IL-18。

横坐标为Pi值,纵坐标为Fst值,分别对应上面和右侧的频率分布图。中部的点代表不同窗口内的相应的Fst和Pi值,其中蓝色和绿色区域为Fst和Pi选择出来的top 5%区域,红色区域为两者共同所选择的top 5%区域The horizontal coordinates is Pi values,and the vertical coordinates are Fst values,which correspond to the frequency distribution chart above and the frequency distribution chart on the right,respectively.The dots in the middle represent the corresponding Fst and Pi values in different windows,where the blue and green areas are the top 5% areas selected by Fst and Pi,and the red area is the top 5% area selected by both together图4 伊犁鹅(A)和霍尔多巴吉鹅(B)Fst & Pi选择消除分析图Fig.4 Fst & Pi selection elimination analysis chart of Yili (A) and Hortobágy (B) geese
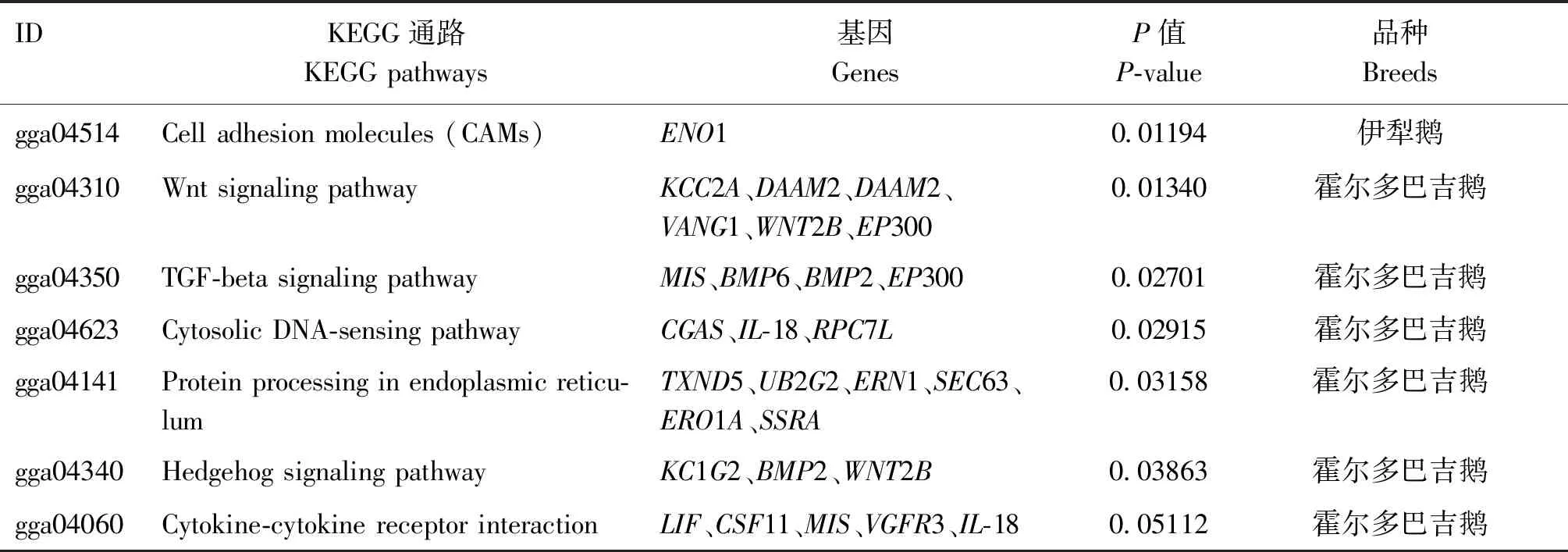
表4 伊犁鹅和霍尔多巴吉鹅群体在KEGG通路中的基因富集情况

续表
3 讨 论
3.1 测序数据分析
本研究对24只伊犁鹅和24只霍尔多巴吉鹅进行全基因组重测序,测序共获得1 066.665 Gb的原始数据,其中Q30平均达到91.80%,群体样本的平均比对率达97.31%,平均测序深度为15.28×,4×覆盖度在83.90%以上,与参考基因组相似性极高,表明测序数据均一性较好,结果可靠。在此基础上,通过注释分析得到SNPs位点共有15 406 790个,大部分(65.35%)SNPs位点分布在基因间区中。SNP的质量一般通过计算Ts/Tv来估计。在人、猪和山羊中,该比值为2.0~2.4[11-13]。本研究中Ts/Tv为2.372,表明潜在随机测序的误差较低,与前人研究结果一致,说明本研究中识别的变异具有较高的准确性。
群体进化树用来表示群体间的进化关系,根据群体的物理或遗传学特征等方面的共同点或差异可以推断出它们的亲缘关系远近,即群体个体间由于共同祖先而产生的相互关系,亲缘关系较近的物种进化分支往往聚成一簇。本试验中2个鹅群体的系统发育进化树表现出较明显的遗传分化。群体遗传结构指遗传变异在物种或群体中的一种非随机分布,按照地理分布或其他标准可将一个群体分为若干亚群,处于同一亚群内的不同个体亲缘关系较高,而亚群与亚群之间则亲缘关系稍远,不同颜色代表不同的亚群体。群体结构分析发现,在K=2时,伊犁鹅群体表现出内部分化,个别个体与其他个体间存在交叉,可能是基因交流的结果,也可能与所选样本有关[14],但整体来看,2个群体之间是可以明显区分开的,与系统发育树结果一致,二者相互印证。从SNP层次来看,2种鹅存在较大的差异性,可能是主导其不同性状的潜在SNP位点造成的。
选择信号是动物经历人工选择或自然选择的过程中在基因组上留下了一些遗传特征的现象[15]。吴旭东[16]研究表明,Pi值越高说明相同基因组区域物种的核苷酸多样性越低。本研究中,霍尔多巴吉鹅作为从欧洲引进的培育品种,经历了很多人工驯化培育的过程,而伊犁鹅相对来说,并未受到过强烈的人工选育过程,其自身还保留着一定的野性,伊犁鹅的Pi值(-0.20393)低于霍尔多巴吉鹅的Pi值(2.05078),说明伊犁鹅的核苷酸多样性较高,推测在选择消除分析中伊犁鹅的受选择区域比霍尔多巴吉鹅少与其自身的遗传信息有关。
3.2 候选基因推测分析
本研究对伊犁鹅和霍尔多巴吉鹅群体进行选择信号分析,筛选出6个与产蛋性状相关的候选基因(BMP2、BMP6、MIS、ENO1、LIF和EP300),以及1个与禽类免疫相关的IL-18基因。
BMP2和BMP6是骨形态蛋白(BMP)家族的成员。BMP2是来源于颗粒细胞的生长因子,在卵泡发育和排卵过程中扮演着重要的角色。研究发现,BMP2及其受体在卵巢内的颗粒细胞、黄体和卵泡液内都被检测到[17-19],说明BMP2可能在调节卵泡发育和黄体功能方面起着重要的作用。Haugen等[20]研究表明,在卵泡发育启动之前,BMP2能够维持鸡颗粒细胞的未分化状态,同时降低鸡颗粒细胞的卵泡刺激素(FSH)敏感性。Selvaraju等[21]研究发现,BMP2可以促进鸡卵巢颗粒细胞内雌二醇的合成,并抑制孕酮的生成。魏强[22]对与猪繁殖相关的基因进行基因分型,结果发现,BMP2基因不同基因型在猪的总产仔数、产活仔数和断奶头数上差异极显著,说明BMP2基因与母猪繁殖力显著相关。BMP6在哺乳动物生殖中也有着重要的作用,BMP6来源于卵母细胞,有刺激颗粒细胞增殖的作用[23]。研究表明,BMP6直接参与了调控繁殖相关激素的合成与分泌,并影响繁殖器官的发育和形成[24]。Sugiura等[25]对敲除BMP6基因的小鼠生殖性能进行测量发现,雌鼠每窝的产仔数只有自然小鼠的78%,推测BMP6可能通过适当增强黄体生成素(LH)信号水平和维持卵母细胞正常功能来增强雌性小鼠的生育能力。姚国瑜[26]研究发现,金寨黑鸡BMP6基因存在A76150G位点突变,该位点与金寨黑鸡体重和340日龄总产蛋数显著相关。本研究中,BMP2和BMP6基因富集到TGF-β信号通路,推测BMP2和BMP6基因可能通过调节禽类颗粒细胞的增殖和分化来影响鹅的卵泡发育情况。
ENO1是糖类代谢中的关键酶之一,在细胞的新陈代谢中发挥着重要作用,广泛存在于动物机体的各个组织和器官之中[27]。张伟宏等[28]研究发现,在体外培养的籽鹅颗粒细胞中,ENO1基因过表达会促进孕酮的分泌,推测ENO1基因过表达可能会促进禽类的卵泡排卵。计红等[29]研究发现,ENO1基因过表达时,可能通过提高籽鹅卵泡颗粒细胞的糖酵解水平从而促进细胞增殖,抑制凋亡,进而改变细胞周期的时相性。本研究发现,ENO1基因在细胞黏附分子信号通路中显著富集,推测ENO1基因可能与鹅的繁殖性能有关。
抗苗勒氏管激素(AMH)又称为苗勒管抵制物(MIS)[30]。研究发现,AMH在雌性动物中主要由卵泡颗粒细胞合成和分泌,是目前发现的体内唯一可以调控卵泡募集的细胞因子,对卵泡的生长发育及优势卵泡的选择起重要作用[31]。吴玉麟等[32]研究发现,在AMH基因第2外显子中检测出的4个SNPs位点与京海黄鸡的繁殖性状均呈显著相关。陈蓉等[33]采用实时荧光定量PCR技术检测了连城白鸭不同等级卵泡中AMH等基因的表达情况,结果发现,AMH基因的表达量随着卵泡的发育而逐渐降低,认为在鸭等级前卵泡中AMH基因的高表达量有助于维持颗粒细胞未分化的状态,抑制原始卵泡的初始募集,进而维持卵泡的发育潜能。葛丽岩等[34]研究发现,AMH基因中存在的g.1731 G>A和g.2044 T>C位点与黑羽番鸭产蛋性状存在相关。本研究中,MIS基因富集到TGF-β信号通路,推测其与鹅产蛋性状相关。
白血病抑制因子(LIF)是一种分泌型糖蛋白,是白细胞介素-6(IL-6)细胞因子家族中的一员,是一个在各种组织中都能发挥生物学作用的多效性细胞因子[35]。研究发现,LIF参与了大鼠原始卵泡的活化[36];LIF可作为颗粒细胞的增殖因子,促进小鼠培养的窦前卵泡生长[37];LIF基因与猪的繁殖性能有关[38-40]。本研究中,LIF基因富集到细胞因子-细胞因子受体相互作用的通路,可能促进原始卵泡的激活和早期卵泡的生长发育,推测LIF可能与鹅繁殖性能有关。
E1A结合蛋白P300(EP300)是一种蛋白质编码基因,该基因编码腺病毒E1A相关细胞p300转录共激活蛋白。EP300作为组蛋白乙酰转移酶,通过染色质重塑调节转录,在细胞增殖和分化过程中起重要作用。陈宇[41]对猪的全基因组SNP筛选时发现,EP300可能是与猪繁殖性状相关的候选基因。本研究中,EP300虽然富集到了Wnt信号通路,但对于EP300是否和鹅产蛋相关还需要进一步的验证。
IL-18编码一种促炎性细胞因子,可增强脾细胞的自然杀伤细胞活性,刺激辅助性T细胞Ⅰ型细胞产生干扰素,是近年发现的一种重要的免疫调节因子[42]。研究发现,IL-18可显著增强禽流感重组禽痘病毒疫苗的保护效力,在鸡中是一种安全的免疫刺激剂[43]。本研究发现,IL-18基因在胞质DNA感应途径和细胞因子-细胞因子受体相互作用通路中都有富集,推测IL-18基因和禽类免疫有关。
4 结 论
本试验采用全基因组重测序的方法收集了伊犁鹅和霍尔多巴吉鹅的全基因组SNP变异情况,通过选择信号分析筛选出了一些受选择的区域,筛选了6个可能与鹅产蛋性状相关的候选基因(BMP2、BMP6、MIS、ENO1、LIF、EP300),以及1个与禽类免疫相关的IL-18基因,为今后在基因组层面选择鹅的产蛋性状提供了有价值的信息。
